Introducción
La percepción de la sociedad acerca del funcionamiento de los sistemas biológicos y propiedades de productos de origen natural o manufacturados, ha cambiado con los avances tecnológicos direccionándolos a la solución de problemas generales o específicos (Duan et al., 2019; Livingston & Risse, 2019). De esta forma surgen las bases de la inteligencia artificial (IA), implementada en la década de 1950s con la máquina de ''Turing'' (Turing, 1936), una propuesta de modelo matemático para una maquina automática, caracterizada principalmente por parámetros de almacenamiento y unidad de control. En 1956, John MacCarthy, aplica el término IA, refiriéndose a una máquina inteligente, capaz de aconsejar, tomar decisiones y razonar como una persona (Morgenstern & Mcilraith, 2011), donde los algoritmos que utiliza la IA para simular el razonamiento humano son llamados aprendizaje automático o Machine Learning (Portugal et al., 2015).
Las redes neuronales son algoritmos que pertenecen al campo de Machine Learning aplicados para la segmentación, detección y clasificación de imágenes de diverso origen y naturaleza (Aguilar-Alvarado & Campoverde-Molina, 2019; LeCun et al., 2015). Estos algoritmos toman las imágenes como entrada y en cada una detectan una serie de características con base en patrones y son capaces de diferenciar unas imágenes de otras (Hafemann et al., 2014; Krizhevsky et al., 2012). El uso de algoritmos de Machine Learning se ha extendido a importantes sectores y especialidades, como en la medicina para el diagnóstico de diferentes enfermedades, por ejemplo, para identificar los síntomas de Covid-19 en radiografías de pacientes. Inclusive, en el ajedrez y StarCraft II se utiliza el algoritmo AlphaZero para contar con estrategias y decisiones en tiempo real (Nouretdinov et al., 2015; Porcelli & Martínez, 2020; Silver et al., 2018; Simić et al., 2018; Vinyals et al., 2019; Ye, 2015).
Los algoritmos de Machine Learning son aplicados en la industria de la madera, principalmente, para la clasificación de especies maderables utilizando imágenes macroscópicas de la madera con redes neuronales artificiales (ANN) (Khalid et al., 2008; Yadav et al., 2013) y redes neuronales convolucionales (CNN) (Fabijańska et al., 2021; Hafemann et al., 2014; Kwon et al., 2017; Siew et al., 2017).
Ante la alta diversidad de especies maderables en los bosques tropicales, existe una creciente necesidad de desarrollar aplicaciones móviles con IA para la identificación de especies maderables desde la extracción, transporte y comercialización. Sin embargo, las limitaciones en la obtención de hojas, flores y/o frutos para la identificación botánica o la obtención de secciones transversales para la identificación anatómica, y el insuficiente número de expertos en identificación de especies a partir de la caracterización anatómica en los puestos de control y fiscalización no permiten avanzar en las políticas de control para reducir la tala ilegal (de Andrade et al., 2020; Filho et al., 2014; Ravindran et al., 2020).
Usualmente, la identificación de especies maderables se realiza con imágenes microscópicas, las cuales demandan una serie de procedimientos, reactivos y equipos sofisticados de laboratorio, cuya alta calidad en la visualización de las características anatómicas, incrementa la precisión de un modelo de red neuronal convolucional (Hafemann et al., 2014; Koch & Haag, 2015; Lens et al., 2020). Sin embargo, la identificación de especies maderables empieza en campo, luego en el puesto de control y finalmente en los centros de comercialización; en estos espacios se requiere una metodología accesible, rápida y práctica. Ante estas condiciones se recurre a la identificación macroscópica de la madera, se realiza un corte con una navaja en la sección transversal y con un lente de 10X se observan las características anatómicas, convirtiéndose en una metodología sencilla y de bajo costo para identificar especies maderables en campo (Filho et al., 2014).
La aplicación de las CNN en la identificación de especies maderables requiere una amplia y representativa base de datos de imágenes macroscópicas de la madera, lo cual se ha incrementado progresivamente desde que se inició el uso de las CNN. Por ejemplo: Apolinario-Lainez et al. (2018) obtuvieron 115 imágenes para describir 7 especies maderables en Perú, Kwon et al. (2017) obtuvieron 187 imágenes de 5 especies maderables en Korea del Sur, Apolinario et al. (2019) utilizaron 355 imágenes de 16 especies, Filho et al. (2010) obtuvieron 1 270 imágenes de 22 especies de Brasil, Ravindran et al. (2019) obtuvieron 2 187 imágenes de 15 géneros, Ravindran et al. (2018) entrenaron un modelo CNN con 2 303 imágenes de 10 especies de la familia Meliaceae; Filho et al. (2014) incrementaron a 2 942 imágenes para 41 especies en Brasil, Yang et al. (2019) presenta 4 800 imágenes de 25 especies, y Mata-Montero et al. (2020) muestran 3 516 imágenes de 146 especies de Costa Rica. Sin embargo, acceder a un banco de imágenes macroscópicas o microscópicas es una recurrente restricción para la aplicación de redes neuronales ANN y CNN en la identificación de especies maderables (Hanssen et al., 2011).
En la captura de imágenes macroscópicas se requiere la preparación de la sección transversal de la madera para visualizar mejor las características anatómicas, para lo cual se utiliza lijas (Rajagopal et al., 2019), cutters (Tang et al., 2018), micrótomos (Khalid et al., 2008) y cuchillos (de Andrade et al., 2020). Sin embargo, se requiere procedimientos adicionales para mejorar la calidad de las imágenes macroscópicas como: estabilizar el sistema de iluminación (Rajagopal et al., 2019), extraer el polvo que obstruye el lumen de los poros después del pulido (Ibrahim et al., 2017) y la selección de áreas sin defectos, grietas, áreas podridas por organismos xilófagos y zonas de anormalidad de crecimiento como nudos, entre otros (da Silva et al., 2017).
El presente estudio se realizó en el puesto de control del SERFOR: ''El Pedregal'' de la provincia Chanchamayo, región Junín; durante las inspecciones a los camiones de carga que transportan madera aserrada se evidenció que no se dispone de tiempo suficiente para preparar la superficie transversal de la madera y obtener imágenes macroscópicas ante el paso constante de camiones de carga. Asimismo, la revisión y verificación de información consignada en la Guía de Transporte Forestal (GTF) no debe exceder 15 min. Por estas razones, es imprescindible contar con equipos y materiales accesibles para obtener imágenes macroscópicas de alta calidad. Se describe la aplicación de tres herramientas de corte en campo y el uso de un microscopio portátil en la captura de imágenes macroscópicas para lograr la clasificación de 25 especies maderables de Selva Central con redes neuronales convolucionales (CNN).
Materiales y métodos
Obtención y preparación de muestras: Las muestras corresponden a 25 especies maderables de Selva Central, se obtuvieron dos tipos de muestras: i. Pequeñas piezas de madera de aserraderos de la provincia Chanchamayo y Satipo, y ii. Rodajas o secciones transversales de los árboles aprovechados por empresas madereras en bosques de comunidades nativas de la provincia Satipo, región Junín (Ferreira et al., 2021).
Las muestras recolectadas fueron etiquetadas con sus nombres comunes, luego fueron llevadas al Laboratorio de Anatomía e Identificación de la Madera de la Universidad Continental campus Incho, Huancayo para realizar la identificación y caracterización anatómica. Se obtuvieron secciones rectangulares (2.5 cm de ancho x 6 cm de largo y 3 cm de alto), evitando áreas con defectos (grietas, rajaduras o áreas podridas por organismos xilófagos) para evitar confusiones en la identificación de patrones anatómicos (da Silva et al., 2017) como se muestra en la Fig. 1.

Fig. 1 A. y B. Preparación de muestras de madera. C. Obtención de imagen macroscópica con el microscopio portátil. D. Imagen macroscópica de Matisia cordata. / Fig. 1. A. and B. Preparation of wood samples. C. Obtaining a macroscopic image with the portable microscope. D. Macroscopic image of Matisia cordata.
Esta etapa es esencial para obtener una metodología adecuada en el proceso de corte y obtención de imágenes. Sin embargo, algunos autores no contemplan la etapa de obtención de muestras y preparación, al contrario, empiezan directamente con la adquisición de imágenes por contar con repositorios de imágenes o muestras de xilotecas en laboratorio. Este estudio describe la importancia del corte en la preparación y propone una serie de etapas para la clasificación, diferente a las reportadas en la Tabla 1.
Tabla 1 Etapas para la clasificación de especies maderables con CNN. / Table 1. Stages for the classification of timber species with CNN.
| Referencia | Etapa 1 | Etapa 2 | Etapa 3 | Etapa 4 | Etapa 5 |
| (Khalid et al., 2008) | Recopilación de muestras | Adquisición de imágenes | Mejoramiento de imágenes | Extracción de características | Clasificación |
| (Filho et al., 2014) | Adquisición de imágenes | Extracción de características | Clasificación | ||
| (Backes & Sá Junior, 2016) (de Andrade et al., 2020) | Adquisición de imágenes | Preprocesamiento de imágenes | Segmentación de imágenes | Extracción de características | Clasificación |
| (Kwon et al., 2017) | Preparación de la muestra | Extracción de características | Clasificación | ||
| (de Andrade et al., 2020) | Preparación de la muestra | Adquisición de imágenes | Preprocesamiento de imágenes | Extracción de características | Clasificación |
| Presente trabajo | Obtención de muestras | Preparación de las muestras | Corte en sección transversal | Adquisición de imágenes | Clasificación |
Obtención de imágenes macroscópicas: Las imágenes macroscópicas fueron obtenidas en la sección transversal, se utilizaron: (i) un cuchillo ''Tramontina'', (ii) un cúter retráctil ''Pretul'' y (iii) una cuchilla ''Ubermann'' con láminas intercambiables. Al realizar el corte, se obtuvieron imágenes de la estructura anatómica macroscópica de las maderas con un microscopio portátil ''Dinolite'', el cual posee un aumento de hasta 250X, presenta un lente de 2.2 cm de diámetro correspondiente a 0.8 x 0.6 cm de la superficie de la madera e iluminación constante. Este microscopio cuenta con fuente de luz propia de 8 leds y mantiene las mismas condiciones de iluminación tanto de día como de noche.
Adicionalmente, su tamaño permite un uso práctico en campo, como en el puesto de control ''El Pedregal'' SERFOR, dónde es necesario subir a los camiones para inspeccionar las piezas de madera aserrada.
Para la captura de imágenes macroscópicas se utilizó el microscopio portátil con un aumento de 50X, este se conecta a un adaptador microUSB o tipo C al celular, luego a través de Play Store se descarga la aplicación denominada ''USB Camera'' para reconocer el microscopio portátil al momento de conectarlo al dispositivo móvil y finalmente obtener una imagen de 640 x 480 pixeles como se observa en la Fig. 1C y Fig. 1D respectivamente.
Se obtuvieron 50 imágenes por especie, estas fueron organizadas en una carpeta y permanecen en la nube con acceso directo para futuras investigaciones. Se obtuvieron 1 250 imágenes macroscópicas por cada herramienta de corte y se dispone de 3 750 imágenes en total, y estas se encuentran disponibles en el siguiente link: https://bit.ly/3K0CrRA.
Clasificación de especies maderables: El entrenamiento de imágenes macroscópicas de las 25 especies maderables se realizó con dos modelos de red neuronal convolucional: ''EfficientNet B0'' en la plataforma Google Colaboratory y ''Custom Vision'' en la plataforma de Microsoft Azure, para contrastar los porcentajes de clasificación.
Resultados
Dimensiones de una muestra: Determinar las dimensiones de una muestra de madera es esencial para una correcta manipulación y corte de la sección transversal, la captura de imágenes, su almacenamiento y conservación. Entre las dimensiones de muestras más utilizadas encontramos desde pequeñas (2 cm de alto x 1.5 cm de ancho x 4 cm de largo) (de Andrade et al., 2020) y medianas (4cm de alto x 5 cm de ancho y 10 cm de largo) (Kwon et al., 2017). Las dimensiones de las muestras se pueden observar en la Fig. 1A, el ancho de 2.5 cm facilita el corte de la superficie con las tres herramientas, en especial con la cuchilla Ubermann que posee una longitud máxima de 3 cm y el ancho de la muestra puede variar de 2 a 3 cm. Con respecto al largo de 6 cm, este estudio propone una longitud de 5 a 8 cm para mayor seguridad en el proceso de corte, ya sea fijado en un tornillo de banco o con las manos como se observa en la Fig. 2. Para el alto de las muestras se sugiere como mínimo 3 cm, dimensiones menores a este, podrían ocasionar accidentes o daño en los dedos. Las dimensiones pequeñas de las muestras permiten su almacenamiento y conservación para la estabilización de humedad y protección contra organismos xilófagos (Fig. 1B).

Fig. 2 Corte de muestras de madera; A. Posición de corte para maderas duras. B. Suaves. C. muestra fijada en un tornillo de banco. / Fig. 2. Cutting of wood samples; A. Cutting position for hardwoods. B. Soft. C. Specimen fixed in a vice.
Obtención de imágenes macroscópicas: Para obtener una alta calidad de imágenes macroscópicas se debe tener en cuenta el corte y el tipo de herramienta utilizada, como se observa en la Fig. 3 con las imágenes macroscópicas de Cedrelinga cateniformis y Brosimum alicastrum. Además, es importante describir las características físicas y ergonómicas de las tres herramientas de corte presentes en la Tabla 2 para evaluar su rendimiento o capacidad en el uso de campo, laboratorio o puesto de control.

Fig. 3 A. Herramientas para el corte de muestras; cuchillo Tramontina, cúter retráctil Pretul y cuchilla Ubermann. B. Imagen macroscópica de Cedrelinga cateniformis. C. Imagen macroscópica de Brosimum alicastrum. / Fig. 3. A. Tools for cutting samples; Tramontina knife, Pretul retractable cutter and Ubermann blade. B. Macroscopic image of Cedrelinga cateniformis. C. Macroscopic image of Brosimum alicastrum.
Tabla 2 Diferencias entre las tres herramientas de corte. / Table 2. Differences between the three cutting tools.
| Herramientas de corte | Descripción | Rendimiento en maderas | |||
| Mango | Lámina | Suaves | Duras | ||
| Cuchillo Tramontina | Policarbonato reforzado con fibra de vidrio | Acero inoxidable | Corta con facilidad | Pierde el filo rápidamente | |
| Cúter retráctil Pretul | Plástico ABS | Acero al carbono intercambiable | Corta con facilidad | No tiene la fuerza para hacer el corte | |
| Cuchilla Ubermann | Goma termoplástica | Acero inoxidable intercambiable | Corta con facilidad | Es resistente y duradera | |
En la Fig. 3, se presenta la calidad de las imágenes con las tres herramientas de corte y corresponden a las especies Cedrelinga cateniformis cuya densidad de 0.50 g/cm3 genera una resistencia blanda al corte y Brosimum alicastrum cuya densidad de 0.70 g/cm3 genera una resistencia medianamente dura al corte (Richter & Dallwitz, 2019), obteniéndose una alta calidad de corte con el estilete retráctil Pretul.
Las imágenes presentes en la Fig. 4 muestran una mejor visualización de sus características anatómicas en orientación vertical en función de sus radios, estas imágenes fueron obtenidas al hacer el corte de la sección transversal de la madera con el estilete retráctil Pretul.

Fig. 4 Imágenes macroscópicas de 25 especies con aumento de 50X. A. Caryocar glabrum. B. Allantoma decantra. C. Hura crepitans. D. Cedrela angustifolia. E. Cedrela odorata. F. Brosimum alicastrum. G. Copaifera paupera. H. Otoba glycycarpa. I. Poulsenia armata. J. Brosimum utile. K. Ocotea aciphylla. L. Aiouea montana. M. Ficus insípida. N. Calophyllum brasiliense. O. Jacaranda copaia. P. Sterigmapetalum obovatum. Q. Schizolobium parayba. R. Protium altissima. S. Hymenopus heteromorphus. T. Ocotea gracilis. U. Matisia cordata. V. Cedrelinga cateniformis. W. Clarisia racemosa. X. Terminalia oblonga. Y. Enterolobium sp. / Fig. 4. Macroscopic images of 25 species with 50X magnification. A. Caryocar glabrum. B. Allantoma decantra. C. Hura crepitans. D. Cedrela angustifolia. E. Cedrela odorata. F. Brosimum alicastrum. G. Copaifera paupera. H. Otoba glycycarpa. I. Poulsenia armata. J. Brosimum utile. K. Ocotea aciphylla. L. Aiouea montana. M. Ficus insipidus. N. Calophyllum brasiliense. O. Jacaranda copaia. P. Sterigmapetalum obovatum. Q. Schizolobium parayba. R. Protium altissima. S. Hymenopus heteromorphus. T. Ocotea gracilis. U. Matisia cordata. V. Cedrelinga cateniformis. W. Clarisia racemosa. X. Terminalia oblonga. Y. Enterolobium sp.
Para una captura efectiva de la imagen se requiere realizar el corte en un área mínima de 2.5 cm de ancho x 2.5 cm de largo en la sección transversal. Incluso, las redes neuronales convolucionales pueden clasificar las imágenes macroscópicas en cualquier ángulo de orientación en relación con sus elementos anatómicos. Esto gracias al aumento de datos (Data Augmentation) que permite girar, incrementar o disminuir las imágenes (zoom).
Clasificación de especies maderables: Con las 3 750 imágenes macroscópicas se entrenaron los modelos de redes neuronales convolucionales EfficientNet B0 en la plataforma de Google Colaboratory y el servicio Custom Vision de Microsoft Azure, las métricas de precisión se pueden observar en la Fig. 5 y Fig. 6, respectivamente.
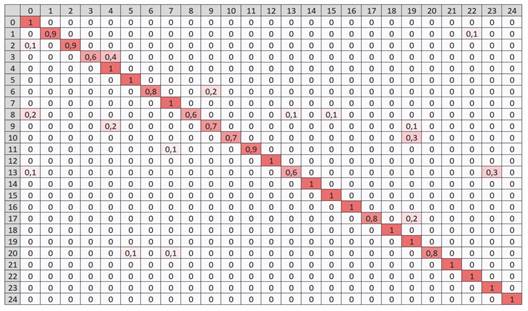
Fig. 5 Matriz de confusión del modelo EfficientNet B0 con imágenes macroscópicas obtenidas con el cúter retráctil Pretul. / Fig. 5. Confusion matrix of the EfficientNet B0 model with macroscopic images obtained with the Pretul retractable cutter.
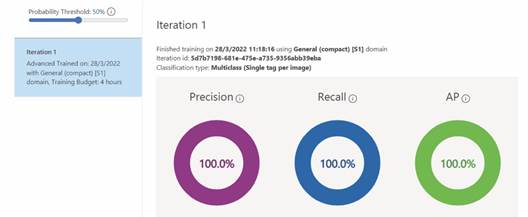
Fig. 6 Métricas de precisión del modelo ''Custom Vision'' en la plataforma de Microsoft Azure con imágenes macroscópicas obtenidas con el cúter retráctil Pretul. / Fig. 6. Accuracy metrics of the ''Custom Vision'' model on the Microsoft Azure platform with macroscopic images obtained with the Pretul retractable cutter.
La numeración de las 25 especies maderables presenta el mismo orden de la Tabla 3 es decir, el número 0 en la matriz de confusión corresponde a la especie número 1: Caryocar glabrum (almendro) de la Tabla 3 y el número 24 de la matriz de confusión corresponde a la especie número 25: Enterelobium sp. (yarahuisca) descritas en la Tabla 3. La línea diagonal representa el porcentaje de clasificación correcta que realiza el modelo en la fase de validación. Por ejemplo, la especie Cedrela angustifolia fue clasificada correctamente con un 60 % de precisión, sin embargo, el modelo lo confunde con Cedrela odorata en un 40 %, esto debido a la similitud de sus características anatómicas como el tipo de porosidad semi-poroso y la demarcación de anillos de crecimiento por bandas de parénquima marginal (Ferreira & Inga, 2022), además, los poros presentan diámetros distintos asociados a una coloración más clara a rosada en C. angustifolia y roja en C. Odorata con poros más grandes cerca del parénquima marginal.
Tabla 3 Lista de 25 especies maderables de Selva Central del Perú (Ferreira et al., 2021). / Table 3. List of 25 timber species from the Central Jungle of Peru (Ferreira et al., 2021).
| N | Nombre común | Nombre científico | Familia |
| 1 | Almendro | Caryocar glabrum (Aubl.) | Caryocaraceae |
| 2 | Cachimbo | Allantoma decantra (Ducke) | Lecythidaceae |
| 3 | Catahua | Hura crepitans L. | Euphorbiaceae |
| 4 | Cedro | Cedrela angustifolia Sessé & Moc | Meliaceae |
| 5 | Cedro | Cedrela odorata L. | Meliaceae |
| 6 | Congona | Brosimum alicastrum Sw. | Moraceae |
| 7 | Copaiba | Copaifera paupera (Herzog) | Fabaceae |
| 8 | Cumala | Otoba glycycarpa (Ducke) | Myristicaceae |
| 9 | Lanchan | Poulsenia armata (Miq.) | Moraceae |
| 10 | Leche Caspi | Brosimum utile (Kunth) Oken | Moraceae |
| 11 | Moena Amarilla | Ocotea aciphylla (Nees & Mart.) | Lauraceae |
| 12 | Moena Blanca | Aiouea montana (Sw.) R. Rohde | Lauraceae |
| 13 | Oje | Ficus insipida Wildenow | Moraceae |
| 14 | Palo Azufre/lagarto caspi | Calophyllum brasiliense Cambess. | Calophyllaceae |
| 15 | Palo Carton | Jacaranda copaia (Aublet) | Bignoneaceae |
| 16 | Palo Verde | Sterigmapetalum obovatum Kuhlm. | Rhizophoraceae |
| 17 | Pino Chuncho | Schizolobium parayba (Vell.) | Fabaceae |
| 18 | Pochotaroque | Protium altissimum (Aubl) Marchand | Burseraceae |
| 19 | Sachapalta | Hymenopus heteromorphus (Benth.) | Chrysobalanaceae |
| 20 | Moena | Ocotea gracilis (Meisn.) Mez | Lauraceae |
| 21 | Sapote | Matisia cordata Ducke | Malvaceae |
| 22 | Tornillo | Cedrelinga cateniformis (Ducke) | Fabaceae |
| 23 | Tulpay | Clarisia racemosa Ruiz & Pav. | Moraceae |
| 24 | Yacushapana | Terminalia oblonga (Ruiz & Pavon) | Combretaceae |
| 25 | Yarahuisca | Enterolobium sp. Mart. | Fabaceae |
Dentro del modelo EfficientNet B0, el 80 % de imágenes fue para entrenamiento y el 20 % para validación y para las pruebas se realizaron con imágenes que no están dentro de la base de datos, mientras que en el modelo ''Custom Vision'' de la plataforma Microsoft Azure no se puede acceder a la distribución de imágenes para dividir imágenes en la fase de entrenamiento y validación. Sin embargo, recomienda el uso de 50 imágenes como mínimo por especie para realizar una buena clasificación. Por esta razón, el entrenamiento se realizó con 50 imágenes por especie en un tiempo de 4 h como se muestra en la Fig. 6, mostrando un 100 % de precisión en la clasificación.
La confusión entre especies que pertenecen a la misma familia es muy común en las maderas debido a la semejanza de sus características anatómicas, es por ello que el modelo de EffficientNet B0 muestra como las especies de la familia Cedrela sp. se confunden entre ellas en porcentajes distintas al entrenar las imágenes macroscópicas con las tres herramientas de corte. La Tabla 4 muestra que solo 25 imágenes de la especie de Cedrela angustifolia fueron clasificadas correctamente por el modelo y 14 imágenes que representa el 27.78 % fueron confundidas con la especie de Cedrela odorata al preparar la base de datos con la herramienta de Cuchillo Tramontina, estos datos se van mejorando con las otras herramientas de corte como se muestra en la Tabla 4.
Tabla 4 Porcentaje de clasificación de imágenes con herramientas de corte distintos para dos especies de Cedrela sp. con el modelo de red neuronal convolucional ''EfficientNet B0''. Table 4. Percentage of classification of images with different cutting tools for two species of Cedrela sp. with the convolutional neural network model ''EfficientNet B0''
| Herramientas de corte | Especies donde el modelo se confunde al clasificar | |
| Cedrela angustifolia | Cedrela odorata | |
| Clasifica correctamente (%) | Clasifica erróneamente (%) | |
| 1. Cuchillo Tramontina | 50 | 27.78 |
| 2. Cúter Retráctil Pretul | 63.63 | 36.36 |
| 3. Cuchilla Ubermann | 72.22 | 27.77 |
La matriz de confusión de la Fig. 6 muestra la similitud que hay entre dos especies de diferentes familias al ser entrenadas con tres bases de datos diferentes y que los tres muestran confusión entre las especies de Calophyllum brasiliense y Terminalia oblonga como se muestra en la Tabla 5, es decir, 28 imágenes de la especie Calophyllum brasiliense de fueron clasificadas correctamente y los otros 22 fueron clasificadas erróneamente y confundidas con las especie de Terminalia oblonga al realiza el entrenamiento con la base de datos preparada con la herramienta de corte Cuchillo Tramontina que mostró solo el 55.56 % de precisión.
Tabla 5 Porcentaje de clasificación de imágenes con herramientas de corte para dos especies con el modelo ''EfficientNet B0'' / Table 5. Percentage of image classification with cutting tools for two species with the ''EfficientNet B0'' model.
| Herramientas de corte | Especies donde el modelo se confunde al clasificar | |
| Calophyllum brasiliense | Terminalia oblonga | |
| Clasifica correctamente (%) | Clasifica erróneamente (%) | |
| 1. Cuchillo Tramontina | 55.56 | 33.33 |
| 2. Cúter Retráctil Pretul | 78.57 | 7.14 |
| 3. Cuchilla Ubermann | 63.63 | 36.36 |
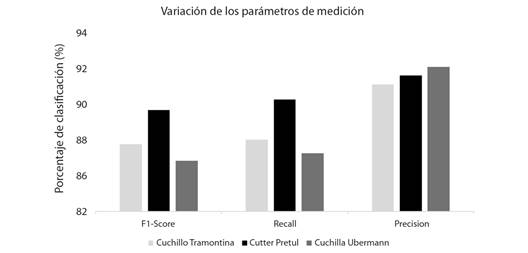
La diferencia en la precisión del análisis de imágenes macroscópicas obtenidas con las tres herramientas de corte es de solo 0.6 % como se visualiza en la Tabla 6, este resultado indica semejanzas en la calidad de corte de las muestras. El F1-Score es el parámetro principal y representa la media armónica entre la precisión y el Recall incorporando en una sola métrica los falsos positivos (precisión) y los negativos (Recall) (Berzal, 2018). El mejor resultado de F1-Score se logró al entrenar las imágenes obtenidas con el cúter retráctil Pretul con 88.8 % y la diferencia entre Recall y precisión es de tan solo 1.6 % en comparación con los valores de 3.7 y 5.8 % para el cuchillo Tramontina y la cuchilla Ubermann.
Tabla 6 Resultados de entrenamiento de los dos modelos CNN con las herramientas de corte. / Table 6. Training results of the two CNN models with the cutting tools
| Herramientas de corte | Modelos CNN | N° de imágenes | |||||
| EfficientNet B0 | Custom Vision | ||||||
| F1-Score (%) | Recall (%) | Precisión (%) | F1-Score (%) | Recall (%) | Precisión (%) | ||
| 1. Cuchilla Tramontina | 86.5 | 86.8 | 90.5 | 100 | 100 | 100 | 1 250 |
| 2. Cúter retráctil Pretul | 88.8 | 89.5 | 91.1 | 100 | 100 | 100 | 1 250 |
| 3. Cuchillo Ubermann | 85.4 | 85.9 | 91.7 | 100 | 100 | 100 | 1 250 |
La Fig. 7 muestra gráficamente la variación de los parámetros de medición que se indicaron en la Tabla 6, se puede ver que la precisión es el valor con mayor porcentaje superando considerablemente a los parámetros de F1-Score y Recall, con las herramientas de cuchillo Tramontina y cuchilla Ubermann. Sin embargo, el estile retráctil Pretul no muestra demasiada amplitud entre los tres parámetros.
Discusión
Las redes neuronales convolucionales (CNN) realizan la etapa de extracción de características de manera automática después de haber obtenido la base de imágenes macroscópicas como se muestra en la Tabla 1 y continua con la clasificación de especies. Anteriormente, diversos autores (Filho et al., 2014; Khalid et al., 2008; Kwon et al., 2017) consideraban la extracción de características de la madera como paso adicional y manual en el uso de métodos tradicionales, como SVM (Suport Vector Machine) (Gu et al., 2010), RF (Random Forest) (Breiman, 2001; Gonçalves et al., 2022) o LDA (Linear Discriminant Analysis) (Lens et al., 2020).
Las herramientas de corte son indispensables para la implementación de base de datos con imágenes macroscópicas de maderas. En condiciones de laboratorio, el cúter retráctil Pretul facilitó la obtención de imágenes macroscópicas con una mayor claridad de los elementos anatómicos. Sin embargo, no todas tienen la misma eficiencia en campo, en el caso del cuchillo Tramontina se pudo observar la necesidad de estar afilando constantemente para renovar el filo y disponer de una piedra de afilar siempre, el cúter retráctil Pretul al ser liviano y tener una envoltura hecha de un material duro de plástico ABS no es recomendable en maderas duras porque se gasta rápido la envoltura del material. Sin embargo, la cuchilla Ubermann resulto ser la más adecuada para usar en campo por tener láminas reemplazables en caso de perder filo y la envoltura de la herramienta es de acero lo cual no se desgasta al realizar cortes en maderas duras.
Al igual que la familia Meliaceae, existen varias especies que conforman una sola familia como Lauraceae, son complejas de identificar mediante la taxonomía botánica y aún más con la anatomía de la madera. Para ello, el modelo ''Custom Vision'' de Microsoft Azure tiene la facilidad de cargar imágenes y al culminar el entrenamiento la plataforma te permite realizar test de prueba, que consiste en cargar solo una imagen y clasificará de acuerdo con la similitud de características encontradas dentro de la base de datos. En la prueba se encontró que la precisión del 100 % disminuye a 89.8 % con la especie Cedrela angustifolia confundiéndose con Cedrela odorata en un 9.8 % como se observa en la Fig. 8A; Brosimum alicastrum disminuye a 53.2 % de probabilidad de reconocimiento confundiéndose en un 43.7 % con Terminalia oblonga presente en la Fig. 8B.

Fig. 8 Prueba rápida de A. Cedrela angustifolia. B. Brosimum alicastrum. C. Imagen de Ficus insipida de buena calidad. D. Imagen de Ficus insipida de baja calidad. / Fig. 8. Rapid test for A. Cedrela angustifolia. B. Brosimum alicastrum. C. Good quality image of Ficus insipida. D. Low-quality image of Ficus insipida.
La calidad de las imágenes es importante para una mayor precisión en la clasificación, la especie Ficus insípida presenta 99.7 % de reconocimiento con una buena calidad de imagen añadiendo características anatómicas peculiares como las bandas de parénquima axial (Ferreira & Inga, 2022) como se visualiza en la Fig. 8C, mientras que la misma especie con una imagen que no presenta un buen corte, solo alcanzó 36.8 % de precisión Fig. 8D.
Las ventajas del modelo EfficientNet B0 es el acceso al código de programación y dentro de ello se pueden hacer los cambios necesarios como aumento de datos, acceder a la base de datos de imágenes, cambiar el número de recorridos o epochs del modelo, seleccionar las imágenes de entrenamiento y validación, entre otros, los resultados del entrenamiento se pueden visualizar en la matriz de confusión de la Fig. 5. Mientras que el modelo Custom Vision de Microsoft Azure solo requiere cargar las imágenes en la plataforma y comenzar con el entrenamiento durante el tiempo que uno desee, desde 1 h hasta 96 h máxima, sin embargo, el costo de entrenamiento por hora es de $10 USD y los resultados de entrenamiento con tiempo mayor a 1 h es de 100 % de precisión. La ventaja principal es el interfaz amigable para entrenar imágenes sin tener conocimiento de programación o redes neuronales, la opción Quick Test del modelo Custom Vision permite subir una imagen y comprobar en tiempo real si reconoce o no la imagen, como se muestra en la Fig. 8.
La variedad de las imágenes es, sin duda, el factor principal para optimizar el rendimiento de cualquier modelo CNN, la calidad de la imagen en la Fig. 8D no es muy buena y por ende su rendimiento es menor, esto debido a que el modelo se entrenó con imágenes de alta calidad, frente a ellos es imprescindible entrenar modelos CNN con imágenes de alta y baja calidad para tener variabilidad en la base de datos. Sin embargo, la calidad de los cortes no es el único factor que podría generar variabilidad en la base de datos, también se debe mencionar a las muestras de madera frescas (muestras que han sido recién extraídas y máximo al quinto día se obtiene las imágenes) como se muestran en la Fig. 9A y Fig. 9B. Las muestras secas (pasaron el proceso de secado al aire libre sin presencia de lluvias) (Fig. 9C) y las muestras húmedas (estuvieron expuestos al sol y la lluvia) (Fig. 9D). Una sola especie puede presentar variedad en su estructura anatómica macroscópica y también en sus características generales como color, distribución de poros, entre otros, según el ambiente en que se encuentra, esto se realizó con la finalidad de generar mayor variabilidad en las imágenes que constituyen la base de datos y observar el comportamiento del modelo al realizar pruebas con estas imágenes.

Fig. 9 Imagen macroscópica de Schizolobium parayba. A.-B. Madera fresca. C. Madera seca. D. Madera húmeda. / Fig. 9. Macroscopic image of Schizolobium parayba. A.-B. Cool wood. C. Dry wood. D. Damp wood.
Declaración de ética: los autores declaran que todos están de acuerdo con esta publicación y que han hecho aportes que justifican su autoría; que no hay conflicto de interés de ningún tipo; y que han cumplido con todos los requisitos y procedimientos éticos y legales pertinentes. Todas las fuentes de financiamiento se detallan plena y claramente en la sección de agradecimientos. El respectivo documento legal firmado se encuentra en los archivos de la revista.












 uBio
uBio