Introducción
La resistencia antimicrobiana (RAM) es la capacidad de las bacterias, hongos, virus y parásitos de sobrevivir a los medicamentos a los que son expuestos. Esto dificulta el tratamiento de las infecciones causadas por estos agentes, propicia el desarrollo de formas más agresivas de las enfermedades (de importancia para la sanidad e inocuidad) e incrementa su propagación y mortalidad. Por ello, la Organización Mundial de la Salud (OMS) ha declarado a la RAM como una de las 10 principales amenazas para la salud pública (OMS, 2021).
De acuerdo con un estudio publicado en el 2019, se estimó que hubo un total de 4,95 (3,62-6,57) millones de muertes asociadas a RAM en bacterias. Las 6 especies reportadas con mayor frecuencia fueron Escherichia coli, Staphylococcus aureus, Klebsiella pneumoniae, Streptococcus pneumoniae, Acinetobacter baumannii, y Pseudomonas aeruginosa; las infecciones con estas contabilizaron un total de 3,57 (2,62-4,78) millones de muertes asociadas a RAM (Murray et al., 2022). Para el año 2050, el número de muertes alcanzarían los 10 millones por año y se espera una reducción del PIB (Producto Interno Bruto) del 2 al 3,5 % (O'Neill, 2014).
Los antibióticos son usados ampliamente en la crianza y en la prevención de enfermedades en animales destinados al consumo humano, sin embargo, el uso excesivo e irresponsable de estos agentes, junto con la administración de dosis en animales sanos ha contribuido al aumento de la RAM y la transmisión de bacterias resistentes a los humanos (van Boeckel et al., 2015). En los Estados Unidos, aproximadamente el 80 % del consumo en agentes antimicrobianos es empleado en animales destinados al consumo humano; muchos de estos agentes son utilizados también para el tratamiento de infecciones en humanos. Debido al incremento de la demanda de proteína de origen animal, se espera que el consumo de antibiócos también aumente. Las predicciones indican que entre el 2010 al 2030, se incrementará el consumo de agentes antimicrobianos en 67 %, de 63 151 ± 1560 toneladas a 105 596 ± 3605 toneladas (van Boeckel et al., 2015).
Uno de los puntos más controversiales en la administración de antibióticos en animales, es que, en lugar de tratar a los individuos enfermos, los medicamentos son aplicados a todo el grupo al cual pertenece el animal infectado (Ferri, et al., 2017). Este proceso es conocido como ''metafilaxis”. Algunos productores aplican esta estrategia incluso cuando no hay animales infectados, pero consideran que las condiciones de crianza podrían incrementar el riesgo de infecciones, por ejemplo, en casos de condiciones sanitarias no adecuadas o hacinamiento (McEwen & Collignon, 2018).
La acuicultura es una industria clave para satisfacer la demanda de alimentos para consumo humano. Tan solo en el 2010 se observó que la producción había aumentado cerca de 30 veces desde el año 1970, con una tasa de crecimiento anual de 7,8 %, pasando de producir 2,6 millones a 60,4 millones de toneladas por año (Troell et al., 2014).
En el Perú, en el año 2021 la producción de la acuicultura fue 144 206 toneladas.
Aproximadamente el 70 % de esta producción corresponde a concha de abanico, langostino, trucha y tilapia, en las regiones de Piura, Tumbes, Puno y San Martín, respectivamente (FAO, 2022; Ministerio de la Producción, 2022). Este crecimiento en la productividad podría estar acompañado de un incremento en el uso de antibióticos para prevenir y tratar infecciones bacterianas; por ello, el estudio tuvo como objetivo analizar la resistencia antimicrobiana en los principales recursos de la acuicultura peruana, para así poder establecer medidas más eficientes para la mitigación de este problema.
Metodología
Diseño del estudio, muestra y muestreo
El estudio fue de tipo observacional y descriptivo. Se realizó un muestreo por conveniencia. Se incluyeron especímenes de todos los centros de producción acuícola (CPA) que aceptaron participar en el estudio.
Los ejemplares de Argopecten purpuratus ''concha de abanico''(clase Bivalvia, familia Pectinidae) fueron obtenidos de 45 CPA en Sechura (37,5 % del total). Se tomaron 103 muestras, cada una estuvo conformada por 5 pools de 25 individuos cada uno.
Los ejemplares de Oncorhynchus mykiss ''trucha arcoíris''(clase Actinopterygii, familia Salmonidae) fueron obtenidos de 54 CPA en Puno (100 % del total). Se tomaron 56 muestras, cada una estuvo conformada por un pool de 5 individuos.
Los ejemplares de Oreochromis niloticus ''tilapia''(clase Actinopterygii, familia Cichlidae) fueron obtenidos de 38 CPA en San Martín (100 % del total). En total, se tomaron 79 muestras de cada uno de los 6 órganos evaluados (bazo, cerebro, hígado, intestino, músculo, riñón) haciendo un total de 474.
Los ejemplares de Litopenaeus vannamei ''camarón blanco''(clase Malacostraca, familia Penaeidae) fueron obtenidos de 33 CPA en Tumbes (44 % del total). Se tomaron 324 muestras, cada una estuvo conformada por un pool de 5 individuos.
Bacterias y asilamiento
Se evaluaron 3 géneros por su importancia en transmisión alimentaria: Enterococcus spp., Salmonella spp. y Escherichia coli. Adicionalmente, se estudiaron 3 géneros por su importancia en la sanidad animal; Vibrio parahaemolyticus fue evaluado en Argopecten purpuratus y Litopenaeus vannamei; Aeromonas spp., fue evaluado en Oncorhynchus mykiss y Oreochromis niloticus; mientras que, Flavobacterium psychrophilum en Oncorhynchus mykiss.
Para el aislamiento de Enterococcus spp., se utilizó el método estándar descrito en la ISO 7899-2:2000 (International Organization for Standardization, 2000); para Salmonella spp., se usó el procedimiento de la ISO 6579-1:2017 (excepto ítems 9.3.3, 9.4.3 porque no se analizan muestras de producción primaria y Anexo D debido a que se usa un agar cromogénico (Chrom Agar Salmonella Plus), que aísla el 100 % de los tipos de Salmonella, incluidos los del anexo D.) (International Organization for Standardization, 2017a) y para Escherichia coli se usó el procedimiento de la ISO 16649-3 (excepto ítems 4.1, 9.1 y 10.1) (International Organization for Standardization, 2015).
Para el aislamiento Vibrio parahaemolyticus se siguió el protocolo de la ISO 21872-1:2017 (International Organization for Standardization, 2017b); mientras que para Flavobacterium psychrophilum se utilizó el protocolo descrito por (Castillo Miranda, 2017).
Respecto a Aeromonas spp., se utilizó la técnica aplicada por Fernández et al., (2016); para ello el hisopado se siembra en agua peptonada alcalina (APA) y se incuba a 25 °C ± 1 °C por 24 horas. Luego se sembró en agar furunculosis (FA) y agar selectivo para Pseudomonas-aeromonas (GSP agar) y se incubó a 22 °C ± 1 °C y 25 °C por 48 horas, respectivamente. Se realizó la tinción Gram, prueba de oxidasa y catalasa y se seleccionaron las muestras positivas. Posteriormente, se efectúo la caracterización bioquímica para confirmar el aislamiento.
Antibiograma (INS, 2002)
Para la preparación del inóculo, se realizó un cultivo puro por 18 a 24 horas, en suspensión directa en 5 ml SSF al 0,85 %. Se estandarizó la turbidez de la suspensión al tubo 0,5 de la escala de Mc Farland (1,5 x 108 UFC/ml), mediante la medición de la turbidez en el espectrofotómetro. Para la inoculación en la placa, se impregnó el hisopo en la suspensión que tenía una concentración de 1,5 x 108 UFC/ml, se removió el exceso de líquido y se sembró en 3 direcciones por hisopado en la superficie de agar Mueller Hinton. Se secó la placa de 3 a 5 minutos y con la ayuda de pinzas estériles o dispensador de discos se colocaron los discos de antibióticos sobre la placa seca. Luego, las placas fueron incubadas a 35 ºC de 16 a 18 horas o por el periodo necesario, de acuerdo con el microorganismo a evaluar. Para la lectura de los antibiogramas, se usó el equipo Lector de zona de inhibición/ Contador de colonias automático (Interscience SCAN 4000). Los antibióticos evaluados, la concentración y rangos para interpretar el antibiograma se muestran en la Tabla 1.
Extracción de ADN y secuenciación
Se seleccionaron muestras que fueron resistentes en el antibiograma para evaluar-las mediante secuenciación masiva y ensamblaje del genoma microbiano completo. Realizado en forma colaborativa en 2 laboratorios, por capacidad operativa y duración del proyecto.
El primer grupo de muestras fue se-cuenciado en el laboratorio de una univer-sidad privada subcontratada para la ex-tracción del ADN, se inocularon colonias individuales en 2ml de medio TSB (Caldo tripticasa soya) para incubación a 37˚C y 300 rpm por 6 horas en un equipo Eppen-dorf Thermomixer (Thermo Fisher). Se centrifugaron los tubos a 10 000 rpm por 60 segundos. El pellet fue lavado 3 veces con PBS (buffer de fosfatos) 1x y resuspendido en 250ul de PBS 1x. Se extrajo el ADN del pellet usando el kit GeneJET Genomic DNA Purification Kit (Thermo Fisher), según las instrucciones del fabricante. El ADN puri-ficado se eluyó en 100ul de agua estéril, se determinó su concentración usando Qubit4 (Invitrogen) y se conservó a -20˚C hasta su procesamiento. Se prepararon librerías ge-nómicas a partir de 2ng de ADN purificado usando el kit Illumina DNA prep (Illumi-na cod 20060060), según instrucciones del fabricante. Las librerías fueron secuencia-das utilizando el Illumina NextSeq 550 y el kit Nextera 550 v2.5 de 300 ciclos (cod 20024908). Las secuencias crudas (en for-mato *.fastq) fueron editadas para eliminar la química de secuenciación y secuencias de mala calidad y ensambladas en la platafor-ma Pathogenwatch v2.0 para generar geno-mas completos. A partir de los ensamblajes, se utilizó la herramienta AMRFinderPlus 3.10 para identificar genes asociados a re-sistencia antimicrobiana.
El segundo grupo fue secuenciado en el Laboratorio de Sanidad Acuícola del SA-NIPES, el cual realizó un cultivo bacteriano a partir de las cepas identificadas y carac-terizadas como resistentes en las pruebas de difusión en disco. Para la extracción del ADN se utilizó el kit PrestoTM Mini gDNA Bacteria (Geneaid). Seguidamente, se veri-ficó la cantidad y calidad del ADN extraído mediante fluorometría y espectrofotome-tría con los equipos Qubit 4 Fluorometer (Invitrogen) y BioTek Epoch 2 Microplate Spectrophotometer (BioTek), respectiva-mente. Se realizó la secuenciación del ADN genómico bacteriano con el uso del kit Ra-pid Barcoding Kit SQK-RBK004 (Oxford Nanopore Technologies (ONT)) y una celda de flujo R9.4.1 (ONT) en la plataforma de
Tabla 1 Concentración y rangos de la zona de inhibición para la evaluación de los antibióticos analizados en el antibiograma
| Antibiótico | Concentración | Zona de inhibición (mm) | ||
|---|---|---|---|---|
| Resistente (R) | Intermedia (I) | Sensible (S) | ||
| Ampicilina | 10µg | ≤ 14 | 15-22 | ≥ 23 |
| Azitromicina | 15µg | ≤ 11 | 12-13 | ≥ 14 |
| Cloranfenicol | 30µg | ≤ 20 | 21-27 | ≥ 28 |
| Colistina | 10µg | ≤ 10 | 11-17 | ≥ 18 |
| Enrofloxacino | 30 mcg | ≤ 14 | 15-21 | ≥ 22 |
| Eritromicina | 15µg | ≤ 12 | 13-23 | ≥ 24 |
| Fosfomicina | 50µg | ≤ 21 | 22-30 | ≥ 31 |
| Oxitetraciclina | 30µg | ≤ 13 | 14-19 | ≥ 20 |
Nota: Fuente base de datos Scan 4000
secuenciación MinION Mk1C (ONT), siguiendo las especificaciones del fabricante. El pipeline bioinformático se ejecutó en la plataforma virtual NanoGalaxy partiendo desde los archivos FASTQ como data inicial. Se concatenaron los múltiples archivos FASTQ generados en cada Barcode (muestra) mediante la herramienta Concatenate dataset (cat). El FASTQ generado que alberga todos los reads de ese Barcode fue analizado con la herramienta NanoPlot v.1.28.2 para obtener las estadísticas de los reads obtenidos. Se filtraron las secuencias con un qscore ≥ 8 y con una longitud de read ≥ 1000 bp con la herramienta Filtlong v.0.2.1. Se procedió a limpiar las secuencias con la herramienta Porechop v.0.2.4. Las secuencias limpias fueron ensambladas de novo usando la herramienta Flye v.2.9. La secuencia ensamblada fue almacenada en formato fasta. Para la visualización del genoma ensamblado se utilizó la herramienta Bandage v.0.8.1 y la evaluación de la calidad del ensamblado fue realizada con la herramienta Quast v.5.2.0. Los genes de resistencia antimicrobiana fueron identificados a partir del genoma ensamblado (contigs contenidos en el archivo fasta). Se utilizaron las herramientas staramr v.0.8.0 y Web portal - RGI 6.0.0, CARD 3.2.5 para la identificación de genes de resistencia a antimicrobianos.
Análisis estadístico
Se calculó la frecuencia absoluta y porcentual, para el análisis estadístico se empleó el lenguaje de programación R versión 4.1.0 mediante el programa RStudio versión 1.4.17.17, asimismo para los datos del secuenciamiento se utilizaron los softwares Guppy y EPI2ME. Además, se estimaron los intervalos de confianza de Wald, intervalos de confianza exactos de Clopper-Pearson, y prevalencia agrupada.
Resultados
Argopecten purpuratus ''concha de abanico”
Se aislaron un total de 11 muestras positivas; de ellas, 9 muestras tuvieron importancia por la inocuidad alimentaria (E. coli); y 2, por sanidad animal (V. parahaemolyticus) (Tabla 2).
De las 9 muestras positivas para E. coli, se reportaron 2 resistentes a antibióticos (22,22 %). Ambas muestras fueron resistentes a oxitetraciclina, y sensibles a ampicilina, azitromicina, cloranfenicol y colistina; respecto a fosfomicina, el resultado fue intermedio. Las dos muestras fueron secuenciadas y se detectaron los GRA: qnrB81 y qnrB19, que confiere resistencia a quinolonas; tet(B), resistencia a tetraciclinas; y blaEC, resistencia a betalactámicos (Data suplementaria).
El 4,44 % de los CPA (n=2) presentaron muestras de E. coli resistentes. Ninguna muestra positiva para V. parahaemolyticus fue resistente y no se identificó Enterococcus spp. ni Salmonella spp.
Oncorhynchus mykiss ''trucha arcoíris”
En total, 10,71 % de las muestras fueron positivas para E. coli (n=6), 16,07 % positivas para Enterococcus spp. (n=9) y 3 muestras fueron positivas para Flavobacterium psychrophilum (5,38 %) (Tabla 2).
El 88,89 % de las muestras positivas para Enterococcus spp. fue resistente (n=8). De las 8 muestras resistentes, todas lo fueron para fosfomicina, 7 de las cuales mostraron, además, resistencia contra oxitetraciclina (87,5 %). Todas las muestras fueron sensibles a ampicilina, azitromicina, cloranfenicol y
Tabla 2 Proporción y prevalencia agrupada estimada para los casos positivos y resistentes, según las bacterias y especies evaluadas
| Especie | Bacteria | n | Aislamientos | % (IC95%) | P pooleed(IC95%) | Resistente | % (IC95%)*** |
|---|---|---|---|---|---|---|---|
| Argopecten purpuratus | Escherichia coli | 103 | 9 | 8,74 | 0,00073 | 2 | 22,22 |
| (3,28 - 14,19)* | (0,0003 - 0,0014) | (2,81 - 60,00)** | |||||
| Vibrio parahaemolyticus | 103 | 2 | 1,94 | 0,00016 | 0 | - | |
| (0,23 - 6,84)** | (0,00002 -0,0006) | ||||||
| Oncorhynchus mykiss | Escherichia coli | 56 | 6 | 10,71 | 0,022 | 0 | - |
| (2,61 - 18,82)* | (0,008 - 0,048) | ||||||
| Enterococcus spp. | 56 | 9 | 16,07 | 0,034 | 8 | 88,89 | |
| (6,45 - 25,69)* | (0,016 - 0,064) | (51,16- 99,72)** | |||||
| Flavobacterium psychrophilum | 56 | 3 | 5,38 | 0,011 | 0 | - | |
| (1,12 - 14,87)** | (0,002 - 0,032) | ||||||
| Oreochromis niloticus | Escherichia coli | 474 | 93 | 19,62 | - | 44 | 47,31 |
| (16,04 - 23,20)* | (37,16 - 57,46)* | ||||||
| Enterococcus spp. | 474 | 47 | 9,92 | - | 42 | 89,36 | |
| (7,23 - 12,61)* | (80,55 - 98,18)* | ||||||
| Salmonella spp. | 474 | 1 | 0,21 | - | 0 | - | |
| (0,01 - 1,17)** | |||||||
| Litopenaeus vannamei | Enterococcus spp. | 324 | 256 | 79,01 | 0,27 | 223 | 87,11 |
| (74,60 - 83,40)* | (0,24 - 0,30) | (83,00 - 91,21)* | |||||
| Vibrio parahaemolyticus | 324 | 311 | 95,98 | 0,47 | 109 | 35,05 | |
| (93,85 - 98,12)* | (0,42 - 0,54) | (29,74 - 40,23)* |
Nota: IC95%: intervalo de confianza al 95 %, n: número de muestras, Ppooled: prevalencia agrupada, *: Intervalos de confianza calculados con el método de Wald, * *: Intervalos de confianza calculados con el método de Clopper-Pearson, ***: respecto a aislamientos.
Fuente: propia de la investigación.
colistina; 4 de las muestras fueron secuenci das y se identificaron 9 GRA; los genes aa c(6')-Ii (n=2), aph(3')-IIIa (n=1) y ant(6)-Ia (n=1), asociados a la resistencia a aminoglu cósidos; tet(L) (n=2) y tet(M) (n=1), a tetraciclinas; msr(C) (n=2) y erm(B) (n=1), a macrólidos; lsa(A) (n=2), a lincosamidas y estreptograminas; y dfrE (n=2), a trimetoprima.
El 12,96 % de los CPA (n=7) presentaron Enterococcus spp. resistentes. Ninguna de las muestras positivas para E. coli o psychrophilum fue resistente y no se identificó Salmonella spp., ni Aeromonas spp.
Oreochromis niloticus ''tilapia”
El 19,62 % de las muestras fueron positivas para E. coli (n=93); 9,92 % para Enterococcus spp. (n=47) y 0,21 % para Salmonella spp. (n=1) (Tabla 2). Tanto para coli como para Enterococcus spp. las muestras positivas fueron mayormente tomadas en intestino. Salmonella spp. solo se aisló de cerebro (Figura 1). De las muestras positivas, 47,31 % de las muestras de E. coli (n= 44) y 89,36 % de las muestras de Enterococcus spp. (n=42) fueron resistentes. Cabe indicar, que para las muestras de tilapia se consideró el muestreo de hisopado por órganos, debido a que las muestras fueron tomadas en el departamento de San Martín (más de 8 horas de traslado por el método convencional) y tuvieron que ser trasladadas a provincia constitucional del Callao, donde se encuentran los Laboratorios de SANIPES, asegurando con ello la conservación de la muestra hasta, como máximo, por 24 horas.
Respecto a E. coli resistente, el 86,36 % (n=38) fueron resistentes a oxitetraciclina, 54,55 % (n=24) a ampicilina y 47,73 % n=21) a cloranfenicol. Además, 2 muestras fueron resistentes a fosfomicina y azitromicina (una por cada antibiótico) (Figura 2A). En referencia a la cantidad de antibióticos;
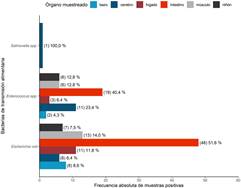
Fuente: propia de la investigación
Figura 1 Distribución de las muestras positivas de bacterias de transmisión alimentaria por el órgano de O. niloticus de donde se aisló.
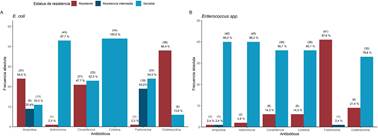
Fuente: propia de la investigación
Figura 2 Resultado del antibiograma de las muestras resistentes de Escherichia coli (A) y Enterococcus spp. (B) aisladas de Oreochromis niloticus.
36,4 % (n=16) fueron resistentes a 3, 20,4 % (n=9) a 2 y 43,2 % (n=19) a 1. Se secuencia ron 22 muestras resistentes y se detectaron 33 GRA. Los más frecuentes fueron tet(A) (n=18, 54,55%), aph(3'')-Ib (n=9, 27,27%), aph(6)-Id (n=9, 27,27 %), qnrB19 (n=9, 27,27%), blaEC (n=8, 24,24%), y sul2 (n=8, 24,24%) (Figura 3A).
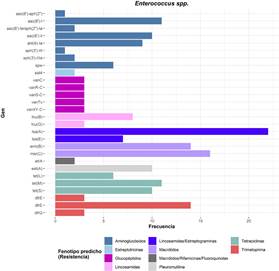
Respecto a las muestras de Enterococcus spp. resistentes (n=42), el 97,62 % (n=41) fue resistente a fosfomicina, mien tras que la resistencia al resto de antibióticos se mostró en un bajo porcentaje (Figura 2B). El 54,80 % (n=23) fueron resistentes a 1 solo antibiótico, 38,1 % (n=16) a 2, 4,8 % (n=2) a 3 y 2,4 % (n=1) a 4. Se secuenciaron 14 muestras resistentes y se detectaron 17 GRA. Los más frecuentes fueron aac(6')-I (n=7, 50,00%), asociado a la resistencia a aminoglucósidos; tet(M) (n=7, 50,00%), a tetraciclinas; y lsa(A) (n=6, 42,86%), a lincosamidas y estreptograminas (Figura 3B).
El 50,00 % de los CPA (n=19) presentaron muestras de E. coli resistentes y 36,84 % (n=14) Enterococcus spp. resistentes. La muestra positiva para Salmonella spp. no fue resistente y no se identificó Aeromonas spp.
Litopenaeus vannamei ''camarón blanco”
En total, 79,01 % de las muestras fueron positivas para Enterococcus spp. (n=256) y 95,90 % para V. parahaemolyticus (n=311). De las muestras positivas, 87,11 % de las muestras de Enterococcus spp. (n=223) y 35,05 % de las muestras de V. parahaemolyticus (n=109) fueron resistentes (Tabla 2).
Respecto a las muestras de Enterococcus spp. resistente a antibióticos, el 92,50 % n=213) fueron resistentes a fosfomicina y el 82,10 % (n=183), a colistina. También se encontraron muestras resistentes a ampicilina, azitromicina, cloranfenicol, enrofloxacino,
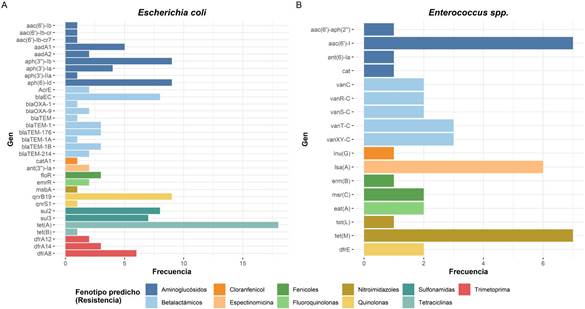
Fuente: propia de la investigación.
Figura 3 Genes de resistencia antimicrobiana encontrados en muestras de E. coli (A) y Enterococcus spp. (B) resistentes aisladas de Oreochromis niloticus.
eritromicina y oxitetraciclina (Figura 4A). La mayoría de las muestras fueron resisten-tes a 2 antibióticos (64,60 %) (Figura 4B). Se secuenciaron 57 muestras y se detectaron 28 GRA. Los más frecuentes fueron lsa(A), msr(C), erm(B), dfrE, tet(M), aac(6')-I, ea-t(A), tet(S) y aac(6')-Ii (Figura 5).
De las muestras con V. parahaemolyti-cus resistente a antibióticos, el 97,20 % (n=106) fueron resistentes a ampicilina. Se evidenció resistencia a fosfomicina, colisti-na, oxitetraciclina y azitromicina, pero con frecuencia muy baja (Figura 4b). El 92,70 % (n=101) de las muestras fueron resisten-tes a un solo antibiótico y el 7,30 % (n=8) fue resistente a 2.
Se secuenciaron 53 muestras resis-tentes y se detectaron 7 GRA; blaCARB-21 (n=33, 62,26%), CARB-20 (n=24, 45,28%), blaCARB-20 (n=19, 35,85%) y blaCARB (n=1, 1,89%), asociados a la resistencia a betalactámicos; tet(34) (n=20, 37,74%) y tet(35) (n=20, 37,74%), a tetraciclinas; y CRP (n=15, 28,30%), a macrólidos.
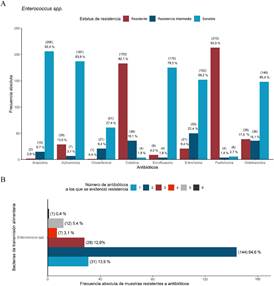
Fuente: propia de la investigación
Figura 4 Resultado del antibiograma (A) y número de antibióticos a los que fueron resistentes (B) las muestras de Enterococcus spp. aisladas de Litopenaeus vannamei.
El 100,00 % de los CPA (n=33) pre-sentaron muestras de Enterococcus spp. resistentes y 78,79 % (n=26) V. parahae-molyticus resistentes. No se identificó E. coli ni Salmonella spp.
Discusión y conclusiones
La RAM es uno de los principales riesgos de salud pública en el mundo y se ha demostrado que las bacterias resistentes pue-den propagarse no solo de persona a persona, sino también de animales a personas (Verraes et al., 2013). La acuicultura es de especial in-terés dado que, usualmente, los antibióticos son administrados con el alimento a toda la población (metafilaxis); sin embargo, estos alimentos medicados podrían ser consumi-dos principalmente por los peces más gran-des y sanos, y no necesariamente por los enfermos. Debido a esto, es difícil controlar que se consuman dosis exactas, a la vez que es necesario aplicar dosis mayores a las recomendadas (Romero, Feijoo, & Navarrete, 2012).
Fuente: propia de la investigación
Figura 5 Genes de resistencia antimicrobiana encontrados en muestras Enterococcus spp. (B) aisladas de Oreochromis niloticus.
Además, los antibióticos no son metabolizados totalmente y entre el 70 % y 80 % de ellos son excretados al agua y después expulsados al sistema de alcantarillas y a otras fuentes de agua (Food and Agriculture Organizations of the United Nations, n.d.). Esto conduce a la acumulación de antibióticos en el medio acuático, sean piscigranjas o sedimentos marinos, lo que favorece la selección de microorganismos resistentes (Hollis & Ahmed, 2014).
En el presente estudio se evaluó la resistencia antimicrobiana en las principales especies cultivadas en la acuicultura peruana. En Piura, solo el 4,44 % de los CPA de concha de abanico (Argopecten purpuratus) presentaron de E. coli resistentes a oxitetraciclina, siendo esta la única bacteria en la que se encontró RAM. En el análisis genómico, se puedo identificar tet(B), que explica la resistencia a tetraciclinas y, adicionalmente, genes asociados a la resistencia a quinolonas y betalactámicos. Solo 2 de los 9 aislados (22,22 %) de E. coli fueron resistentes, lo que es comparativamente menor a lo reportado antes, donde el 55,56 % de los aislados fueron resistentes a cefotaxima, ceftazidima, cefepima, ciprofloxacina, meropenem o cotrimoxazol (Alejos, 2017).
Por otro lado, en Puno, el 12,96 % de los CPA de trucha arcoíris (Oncorhynchus mykiss) y 88,89 % de los aislados presentaron Enterococcus spp. resistentes a fosfomicina y oxitetraciclina. Aunque no se detectó Aeromonas spp., en estudio realizado en el año 2019 en 4 regiones del Perú, se pudieron identificar los genes de resistencia sul1 y tetA en el 6,6 % (2/30) y 3,3 % (1/30) de los aislados, respectivamente (Hurtado, 2019).
Es importante recalcar que en San Martín y en Tumbes la frecuencia de RAM fue mayor. En San Martín, el 50,00 % y 36,84 % de los CPA de tilapia (Oreochromis niloticus) presentaron muestras de E. coli y Enterococcus spp. resistentes, respectivamente; lo que, a su vez, correspondía al 47,31 % de aislados de E. coli y 89,36 % de Enterococcus spp. Mientras que en Tumbes el 100,00 % y 78,79 % de los CPA de camarón blanco (Litopenaeus vannamei) presentaron muestras de Enterococcus spp. y Vibrio parahaemolyticus resistentes, respectivamente. Los resultados de frecuencia de Vibrio spp. en camarón blanco fueron similares a lo reportado por Grande FJ. (2020), quien encontró que el 76,46 % de los aislados mostró resistencia a ampicilina, 35,29 % a tetraciclina y oxitetraciclina, 0,84 % a trimetropin/sulfametoxazol y 0,84 % a enrofloxacina; además de encontrar GRA asociados a la resistencia a tetraciclinas.
En este estudio se pudo detectar GRA en las 4 especies evaluadas. Estos genes son importantes ya que además de infectar los peces y otros alimentos de origen marino, pueden transmitir dicha resistencia a patógenos que infectan a los humanos. Un experimento in vitro demostró que es posible transmitir genes de resistencia antimicrobiana (GRA) de Vibrio cholerae a Vibrio parahaemolyticus (Sørum, 2006). Adicionalmente, existen varios estudios que apoyan la existencia de una asociación entre el desarrollo de RAM en acuicultura y RAM en humanos. Se ha descrito el mismo perfil de genes de resistencia antimicrobiana en bacterias de peces y aislados clínicos humanos, y alrededor de la mitad de los GRA identificados en patógenos de peces son comunes a los identificados en patógenos humanos (Furushita et al., 2003; Rhodes et al., 2000; Sørum, 2006). En Japón, se encontró que, los genes que codifican para la resistencia a la tetraciclina en las bacterias de peces cultivados y en aislamientos clínicos tienen una gran similitud, lo que sugiere que pueden tener su origen en la misma fuente. Esta observación fue reforzada por experimentos de laboratorio en los que la resistencia a la tetraciclina de cepas marinas de Photobacterium, Vibrio, Alteromonas y Pseudomonas fueron transferidos a E. coli por conjugación, lo que apunta a la viabilidad de transferir determinantes de resistencia de bacterias marinas a bacterias asociadas con los humanos (Furushita et al., 2003).
En conclusión, se pudo identificar que existe resistencia antimicrobiana en la acuicultura peruana, siendo especialmente alta en los CPA de O. niloticus (San Martín) y L. vannamei (Tumbes). Es necesario realizar estudios que permitan establecer los factores asociados a esta situación y establecer medidas que prevengan y mitiguen el impacto que podría causar.
Agradecimiento
Agradecemos a todos los centros de producción acuícola que nos facilitaron el acceso a las muestras.
Declaración de la contribución de los autores
Todos los autores afirmamos que se leyó y aprobó la versión final de este artículo.
El porcentaje total de contribución para la conceptualización, preparación y corrección de este artículo fue el siguiente: C.D.C 12 %, E.H.R. 12 %, J.C.S. 12 %, R.Z.V 12 %, L.R. 10 %, D.R.A. 10 %, C.A. LL. 10 %, L.A.S. 10 %, C.A.V. 12 %.















