Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista de Biología Tropical
On-line version ISSN 0034-7744Print version ISSN 0034-7744
Rev. biol. trop vol.59 n.3 San José Sep. 2011
Caracterización fenotípica y genotípica de doce rizobios aislados de diversas regiones geográficas de Venezuela
María Eugenia Marquina, Néstor Enrique González & Yulimar Castro
Laboratorio de Fijación Biológica de Nitrógeno y Cultivos de Tejidos Vegetales in vitro, Departamento de Biología, Facultad de Ciencias, Universidad de Los Andes. Núcleo Pedro Rincón Gutiérrez, Edificio A, 2º piso, La Hechicera, Mérida, Venezuela; martu@ula.ve, ngonzalezv2007@gmail.com, yulica@gmail.com
Dirección para correspondencia
Laboratorio de Fijación Biológica de Nitrógeno y Cultivos de Tejidos Vegetales in vitro, Departamento de Biología, Facultad de Ciencias, Universidad de Los Andes. Núcleo Pedro Rincón Gutiérrez, Edificio A, 2º piso, La Hechicera, Mérida, Venezuela; martu@ula.ve, ngonzalezv2007@gmail.com, yulica@gmail.com
Dirección para correspondencia
Abstract
Phenotypic and genotypic characterization of twelve rhizobial isolates from different regions of Venezuela. Rhizobial taxonomy and systematics have progressed substantially, nevertheless, few studies have been developed on venezuelan species. This study evaluated the phenotypic and genetic variation between 12 venezuelan indigenous rhizobial isolates and 10 international referential strains, by phenotypical traits and DNA molecular markers. In this regard, a PCR-RFLP of the 16S rDNA gene, the presence of large plasmids, metabolic assays in solid media, salinity resistance, pH and temperature growth conditions, and intrinsic antibiotic resistance were assayed. In reference to the phenotypic attributes, we recognized three main groups: A group I, which comprised all the strains metabolizing between 67.5%-90% of the C and N sources. They were also acid-tolerant, as well as acid producers, capable of growing at 40ºC and in high salinity conditions (2-2.5% NaCl). With regard to the antibiotic sensitivity, this group was susceptible to a 30% of the antibiotic assayed. Strains belonging to Group II exhibited a lower salt tolerance (0.1-1.5%NaCl), as well as a lower acid tolerance, since they grew well at pH values equal or higher than 5.0. This group appeared to be resistant to all of the antibiotics assayed and only metabolized between 52.5%-82.5% of the C and N sources. Group III was represented by a single bacterial strain: it has a extremely low salt tolerance (0.1% NaCl). This strain grew at a pH equal or higher than 5.6, was susceptible to 50% of the antibiotics assayed and metabolized 72% of the C and N sources. On the basis of a PCR- RFLP of the 16S rDNA, three groups were also obtained. Members of the group A showed a close resemblance to Rhizobium tropici CIAT 899 and Sinorhizobium americanum CFN-EI 156, while Group B was closely related to Bradyrhizobium spp. Group C, was also represented by only one isolate. The Trebol isolate, was the only one strain able to form nodules and does not appear to be related to any of the referential rhizobial strains, suggesting a possible symbiotic horizontal gene transfer. Finally, in this work, there are evidences of a genetic diversity in the venezuelan rhizobial strains. A different geographical origin is perhaps an important factor affecting the diversity of the indigenous rhizobia in this study. Rev. Biol. Trop. 59 (3): 1017-1036. Epub 2011 September 01.
Key words: Rhizobium, Bradyrhizobium, phenotypic characterization, PCR-RFLP, 16S rDNA.
Resumen
Rasgos fenotípicos y marcadores moleculares de ADN se utilizaron para investigar la variación fenotípica y genética entre 12 aislados rizobianos venezolanos y 10 cepas de referencia internacionales. Para ello, se realizó un PCR-RFLP del gen rDNA 16S, la presencia de plásmidos grandes, análisis metabólicos en medios sólidos, resistencia a la salinidad, condiciones del crecimiento a diferentes pH y temperaturas y la resistencia intrínseca a antibióticos. En referencia a las cualidades fenotípicas, se diferenciaron tres grupos principales, un grupo I que abarcó a todas aquellas cepas que metabolizaban entre 67.5% y 90% de las fuentes de C y de N. También eran tolerantes a la acidez y productoras de ácido, capaces de crecer a 40ºC y a altas condiciones de salinidad (NaCl 2-2.5%). Con respecto a la sensibilidad a antibióticos, este grupo era susceptible a un 30% de los antibióticos empleados. Las cepas que pertenecen al grupo II exhibieron una tolerancia salina más baja (0.1- 1.5%NaCl), así como una menor tolerancia a la acidez, puesto que crecieron bien en valores de pH iguales o mayores a 5.0. Este grupo era resistente a todos los antibióticos probados y metabolizaban solamente entre 52.5%-82.5% de las fuentes de C y de N. Una sola cepa bacteriana representó al grupo III, con una baja tolerancia salina (0.1% NaCl). Este aislado creció a un pH mayor o igual a 5.6, era susceptible a 50% de los antibióticos probados y metabolizó el 72% de las fuentes de C y de N. Al tener como base el PCR-RFLP del 16S rDNA, se diferenciaron también tres grupos. Los miembros del grupo A demostraron una estrecha relación con Rhizobium tropici CiAT 899 y Sinorhizobium americanum CFN-Ei156, mientras que el grupo B está estrechamente vinculado a Bradyrhizobium spp. El grupo C, está representado por solo un aislado. El aislado Trebol, fue la única cepa capaz de formar nódulos y no aparece relacionado con ninguna cepa de referencia, y sugiere una transferencia horizontal de genes simbióticos. Finalmente, en este trabajo se evidencia una diversidad genética en las cepas rizobianas venezolanas. El origen geográfico diverso de estas cepas, quizás sea un factor importante que influencie la diversidad de los rizobios indígenas utilizados en este estudio.
Palabras clave: Rhizobium, Bradyrhizobium, caracterización fenotípica, PCR-RFLP, ADNr 16S.
Las bacterias denominadas comúnmente rizobios presentan varias formas de vida; pueden comportarse como saprófitos en el suelo establecer una asociación simbiótica y formar nódulos con las raíces y tallos de las leguminosas, donde tiene lugar la reducción del nitrógeno atmosférico en amonio, el cual es transportado a la planta y convertido en biomoléculas esenciales, o bien estar presentes como endófito en raíces de diferentes especies vegetales, donde ejercen efectos promotores del crecimiento (Wang et al. 2001).
La taxonomía rizobiana basada tanto en técnicas moleculares como en las aproximaciones polifásicas ha permitido reconocer seis géneros hasta la actualidad, los cuales son: Allorhizobium, Mesorhizobium, Azorhizobium, Sinorhizobium, Bradyrhizobium y Rhizobium (Wang et al. 2001), los mismos constituyen un grupo filogenéticamente heterogéneo en el que la taxonomía es continuamente reexaminada, como en el caso del género Sinorhizobium, cuyo nombre fue cambiado por la denominación más antigua Ensifer (Young 2003) y los géneros Allorhizobium y Agrobacterium, que han sido incluidos dentro del género Rhizobium (Young 2001).
La taxonomía y sistemática rizobiana ha progresado notablemente por el análisis comparativo del ADNr 16S (Young 2001) y las aproximaciones taxonómicas polifásicas, las cuales incluyen características morfológicas, bioquímicas, fisiológicas y genéticas, que junto a los ensayos de infectividad en un rango de huéspedes, permiten que la taxonomía tenga una base más natural y confiable (Wang et al. 2001).
El ADN ribosómico 16S constituye una unidad genética de gran interés evolutivo, presente en los organismos eucariotas y procariotas: funciona como un cronómetro molecular pues preserva la historia evolutiva (Olsen & Woese 1993). El grado de divergencia de las secuencias del ADNr presentes en dos organismos, dan un estimado de las distancias evolutivas entre ellas (Ludwig & Schleifer 1994), lo que ha permitido estudiar la diversidad genética de los miembros de una población. Al respecto se ha señalado que el análisis del polimorfismo de la longitud de los fragmentos de restricción del amplificado del ADNr 16S digerido con enzimas de restricción (RFLP), ha permitido una identificación rápida de los aislamientos nodulares y la detección de nuevos taxa (Laguerre et al. 1994). Los mismos autores han demostrado que la combinación de cuatro enzimas (Cfo, HinfI, MspI y NdeII), tienen el mismo poder discriminativo que la combinación de nueve para separar cepas de Rhizobium en especies.
Los estudios de diversidad rizobial, particularmente en Venezuela, son escasos; entre ellos se puede señalar un trabajo realizado con aislados autóctonos de Bradyrhizobium sp., obtenidos de Lupinus spp. del estado Mérida, con diversos criterios de tipificación. Sus hallazgos dan cuenta de la diversidad de las poblaciones bacterianas fijadoras de nitrógeno (Vielma 1999). Los autores Sicardi-Mallorca et al. (1998) estudiaron la diversidad de rizobios nativos aislados de especies autóctonas de Sesbania en diferentes zonas de Venezuela, los cuales analizan características fenotípicas y genéticas. En el presente trabajo, se presenta una caracterización fenotípica, un análisis por PCR- RFLP y un perfil plasmídico de aislados rizobianos de diversas regiones de Venezuela, como un aporte al conocimiento de la diversidad rizobial del país.
Materiales y métodos
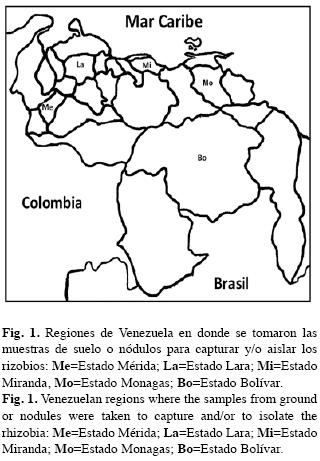
Aislados, cepas bacterianas de referencia y medios de cultivo: La ubicación geográfica de las regiones donde se tomaron las muestras de suelo o nódulos para capturar y/o aislar los rizobios se muestran en la Fig. 1. Todos los aislados y cepas de referencia (Cuadro 1, 2) han sido conservadas en glicerol (20%) a -20°C y cultivados en medio YM líquido modificado (manitol 2.5g/L, sacarosa 7.5g/L) (YMLm) (Vincent 1975). La pureza de dichos cultivos fue verificada mediante siembra por agotamiento en medios agarizados YM modificado con rojo congo (25μg/mL) (YMAmrc) y medio glucosa peptona más púrpura de bromocresol (100μg/mL) (GPA) (Rodríguez-Mendoza & Ferrera-Cerrato 1984).
Todos los ensayos se realizaron por triplicado con cultivos de 108cel/mL, incubados a 30°C, y en agitación continua (200rpm) cuando se trate de cultivos líquidos, salvo se indique lo contrario.
Prueba de infectividad: La autenticación de los aislados se realizó en Macroptilium lathyroides (Siratro) (Bromfield & Barran 1990) y Crotalaria sp. Previamente se escarificaron las semillas con papel lija Nº 280, y se esterilizó con hipoclorito 0.5% durante 7min; se germinaron a 26ºC en medio TY agarizado (Beringer 1974) y se sembraron en recipientes plásticos (7 x 15cm) con arena estéril, impregnada inicialmente con solución Hoagland (Taiz & Zeiger 1998) con el nitrógeno diluido a la mitad (nitrógeno de arranque). El ensayo contaba con 5 plantas/aislado y 5 plantas control sin inocular, estas últimas se fertilizaron semanalmente con solución de Hoagland original. Después de 35 días de inoculadas (5mL/planta) se reaislaron los rizobios a partir de nódulos mediante el procedimiento señalado en el Manual de Rhizobiología (CiAT 1988).
Caracterización fenotípica: Primero se determinó las características morfológicas de las colonias rizobianas (Cuadro 3) (CIAT 1988), el crecimiento en el medio Luria Bertani (LB) y la producción de ácido o base, de las cepas de referencia y aislados rizobianos (Vincent 1975, Rodríguez-Mendoza 1993). Para probar la tolerancia a varias temperaturas (20º, 27º, 30º, 35º y 40ºC) y a medios salinos con NaCl al 0.1, 1.0, 1.5, 2.0 y 2.5% (Wang et al. 1998) se colocaron 5μL de cada cultivo de rizobios en el medio YMAmrc.
La capacidad para tolerar pH ácidos se efectúo según Ayanaba et al. (1983) con modificaciones. Para ello se prepararon cajas de Petri con el medio de Keyser & Munns (1979) modificado, a pH 4.0, 4.4, 4.6, 5.0, 5.2, 5.6, 6.0 y 6.8. También se comprobó la resistencia intrínseca a antibióticos, los cuales fueron esterilizados por filtración y adicionados al medio YMAmrc, donde se sembraron alícuotas de 5μL de cada cultivo de rizobio. Los antibióticos ensayados fueron los siguientes (µg/ mL): gentamicina (Gmc.) 2.5, 5, 25, 50 y 100. Lincomicina (Lmc.), cloranfenicol (Cam.) 50, 100, 200, 300. Amikacina (Amkc.), ampicilina (Amp.), doxiciclina (Doxi.), ácido nalidíxico (Nal.) y tetraciclina (Tc.) 5, 25, 50, 100 y 200, kanamicina (Kan.) 5, 10, 25, 50, 8 y estreptomicina (Str.) 5, 10, 25, 50, 100 y 200 (Wang et al. 1998).
Para el ensayo de crecimiento en diferentes fuentes carbonadas y nitrogenadas, se cultivaron las bacterias en 5mL de medio YMmrc, en agitación continua (200rpm), durante 2-5días. Después se efectúo el lavado de las células con NaCl 0.8% y se permitió el crecimiento en medio basal salino M9 (Millar 1972) con biotina, sin fuentes carbonadas y nitrogenadas (carenciamiento) durante 24h. Posteriormente, se tomaron alícuotas de 5μL de cada una de los cultivos bacterianos y se sembraron en medio YM agarizado con azul de bromotimol (0.5mg/200mL) y las siguientes fuentes carbonadas esterilizadas por filtración: L-arabinosa, dulcitol, esculina, D-fructosa, D-galactosa, glicerol, glucosa, inositol, D-lactosa, maltosa, D- manosa, rhamnosa, rafinosa, sacarosa, L-sorbitol, L-sorbosa, D-xilosa y las sales de los ácidos orgánicos: succínico, cítrico, málico, pirúvico, α- cetoglutarato (todas a una concentración del 0.1%). El ácido nicotínico y ácido ascórbico se utilizaron a una concentración de 100 μg/mL (Wang et al. 1998). También se ensayaron 14 fuentes nitrogenadas esterilizadas por filtración: L-alanina, L-arginina, L-asparagina, L-fenilalanina, L- glicina, L-histidina, L-homoserina, L-leucina, L-metionina, L-treonina, L-triptofano, L-tirosina, L-valina y urea (todas a una concentración 0.5g /L) (Wang et al. 1998). Los cultivos bacterianos se incubaron a la misma temperatura que los ensayos anteriores. En todos los ensayos el crecimiento fue registrado de manera cualitativa durante 2-9 días.
Caracterización genotípica de rizobios
Perfil plasmídico: Se empleó una modificación del método de lisis alcalina para aislar plásmidos de alto peso molecular (Casse et al. 1979), para ello se utilizó cultivos frescos crecidos en agitación (200 rpm) durante 1-3 días en 10mL de medio YMLm.
La presencia de plásmidos de alto peso molecular fue verificada mediante electroforesis en gel de agarosa (0.9%) con tampón de corrida TBE 1X pH 8.0, 2h con 63 voltios y 100mA. Se utilizaron como marcadores de peso molecular cepas de E. coli K-12, portadoras de cinco plásmidos de referencia provenientes del Centro Venezolano de Colecciones de Microorganismos (CVCM) 1- CVCM 134, (pR478, 279 Kb); 2- CVCM 132 (pR27, 188 Kb); 3- CVCM 153 (pR1, 104 Kb), 4- CVCM 164 (pR64, 121 Kb), 5-CVCM 194 (pRP4, 60 Kb).
Extracción y purificación del ADN cromosómico: En el procedimiento de extracción y purificación del ADN, se utilizó el Kit Wizard Genomic DNA Purification (Promega) ®, lavando previamente las células con NaCl 0.8%.
Amplificación del ADNr 16S, mediante la Reacción en Cadena de la Polimerasa (PCR): La reacción de amplificación fue llevada a cabo con los oligonucleótidos cebadores fD1 y rD1 descritos por Weisburg et al. (1991).
La reacción de PCR se realizó de acuerdo a Laguerre et al. (1994) con modificaciones en un volumen de 50µL, donde se emplearon entre 50 y 100ng de ADN genómico y 1.25 U de Taq Polimerasa (Promega)®. La amplificación fue realizada en un termociclador Gene Cycler marca BiO-RAD modelo 170-6700-170-6701. Los amplificados (5-6µL) fueron visualizados mediante electroforesis en geles de agarosa de uso rutinario para PCR, al 0.9% (12cm x 20cm) en tampón TBE 1X pH 8.0, bajo las siguientes condiciones: 100 Voltios, 80mA, durante 4h.
Ensayos de restricción (RFLP): Alícuotas de 8-12µL de amplificados fueron digeridos con 5 U de endonucleasas de restricción (Promega) ® en un volumen de reacción de 20µL a 37ºC durante 4h. Además se utilizaron las siguientes enzimas de restricción: Mspi, Rsai, Sau 3AI, y HhaI, éstas dos últimas corresponden a endonucleasas, y a los isoesquizómeros de las enzimas CfoI y NdeII.
Los digeridos (10μL c/u) fueron analizados en geles de poliacrilamida (8%), de grosor 0.75mm, en un sistema de electroforesis vertical, Mini-Protean II cell (Bio Rad), durante 2h aproximadamente. Luego se empleó un tampón TBE 1X pH 8.0 a 19mA, 72 V. Los geles fueron teñidos por el método de tinción con nitrato de plata (Bassam et al. 1991).
Las características fenotípicas y los patrones de restricción del ADNr 16S de los rizobios fueron codificados en forma binaria (1, presencia de la característica o de la banda; 0 ausencia de las mismas) y analizados por Taxonomía Numérica, empleando el programa estadístico SPSS, versión 13 para construir las matrices de similaridad de enlace simple y luego obtener el índice de Disimilaridad (Dsm) representado en los dendrogramas, los cuales fueron elaborados mediante el algoritmo Unweighted Pair-Group Method using Aritmethic Averages (UPGMA) (Programa Statistica, versión 6.0).
Resultados
Prueba de infectividad: La presencia de nódulos de color marrón rojizo, permitió considerar a todos los aislados rizobianos como efectivos y eficientes.
Macroptilium lathyroides (Siratro), considerada una especie promiscua (Bromfiel & Barran 1990) no fue nodulada por el rizobio ME 01 pero sí formó nódulos eficientes en Crotalaria sp. y en Phaseolus vulgaris L. (datos no publicados).
Caracterización fenotípica
Características morfológicas de las colonias de rizobios autóctonos y tiempo de aparición de las colonias: Las colonias de los aislados rizobianos estudiados presentaron morfologías muy diversas y tiempos de aparición diferentes. Los microsimbiontes NB 1.1, NB 2.1, NB 4.1, NB 9.1, y NB 15.1, originaron colonias de crecimiento lento y extralento (tiempo de aparición de las colonias 5-6 días y de 7-9 días respectivamente), de morfología muy similar (Cuadro 3). El aislado Trébol, a pesar de presentar crecimiento extralento se diferenció de los anteriores por formar colonias de aspecto translúcido y brillante de consistencia mucilaginosa, y el aislado NB 23.1 con morfología similar al primer grupo, presentó crecimiento rápido (colonias apreciables a los 3-4 días) (Cuadro 3).
El aislado de Phaseolus vulgaris L., Nod 2Rojo (Nod 2R), de crecimiento lento, originó colonias muy similares a Bradyrhizobium elkanii y B. japonicum, lo que contrasta con las del simbionte ME 01, aislado también de nódulos de la misma especie vegetal, pero con características similares a R. tropici CIAT 899 (Cuadros 3, 4). Las colonias de Rm Vivero (RmV) y Rm Borbollón (RmB), aislados de alfalfa, se diferenciaron por el color blanco crema y blanco lechoso respectivamente y se constató que ambas son de crecimiento rápido. El aislamiento proveniente de Leucaena sp. [Leu 2A (1)2], también presentó crecimiento rápido, pero difería de las anteriores, en cuanto a la textura gomosa de las colonias y el aspecto semi-translúcido.
La caracterización fenotípica de 12 aislados rizobianos, provenientes de diversas regiones geográficas de Venezuela, mostró una población heterogénea y se catalogó cuatro categorías en relación al tiempo en que se apreció el crecimiento de las colonias, de acuerdo al criterio relacionado a la velocidad de crecimiento de los rizobios, señalado por Jordan (1984) y Rodríguez-Mendoza (1993):
Rizobios de crecimiento rápido (colonia apreciable en 3-4 días). En este grupo se incluyó los aislados NB 23.1, Leu 2A (1)2, Rm V, Rm B, de condición semejante a la cepas, Sinorhizobium meliloti 41 y Rhizobium etli CFN42 (Cuadros 3, 4).
Rizobios de crecimiento muy rápido (colonia apreciable en 1-2 días), donde se incluyó la cepa ME 01, muy similar a las cepas de referencia R. tropici CIAT 899, S. americanum CFNEi 156 y A. tumefaciens C58 (Cuadros 3, 4).
Rizobios de crecimiento lento (colonia apreciable en 5-6 días), señalando los aislamientos, Nod 2R, NB 15.1 y Bradyrhizobium sp. CIAT 2453.
Rizobios de crecimiento extralento (colonia apreciable en 7-9 días), agrupó los aislados NB 1.1, NB 2.1, NB 4.1, NB 9.1 y Trébol, similares a las cepas de referencia B. elkanii USDA 76 y B. canariense BTA-1 y B. japonicum DSMZ 30131 (Cuadros 3, 4).
Características bioquímicas: El 41.6% de los aislados produjeron reacción ácida (ME 01, Trébol, Rm V, Rm B y NB 23.1), todos de crecimiento rápido-más rápido, con excepción del aislado Trébol que es una bacteria de crecimiento extralento, mientras el 58.3% restante que originaron una reacción básica, eran de crecimiento lento-extralento, e incluían el aislado Leu 2A(1) 2, a pesar de presentar crecimiento rápido (Cuadro 5).
En relación al crecimiento en medio LB, el 58.3% de los aislados mostraron resultados negativos, representados por los simbiontes de crecimiento lento-extralento a diferencia del resto o sea el 41.6% que si crecieron en este medio de cultivo, de manera que incluían los aislados de crecimiento rápido-muy rápido (Cuadro 5).
Todos los aislados analizados utilizaron D-fructosa, inositol, glicerol, D(+) manosa, rafinosa y sorbitol, además del manitol y de menor preferencia fueron los sustratos tales como: glucosa, esculina, D-xilosa y L(-) sorbosa, no metabolizados por la mayoría de los simbiontes (66%-83%), mientras la D(+)galactosa, arabinosa, D-lactosa, sacarosa, maltosa y rhamnosa no fueron aprovechados por un grupo de microorganismos (16%-50%).
Los ácidos orgánicos (citrato, α-cetoglutarato, glutamato, L-malato, piruvato, succinato) fueron metabolizados como fuente carbonada por todos los aislados y el ácido ascórbico no fue utilizado por el simbionte NB 23.1.
Todos los aislados metabolizaron la L-asparagina; mientras que los aminoácidos L-valina y L-fenilalanina fueron los sustratos de menor preferencia como fuente nitrogenada, metabolizados respectivamente por el 25% y 33% de los aislados rizobianos y la L-homoserina no fue utilizada por ninguno de los microsimbiontes autóctonos ni cepas de referencia.
El dendrograma A de la Fig. 2 elaborado con base a 40 atributos, relacionados a la utilización de fuentes de carbono, nitrógeno, la producción de ácido o álcali y crecimiento en medio LB, permitió reconocer cuatro perfiles principales de actividad metabólica.
El Grupo I incluyó una mezcla heterogénea de siete aislados autóctonos y siete cepas de referencia, pertenecientes a diversos géneros (Rhizobium, Bradyrhizobium, Sinorhizobium), los cuales se caracterizaron por utilizar L(+) arabinosa a excepción de NB 2.1. Este grupo de bacterias metabolizaron la mayor cantidad de sustratos carbonados y nitrogenados suministrados (entre el 75% y 90%) y el Grupo II conformado por 3 aislados autóctonos, no utilizaron L(+) arabinosa como fuente de carbono.
Dentro del Grupo I se separaron los siguientes subgrupos: el subgrupo Ia (ME 01) que metabolizó D(+)lactosa y L-fenilalanina y está relacionado con un nivel de disimilaridad Dsm=0.1 (90% similaridad) a Bradyrhizobium sp. CIAT 2453, R. etli CFN42 y R. tropici CIAT 899. El subgrupo Ib (Nod 2 R, NB 1.1, NB 4.1 y NB 23.1) que no utilizaron estos compuestos con excepción del aislamiento NB 4.1 que creció en presencia del carbohidrato. El subgrupo Ic (S. meliloti 41) fue incapaz de utilizar el α-cetoglutarato. El subgrupo Id (NB 15.1) no utilizó sacarosa y tres aminoácidos, L-leucina, L- metionina y L-tirosina, pero si L-fenilalanina, y finalmente el subgrupo Ie (NB 2.1), caracterizado por no aprovechar L-alanina y L-leucina.
El Grupo II incluyó a Rm V y Trébol que emplearon el 67.5% de la totalidad de fuentes suministradas. Este último con 85% de similaridad (Dsm=0.15) a Agrobacterium tumefaciens C58 y Rm B utilizó el 77.5% con 87% de similaridad a Sinorhizobium americanum CFN-EI 156 (Dsm=0.13). Los Grupos III y IV, conformados por NB 9.1 y Leu 2A (1)2 respectivamente, presentaron un estrecho rango de auxotrofía. El primero de ellos metabolizó el 65% de los sustratos y apenas el 52.5% de los compuestos carbonados y nitrogenados fue aprovechado por Leu 2A(1)2, y se diferenció de los restantes por no emplear urea.
Todos los aislados fueron resistentes a altos niveles de lincomicina (300µg/mL), gentamicina (2.5-100 µg/mL) y ácido nalidíxico (5-100µg/mL). La mayor susceptibilidad se encontró con amikacina, doxiciclina y tetraciclina, inhibiendo el crecimiento en el 19%, 33% y 28.6% respectivamente de las cepas estudiadas.
El dendrograma B de la Fig. 2 separó cuatro grupos con base en la resistencia intrínseca a 10 antibióticos. El Grupo I conformado por los siguientes subgrupos: Subgrupo Ia, ME 01 y la cepa de referencia R. tropici CIAT 899, las cuales presentaron un bajo Dsm=0.12 correspondiente a 88% de similaridad en relación a la resistencia (μg/mL) a los siguientes antibióticos: gentamicina (25), lincomicina (300), ampicilina (50), amikacina (5), estreptomicina (200), cloranfenicol (100) y ácido nalidíxico (100). El subgrupo Ib (Rm V y Rm B) se separó del grupo anterior con un Dsm 0.24 (76% de similaridad) y junto a S. meliloti 41 y S. americanum CFN-Ei156 mostraron en común resistencias a gentamicina (5μg/mL), ampicilina, kanamicina (25), ácido nalidíxico (100), estreptomicina (200), lincomicina, cloranfenicol (300). Todas fueron susceptibles a doxiciclina. El subgrupo Ic lo integró R. etli CFN42 resultando susceptible a doxiciclina, ampicilina, cloranfenicol y tetraciclina.
El grupo II (Fig. 2B) lo integraron la mayoría de los aislados autóctonos: Nod 2R, NB 2.1, NB 4.1, NB 9.1, NB 15.1, NB 23.1, Leu 2A (1) 2 y NB 1.1, con una similitud del 60% (Dsm=0.4) respecto al Grupo I, incluyendo cuatro cepas de referencia: B. japonicum DSMZ 3011, B. canariense BTA-1, Bradyrhizobium sp. CIAT 2453 y B. elkanii USDA 76. Todos mostraron resistencia a los 10 antibióticos ensayados, excepto NB 1.1 (subgrupo IIb), el cual fue susceptible a amikacina. En total el 66% de los aislamientos fueron resistentes a todos los antibióticos ensayados.
El Grupo III (Fig. 2B) integrado por A. tumefaciens C58, se separó de los grupos anteriores con un Dsm=0.53 (47% de similaridad), siendo susceptible a cloranfenicol, amikacina y kanamicina, pero resistente a ampicilina (200µg/mL).
El Grupo IV, presentó un Dsm=0.85 apenas con un 15% de similaridad respecto a los grupos restantes y lo conformó únicamente el aislado Trébol (Fig. 2B), susceptible a los antibióticos amikacina, doxiciclina, kanamicina streptomicina y tetraciclina; solo presentó resistencia a las concentraciones más bajas de gentamicina (2.5µg/mL), ampicilina (5µg/mL) cloranfenicol (50µg/mL) y ácido nalidíxico (5-25µg/mL).
Análisis fenético general de las cepas: Se elaboró un dendrograma con 94 caracteres referentes a la resistencia intrínseca a varios antibióticos, la tolerancia al pH, salinidad y temperatura, la utilización de fuentes de carbono y nitrógeno, la producción de ácido o base y el crecimiento en medio LB, donde se distinguieron tres grupos (Fig. 2C):
El Grupo I (25% de rizobios productores de ácido) se subdividió en tres subgrupos. El subgrupo Ia (ME 01), proveniente de P. vulgaris L., (Dsm=0.07), mostró un alto grado de similaridad (93%) a R. tropici CIAT 899.
Los microsimbiontes Rm V, Rm B (subgrupo Ib), presentaron características similares a los rizobios S. meliloti 41 y S. americanum CFN-EI 156, con una similitud al subgrupo I del 80% Dsm=0.20, excepto que Rm B no fue tolerante a pH inferiores a 5.0 (Cuadro 4 y 5). El subgrupo Ic incluyó solo a A. tumefaciens C58.
El Grupo II (66%), conformado por los aislados de crecimiento lento-extralento, excepto NB 23.1 y Leu 2A(2)1 de crecimiento rápido, los restantes mostraron afinidad al género Bradyrhizobium y se subdividió en 2 subgrupos: El subgrupo IIa incluyó a Nod 2R con 85% de similaridad (Dsm=0.15) (Fig. 2C) a B. japonicum DSMZ 30131 y B. canariense BTA-1 (Cuadro 4, 5), así como también a los aislados de diversos suelos del país (NB 4.1, NB 15.1, NB 23.1 y NB 2.1, NB1.1 (Dsm=0.13-0.27), con una similaridad mayor del 70% a las cepas de referencia menos tolerantes a las condiciones salinas, Bradyrhizobium sp. CIAT 2453 y B. elkanii USDA 76 (Fig. 2C), (Cuadro 5). El subgrupo II b, conformado por Leu 2A (1)2 y NB 9.1, se separó de los subgrupos anteriores por presentar una similaridad igual o menor al 70% (Dsm=0.3). Los aislados que lo integran se caracterizaron por crecer en medios con un pH entre 5.0-6.6 y no en concentraciones de NaCl mayores a 0.1 y 1% respectivamente (Cuadro 5).
El Grupo III (8.3%), separó el aislado Trébol con el más bajo nivel de similaridad respecto a los demás grupos 44% (Dsm=0.56) (Fig. 2C), el cual se diferenció de los restantes por poseer los límites de tolerancia más estrechos a condiciones extremas; creció a pH iguales o superiores a 5.6, condiciones de salinidad (0.1% NaCl), temperaturas entre 22°C y 35°C (Cuadro 5), además fue el aislado que mostró susceptibilidad a 50% de los antibióticos ensayados.
Caracterización molecular: También se detectó la presencia de un megaplásmido de tamaño aproximado a 279 Kb, en siete cepas rizobianas de las doce analizadas, correspondientes a seis aislamientos autóctonos: NB 2,1, NB 4.1, Nod 2R y Trébol de crecimiento lentoextralento y NB 23.1 ME 01de crecimiento rápido-muy rápido y la cepa tipo Rhizobium tropici CIAT 899 (Fig. 3). La línea cinco de esta figura correspondió a Rm B, donde no se detectó megaplásmidos con el método utilizado.
Los amplificados del ADN ribosómico 16S de los doce aislados autóctonos, nueve cepas de referencia y Pseudomonas fluorescens BIOMI ALQ 7, originaron una banda de 1 500pb aproximadamente.
La restricción con: Msp I, Rsa I, Hha I y Sau 3A I, generó un patrón de 5, 6, 10 y 10 bandas, respectivamente. En el análisis sólo se incluyeron las bandas apreciables de tamaño comprendido entre 125pb y 800pb (Fig. 4B).
Los perfiles de restricción, están constituidos por una combinación de bandas, que representan genotipos diferentes, 16 en 21 cepas analizadas (12 aislados autóctonos y 9 cepas de referencia) (Cuadros 5, 6). El orden numérico de los genotipos se hizo con base al dendrograma (Fig. 4A).
Las enzimas de restricción, Msp I, Hha I, y Sau 3AI(i-ii), permitieron detectar un mayor polimorfismo en el gen ribosómico 16S, lo cual logró definir diversos genotipos (Fig. 4B).
El análisis estadístico de los ensayos de restricción realizados con los amplificados, permitió la separación de tres grupos (Fig. 4A): El Grupo A conformado por el 42%, de los microsimbiontes de crecimiento rápido-muy rápido, en su mayoría productores de ácido, excepto Leu 2A(1)2. Se evidenció que ME 01era 100% similar a R. tropici CIAT 899 y que los aislados Rm V y NB 23.1, eran idénticos a S. americanum CFN-EI 156. El rizobio nativo Leu 2A(1)2, se presentó como un genotipo diferenciado, muy relacionado en un 97% (Dsm=0.03) a los dos aislados anteriores. Finalmente, Rm B presentó un perfil polimórfico característico generado por Sau 3AI, Rsa I, y Msp I, relacionado en un 80% (Dsm=0.20) a las especies tipo de crecimiento rápido, R. tropici CIAT 899 y S. americanum CFN-EI 156.
En el Grupo B se incluyeron 50% de los aislados analizados conformado por microsimbiontes de crecimiento lento productores de álcali, donde dos aislados autóctonos NB 2.1 y NB1.1 y la cepa de referencia B. canariense BTA-1 conformaron una terna 100% similar entre sí. En este grupo se identificó el genotipo, NB 9.1, muy relacionado con la cepa de referencia citado anteriormente con un bajo índice de disimilaridad (Dsm=0.1), lo ual representa un 90% de similaridad respecto a la terna anterior.
El microsimbionte Nod 2R, desde el punto de vista fenotípico, se mostró afín a B. japonicum DSMZ 30131 y Bradyrhizobium canariense BTA-1 y el análisis de polimorfismo del gen 16Sr (PCR-RFLP) también reveló que está relacionado con un alto grado de similaridad 97% (Dsm=0.03) con Bradyrhizobium japonicum DSMZ. La bacteria nativa NB15.1, se relacionó con los integrantes del grupo B citados anteriormente en un 75% de similaridad (Dsm=0.25) y el microsimbionte NB 4.1, mostró un 80% de similaridad (Dsm=0.2), con Bradyrhizobium elkanii USDA 76.
El Grupo C, separó individualmente al aislado Trébol como uno de los rizobios con el más alto coeficiente de disimilaridad (0.45) respecto a los restantes, el cual muestra apenas 55% de similaridad con las cepas de referencia analizadas desde el punto de vista genotípico.
Discusión
Las cuatro categorías establecidas en relación a la velocidad de crecimiento de los 12 aislados, basadas en el tiempo en que fueron apreciables las colonias, mostraron divergencias con los resultados planteados por Jordan (1984) & Rodríguez-Mendoza (1993), quienes señalaron solo 2 categorías: cepas de crecimiento rápido y lento, a pesar de emplear el mismo criterio. En este sentido, la clasificación propuesta por Jordan en 1984, no indicó formas extremas o intermedias, probablemente debido al muestreo reducido de leguminosas, el cual no fue representativo de la población total (Martínez-Romero & Caballero-Mellado 1996). Por lo tanto, se ha señalado la inconveniencia de tomar en cuenta sólo los aislamientos nodulares, porque se pasa por alto la gran diversidad de los grupos de rizobios existentes en el suelo (Bromfield et al. 1995), además, se conoce la existencia de un grupo de rizobios no simbióticos presentes en él (Sullivan et al. 1996). Este criterio es relativo y subjetivo (Bécquer et al. 2000), puesto que va a depender en parte de las condiciones del medio de cultivo, las cuales son: composición, pH, temperatura y origen de la cepa, sean provenientes de un aislamiento nodular o de repiques frecuentes de cepas ya aisladas y conservadas en el laboratorio, factores que modifican la velocidad de crecimiento. En general, a medida que se avanza en los estudios de diversidad rizobiana, se reconocen más categorías (intermedias y más extremas) en cuanto a la velocidad de crecimiento (Odee et al. 1997), lo cual indica la heterogeneidad en los patrones de crecimiento.
El análisis fenotípico incluyó los aislados NB 23.1 y Leu 2A (1)2 de crecimiento rápido en el grupo Bradyrhizobium de crecimiento lento. Esta contradicción se explicaría por no incluirse en estos análisis la velocidad de crecimiento como característica diferencial. Sin embargo, el aislado Leu 2A (1)2 desde el punto de vista genotípico, se encontró muy relacionado con Sinorhizobium. americanum CFN-EI 156, también de crecimiento rápido (Fig. 4A) y podría referirse al simbionte Leu 2A(1)2, aislado de Leucaena leucocephala (planta introducida en Venezuela), como un rizobio propio de la zona tropical de crecimiento rápido, esta última característica también ha sido referida por Trinick 1968, (citado por Allen & Allen 1981). También, se ha sugerido que esta leguminosa arbórea, una planta con una relativa promiscuidad, es capaz de asociarse con diversos géneros rizobianos autóctonos de crecimiento rápido o menos rápido, Rhizobium, Mesorhizobium, Sinorhizobium y Agrobacterium, lo cual ha sido determinado mediante análisis de PCR-RFLP del ADNr 16S y el ITS de la región espaciadora 16S-23S. Esta propiedad le ha conferido el éxito de su establecimiento en suelos tropicales según Bala et al. (2003).
En el presente estudio, se demostró que los aislados rizobianos de crecimiento rápido provenientes de diversas regiones de Venezuela, fueron tolerantes a pH ácidos, resultados que presentaron una incongruencia respecto al señalamiento que consideró a las cepas de crecimiento rápido, especialmente Rhizobium meliloti (Sin. S. meliloti) (Munns & Keyser 1981), menos tolerantes a pH ácidos, en comparación a las bacterias de crecimiento lento del género Bradyrhizobium. No obstante, se ha evidenciado que especies de crecimiento rápido tales como R. loti y R. tropici pueden crecer a pH 4.0 (Martínez-Romero et al. 1991). Los resultados de este trabajo también demostraron que R. tropici y R. etli CFN42 toleraron un pH entre 4.0 y 4.4 respectivamente (Cuadro 5). Valores similares se registraron en un estudio con varias cepas (7) de R. etli aisladas de suelos de Egipto, en donde la mayoría eran capaces de sobrevivir a pH 4.7 y R. tropici CIAT 899 a pH 4 (Shamseldin & Werner 2005). Este hecho fue valedero para las cepas de referencia S. americanum CFN-EI 156 y S. meliloti 41, todas de crecimiento rápido al igual que los rizobios autóctonos Rm V y Rm B capturados respectivamente de nódulos y suelo con pH 4.7.
La tolerancia a la salinidad es otra propiedad presente en las cepas autóctonas de crecimiento rápido pertenecientes al género Rhizobium en relación a los simbiontes de crecimiento lento del género Bradyrhizobium, de tal manera que coincide con lo señalado por Elsheikh & Wood (1995), Odee et al. (1997) y Shamseldin & Werner (2005). Los dos últimos autores señalaron que dos de las cepas de R. etli (EBRI 21 y EBRI 26), aisladas de los suelos de Egipto mostraron ser tolerantes a elevadas concentraciones salinas (4% NaCl) y estaban adaptadas a altas temperaturas (42ºC).
El 25% de los aislados fueron tolerantes a temperaturas altas (40ºC), siendo 27ºC- 35ºC el rango óptimo para el crecimiento de la mayoría de estos simbiontes, diferente al señalado por Ek-Jander & Fahraeus (1971), (28ºC-31ºC), sin embargo, se ha citado que el óptimo para R. meliloti es 35ºC (Allen & Allen 1950). Inclusive se ha demostrado la tolerancia de rizobios aislados de P. vulgaris a 45ºC (KüçüK et al. 2006) y de cepas tipo como R. tropici a 40ºC (Martínez-Romero et al. 1991).
Los tres aislados autóctonos, ME 01, Rm V y Rm B, se pueden considerar como cepas que poseen amplios límites de tolerancia a determinadas condiciones ambientales (pH ácidos, salinidad, altas temperaturas) y gran capacidad de metabolizar diversas fuentes nutricionales además de presentar susceptibilidad a los antibióticos amikacina, doxiciclina y tetraciclina. Todos los aislados autóctonos presentaron altos niveles de resistencia intrínseca a los antibióticos, particularidad que también fue señalada por Bromfield et al. (2010) para especies del género Rhizobium, Ensifer y Phyllobacterium. Según Jordán (1984), la mayor susceptibilidad es característica del género Rhizobium a diferencia de las cepas de crecimiento lento del género Bradyrhizobium, las cuales han mostrado un espectro más amplio de resistencia a los antibióticos en cuanto al número y concentraciones ensayadas. Estos resultados son semejantes a los registrados en este trabajo, donde el 66.6% de los aislados afines al género Bradyrhizobium, presentaron resistencia a los 10 antibióticos aplicados, sin embargo, se contraponen a lo señalado por Odee et al. (1997).
Por otra parte, los análisis fenotípicos revelaron un alto porcentaje de similaridad con el análisis del polimorfismo de los RFLP del ADN ribosómico 16S, conformado por tres grupos en ambos casos, y muestran una población heterogénea compuesta de diversos géneros rizobianos. Sin embargo, algunos estudios de biodiversidad, no muestran coincidencia en el número de grupos conformados mediante el análisis fenotípico y el de la longitud de los RFLP del ADNr 16S, tal es el caso del trabajo realizado a 48 aislados rizobianos formadores de nódulos en Cicer arietinum L., donde los autores registraron tres grupos al analizar numéricamente las características fenotípicas e identificaron cuatro ribotipos en los ensayos de los RFLP del ADNr 16S, en su mayoría pertenecientes al género Mesorhizobium (Maâtallah et al.2002).
De esta forma, en este análisis no se encontraron diferencias en cuanto al número y tamaño de los plásmidos de los rizobios aislados, sin descartar la probabilidad de que existan plásmidos de mayor tamaño, no detectados por la metodología empleada en este estudio y que podrían estar relacionados con la diversidad genética (Martínez-Romero & Caballero-Mellado 1996).
Asimismo, se detectó un plásmido de aproximadamente 279Kb en la cepa tipo B (R. tropici CIAT 899), mediante la técnica de Casse et al. (1979) (Fig. 3), la cual nos permite identificar plásmidos cercanos a los 300 Kb. Sin embargo, en los 2 subgrupos A y B de R. tropici, CFN299 y CIAT 899 respectivamente, ha sido visualizado mediante el método de Eckhardt (1978), un megaplásmido mayor de 1000Kb no homólogo (subgrupo específico) (Geniaux et al. 1995). De igual manera se ha evidenciado un plásmido de 200Kb en la cepa tipo A (CFN 299), pero no en la tipo B (CIAT 899) (Martínez-Romero 2003).
El análisis del Polimorfismo de los RFLP del ADN ribosómico 16S, arrojó los siguientes resultados: el 50% de los 12 aislados eran similares al género Bradyrhizobium, un 42 % estaban relacionados con los géneros Rhizobium y Sinorhizobium, de ellos un 8% era idéntico al primer género, un 25% similares al segundo y el 8.3% restante era afín a ambos. Esta es una técnica adecuada para agrupar cepas a nivel de especies o géneros, además puede ser usada para encontrar nuevos taxa potenciales o clasificar filogenéticamente un vasto número de rizobios (Vinuesa et al. 1998). Sin embargo, los patrones genómicos de un solo loci, en este caso el gen 16Sr, no reflejan la complejidad del genoma de Rhizobium, el cual está generalmente fraccionado en varios replicones.
El aislado Trébol (8.3%) presentó el más bajo porcentaje de similaridad respecto a las bacterias rizobianas, se mostró más relacionada con la cepa seleccionada como foránea Pseudomonas fluorescens BIOMI AlQ 7. Este microsimbionte reveló un patrón de restricción característico, y sugiere la probabilidad de tratarse de una bacteria endémica de los suelos venezolanos, que adquirió los genes simbióticos por transferencia horizontal, capaz de nodular a Trifolium sp., una planta introducida en Venezuela. En este sentido, Liu et al. (2007) demostraron que varias especies de Trifolium que crecían en la región subtropical y tropical de China, se asociaban a diversos especies rizobianas pertenecientes a Bradyrhizobium, Mesorhizobium, Rhizobium y Sinorhizobium, sugiriendo la posibilidad que los genes específicos para la nodulación de Trifolium, hayan sido transferidos desde R. leguminosarum a otras cepas rizobianas, contrario a lo afirmado por Jordan (1984), quien señaló en ese momento que R. leguminosarum bv. trifolii era el único grupo rizobial asociado a Trifolium spp.
La transferencia horizontal de genes simbióticos es un evento que ha sido evidenciado entre rizobios, que le confiere una ventaja ecológica a aquellas que lo han adquirido, para establecer una simbiosis efectiva con especies de leguminosas introducidas o exóticas (Barcillos et al. 2007). También se ha sugerido que la transferencia horizontal de genes simbióticos puede ocurrir entre rizobios y géneros bacterianos filogenéticamente relacionados no simbióticos, tal como se ha evidenciado en el análisis del ADNr 16S de bacterias que forman nódulos y fijan nitrógeno, provenientes de Crotalaria spp., pertenecientes al género Methylobacterium y donde se ha mostrado que el gen Nod A de Methylobacterium nodulans está estrechamente relacionado a Nod A de Bradyrhizobium (Sy et al. 2001).
Las características fenotípicas y genotípicas tan particulares que presentó el aislado Trébol, con baja similaridad a las cepas rizobianas de referencia bajo estudio, aunado al hecho de ser un microsimbionte capaz de crecer en YMm líquido con NH4NO3 y metanol (0.1%) como única fuente carbonada durante 96h, en ausencia de luz y en agitación (200rpm) (datos no publicados), han permitido sugerir que se trata de una bacteria metilotrófica facultativa aeróbica, capaz de nodular a la leguminosa Trébol y formar colonias no pigmentadas en medio YMAm con Rojo Congo, con algunas características fenotípicas similares a las señaladas por Sy et al. (2001).
El análisis del polimorfismo de los fragmentos de restricción permitió reconocer que P. vulgaris L. puede ser nodulada por 2 géneros rizobianos con características fenotípicas y genotípicas diferentes: un simbionte muy similar a R. tropici CIAT 899 y otro a B. japonicum DSMZ 30131(ME 01, Nod 2R respectivamente). Esto no es extraño, debido a que la asociación simbiótica establecida entre esta especie vegetal y B. japonicum además de otros géneros rizobianos que incluyen R. etli, R. tropici, S. fredii, M. loti, R. leguminosarum bvs. trifolii y viciae, S. meliloti y Rhizobium spp., ya han sido señaladas por Bromfield & Barran (1990) y Michiels et al.(1998) como un hospedador no selectivo, aunque la mayoría de estas asociaciones no eran efectivas. Sin embargo, el aislado Nod 2 R resultó ser un microsimbionte eficiente en cuanto a la capacidad fijadora de nitrógeno (datos no publicados).
Resultados negativos en la nodulación, como el que obtuvimos, también han sido señalados en la simbiosis Rhizobium-P. vulgaris L. considerado este último también como un hospedador promiscuo (Michiels et al. 1998), aunque con un grado de preferencia por ciertos rizobios (Pacovsky et al. 1984, Aguilar et al. 2001), donde se sugiere que la baja infectividad en esta asociación simbiótica, posiblemente se deba a fallas en la interacción entre la bacteria y planta huésped, tal como ha sido señalado por Bernal & Graham (2001).
En diversas regiones de la China han sido identificados varios aislados pertenecientes a Agrobacterium tumefaciens y tres grupos del género Bradyrhizobium, uno de ellos B. japonicum, además de R. leguminosarum bv. phaseoli, en P. vulgaris, Campylotropis spp. y Cassia spp. Los mismos autores han señalado este hecho como un caso de evolución convergente entre los rizobios y las leguminosas, puesto que las poblaciones rizobianas asociadas con P. vulgaris en los suelos de China eran completamente diferentes a las de México, el sitio de origen de P. vulgaris (Han et al.2005).
Los dos aislados autóctonos provenientes de Medicago sativa (alfalfa), Rm V y Rm B, también mostraron diferencias entre ellos. Fenotípicamente eran muy similares a Sinorhizobium meliloti 41 (82%) y a Sinorhizobium americanum CFN-EI 156 (88%) respectivamente. Sin embargo, el perfil de restricción incluyó a Rm V en el grupo de S. americanum CFN-EI 156 (100% similares) y a Rm B en un genotipo diferente con 80% de similaridad a las cepas de referencia de crecimiento rápido R. tropici CIAT 899 y S. americanum CFNEI 156. Estos resultados son apoyados por los encontrados por Bromfield et al. (2010), quienes han señalado a Medicago sativa como una leguminosa, cuyos nódulos estaban ocupados por varios genotipos bacterianos, pertenecientes a los géneros Ensifer, Rhizobium y Phyllobacterium, este último considerado como oportunista.
Los resultados de nuestro trabajo concuerdan con algunos estudios realizados en regiones tropicales por varios autores, en el sentido de hallar una diversidad de especies rizobianas formadoras de nódulos en varias leguminosas, presentes en diferentes localidades. Ellos han indicado que los trópicos constituyen un importante reservorio de genes de fijación de N2 (Menna et al. 2006), y surge la idea de considerarlos como un centro de biodiversidad y diversificación de bacterias que se asocian simbióticamente con las leguminosas (Lafay & Burdon 2007).
Finalmente, en suelos y nódulos radicales de leguminosas provenientes de varias regiones de Venezuela, se encontró una diversidad de especies rizobianas pertenecientes a diferentes géneros tales como Rhizobium, Bradyrhizobium y Sinorhizobium, adaptados a distintas condiciones ambientales, que abarcan un amplio rango de pH del suelo desde 4.7 a 8.2 (datos no publicados), lo que representa una extensa distribución geográfica que comprende la región centro occidental y sur oriental del país. Por otra parte, se destacó el hecho que en una misma especie de leguminosa, se pueden formar nódulos por la infección de diferentes especies de rizobios. En conclusión, se evidenció que existen bacterias formadoras de nódulos no relacionadas genéticamente a las cepas de referencia, que sugiere una posible transferencia horizontal de los genes simbióticos que confieren esta propiedad.
Agradecimientos
Este trabajo fue financiado por el Consejo de Desarrollo Científico Humanístico y Tecnológico (CDCHT) de la Universidad de Los Andes, Mérida- Venezuela, proyecto C-725-95-03-D. Agradecemos A Esperanza Martínez-Romero por donar gentilmente las seis cepas rizobianas de referencia. Al laboratorio de Biotecnología de Microorganismos (BIOMI) de la Universidad de Los Andes, Mérida, Venezuela, por facilitarnos la cepa de Pseudomonas fluorescens BiOMi ALQ 07. A Pablo García, Guillermo López Corcuera†, María Fernanda Segovia†, Jesús Pacheco, Guillermo Bianchi y Armando Briceño.
Referencias
Aguilar, O.M., M.V. López & P.M. Riccilio 2001. The diversity of rhizobia nodulating beans in northwest Argentina as a source of more efficient inoculant strain. J. Biotechnol. 91: 181-188. [ Links ]
Allen, O.N. & E.K. Allen.1950. Biochemical and symbiotic properties of the rhizobia. Bacteriol. Rev. 14: 273-330. [ Links ]
Allen, O.N. & E.K. Allen. 1981. The leguminosae. A source book of characteristics. Uses and nodulation. Macmillan, Londres, Reino Unido. [ Links ]
Ayanaba, A., S. Asanuma & D.N. Munns. 1983. An agar plate method for the rapid screening of Rhizobium for tolerante to acid - aluminium stress. Soil Sci. Soc. Am. J. 47: 256-258. [ Links ]
Bala, A., P. Murphy & K.E. Giller. 2003. Distribution and diversity of rhizobia nodulating agroforestry legumes in soils from three continents in the tropic. Mol. Ecol. 12: 917-929. [ Links ]
Barcellos, G.F., P. Menna, J.S. da Silva Batista & M. Hungria. 2007. Evidence of horizontal transfer of symbiotic genes from a Bradyrhizobium japonicum inoculant strain to indigenous diazotrophs Sinorhizobium (Ensifer) fredii and Bradyrhizobium elkanii in a Brazilian Savannah Soil. Appl. Environ. Microbiol. 73: 2635-2643. [ Links ]
Bassam, B.J., G. Caetano-Anolles & P.M. Gresshoff. 1991. Fast and sensitive silver staining of DNA in Polyacrylamide Gels. Anal. Biochem. 196: 80-83. [ Links ]
Bécquer, C.J., D. Prévost & A. Prieto. 2000. Caracterización fisiológica-bioquímica de cepas de rizobios, aislados en leguminosas forrajeras. Rev. Biol. 14: 57-65. [ Links ]
Beringer, J.E. 1974. R Factor transfer in Rhizobium leguminosarum. J. Gen. Microbiol. 84: 188-198. [ Links ]
Bernal, G. & P.H. Graham. 2001. Diversity in the rhizobia associated with Phaseolus vulgaris L. in Ecuador, and comparisons with Mexican bean rhizobia. Can. J. Microbiol. 47: 526-534. [ Links ]
Bromfield, E.S.P & L.R. Barran. 1990. Promiscuous nodulation of Phaseolus vulgaris, Macroptilium atropurpureum and Leucaena leucocephala by indigenous Rhizobium meliloti. Can. J. Microbiol. 36: 369-372. [ Links ]
Bromfield, E.S.P., L.R. Barran & R. Wheatcroft. 1995. Relative genetic structure of a population of Rhizobium meliloti isolated directly from soil and from nodules of alfalfa (Medicago sativa) and sweet clover (Melilotus alba). Mol. Ecol. 4: 183-188. [ Links ]
Bromfield, E.S.P., J.T. Tambong, S. Cloutier, D. Prévost, G. Laguerre, P. van Berkum, T.V. Tran Thi, R. Assabgui & L.R. Barran. 2010. Ensifer, Phyllobacterium and Rhizobium species occupy nodules of Medicago sativa (alfalfa) and Melilotus alba (sweet clover) grown at a Canadian site without a history of cultivation. Microbiology 156: 505-520. [ Links ]
Casse, F., C. Boucher, J.S Julliot, M. Michel & J. Denarie. 1979. isolation and Characterization of Large Plasmids in Rhizobium meliloti Using Agarose Gel Electrophoresis. J. Gen. Microbiol. 113: 229-242. [ Links ]
CIAT (Centro internacional de Agricultura Tropical). 1988. Simbiosis leguminosa-Rhizobium.Manual de Métodos de Evaluación, Selección y Manejo Agronómico Proyecto CiAT-UNDP de Evaluación, selección y manejo de la simbiosis leguminosa-rhizobio para aumentar la fijación de nitrógeno. [ Links ]
De Lajudie, P., A.Willems, B. Pot, D. Dewettinck, G. Maestrojuan, M. Heyra, M.D.Collins, B. Dreyfus, K. Kersters & M. Gillis. 1994. Polyphasic taxonomy of rhizobia: Emendation of the genus Sinorhizobium and description of Sinorhizobium meliloti comb. nov., and Sinorhizobium teranga sp. comb.nov. int. J. Syst. Bacteriol. 44: 715-733. [ Links ]
Eckhardt, T. 1978. A rapid method for the identification of plasmid deoxyribonucleic acid in bacteria. Plasmid 1: 584-588. [ Links ]
Ek-Jander, J. & G. Fahareus.1971.Adaptation of Rhizobium to subartic environment in Scandinavia. Plant Soil (spec. Vol): 129-137. [ Links ]
Elsheikh, E.A.E. & M. Wood. 1995. Nodulation and N2 fixation by soy bean inoculated with sal-tolerant rhizobia o salt–sensitive Bradyrhizobia in saline. Soil. Biol. Biochem. 27: 656-661. [ Links ]
Geniaux, F., M. Flores, R. Palacios & E. Martínez-Romero. 1995. Presence of Megaplasmids in Rhizobium tropici and further evidence of differences between two R. tropici subtypes. int. J. Syst. Bacteriol. 45: 392-394. [ Links ]
Han, S.Z., E.T. Wang & W.X. Chen. 2005. Diverse bacteria isolated from root nodules of Phaseolus vulgaris and species within the genera Campylotropis and Cassia grown in China. Syst. Appl. Microbiol. 28: 265-76. [ Links ]
Jordan, D.C. 1982. Transfer of Rhizobium japonicum Buchanan 1980 to Bradyrhizobium gen. nov., a genus of slow-growing, root nodule bacteria from leguminous plants. int. J. Syst. Bacteriol. 32: 136-139. [ Links ]
Jordan, D.C. 1984. Rhizobiaceae, p. 234-256. In N.R. Krieg & J.G. Holt (eds.). Bergey´s manual of systematic bacteriology. Williams and Wilkins, Baltimore, EEUU. [ Links ]
Keyser, H.H. & D.N. Munns. 1979. Tolerance of rhizobia to acidity, aluminium and phosphate. Soil Sci Soc. Am. J. 43: 519-523. [ Links ]
Küçük, C., M. Kivanç & E. Kinaci. 2006. Characterization of Rhizobium sp. isolated from bean. Turk. J. Biol. 30: 127-132. [ Links ]
Kuykendall, L.D., B. Saxena, T.E. Devine & S.E. Udell. 1993. Genetic diversity in Bradyrhizobium japonicum Jordan 1982 and a proposal for Bradyrhizobium elkanii sp. nov. Can. J. Microbiol. 38: 501-505. [ Links ]
Lafay, B. & J.J. Burdon. 2007. Molecular Diversity of Legume Root-Nodule Bacteria in Kakadu National Park, Northern Territory, Australia. PLoS ONE 2: e277. [ Links ]
Laguerre, G., M.R. Allard, F. Revoy & N. Amarger. 1994. Rapid identification of rhizobia by Restriction Fragment Length Polymorphism analysis of PCR-amplified 16S rRNA genes. Appl. Environ. Microbiol. 60: 56-63. [ Links ]
Liu, X.Y., E.T. Wang, Y. Li. & W.X. Chen. 2007. Diverse bacteria isolated from root nodules of Trifolium, Crotalaria and Mimosa grown in the subtropical regions of China. Arch. Microbiol. 188: 1-14. [ Links ]
Ludwig, W. & K.H. Schleifer. 1994. Bacterial phylogeny based on 16S and 23S rRNA sequence analysis. FEMS Microbiol. Rev. 15: 155-173. [ Links ]
MaÂtallah, J., E.B. Berraho, S. Muñoz, J. Sanjuán & C. Lluch. 2002. Phenotypic and molecular characterization of chickpea rhizobia isolated from different areas of Morocco. J. Appl. Microbiol. 93: 531-540. [ Links ]
Martínez-Romero, E., L. Segovia, M. Martins, A. Franco, P. Graham & M. Pardo. 1991. Rhizobium tropici, a novel species nodulating Phaseolus vulgaris L. beans and Leucaena sp. trees. int. J. Syst. Bacteriol. 41: 417-426. [ Links ]
Martínez-Romero, E. & J. Caballero-Mellado. 1996. Rhizobium phylogenies and bacterial genetic diversity. Crit. Rev. Plant Sci. 15: 113-140. [ Links ]
Martínez-Romero, E. 2003. Diversity of Rhizobium-Phaseolus vulgaris symbiosis: overview and perspectives. Plant Soil. 252: 11-23. [ Links ]
Menna, P., M. Hungria, F.G. Barcellos, E.V. Bangele, P.N. Hess & E. Martínez-Romero. 2006. Molecular phylogeny based on the 16S rRNA gene of elite rhizobial strains used in Brazilian commercial inoculants. Syst. Appl. Microbiol. 29: 315-332. [ Links ]
Michiels, J., B. Dombrecht, N. Vermeiren, X. Chuanwu, E. Luyten & J. Vanderleyden. 1998. A Phaseolus vulgaris is a non selective host for nodulation. FEMS Microbiol. Ecol. 26: 193-205. [ Links ]
Miller, J.H. 1972. Experiments in molecular genetics. Cold Spring Harbor Laboratory, Cold Spring Harbor, Nueva York, EEUU. [ Links ]
Munns, D.N. & H.H. Keyser. 1981. Response of Rhizobium strains to acid and aluminum stress. Soil. Biol. Biochem. 13: 115-118. [ Links ]
Odee, D.W., J.M. Sutherland, E.T. Makatiani, S.G. Mcinroy & J.I. Sprent. 1997. Phenotypic characteristics and composition of rhizobia associated with woody legumes growing in diverse Kenyan conditions. Plant Soil 188: 65-75. [ Links ]
Olsen, G.J. & C.R. Woese. 1993. Ribosomal RNA: a key to phylogeny. Faseb. J. 7: 113-123. [ Links ]
Rodríguez-Mendoza, M.N. & R. Ferrera-Cerrato. 1984. Estudio y caracterización de la relación simbiótica de Rhizobium phaseoli en frijol (Phaseolus vulgaris) de distintos hábitos de crecimiento. Memorias XII Reunión Latinoamericana sobre Rhizobium, Campinas, Sao Paulo, Brazil. [ Links ]
Rodríguez-Mendoza, M.N. 1993. Asociación Rhizobium-leguminosa, p. 11-53. Composición de las soluciones empleadas, p. 21-30. In R. Ferrera- Cerrato, M.C.A. González-Chávez & M.N. Rodríguez-Mendoza (eds.). Manual de Agromicrobiología. Trillas, México. [ Links ]
Segovia, L., J.P.W. Young & E. Martínez-Romero. 1993. Reclassification of American Rhizobium leguminosarum biovar phaseoli type I Strains as Rhizobium etli sp. nov. int. J. Syst. Bacteriol. 43: 374-377. [ Links ]
Shamseldin, A. & D. Werner. 2005. High salt and high pH tolerance of new isolated Rhizobium etli strains from Egyptian soils. Curr. Microbiol. 50: 11-6. [ Links ]
Sicardi-Mallorca, M., E.J. Bedmar, P. Vinuesa-Fleischmann & M.L. Izaguirre-Mayoral. 1998. Diversidad de rizobios nativos aislados de especies de Sesbania en diferentes zonas de Venezuela. Memorias XIX Reunión Latinoamericana de Rhizobiología. Monagas, Venezuela. [ Links ]
Sullivan, J.T., H.N. Patrick, W.L. Lowther, D.B. Scott & C.W. Ronson. 1996. Nodulating strains of Rhizobium loti arise through chromosomal symbiotic gene transfer in the environment. Proc. Natl. Acad. Sci. Unit States Am. 92: 8985-8989. [ Links ]
Sy, A., E. Giraud, P. Jourand, N. Garcia, A. Willems, P. De Lajudie, Y. Prin, M. Neyra, M. Gillis, C. Boivin-Masson & B. Dreyfus. 2001. Methylotrophic Methylobacterium bacteria nodulate and fix nitrogen in symbiosis with legumes. J. Bacteriol. 183: 214-220. [ Links ]
Taiz, L. & E. Zeiger. 1998. Mineral Nutrition, p. 103-124. In Plant Physiology. Sinauer Associates, Massachusetts, EEUU. [ Links ]
Toledo, I., L. LLoret & E. Martínez-Romero. 2003. Sinorhizobium americanum sp. nov.,a new Sinorhizobium species nodulating native Acacia spp. in Mexico. Syst. Appl. Microbiol. 26: 54-64. [ Links ]
Vielma, M. 1999. Caracterización de cepas autóctonas de Bradyrhizobium sp. (Lupinus) aisladas de Lupinus spp. Rev. Fac. Agron. (LUZ). 16: 495- 508. [ Links ]
Vincent, J.M. 1975. Cultivo, aislamiento y conservación de los rhizobios, p. 1-17. In J.M. Vincent (ed.). Manual práctico de rizobiología. Hemisferio Sur, Buenos Aires, Argentina. [ Links ]
Vinuesa, P., J.L.W. Rademaker, F.J. De Bruijn & D. Werner. 1998. Genotypic characterization of Bradyrhizobium strains nodulating endemic woody legumes of the Canary islands by PCR- Restriction Fragment Length Polymorphism analysis of genes encoding 16S Rrna (16S rDNA) and 16S-23S rDNA intergenic spacers, repetitive extragenic palindromic PCR genomic fingerprinting and partial 16S rDNA sequencing. Appl. Environ. Microbiol. 64: 2096-2104. [ Links ]
Wang, E.T., P. Van Berkum, D. Beyene, X.H. Sui, O. Dorado, W.X. Chen & E. Martínez- Romero. 1998. Rhizobium huatlense sp, nov., a symbiont of Sesbania herbacea that has a close phylogenetic relationship with Rhizobium galegae. int. J. System. Bacteriol. 48: 687-699. [ Links ]
Wang, T., J. Romero-Martínez & I. López-Lara. 2001. Rhizobium y su destacada simbiosis, cap. 8. In E. Martínez-Romero & J. Martínez-Romero (eds.). Microbios. Centro de investigaciones sobre Fijación de Nitrógeno. Universidad Nacional Autónoma de México, México. [ Links ]
Weisburg, W.G., S.M. Barns, D.A Pelletier & D.J. Lane. 1991. 16S ribosomal DNA amplification for phylogenetic study. J. Bacteriol. 173: 697-703. [ Links ]
Young, J.M., L.D. Kuykendall, E. Martínez-Romero, A. Kerr & H. Sawada. 2001. A Revision of Rhizobium Frank 1889, with an emended description of the genus, and the inclusion of all species of Agrobacterium Conn 1942 and Allorhizobium undicola de Lajudie et al. 1998 as new combinations: Rhizobium radiobacter, R. rhizogenes, R. rubi, R. undicola and R. vitis. int. J. Evol. Microbiol. 51: 89-103. [ Links ]
Young, J.M. 2003. The Genus Ensifer Casida. 1982 take priority over Sinorhizobium Chen et al. 1988, and Sinorhizobium morelense Wang et al. 2002 is a later synonym of Ensifer adhaerens Casida 1982. is the combination ‘Sinorhizobium adhaerens’ (Casida 1982) Willems et al. 2003 legitimate? Request for an opinion. int. J. Syst. Evolu. Microbiol. 53: 2107-2110. [ Links ]
Correspondencia a: María Eugenia Marquina, Néstor Enrique González & Yulimar Castro. Laboratorio de Fijación Biológica de Nitrógeno y Cultivos de Tejidos Vegetales in vitro, Departamento de Biología, Facultad de Ciencias, Universidad de Los Andes. Núcleo Pedro Rincón Gutiérrez, Edificio A, 2º piso, La Hechicera, Mérida, Venezuela; martu@ula.ve, ngonzalezv2007@gmail.com, yulica@gmail.com
Recibido 10-VIII-2010. Corregido 08-I-2011. Aceptado 08-II-2011.