Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista de Biología Tropical
On-line version ISSN 0034-7744Print version ISSN 0034-7744
Rev. biol. trop vol.56 n.3 San José Sep. 2008
Genética poblacional de cobayas de Colombia, Cavia spp. (Rodentia: Caviidae) con marcadores moleculares RAPD
Héctor Aníbal Campos1 & Manuel Ruiz-García2
1. Departamento de Biología, Facultad de Ciencias, Universidad Nacional de Colombia, A. A. 14490. Bogotá D.C., Colombia; hacamposm@unal.edu.co
2. Laboratorio de Genética de Poblaciones Molecular-Biología Evolutiva, Departamento de Biología, Facultad de Ciencias, Pontificia Universidad Javeriana. Cra 7a No 43-82. Bogotá DC., Colombia; mruiz@javeriana.edu.co
Abstract: Population genetics of Colombian Guinea Pigs, Cavia spp. (Rodentia: Caviidae) with RAPD molecular markers. The genus Cavia occurs in South America, mainly in grasslands.. We collected blood samples from 97 individuals in six field populations and analyzed them with RAPD molecular markers. One wild type (C. anolaimae) was differentiated from the domestic form (C. porcellus), in agreement with other authors who used morphological, osteological and karyotipic results. Genetic diversity was considerable in both species, but higher in C. porcellus. The levels of genetic heterogeneity were also higher among the populations of C. porcellus (FST = 0.254) than among the populations of C. anolaimae (FST = 0.118). These significant levels of genetic heterogeneity, and the low levels of gene flow, were consistent with a complex domestication process for Cavia porcellus. Rev. Biol. Trop. 56 (3): 1481-1501. Epub 2008 September 30.
Key Words: Cavia, population genetics, RAPD, Colombia, domestication process.
El género Cavia (familia Caviidae) posee ámbito de distribución en el Sur de América, con hábitats restringidos a los pastizales. En Colombia este género ha estado tradicionalmente representado por dos especies: una silvestre C. aperea, y otra doméstica, C. porcellus, conocido como conejillo de indias, cobaya, curí o cuyes (MacDonald 1993). Sin embargo, en los estudios más recientes respecto a este género en Colombia, Zúñiga (2000) y Zúñiga et al. (2002), utilizando caracteres osteológicos, morfométricos, cariológicos, ecológicos y de distribución, plantean la presencia de tres especies de Cavia válidas para el citado país: dos silvestres, C. anolaimae y C. guianae, y una doméstica: C. porcellus.
Osteológicamente, las tres especies difieren en medidas del cráneo, mandíbula, cintura pélvica, fémur, escápula, húmero y número de vértebras caudales; por otra parte, los análisis multivariantes registraron dimorfismo sexual, y también separaron claramente las tres formas estudiadas; adicionalmente, el análisis citogenético mostró un número diploide de 64 cromosomas, pero con números fundamentales diferentes: C. anolaimae (48), C. guianae (51) y C. porcellus (40, 42 y 43). Por otra parte, las diferencias observadas en la estructura del cariotipo sugieren que puedan corresponder a especies distintas entre las cuales es improbable la producción de híbridos fértiles (Zúñiga 2000, Zúñiga et al. 2002).
Las dos especies silvestres son alopátricas, encontrándose la primera de ellas, C. anolaimae, en zonas altas y húmedas de la Cordillera Oriental (por ejemplo en los humedales de la ciudad de Bogotá), en los departamentos de Boyacá, Cundinamarca y sur de Santander, desde alturas de 1700 a 4000 msnm en terrenos saturados de agua y en áreas de pajonal; C. guianae se localiza en las sabanas, principalmente de gramíneas, de la Orinoquía, en los departamentos de Meta, Vichada y posiblemente Arauca y Casanare, por debajo de los 500 msnm.
Estos organismos son herbívoros, con hábitats más restringidos a los pastizales, en los que C. anolaimae en ocasiones se alimenta de los cultivos propios de las zonas frías como la papa, debido a la interacción del hombre con sus nichos para dar lugar a procesos agrícolas. Este hecho, aunado a la cacería con fines de subsistencia, o deportivos, ha llevado a que el hombre sea uno de sus principales depredadores al igual que animales domésticos como el perro y el gato. Desde tiempos prehistóricos los cuyes han sido fuente de proteína animal y su consumo llegó a ser tan significativo para los habitantes de los Andes, desde Colombia hasta Bolivia, que llegó a domesticarse (Cabrera 1953), siendo por ello un marcador de los procesos de domesticación en poblaciones prehispánicas (Pinto et al. 2001). De hecho, fue una de las pocas especies de mamíferos domesticados en tiempos pre-hispanos por pueblos amerindios, en el único centro de domesticación de mamíferos en el Nuevo Mundo (Andes) (Wing 1986). Actualmente, el impacto de los cuyes domésticos en estos países es tal que contribuye a la seguridad alimentaria de la población rural de escasos recursos; allí existe una población estable de más o menos 36 millones de cuyes domésticos, siendo el Perú el país con la mayor población y consumo, con un registro de producción anual cercano a las 16.500 toneladas de carne. La distribución de C. porcellus en Perú y en Ecuador es tal que se encuentra en la casi totalidad de sus territorios, mientras que en Bolivia y Colombia su distribución es regional y con poblaciones menores (Chauca 1997). De hecho, en Colombia es importante su presencia en comunidades de los departamentos de Nariño, Putumayo y Cundinamarca.
Por otra parte, C. porcellus es un modelo biológico básico como material en laboratorios para investigación médica y biológica, siendo utilizado en estudios que incluyen estructura social y comportamiento (Sachser et al. 1999; Hohoff et al. 2001, Touma et al. 2001a), historia de vida (Trillmich y Linke 2001), fisiología de hormonas (Touma et al.2001b), procesos infecciosos (Verma et al. 2000), selección de pareja (Adkins et al. 2001), morfología comparada (Drees et al. 2001, Vieytes et al. 2001), filogenia y sistemática molecular (Da Silva Neto 2000, Atkins et al. 2001, Gallardo y Kirsch 2001, Mouchaty et al. 2001), ecogenética (Kuenkele 2000a, b, Trillmich 2000), estudios sobre el sueño (Darchia et al. 2000, Gvilla et al. 2000), visión de los colores (Yokohama y Radlwimmer 1999), estrés (Sachser et al. 1998) y procesos de domesticación (Sachser 1998, Spotorno et al. 2006).
Hasta la fecha, en Colombia no se ha realizado un estudio molecular en las poblaciones de estos cávidos en aras de determinar su estatus genético. Por ello en esta investigación se han usado marcadores moleculares RAPD (Random Amplified Polymorphic DNA) (Williams et al. 1990, Welsh y McClelland 1990), los cuales detectan el polimorfismo existente entre los individuos analizados con base en la secuencia del ADN molde. Estos marcadores son detectados a través de la amplificación de regiones arbitrarías de un genoma aplicando la técnica de la PCR (reacción en cadena de la polimerasa).
Estos marcadores tienen algunas ventajas entre las que se destacan que la técnica es relativamente sencilla de aplicar y son de bajo costo comparado con otros marcadores moleculares; por otra parte, los cebadores son de fácil adquisición dado que no se requiere un conocimiento previo de la secuencia del ADN molde, y, adicionalmente, se necesita una pequeña cantidad de este para la amplificación (Hadrys et al. 1992).
La principal limitante que presentan los RAPD es que segregan como marcadores dominantes (presencia de banda-alelo dominante; ausencia de banda-alelo recesivo). Este hecho tiene profundas implicaciones en ciertos análisis: el homocigoto dominante es indiferenciable del heterocigoto y por tanto no es posible probar si la estructura de cruzamientos dentro de las poblaciones es al azar o no. También algunos estudios han presentado problemas de reproducibilidad (Pérez et al. 1998) con este tipo de marcadores moleculares. Sin embargo, en el presente estudio se observó una alta reproducibilidad en los resultados .
A pesar de ello, los RAPD han sido ampliamente usados en estudios genético poblacionales, como, por ejemplo, para caracterización genética y detección de híbridos en plantas (Divakaran et al. 2006) o para la determinación del sexo en palomas (Yan-Ming et al. 2006). También han servido para detectar cuellos de botella en el caso de dos poblaciones de Phoca vitulina en Holanda y Escocia (Kappe et al. 1995), para estrategias de conservación (Gibbs et al. 1994), para determinar patrones de colonización después de la última glaciación (Saxifraga oppositifolia) en el Norte de Europa (Gabrielsen et al. 1997) o para la discriminación de "stocks" en peces (Gomes et al. 1998). Incluso, los RAPD han servido para determinar correlaciones con variables eco-geográficas poniendo de manifiesto la acción de la selección natural. Nevo et al. (1998), estudiando Hordeum spontaneum en Israel, Turquía e Irán, determinaron que las poblaciones con mayor diversidad genética fueron aquellas con ambientes más estresantes como en desiertos tanto fríos como cálidos. Lo mismo fue determinado con RAPD para Triticum dicoccoides (Fahima et al. 1999 y Li et al. 1999, 2000). También determinaron niveles de polimorfismo y de diversidad genética mas elevados en las poblaciones en suelos de basalto que en suelos de tierra roja (Owuor et al. 1999). Finalmente, los RAPD han sido importantes como herramienta en la resolución de problemas sistemáticos y filogenéticos, como en el caso de los cérvidos (Comincini et al. 1996).
Los objetivos del presente estudio fueron:
1. Determinar los niveles de variabilidad genética en las poblaciones analizadas de cávidos.
2. Estimar el grado de heterogeneidad genética entre las diversas poblaciones de Cavia anolaimae y entre las poblaciones de C. porcellus analizadas en Colombia, y,
3. Contextualizar los resultados con las relaciones filogenéticos existentes entre diversas especies de Cavia evaluadas por otros autores y la domesticación de C. porcellus.
Materiales y métodos
Para el presente estudio se recolectaron muestras de sangre de 97 individuos de seis poblaciones, para dos de las especies descritas por Zúñiga et al. (2002), distribuidas como sigue: 1- Cavia anolaimae – se muestrearon dos poblaciones naturales: Cogua - Zipaquirá (5° 5 24.42"N, 74° 1 9.93"W; Departamento de Cundinamarca), con 30 individuos, y Sumapaz (3°5427.68"N, 74°19 6.90"W; Departamento de Cundinamarca), con 16 individuos. 2- C. porcellus – se muestrearon cuatro poblaciones discriminadas así: Botana, con 26 individuos, Mejorados, 15 individuos y Mercado, 5 individuos, representando tres variedades diferentes que se encuentran en la ciudad de Pasto (1o 6 48.35 N, 77o 0 29.78 W; Departamento de Nariño), y Alto Santiago (1o 8 28.07 N, 77o 0 28.11 W; Departamento del Putumayo), con 5 individuos.
Con el material mencionado, se realizó la extracción del ADN siguiendo el protocolo de Sambrook et al. (1989) con fenol-cloroformo y el ADN obtenido fue almacenado en tubos Eppendorf a -20 oC. Catorce cebadores fueron inicialmente probados, de los cuales se escogieron diez por la obtención de bandas discernibles en las amplificaciones, los cuales fueron A-7, A-17, A-67, OPH-03, OPA-09, OPA-12, OPA-18, OPG-04, OPG-10 y S-1. Las secuencias de los mismos se registran en el cuadro 1.
El protocolo para la amplificación de los RAPD aplicando la PCR fue tomado de Ruiz-García et al. (2003) y otros ensayos previos realizados en el laboratorio de Genética de Poblaciones Molecular-Biología Evolutiva de la Pontificia Universidad Javeriana con otras especies. Las condiciones de amplificación aplicadas a volúmenes totales de 25 µL contenían: 20ng de ADN genómico, 1X de buffer libre de MgCl2 y DNA polimerasa, 2 nM de MgCl2, 250µM de cada dNTP, 25 pM de cebador y 0.25 U de Taq DNA-polimerasa. Las PCR se llevaron a cabo en un termociclador Perkin-Elmer 9600 usando las siguientes condiciones: desnaturalización a 95oC por cinco minutos, seguida por 40 ciclos de 1 minuto a 94 oC, anillamiento a 36 oC por 2 minutos y elongación a 72 oC por 2 minutos. Se aplicó un periodo adicional de 5 minutos de elongación a 72 oC después del último ciclo. Los productos amplificados fueron sometidos a electroforesis en geles de agarosa al 1.5% en TBE, y las bandas fueron detectadas por tinción con bromuro de etidio o SYBER Safe; como marcador de tamaño molecular se usó el DNA Step- Leadder de 100 bp de Perkin Elmer.
La determinación de las bandas se constató con la aplicación de los programas informáticos Microsoft ® Paint, Picasa 2 y Corel Draw Graphics Suite 12®; bandas con la misma movilidad fueron consideradas homólogas y representan un "locus" independiente. Los fragmentos fueron contados y organizados en matrices binarias donde 0 indica que la banda está ausente y 1 que la banda está presente. Solamente aquellas bandas que se registraron con nitidez, y que en varias PCR fueron nuevamente obtenidas, fueron tomadas para los análisis, obteniéndose un total de 72 loci RAPD para las dos especies de Cavia.
Las estimas de las frecuencias alélicas fueron calculadas siguiendo a Lynch y Milligan (1994). Con esas frecuencias se realizaron comparaciones locus a locus para establecer el grado de heterogeneidad genética entre las poblaciones en estudio, mediante tablas de contingencia con la corrección de Yates (Sokal y Rohlf 1995). Así mismo, se aplicaron análisis de estructura poblacional estimando el estadístico FST con los estadisticos θ de Weir y Cockerham (1984) y de Lynch y Milligan (1994) con la aplicaciσn de los programas creados por Black (1995). Adicionalmente, se estimaron los niveles de flujo génico, siguiendo el modelo de isla infinito de Wright (1965), en el cual el flujo génico (Nm) es Nm = (1/FST – 1) / 4. También se determinó el flujo génico para un modelo isla n-dimensional donde Nm = (1/ – 1)/4[(s/(s – 1))2], donde s es el número de poblaciones analizadas (Ruiz-García 1993, Ruiz-García y Alvarez 2000). Debido a las características propias de los datos, se asume que las poblaciones muestreadas están en Equilibrio Hardy – Weinberg; sin embargo, para el presente estudio es posible que esta asunción no se cumpla, particularmente, en las poblaciones de cávidos domésticos, C. porcellus porque al parecer en éstas se da una estructura de cruzamientos endogámica.
Por otra parte, se estimaron las distancias genéticas de Nei (1972) con la corrección de Lynch y Milligan (1994), usando el programa RAPDDIST (Black 1995), entre las poblaciones analizadas. Con esta matriz de distancias, se construyeron árboles fenéticos aplicando los algoritmos UPGMA (Sneath y Sokal 1973) y Neighbour-Joining (Saitou y Nei 1987).
Resultados
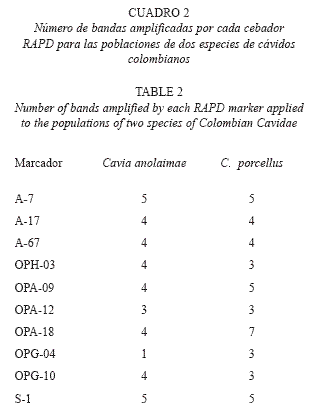
Un total de 38 loci fácilmente discernibles fueron obtenidos con los diez primers en las dos poblaciones de Cavia anolaimae. El total de loci obtenidos en las cuatro poblaciones de C. porcellus fue de 42, discriminados como se muestra en el Cuadro 2.
Como se observa, A-7 y S-1 registraron el más alto número de fragmentos (5) entre los cebadores aplicados en C. anolaimae, mientras que OPG-04 produjo apenas un fragmento. Para C. porcellus, OPA-18 produjo el número más alto de fragmentos (7) mientras que OPH-03, OPA-12, OPG-04 y OPG-10 produjeron cada uno tres fragmentos. El tamaño molecular de las bandas producidas en los cávidos silvestres estuvieron en el ámbito de 470 bp para A-67 hasta 1070 bp para OPA-18; en los cávidos domésticos este ámbito estuvo entre 470 bp para OPA-09 hasta 1060 bp para OPH-03. Todas las bandas producidas fueron polimórficas para cada especie en estudio, y los resultados en cada una de las poblaciones se muestran en el Cuadro 3. Es observable que los niveles de polimorfismo son más altos en las dos poblaciones silvestres, Cogua con 94,73% y Sumapaz con 83,78%, mientras que en las poblaciones domésticas, este valor oscila entre el 78,04%, para Botana, hasta el 45.16% para la población de Mercados. Por lo tanto, esta es la población con menor polimorfismo.
Frecuencias alélicas:
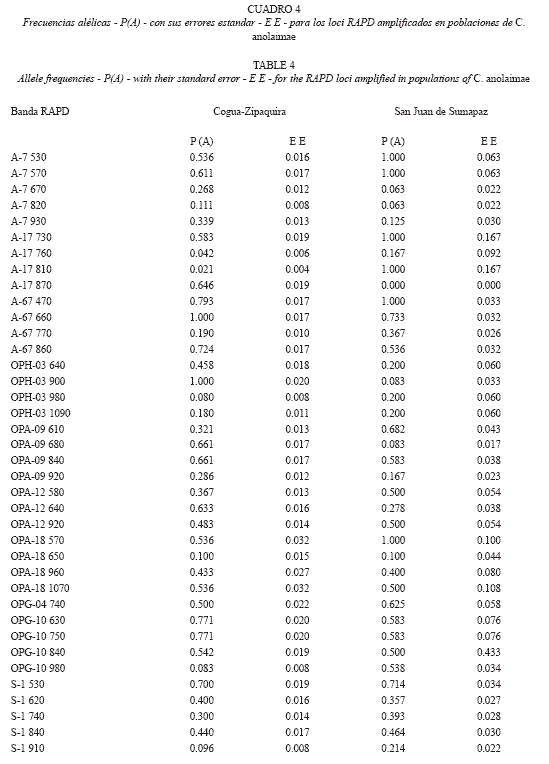
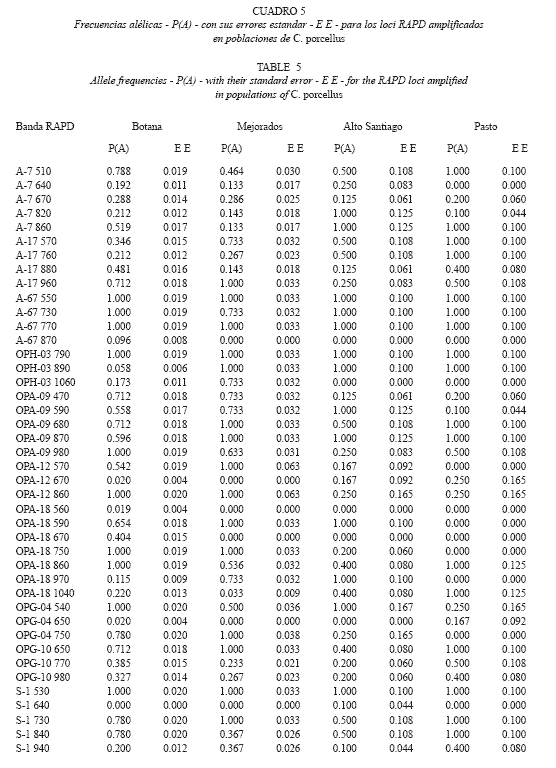
Las frecuencias alélicas para cada locus RAPD presente en las poblaciones de C. anolaimae se presentan en el Cuadro 4 y para C. porcellus en el Cuadro 5.
Como se observa en el Cuadro 4, las frecuencias para el alelo dominante (presencia) para C. anolaimae en la población de Cogua-Zipaquira oscilaron entre 0,021 para el marcador A-17 810, hasta 1,000 para los marcadores A-67 660 y OPH-03 900. En la población de San Juan de Sumapaz las frecuencias oscilaron con un valor mínimo de 0,000 para el marcador A-17 870, seguido por los marcadores A-7 670 y A-7 820 con una frecuencia de 0,063, siendo el valor máximo de 1,000 para los marcadores A-7 530, A-7 570, A-17 730, A-17 810, A-67 470, y OPA-18 570. Por otra parte, se observa que los errores estándar son bastante pequeños, lo cual da confianza respecto al muestreo realizado y, por ende, respecto a las estimas de las frecuencias alélicas obtenidas.
Al realizar las comparaciones locus a locus entre las dos poblaciones de C. anolaimae, se encontró que en el 84,21% de todas las posibles comparaciones se registraron diferencias estadísticas altamente significativas (p < 0,01) como en los loci A-7 530 y A-7 570, y extremadamente significativas (p < 0,001) como en los loci A-17 810, A-17 870, OPH-03 900, OPA-09 680 y OPG-10 980. Las excepciones a este comportamiento fueron los siguientes loci: OPA-12 920, OPA-18 650, OPA-18 960, OPA-18 1070, S-1 530 y S-1 840.
Por lo tanto, existe un fuerte indicio de heterogeneidad genética entre las dos poblaciones silvestres de C. anolaimae.
Las frecuencias de los alelos dominantes de las cuatro poblaciones de C. porcellus (Cuadro 5) oscilaron entre 0 para algunos de los marcadores, como S-1 640 en Botana, Mejorados y Mercado, hasta marcadores con la máxima frecuencia como A-67 730, el cual registró este valor en tres de las cuatro poblaciones, Botana, Alto Santiago y Mercado. Es destacado el comportamiento de cuatro de los 42 marcadores (A-67 550, A-67 770, OPH-03 790 y S-1 530), los cuáles registraron la fijación del alelo dominante para todas las poblaciones.
Para las comparaciones locus a locus en C. porcellus, se registraron 48 casos (19%) que no presentaron diferencias genéticas significativas (p>0,05). Este valor es ligeramente superior a los registrados entre poblaciones de C. anolaimae (15,19%); adicionalmente las diferencias entre los loci no presentan los niveles asociados de probabilidad tan contundentes como los registrados entre los cávidos silvestres. Por otra parte, solamente 17 loci presentaron diferencias estadísticas significativas locus a locus entre todos los posibles pares de poblaciones; a pesar de ello, estas comparaciones muestran un nivel estadísticamente significativo de heterogeneidad genética entre las poblaciones domésticas.
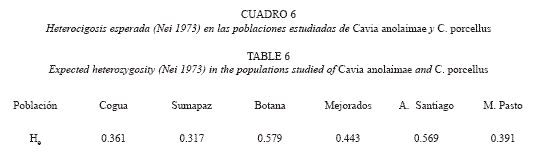
Por otra parte, a partir de las frecuencias alélicas se estimaron los niveles de heterocigosis esperada en las poblaciones de acuerdo a Nei (1978). Este metodo permite obtener estimaciones insesgadas de ese estadistico por lo que el tamaño muestral no tiene una importante influencia en los resultados, aun cuando algunos tamaños muestrales fueron muy pequeños. Se encontro que para las poblaciones silvestres estos niveles son más bajos que para las domésticas (Cuadro 6), con una heterocigosis esperada promedio de 0,339 para C. anolaimae y 0,495 para C. porcellus. Sin embargo, dentro de cada especie estos niveles de heterocigosis no difieren estadísticamente (t = 1.079, p > 0.05 para C. anolaimae, y t = 2,715; p>0.05 entre Botana y Mercados para C. porcellus). El valor claramente menor fue el de Mercados (H = 0.4516). El tamaño muestral de esta poblacion fue muy pequeño (n = 5). Sin embargo, ese menor valor no puede aducirse al pequeño tamaño de la muestra. Tambien Alto Santiago constó de una muestra de 5 animales pero su diversidad genetica fue similar al de otras poblaciones.
Estructura genética:
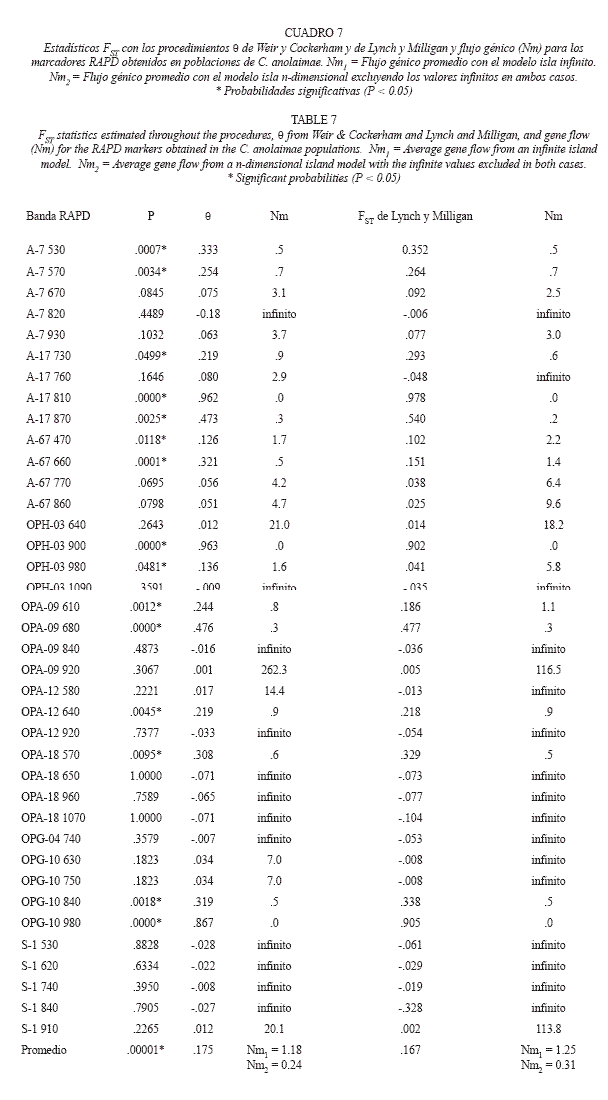
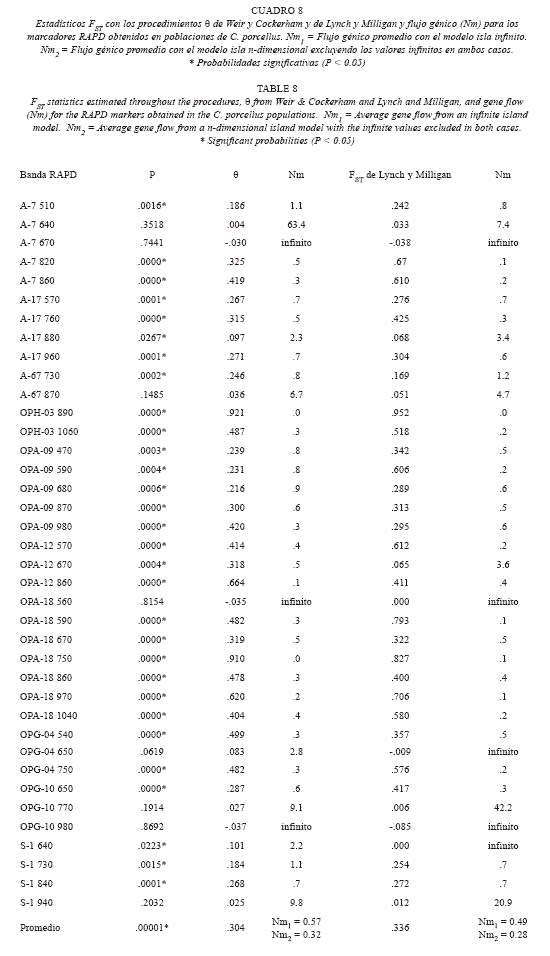
Con el objeto de establecer si cada una de las poblaciones de las dos especies de cávidos están estructuradas genéticamente, se aplicaron análisis utilizando los estadísticos θ de Weir y Cockerham (1984) y FST de Lynch y Milligan (1994). Los resultados para las dos poblaciones de C. anolaimae con los estadísticos mencionados se muestran en el Cuadro 7, y los de los C. porcellus en el Cuadro 8.
Los estadisticos de heterogeneidad obtenidos en C. anolaimae muestran 14 loci con probabilidades estadísticamente significativas (36.84%, siendo este porcentaje superior al error tipo del 5%; x2 = 11.64, 1 gdl, P < 0.0001). Con los valores del estadístico θ de Weir y Cockerham se observo que los valores oscilaron entre -0,18 (≈ 0) (Nm = ∞) para A7 820 y -0,071 (Nm = ∞) para OPA-18 650 y OPA-18 1070 hasta 0,962 (Nm = 0,0) para A-17 810 y 0.963 (Nm = 0,0) para OPH-03 900. Con el estadístico FST de Lynch y Milligan (1994) se registraron valores ligeramente superiores a los del estadístico θ de Weir y Cockerham, variando desde –0,328 (≈ 0) (Nm = ∞) para S-1 840 hasta 0,978 (Nm = 0,0) para A-17 810.
Como se puede observar, las estimas promedio de flujo génico son bastante pequeñas independientemente del modelo o estadístico empleado, lo que enfatiza la relevante heterogeneidad genética encontrada entre las dos poblaciones estudiadas de C. anolaimae (FST = 0.118, P < 0.0001).
Para C. porcellus (Cuadro 8), el análisis de los estadísticos de estructura genética muestra los resultados de 38 de los 42 marcadores iniciales, dado que cuatro de ellos presentaron las frecuencias fijas en todas las poblaciones. Un 78.94% (30/38) de los marcadores presentaron heterogeneidad significativa, valor estadísticamente superior al error del 5% (x2 = 42.66, 1 gdl, P < 0.00001). Este porcentaje de marcadores significativamente heterogéneo en C. porcellus es significativamente superior al encontrado en C. anolaimae (x2 = 13.82, 1 gdl, P < 0.0001). Esto muestra una muy elevada heterogeneidad genética entre las poblaciones domésticas de Cavia procedentes del sur de Colombia.
Los valores para el estadístico de Weir y Cockerham están entre -0,037 (Nm = ∞ ) para OPG-10 980 y –0,035 (Nm = ∞) para OPA-18 560 hasta 0,910 (Nm = 0,0) con OPA-18 750 y 0,921 (Nm = 0,0) para OPH-03 890. Con el estadístico FST de Lynch y Milligan se registraron valores desde 0,000 (Nm = ∞) para OPA 18 560 y S-1 640 hasta 0,952 (Nm = 0,0) para OPH-03 890.
De esta forma, la magnitud de la heterogeneidad genética encontrada en C. porcellus (FST = 0.254, p = 0.00001) es, incluso, superior a la registrada para C. anolaimae. El flujo génico entre las cuatro poblaciones domésticas es en extremo bajo. Ni siquiera alcanza la unidad, denotando la posible existencia de diversos linajes genéticamente diferenciados en la forma doméstica prácticamente aislados entre sí.
Análisis de agrupación:
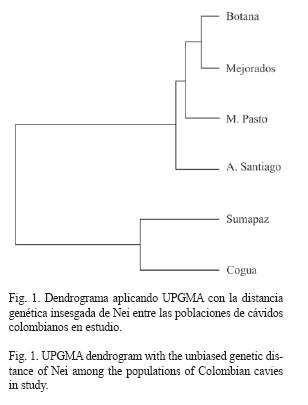
A las seis poblaciones analizadas se les aplicó un análisis de agrupación en busca de dilucidar las relaciones moleculares entre estos cávidos. Para ello se halló la distancia genética de Nei (1972) con la corrección de Lynch y Milligan (1994). Los resultados se registran en el Cuadro 9.
Las distancias genéticas más pequeñas se registraron, en general, entre las poblaciones de C. porcellus, siendo la de menor magnitud entre Botana y Mejorados (D=0,0304). Las dos poblaciones de C. anolaimae son más similares entre ellas que con cualquier otra de las poblaciones de C. porcellus, formando una agrupación que se diferencia claramente de las cuatro poblaciones de cávidos domésticos.
Estos resultados se reflejan en el dendrograma con el método UPGMA (Fig. 1). Se registraron dos agrupaciones, uno en el que los cávidos domésticos están agrupados entre sí y donde Botana y Mejorados son los más similares, y Alto Santiago es la población más divergente respecto a las demás poblaciones de C. porcellus. Las dos poblaciones silvestres forman la otra agrupación, siendo la similitud entre ellas menor que la registrada entre las formas domesticas.
Discusión
El primer estudio del género Cavia en Colombia fue llevado a cabo por Allen (1916). Un ejemplar capturado en la laguna de la Florida en la sabana de Bogotá fue denominado por este autor, C. anolaimae. Desde ese instante, la discordancia entre los autores ha sido manifiesta. Algunos autores aceptaron la nomenclatura de Allen, como en el caso de Ellerman (1940). Otros autores han considerado que C. anolaimae era simplemente una subespecie de la forma doméstica, C. porcellus o un sinónimo (Cabrera 1961, Woods 1993, Rodríguez et al. 1995). Otros autores han considerado la existencia de dos especies en Colombia. Cuervo et al. (1986) consideraron la existencia de C. aperea en el norte de Colombia y C. porcellus en la Orinoquía. Posteriormente, Peña y Pinto (1996) y Alberico et al. (2000) sugirieron, también, la existencia en Colombia de dos especies, C. aperea (silvestre) y C. porcellus (doméstica). Pero son los trabajos de Zúñiga (2000) y Zúñiga et al. (2002) los que definen, quizá, la situación más precisa respecto al género Cavia en territorio colombiano. Estos autores determinan tres especies en dicho país a partir de resultados craneométricos, osteológicos, de la coloración y características del pelaje y de resultados cariotípicos: C. anolaimae, silvestre y de zonas elevadas de Cundinamarca, Boyacá y sur de Santander; C. porcellus, la forma doméstica y C. guianae, otra forma silvestre en la Orinoquía. Esta especie fue determinada por primera vez por Thomas (1901) en la Guyana Británica. Tate (1939) reportó el hallazgo de la misma especie en las sabanas de Caramanta (Estado Bolivar) en Venezuela.
En el presente estudio se ha realizado el primer análisis genético molecular a poblaciones de C. anolaimae y C. porcellus que habitan en Colombia, aplicando marcadores RAPD que han demostrado su utilidad al registrar gran variabilidad en las poblaciones (Caccone et al. 1997). El número de loci amplificados para estos marcadores RAPD muestran que solamente ocho de los 72 loci (11,11%) amplificados son compartidos por las poblaciones de cávidos domésticos y silvestre, lo que nos da un indicio de las diferencias genéticas existentes entre estas dos formas de cávidos al presentarse los demás marcadores como únicos en las poblaciones de C. anolaimae o de C. porcellus. Aun así, en dos de estos marcadores compartidos, el A-67 770 y el S-1 530, las estimaciones de las frecuencias alélicas para las poblaciones de C. porcellus registraron la fijación del alelo dominante en todas ellas, mientras que para las dos poblaciones silvestres (Cogua y Sumapaz) son significativamente mucho más bajas en el primer marcador (P(A)=1,000 vs. 0,190 y 0,367, respectivamente; p < 0,001) y también significativamente más bajas en el segundo (P(A)=1,000 vs. 0,700 y 0,714, respectivamente; p < 0,01). Adicionalmente, es destacable la presencia exclusiva del alelo A-17 870 en la población de Cogua-Zipaquirá, el cual podría servir como marcador poblacional especifico en C. anolaimae. De este modo, se ratifica, a nivel molecular, la diferenciación específica de la forma doméstica (C. porcellus) de una de las formas silvestres del mismo género tal como morfológicamente determinaron Zúñiga et al. (2002). Estos resultados también dan soporte a lo encontrado por Spotorno et al. (2004, 2006) al emplear secuencias del gen mitocondrial citocromo-b. Aunque algunos autores, que abogan por la sinonimia entre C. aperea (= C. anolaimae) y C. porcellus, se basan en la existencia de híbridos fértiles entre ambas formas (Sachser 1998; Künzl y Sachser 1999) y que el primero puede haber dado origen al segundo, o Trillmich et al. (2004) quienes, con secuencias de ADN mitocondrial, plantearon que la forma doméstica no amerita el estatus de especie y debería ser clasificada como C. aperea f. porcellus, Spotorno et al. (2004) mostraron que la relación entre C. tschudii y C. porcellus es mucho mayor que la registrada entre C. aperea y C. porcellus. De hecho, C. tschudii y C. porcellus también dan híbridos fértiles procedentes de su cruzamiento (Castle y Wright 1916). Además, ese resultado es consistente con los registros arqueológicos procedentes de Perú y de Chile ya que ambas formas se han encontrado en las mismas zonas geográficas (Sandweiss y Wing 1997, Marcus et al. 1999). Otros autores afirmaron que, en realidad, C. aperea y C. tschudii formaban un único taxon (Huckinghaus 1961), aunque Cabrera (1953) contradice esta información, porque también son interfértiles. No obstante, el resultado de Spotorno et al. (2004) es contundente. La distancia genética entre C. tschudii y C. aperea es muy elevada (7,7%) y, siguiendo a Bradley y Baker (2001), para citocromo-b, diferencias entre el 2% y el 11% son típicas de especies diferentes. Algunos autores también han abogado por la posibilidad que C. fulgida pueda ser el ancestro de C. porcellus (Weir 1974). Sin embargo, los cruzamientos entre estas dos formas son inviables (Detlefsen 1914). Por lo tanto, los resultados con RAPD ponen de manifiesto que C. anolaimae y C. porcellus representan dos acervos genéticos bien diferenciados y que las diferencias son lo suficientemente notable para pensar que C. anolaimae no está en el origen directo de C. porcellus confirmando lo establecido por Spotorno et al. (2004, 2006).
Un asunto a discutir sería el efecto del tamaño muestral de algunas de las poblaciones consideradas tanto en C. anolaimae (San Juan de Sumapaz), como en C. porcellus (Mejorados, Alto Santiago y Mercado) en las estimas de las frecuencias alélicas. Sobre esto es de señalar que, aunque las mencionadas poblaciones registran números muestrales menores a 16 individuos, las estimas de los errores muestrales presentaron una magnitud pequeña, lo cual ofrece confiabilidad en el muestreo realizado y, por tanto, en las estimas obtenidas de las frecuencias alélicas.
A nivel intra específico también es de destacar las diferencias estadísticas registradas, en el 84,21% de las comparaciones locus a locus, para los marcadores analizados entre las dos poblaciones de C. anolaimae, y del 81% entre las poblaciones de C. porcellus. En este sentido, los marcadores A-17 810, A-17 870, OPH-03 900, OPA-09 680 y OPG-10 980 muestran diferencias genéticas estadísticamente contundentes (p < 0,001) entre las dos poblaciones C. anolaimae. Este es un primer indicio consistente a cerca de la acción de la deriva genética actuando en las poblaciones de C. anolaimae debido a la fuerte fragmentación de hábitat y a la presión cinegética (de caza) a la que está sometida esta especie, habiendo reducido, probablemente, de forma drástica sus números efectivos durante el siglo XX. Entre las poblaciones de C. porcellus para dicho análisis, los resultados no son tan dicientes pero existen marcadores como OPA-09 980 y OPG-04 750 quienes registran diferencias robustas (p < 0,001) entre las cuatro poblaciones. Sin embargo, de acuerdo con los valores de FST promedios de Wright hallados entre las poblaciones de los dos especies de cávidos, = 0,118 (p < 0,0001) para C. anolaimae, y FST = 0,254 (p < 0,0001) para C. porcellus, nos FST encontramos con una diferenciación genética extremadamente significativa entre las poblaciones muestreadas.
Esta fuerte heterogeneidad genética estaría en concordancia con lo esperado en las dos especies. Si bien no conocemos cuál es la capacidad de dispersión en C. anolaimae, la población de Cogua-Zipaquirá está en la zona montañosa al noroccidente de la Sabana de Bogotá y de la ciudad del mismo nombre; San Juan de Sumapaz se encuentra al sur de Bogotá, en la región geográficamente conocida como Páramo del Sumapaz, y su transito hacia esa región es complejo por las circunstancias socio políticas que prevalecen en dicha zona, con una distancia geográfica entre las dos poblaciones silvestres de, al menos, 140 km en línea recta. Esta distancia es considerable para organismos que son bastante territoriales y cuya estructura social, estrategias de comportamiento y éxito reproductivo han mostrado un comportamiento poligínico, en la que se da una incompatibilidad casi extrema entre los machos adultos y de estos con los jóvenes, teniendo como consecuencia el que solamente un macho adulto pueda estar junto a varias hembras (Sachser et al. 1999, Asher et al. 2004). Con ello podemos esperar unas poblaciones estructuradas genéticamente en las que unos pocos machos son los responsables de pasar sus genes a las siguientes generaciones, lo cual hace decrecer notablemente el número efectivo potencial de esas poblaciones e incrementa notablemente los efectos de la deriva genética. De hecho, en otra especie silvestre como C. tschudii se encuentra una considerable heterogeneidad genética. Spotorno et al. (2006) mostraron que ejemplares de esta especie procedentes de Chile (5 ejemplares) y del sur de Perú (1 ejemplar) formaban una agrupación muy sólida al utilizar el citocromo-b mitocondrial ("bootstrap" del 99%), mientras que otros dos especimenes se agrupaban más cercanos a ejemplares domésticos de Cuzco y Puno (Perú). Desde el punto de vista sistemático corresponderían a C. tschudii arequipae y a C. tschudii osgoodi (Tate 1935).
Lo anterior es todavía más contundente para las poblaciones de C. porcellus en los que el 78.94% de los casos los FST mostraron diferenciación genética entre las cuatro poblaciones analizadas. Entre las poblaciones colombianas de C. porcellus, el intercambio genético es muy escaso; de hecho, se reporta que en dos de esas poblaciones, Botana y Alto Santiago, separadas por unos 29 km, no ha ocurrido introducción de individuos por varias generaciones. Adicionalmente, se registró, en buena parte de las poblaciones visitadas, una estructura de cruzamientos endogámicos que contribuye junto con el efecto de deriva genética, y eventualmente de selección, a la diferenciación de grupos o poblaciones de esta especie. Spotorno et al. (2004, 2006) presentaron otros resultados moleculares que son altamente compatibles con los aquí mostrados haciendo referencia a una extrema heterogeneidad genética en la forma domestica. Spotorno et al. (2006) constataron la posible existencia de tres pasos en la domesticación de Cavia. El primer paso habría consistido en una única domesticación antigua (Wing 1986) de C. tschudii (de pequeño peso, 295 gramos) en la época precolombina que habría generado variedades criollas, todavía de bajo peso (639 gramos), como, por ejemplo, la Nativa de Bolivia o la Andina criolla de Chile. Esta domesticación habría sido única porque todos los ejemplares de C. porcellus analizados para el citocromo-b mitocondrial forman un único linaje caracterizado por 13 substituciones nucleotídicas (dos no silenciosas) que no comparten con especies silvestres del mismo genero. La segunda fase se habría dado en el siglo XVI, después de la llegada de los europeos a América. A partir de alguna de esas variedades domesticas de la primera fase, como la Andina Criolla, se generó todo el linaje que dio lugar a los actuales cobayas que encontramos en los laboratorios y tiendas de animales domésticos de todo el mundo al ser exportados a Europa. El peso de esos animales se habría elevado nuevamente (900 gr). Todos esos cuyes que se constituyeron a partir de los animales enviados a Europa se caracterizan molecularmente por 6 substituciones nucleotídicas únicas para el citocromo-b mitocondrial (una no silenciosa) y que ofrecería una condición sinapomórfica de una serina en el sitio 26 (Spotorno et al. 2004). Esto junto a ciertas características morfológicas, osteológicas y craneométricas (Cabrera 1953, Zuñiga et al. 2002) y, también, fisiológicas y de comportamiento (Wagner y Manning 1976) y pese al abundante polimorfismo y diferencias de coloración y textura del pelaje que encontramos en las mascotas domésticas de todo el mundo, muestran la existencia de un único linaje genético. Sin embargo, encontramos una tercera fase de domesticación en Sudamérica. Esta fase estaría constituida por las variedades de cuyes que encontramos actualmente en Sudamérica. De hecho, Spotorno et al. (2004, 2006) no encontraron un grupo monofilético para esos cuyes domésticos. Unos son animales que se encuentran en medios rurales, de tamaño pequeño (como los Nativa o Andina citados anteriormente), nerviosos y denominados "criollos" (Archetti 1997) y otros son de gran porte (unos 1200 gr; Tamborada de Perú o Auqui de Ecuador), prolíficos, de comportamiento manso y son denominados "mejorados". Estos provienen de la selección de los "criollos" en el siglo XX para la producción de carne, sobre todo en Perú y Ecuador (Morales 1994, 1995, Chauca 1997). Desde el punto de vista molecular este grupo presenta una enorme diversidad genética que origina una pérdida de cohesividad en el mismo. Por ejemplo, dentro de los "criollos" se encuentran distancias genéticas elevadas (d = 7.3%), mientras que en los mejorados, esas distancias son más pequeñas (d = 3.6%). Sin embargo, dentro de los mejorados se observan diferentes grupos. Independientemente del tamaño, se observa la asociación de formas norteñas (Auqui de Ecuador con Puno, Cuzco, Cajamarca del Perú; con una substitución nucleotídica única; "bootstrap" del 84%) y otro grupo con un origen más sureño (Tamborada, Nativa, Arequipa de Perú y Bolivia; con 4 substituciones nucleotídicas, una no silenciosa; "bootstrap" del 86%). La enorme diversidad genética del grupo de "criollos" es la probable consecuencia de una larga historia de divergencia entre ellos debido a la cría por parte de diferentes grupos indígenas de zonas muy diversas, lo cual es coherente con los registros históricos y arqueológicos reconocidos (Marcus et al. 1999) y de los diferentes sistemas de producción de las múltiples comunidades humanas rurales andinas. Los valores tan elevados de FST encontrados en el presente estudio para los marcadores RAPD en las 4 poblaciones de C. porcellus en el sur de Colombia responde seguramente al hecho que en ellas encontramos ejemplares que pertenecen a variedades "mejoradas", mientras que los animales de Alto Santiago pueden representar uno de esos linajes "criollos" más primitivos con una elevada diversidad genética (H = 0.569). Eso explicaría la considerable heterogeneidad genética entre las poblaciones domésticas del sur de Colombia.
Lo comentado justifica las bajas estimas encontradas de flujo génico tanto en el seno de C. anolaimae como de C. porcellus. Para la primera especie, las estimas de Nm oscilaron entre 0.24 a 1.18. Para la segunda especie, las estimas de Nm oscilaron entre 0.32 y 0.57. Para un modelo isla, se considera que valores de Nm > 1 deberían ser lo suficientemente elevados para prevenir diferenciación por deriva genética. Valores entre 1 > Nm > 0.5, mostrarían poblaciones bastante aisladas y Nm < 0.5 denotaría la existencia de poblaciones totalmente aisladas desde el punto de vista reproductivo. En un modelo "stepping-stone", un flujo génico elevado debería consistir en Nm > 4. Excepto el valor de 1.18, todas las otras estimas de Nm fueron menores a 1, lo que revela un fuerte aislamiento genético entre las poblaciones analizadas.
Respecto a las distancias genéticas, éstas confirman estadísticamente la diferenciación genética existente entre las poblaciones de cada especie, siendo la distancia genética promedio de D = 0.0589 para las poblaciones de C. porcellus y de D = 0.1385 para las de C. anolaimae; además, ínter específicamente la diferencia entre las dos especies de cávidos, con una distancia genética promedio de D = 0,327, es mucho mayor. De acuerdo a Thorpe y Sole-Cava (1994), las magnitudes de distancias genéticas de Nei (1972) entre 0.002 y 0.07 corresponden a poblaciones con-específicas y entre 0.03 y 0.61 para especies co-genéricas; los resultados encontrados en este estudio corresponden a lo reportado por estos autores, y corroborarían la propuesta de Zúñiga (2000) y Zúñiga et al. (2002) de la existencia de especies válidas, al menos, para los dos grupos de cávidos analizados.
Por otra parte, el dendrograma nos indican que C. anolaimae y C. porcellus son acervos bien diferenciados confirmando, una vez más, los resultados moleculares encontrados por otros autores (Spotorno et al. 2004, 2006).
Con todo lo anterior podemos decir que los marcadores RAPD son una técnica que ha mostrado su utilidad en la determinación de las diferencias genéticas entre estos organismos y que esperamos contrastar estos resultados utilizando otro tipo de marcadores como ADN mitocondrial y microsatélites, al igual que analizando la tercera forma colombiana, C. guianae y otras posibles formas del resto de Sudamérica.
Agradecimientos
Los autores agradecen a A. Rodríguez y A. Rico por su apoyo para obtener algunas de las muestras analizadas. El presente estudio fue parcialmente financiado por la División de Investigaciones Bogotá de la Universidad Nacional de Colombia, con código DIB-803848.
Resumen
En el presente estudio, mostramos los primeros resultados moleculares de formas colombianas de Cavia. Claramente, la población silvestre de C. anolaimae fue genéticamente diferenciada de la forma doméstica, C. porcellus, tal como ha sido demostrado por otros autores utilizando resultados morfométricos, osteológicos y cariotípicos. Ambas especies mostraron un considerable nivel de diversidad genética, aunque el segundo taxon mostró niveles mayores de esta diversidad. Los niveles de heterogeneidad genética también fueron mayores entre las poblaciones de C. porcellus (FST = 0.254) que entre las poblaciones de C. anolaimae (FST = 0.118). Esos niveles significativos de heterogeneidad genética, y los consiguientes bajos niveles de flujo génico, fueron discutidos comparativamente con los resultados por otros autores analizando otros marcadores moleculares (citocromo-b mitocondrial). Los resultados aquí mostrados son coherentes con un complejo proceso de domesticación en Cavia porcellus.
Recibido 14-IX-2007. Corregido 29-IV-2008. Aceptado 26-V-2008.
Referencias
Adkins, R.M., E.l. Gelke, D. Rowe & R.L. Honeycutt. 2001. Molecular phylogeny and divergence time estimates for major rodent gropups: Evidence from multiple genes. Molec. Biol. Evol. 18: 777-791. [ Links ]
Alberico, M., A. Cadena, J. Hernandez-Camacho & Y. Muñoz. 2000. Mamiferos (Synapsida: Theria) de Colombia. Biota Colomb. 1: 43-75. [ Links ]
Allen, J.A. 1916. List of mammals collected in Colombia by the American Museum of Natural History Expeditions, 1910-1915. Bull. Amer. Mus. Nat. Hist. 35: 191-238. [ Links ]
Apostol, B.L., W.C.IV. Black, P. Reiter & B.R. Miller. 1996. Population genetics with RAPD-PCRmarkers: the breeding structure of Aedes aegypti in Puerto Rico. Heredity 76: 325-334. [ Links ]
Archetti, E. 1997. Guinea Pigs: food, symbol and conflicts of knowledge in Ecuador (Global Issues). Berg, Nueva York, EEUU. [ Links ]
Asher, M., D.O.E. Spinelli & N. Sachser. 2004. Social system and spatial organization of wild guinea pigs (Cavia aperea) in a natural population. J. Mammal. 85: 788-796. [ Links ]
Black, W.C.IV. 1995. FORTRAN programs for the analisis of RAPD-PCR markers in populations. Colorado State University, Fort Collins, Colorado, EEUU. [ Links ]
Bradley, R.D. & R.J. Baker. 2001. A test of the genetic species concept: cytochrome b sequences and mammals. J. Mammal. 82: 960-973. [ Links ]
Cabrera, A. 1953. Los roedores de la Familia Caviidae. Ministerio de Educación. Facultad de Agronomía y Veterinaria, Universidad de Buenos Aires. Escuela de Veterinaria. Buenos Aires, Argentina. [ Links ]
Cabrera, A. 1961. Catalogo de los mamiferos de America del Sur. Revista del Museo Argentino de Ciencias Naturales "Bernardino Rivadavia" e Instituto Nacional de Investigación de Ciencias Naturales 4: 309-732. [ Links ]
Caccone, A., G. Allegrucci, C. Fortunato & V. Sbordoni. 1997. Genetic differentiation within the European Sea Bass (D. Labrax) as revealed by RAPD-PCR assays. J. Heredity 88: 316-324. [ Links ]
Castle, W.E. & S. Wright. 1916. Studies of inheritance in guinea pigs and rats. Carnegie Institution of Washington. EEUU 241: 1-192. [ Links ]
Chauca, L. 1997. Producción de cuyes (Cavia porcellus) Estudio FAO producción y sanidad animal 138. Roma, Italia. [ Links ]
Comincini, S., M. Sironi, C. Bandi, C. Giunta, M. Rubini & F. Fontana. 1995. RAPD analysis of systematic relationships among the Cervidae. Heredity 76: 215-221. [ Links ]
Cuervo, A., J. Hernandez-camacho & A. Cadena. 1986. Lista actualizada de los mamíferos de Colombia: anotaciones sobre su distribucion. Caldasia 15: 471-501. [ Links ]
Darchia, N., I. Gvilia & T. Oniani. 2000. The sleep-wakefulness cycle of guinea pigs as a biosensor for anxiolytics action. J. Sleep Research 9: 47. [ Links ]
Da Silva Neto, E.J. 2000. Morphology of the regions ethmoidalis and orbitotemporalis in Galea muesteloides (Meyen 1832) and Kerododn rupestris (Wied-Neuwied 1820) (Rodentia: Caviidae) with comments on the phylogenetic systematics of the Caviidae. J. Zool. Syst. Evol. Research 38: 219-229. [ Links ]
Detlefsen, J.A. 1914. Genetic studies on a cavy species cross. Publications of the Carnegie Institution Washington, EEUU 205: 1-134. [ Links ]
Divakaran, M., K. Nirmal-Babu, P.N. Ravindran & K.V. Peter. 2006. Interspecific hybridization in vanilla and molecular characterization of hybrids and selfed progenies using RAPD and AFLP markers. Scie. Hort.108: 414-422. [ Links ]
Drees, E., G.J. Klauer, D. Bachteler & G. Hilken. 2001. Comparative morphology of the follicle-sinus complex (Vibrissae) in wild and domestic guinea pigs. J. Morphol. 248: 225. [ Links ]
Ellerman, J.R. 1940. The families and genera of living rodents. Vol I. British Museum (Natural History). Londres, Reino Unido. [ Links ]
Fahima, T., G.L. Sun, A. Beharav, T. Krugman, A. Beiles & E. Nevo. 1999. RAPD polymorphism of wild emmer wheat populations, Triticum dicoccoides, in Israel. Theoretical Applied Genetics 98: 434-447. [ Links ]
Gabrielsen, T.M., K. Bachmann, K.S. Joacobsen & C. Brochman. 1997. Glacial survival does not matter: RAPD phylogeography of nordic Saxifraga oppositifolia. Molec. Ecol. 6: 831-842. [ Links ]
Gallardo, M.H. & J.A.W. Kirsch. 2001. Molecular relatioships among Octodontidae (Mammalia: Rodentidae: Caviomorpha). J. Mammal. Evol. 8: 73-89. [ Links ]
Gibbs, H.L., K.A. Prior & P.J. Weatherhead. 1994. Genetic analysis of populations of threatened snake species using RAPD markers. Molec. Ecol. 3: 329-337. [ Links ]
Gomes, C., R.B.G. Dales & H.A. Oxenford. 1998. The application of RAPD markers in stock discrimination of the four-wing flyingfish, Hirundichthys affinis in the central western Atlantic. Molec. Ecol. 7: 10291039. [ Links ]
Gvilla, I., N. Darchia & T. Oniani. 2000. The animal model of insomnia. J. Sleep Research 9: 76. [ Links ]
Hadrys, H., M. Balick & B. Schierwater. 1992. Applications of random amplified polymorphic DNA (RAPD) in molecular ecology. Molec. Ecol. 1: 55-64. [ Links ]
Hammer, Ø., D.A.T. Harper & P. D. Ryan. 2001. PAST: Paleontological Statistics Software Package for Education and Data Analysis. Palaeontologia Electronica 4(1): 9pp. [ Links ]
Hohoff, C., K. Franzen, K. Solmsdorff, J.T. Epplen & N. Sachser. 2001. A comparative study on mate preferences and paternities in three species of South American rodents. Zool. (Jena) 103: 44. [ Links ]
Huckinghaus, F. 1961. Vergleichende Untersuchungen uber die Formenmannigfalttigkeit der Ubterfamilie der Caviinae Murray 1886. Z. Wiss. Zool. 166: 1-98. [ Links ]
Kappe, A.L., L. van de Zande, E.J. Vedder, R. Bijlsma & W. van Delden. 1995. Genetic variation in Phoca vitulina (the harbour seal) revealed by DNA fingerprinting and RAPDs. Heredity 74: 647-653. [ Links ]
Kuenkele, J. 2000a. Does primiparity affect the efficiency of converting energy to offspring production in the guinea pig? Canadian J. Zool. 78: 300-306. [ Links ]
Kuenkele, J. 2000b. Effects of litter size on the energetics of reproduction in a highly precocial rodent, the guinea pig. J. Mammal. 81: 691-700. [ Links ]
Künzl, C. & N. Sachser. 1999. The behavioral endocrinology of domestication: a comparison between the domestic guinea pig (Cavia aperea f. porcellus) and its wild ancestor, the cavy (Cavia aperea). Hormones and Behavior 35: 28–37. [ Links ]
Li, Y.C., T. Fahima, A. Beiles, A.B. Korol & E. Nevo. 1999. Microclimatic stress and adaptative DNA differentiation in wild emmer wheat, Triticum dicoccoides. Theoretical and Applied Genetics 98: 873-883. [ Links ]
Li Y.C., T. Fahima, T. Krugman, A. Beiles, M.S. Roder, A.B. Korol & E. Nevo. 2000. Parallel microgeographic patterns of genetic diversity and divergence revealed by allozyme, RAPD, and microsatellites in Triticum dicoccoides at Ammiad, Israel. Conservation Genetics 1: 191-207. [ Links ]
Lynch, M. & B.G. Milligan. 1994. Análisis of population genetic structure with RAPD markers. Molec. Ecol. 3: 91-99. [ Links ]
MacDonald, D. (Ed). 1993. The encyclopedia of mammals. Facts on File. Nueva York, EEUU. [ Links ]
Marcus, J., J.D. Sommer & C.P. Glew. 1999. Fish and mammals in the economy of an ancient Peruvian kingdom. Proc. Nat. Acad. Sciences EEUU. 96: 6564-6570. [ Links ]
Morales, E. 1994. The guinea pig in the Andean economy: from household animal to market commodity. Latin American Res Rev 29: 129-142. [ Links ]
Morales, E. 1995. The guinea pig. Healing, food, and ritual in the Andes. The University of Arizona, Tucson, Arizona, EEUU. [ Links ]
Nei, M. 1972. Genetic distance between populations. American Naturalis 106: 283-292. [ Links ]
Nei, M. 1973. Analysis of gene diversity in subdivided populations. Proc. Natl. Acad. Sci. EEUU 70: 33213323. [ Links ]
Nevo, E., B. Baum, A. Beiles & D.A. Jonson. 1998. Ecological correlates of RAPD DNA diversity of wild barley, Hordeum spontaneum, in the Fertile Crescent. Genetic Resources Crop Evol. 45: 151-159. [ Links ]
Owuor, De., T. Fahima, A. Beharav, A. Korol & E. Nevo. 1999. RAPD divergence caused by microsite edaphic selection in wild barley. Genetica 105: 177-192. [ Links ]
Peña, L.G. & M. Pinto 1996. Mamíferos mas comunes en los sitios preceramicos de la Sabana de Bogota: guía ilustrada para arqueólogos. Academia Colombiana de Ciencias Exactas, Físicas y Naturales. Colección Julio Carrizosa Valenzuela No 6, Bogota, Colombia. [ Links ]
Pérez, T., J. Albornoz & A. Dominguez. 1998. An evaluation of RAPD fragment reproducibility and nature. Molec. Ecol. 7: 1347-1357. [ Links ]
Pinto, M., H. Zúñiga & O. Torres. 2001. Estudio sistemático del género Cavia Pallas, 1766 (Rodentia: Cavidae) en Colombia. Revisión del registro arqueológico Colombiano. Instituto de Ciencia Naturales, Universidad Nacional de Colombia – Fundación para la investigación y la tecnología Banco de la Republica. División de Investigación sede Bogotá, Universidad Nacional de Colombia. [ Links ]
Rodríguez, J.V., J.I. Hernandez-Camacho, T.R. Defler, M. Albericho, R.B. Mast, A. Mittermeier & A. Cadena. 1995. Mamíferos colombianos: sus nombres comunes e indigenas. Ocasional Papers N 3, Conservation Inter. 3: 1-56. [ Links ]
Ruiz-García, M. 1993. Analysis of the Evolution and Genetic Diversity within and between Balearic and Iberian cat populations J. Heredity 84:173-180. [ Links ]
Ruiz-García, M. & D. Alvarez. 2000. Genetic microstructure in two Spanish cat populations. I: Genic diversity, gene flow and selection. Genes Genetic Syst 75: 269-280. [ Links ]
Ruiz-García, M., E. Banguera, H. Gálvez & D. Alvarez. 2003. Discriminación de especies de los géneros Saimiri y Aotus mediante las técnicas de RAPDs y STRPs: Un estudio preliminar para la identificación de diferencias interespecíficas. Capítulo 7: 207-218. Primatología del Nuevo Mundo: Biología, Medicina, Manejo, Conservación. F. Nassar, V. Pereira, A. Savage (Eds. [ Links ]).
Sachser, N. 1998. Of domestic and wild guinea pigs: Studies in sociophysiology, domestication and social evolution. Naturwissenschaften 85: 307-317. [ Links ]
Sachser, N., M. Duerschlag & D. Hirzel. 1998. Social relationships and the management of stress. Psychoneuroendocrinology 23: 891-904. [ Links ]
Sachser, N., E. Schwarz-Weig, A. Keil & J.T. Epplen. 1999. Behavioral strategies, testis size, and reproductive success in two caviomorph rodents with different mating systems. Behaviour 136: 1203-1217. [ Links ]
Saitou, N. & M. Nei. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molec. Biol. Evol. 4: 406-425. [ Links ]
Sandweiss, D.H. & E.S. Wing. 1997. Ritual rodents: the guinea pigs of Chincha, Peru. J. Field Archaeology
24: 47-58.
Sambrook, J., E.F. Fritsch & T. Maniatis. 1989. Molecular cloning: a Laboratory Manual. Cold Spring Harbor Laboratory, Nueva York, EEUU. [ Links ]
Slatkin, M. & N.H. Barton. 1989. A comparison of the three indirect methods for estimating average levels of gene flow. Evolution 43: 1349-1368. [ Links ]
Sneath, P.A. & R.R. Sokal. 1973. Numerical Taxonomy. Freeman. San Francisco, EEUU. [ Links ]
Sokal, R.R. & F.J. Rohlf. 1995. Biometry. Freeman. San Francisco. EEUU. [ Links ]
Spotorno, A.E., J.P. Valladares, J.C. Marin & H. Zeballos. 2004. Molecular diversity among domestic guineapigs (Cavia porcellus) and their close phylogenetic relationship with the Andean wild species Cavia tschudii. Rev. Chilena Historia Nat. 77: 243-250. [ Links ]
Spotorno, A.E., J.C. Marin, G. Manriquez, J.P. Valladares, E. Rico & C. Rivas. 2006. Ancient and modern steps during the domestication of guinea pigs (Cavia porcellus L.). J. Zool. 270: 57-62. [ Links ]
Tate, G.H.H. 1935. The taxonomy of the genera of neotropical hystricoid rodents. Bull. Amer. Mus. Nat. Hist. 68: 295-447. [ Links ]
Tate, G.H.H. 1939. The Mammals of Guiana Region. Bull. Amer. Museum. Nat. Hist. 76: 151-229. [ Links ]
Thomas, O. 1901. On a collection of Mammals from the Kanuku Mountains, British Guiana. Ann. Mag. Nat. Hist. 7: 139-154. [ Links ]
Thorpe, J.P. & A.M. Sole-Cava. 1994. The use of allozyme electrophoresis in vertebrate systematics. Zoologica Scripta 23: 3-18. [ Links ]
Touma, C., S. Kaiser, R. Palme & N. Sachser. 2001a. Different types of oestous cycles in two closely related rodent species differering in social and mating systems. Zoology (Jena) 103: 37. [ Links ]
Touma, C., R. Palme & N. Sachser. 2001b. Different types of oestrous cycle in two closely related South American rodents (Cavia aperea and Galea musteloides) with different social and mating systems. Reproduction (Cambridge) 121: 791-801. [ Links ]
Trillmich, F. 2000. Effects of low temperature and photoperiod on reproduction in the female wild guinea pig (Cavia aperea). J. Mammal. 81: 586-594. [ Links ]
Trillmich, F. & S. Linke. 2001. Towards urderstanding guinea pig life history: Factors influencing reproduction and maturation in the female wild guinea pig, Cavia aperea. Zoology (Jena) 103: 53. [ Links ]
Trillmich, F., C. Kraus, J. Künkele, M. Asher, M. Clara, G. Dekomien, J.T. Epplen, A. Saralegui & N. Sachser. 2004. Species-level differentiation of two cryptic species pairs of wild cavies, genera Cavia and Galea, with a discussion of the relationship between social systems and phylogeny in the Caviinae. Canadian J. Zool. 82: 516-524. [ Links ]
Verma, S., R.C. Katoch & V.K. Gupta. 2000. Infection of Brucella melitensis in guinea pig (Cavia porcellus). Indian J. Animal Sci. 70: 269-270. [ Links ]
Vieytes, E.C., D.H. Verzi & M.G. Vucetich. 2001. Enamel evolution in molariforms of Cavioidea (Rodentia: Caviomorpha). J. Morphology 248 (3): 296. Wagner, J.E. & P.J. Manning. 1976. The biology of the guinea pig. Academic, Nueva York, EEUU. [ Links ]
Weir, B.J. 1974. Notes on the origin of the domestic guinea-pig. Symposia of the Zoological Society of London 34: 437-446. [ Links ]
Weir, B.S. & C.C. Cockerham. 1984. Estimating F-statistics for the analysis of population structure. Evolution 38: 1358-1370. [ Links ]
Welsh, J. & M. McClelland. 1990. Fingerprinting genomes using PCR with arbitrary primers. Nucleic Acids Research 18: 7213-7218. [ Links ]
Williams, J.G.K., A.R. Kubelik, K.J. Livak, J.A. Rafalski & S.V. Tingey. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research 18: 6535-6541. [ Links ]
Wing, E. 1986. Domestication of Andean mammals. En High altitude tropical biogeography: 246-264. Vuilleumier F & M. Monaterior (Eds.). Oxford: Oxford University, Oxford, Reino Unido. [ Links ]
Woods, C.A. 1993. Suborden Hystricognathi. Pp 771-806. En Wilson DE & CM Reeder (Eds). Mammalian species of the World. A taxonomic and geographic reference. Smithsonian Institution, Washington DC, EEUU. [ Links ]
Wright, S. 1965. The interpretation of population structure by F-statistics with special regards to system of mating. Evolution 19: 395-420. [ Links ]
Yan-Ming, H., W. Chean-Ping, W. Yng-Chyu & H. Mu-Chiou. 2006. A novel molecular genetic marker for gender determination of pigeons. Theriogenology 65: 1759-1768. [ Links ]
Yokohama, S. & B.F. Radlwimmer. 1999. The molecular genetics of red and green color vision in mammals. Genetics 153: 919-932. [ Links ]
Zúñiga, H., M. Pinto, J. Hernández-Camacho & O. Torres. 2002. Revisión taxonómica de la especies del género Cavia (Rodentia: Caviidae) en Colombia. Acta Zoológica Mexicana (87): 111-123. [ Links ]
Zúñiga, H. 2000. Estudio sistemático del género Cavia Palla, 1766 (Rodentia: Caviidae) en Colombia. Tesis de Maestría. Universidad Nacional de Colombia. Facultad de Ciencias, Bogotá, Colombia. [ Links ]