Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista de Biología Tropical
On-line version ISSN 0034-7744Print version ISSN 0034-7744
Rev. biol. trop vol.56 n.1 San José Mar. 2008
Diversidad genética y relaciones de parentesco de las poblaciones silvestres y cultivadas de pejibaye (Bactris gasipaes, Palmae), utilizando marcadores microsatelitales
José Alfredo Hernández Ugalde1,2, Jorge Mora Urpí1 & Oscar Rocha Nuñez1,3
1 Escuela de Biología, Universidad de Costa Rica, Ciudad Universitaria "Rodrigo Facio", San Pedro de Montes de Oca, San José, Costa Rica. Tel: (506)207-53-75, Fax: (506)207-53-92; pejibaye@cariari.ucr.ac.cr
2 Oficina Técnica de la Comisión Nacional para la Gestión de la Biodiversidad (CONAGEBio), Ministerio del Ambiente y Energía, San Pedro de Montes de Oca, San José, Costa Rica; johernan@costarricense.cr
3 Department of Biological Sciences, Kent State University, Kent, Ohio, 44242, USA; orocha@kent.edu
Abstract: Genetic diversity and kin relationships among wild and cultivated populations of the pejibaye palm (Bactris gasipaes, Palmae) using microsatellite markers. The genetic diversity of the peach palm (Pejibaye, Bactris gasipaes Kunth) was evaluated using four nuclear DNA microsatellites in an effort to elucidate the evolution and domestication of this crop. A total of 258 samples from seven wild populations and eleven races were analyzed. All loci were polymorphic and a total of 50 alleles were identified. Average genetic diversity (0.67) and genetic differentiation among populations (Fst=0.16) were high when all populations were considered. Genetic differentiation was lower when the populations were grouped according to their origin into Western and Eastern populations (Fst=0.13 for both). Gene flow was slightly higher among Western populations (Nm=1.71) than among Eastern populations (Nm=1.62). The Putumayo, Yurimaguas, vaupés, Tucurrique and Guatuso races seem to have been subjected to intense human selection. Hybrid populations exist in Azuero, Tuira, Cauca, vaupés, Puerto Ayacucho and Solimões, probably resulting from exchange and introgressions among sympatric wild and cultivated populations. Genetic distance (Dm) was estimated to determine the degree of relationship among populations using the neighbor-joining method; the wild populations from Maracaibo were used as the outgroup. The populations were divided into three general groups: Maracaibo (B. caribaea, B. macana var veragua and B. macana var arapuey), Eastern Amazon (Tembe, Pará and Acre) and a third group with two subgroups, Western (Azuero, Chontilla, Tuira, Cauca, Tucurrique and Guatuso) and Upper Amazon (B. dahlgreniana, Puerto Ayacucho, Solimões, vaupés and Putumayo). The genetic relationships strongly support the hypothesis that peach palm was brought into cultivation independently in no less than three areas: the Western Andes (extending into lower Central America); Upper Amazon (extending into the Solimões and its tributaries), and the Eastern Amazon (extending from Bolivia to the lower Amazon through the Madeira River). Rev. Biol. Trop. 56 (1): 217-245. Epub 2008 March 31.
Keywords: Bactris gasipaes, pejibaye, peach palm, genetic diversity, evolution, microsatellites, races, wild relatives.
El pejibaye (Bactris gasipaes Kunth) es la palmera de mayor importancia económica de origen americano. Es una planta alógama, autoincompatible, con número de cromosomas n=14 (Mora-Urpí 1992). Tiene una amplia distribución geográfica en el Neotrópico húmedo. Los parientes silvestres y variedades cultivadas de pejibaye se extienden en el ámbito comprendido entre los paralelos 16° N y 17° S (Mora-Urpí et al. 1997). A través de este inmenso territorio, esta planta ha sido utilizada como hortaliza (palmito), como fuente de energía para consumo humano y animal (fruta), para producción de aceite, y como fuente de madera para construcciones y la elaboración de herramientas (Mora-Urpí et al. 1997).
El pejibaye se cultivaba extensa e intensamente en las Américas antes de la llegada de los españoles en el siglo XV (Patiño 1963, Clement y Mora-Urpí 1982, Corrales y Mora-Urpí 1990). Los indígenas que habitaban la región de Talamanca (Costa Rica) durante la época precolombina consideraban que la fruta del pejibaye era más importante en su dieta que el maíz y la yuca (Ferrero 1981, Corrales y Mora-Urpí 1990). Otras culturas indígenas tanto de Suramérica como de Centro América utilizaban la madera del pejibaye para fabricar herramientas y armas (Patiño 1963). El valor de este cultivo trascendía hasta lo espiritual por su importancia para la supervivencia y el desarrollo de muchos grupos indígenas (Patiño 1963, Pellizzaro 1978).
La taxonomía del pejibaye es todavía motivo de controversia. Henderson (2002) propone reunir varias poblaciones tanto silvestres como cultivadas, designadas originalmente como especies diferentes bajo un solo taxón, Bactris gasipaes. El mismo autor también propone subdivididir esta especie en dos variedades, B.g. var chichagui (silvestre) y B.g. var gasipaes (cultivada), lo cual es aún motivo de controversia. En este trabajo se utilizan los nombres antiguos para algunas de las poblaciones silvestres por la amplia difusión que tuvieron en el pasado y su fácil relación geográfica. Por otra parte, las poblaciones silvestres descubiertas recientemente se citan identificándolas únicamente por el nombre de la región donde fueron colectadas. Las poblaciones cultivadas ó razas se agruparon originalmente en Occidentales y Orientales tomando como referencia el lado de la cordillera de Los Andes donde estaban localizadas (Mora-Urpí et al. 1993).
En este trabajo se adopta, la acepción general de "raza", como grupo de individuos que poseen un conjunto de características morfológicas y funcionales transmitidas por herencia (Font Quer 1963). Específicamente se aplica en el trabajo el término "raza", como sinónimo de "raza artificial o variedad primitiva", esto es, aquella población primitiva establecida con la participación del hombre; y se utiliza "población silvestre" como sinónimo de "raza natural" o sea aquella surgida sin participación del hombre. Ambos casos implican una localización geográfica de origen.
Las poblaciones, tanto silvestres como cultivadas, presentan gran diversidad genética entre y dentro de ellas; y en algunas de ellas el alto grado de variabilidad acusa su condición híbrida (Clement 1986, Mattos 1992, Mora-Urpí et al. 1993). Las Razas han evolucionado a partir de uno o varios ancestros silvestres con frutos pequeños con pesos menores de 10g, hasta alcanzar, con la participación humana frutos mayores de 100 g (Zúñiga 2004). Las diferentes razas, se distinguen entre sí, y de sus ancestros silvestres, con relativa facilidad por el tamaño, forma de frutos y semillas, tamaño y forma de las inflorescencias antes de su apertura, morfología de las flores, características de las espinas, tallos y hojas, así como por las condiciones ecológicas de la región que habitan, entre otros descriptores reconocidos (Clement 1986, Mattos 1992, Mora-Urpí et al. 1993, Ferreira 1999, Henderson 2000).
Debe tenerse presente que no todas las poblaciones silvestres y razas de pejibaye han sido identificadas, por lo que su número se desconoce. Por otra parte, la amplia y rápida erosión genética que ha tenido lugar durante el último siglo, hace posible que algunas de las razas hayan desaparecido. Además, el gran número de ellas se encuentran altamente contaminadas por el trasiego de material genético, lo cual dificulta la obtención de muestras válidas representativas de las poblaciones originales para su estudio.
Se han formulado varias hipótesis que tratan de explicar el origen de la domesticación de esta palmera. Se ha propuesto que este cultivo se originó en la región occidental de la cuenca amazónica (Oriente de los Andes), desde donde luego se difundió por todo su ámbito de distribución actual (Huber 1904, Vavilov 1951, Prance 1982, Clement et al. 1989, Ferreira 1999, Rodrigues et al. 2004). Cada uno de estos autores presenta aspectos particulares propios de su planteamiento. Por otra parte, Patiño (1989) sugiere que el cultivo del pejibaye tuvo su origen en el occidente de la Cordillera de los Andes y su uso original fue la madera para la fabricación de herramientas y armas de guerra; y solamente después se inició el uso de la fruta como alimento. Una tercera posición plantea que su origen es el resultado de la domesticación independiente de varias poblaciones silvestres situadas tanto al occidente como al oriente de la cordillera de Los Andes (Mora-Urpí 1993).
Además de estudios morfológicos, se han realizado varios trabajos con marcadores moleculares para establecer la diversidad presente y el grado de parentesco entre razas y poblaciones silvestres que permitieran fundamentar las hipótesis sobre el origen de su domesticación. Entre estas investigaciones se encuentran trabajos utilizando: isoenzimas (Rojas et al. 1999), RAPDs (Sousa et al. 2001, Rodrigues et al. 2004), AFLPs (Clement et al. 2002, Adin et al. 2004), y microsatélites (Martínez et al. 2002, Couvreur et al. 2006, Cole et al. 2007). Con estos marcadores se obtuvieron altos valores de polimorfismos y diversidad genética, lo cual es reflejo de la diversidad morfológica del cultivo, así como relaciones de parentesco. Todas estas investigaciones han tenido la limitación de no contar con muestras representativas de todas las poblaciones silvestres y cultivadas conocidas, dada la enorme dificultad de reunirlas para su estudio conjunto.
El objetivo de esta investigación es determinar los niveles de variación genética y establecer las relaciones de parentesco entre poblaciones silvestres y cultivadas mediante el uso de marcadores microsatélites descritos por Martínez et al. (2002), y contribuir a dilucidar la validez de los agrupamientos de poblaciones basados en su distribución geográfica y la similitud de caracteres morfológicos (Mora-Urpí 1993).
Los resultados de esta investigación constituyen un estudio preliminar, debido al bajo número de microsatélites y pequeño tamaño de las muestras utilizadas de algunas poblaciones. Sin embargo, al mismo tiempo tiene la fortaleza de la amplitud del territorio representado en las muestras usadas que le confieren solidez a otros aspectos.
Materiales y métodos
Recolección de muestras: para realizar este trabajo se utilizó, en su mayor parte, material genético del banco de germoplasma de pejibaye, situado en la región Caribe de Costa Rica. Esta colección se inició en 1972 y en la actualidad es la más grande a nivel internacional con 1 207 accesiones procedentes de Bolivia, Brasil, Colombia, Costa Rica, Ecuador, Nicaragua, Panamá, Perú y Venezuela. Se recolectaron 258 muestras de hojas de plantas silvestres y razas de pejibaye de los bancos de germoplasma de la Estación Experimental los Diamantes (Guápiles, Costa Rica) y de CATIE (Turrialba, Costa Rica). Las muestras utilizadas en este trabajo incluyen once poblaciones de pejibaye cultivado y siete poblaciones de pejibaye silvestre (Cuadro 1).

Extracción de ADN: El ADN genómico fue extraído de hojas utilizando el protocolo de Lodhi et al. (1994) y se resuspendió en 100 µl de TE con 3 µl ARNasa de 5 unidades/µl. La cantidad de ADN obtenida de cada muestra se cuantificó usando un fluorómetro Hoefer Dyna Quant 200. Finalmente, las muestras se diluyeron hasta una concentración de 10 ng/µl.
Análisis genético: se estudiaron los niveles de variación genética usando cuatro marcadores microsatélites desarrollados por Martínez et al. (2002). En particular se utilizaron los loci Bg6, Bg17, Bg51 y Bg63, debido a que fueron los únicos que amplificaron eficazmente para todas las muestras, descartando así 4 loci (Bg1, Bg21, Bg24, Bg29) de 8 loci que inicialmente se habían escogido para esta investigación. La reacción de PCR fue ejecutada en un termociclador PTC-200TM (MJ Research, Inc., Waltham, MA, USA). La mezcla de los reactivos, las condiciones de tiempo y temperatura de la reacción de la cadena de la polimerasa (PCR) para cada locus se describen en el cuadro 2.
Los productos de PCR se separaron en un gel de secuenciación de acrilamida-urea (7 M) al 5 % de 39 cm, con un campo eléctrico de 55 watt y a una temperatura de 55 °C. Una vez separadas las bandas amplificadas de ADN, estas fueron visualizadas usando el protocolo de tinción de plata para acrilamida (PROMEGA USA). Los diferentes alelos para cada locus fueron identificados según su tamaño. El tamaño de los alelos se determinó por interpolación, utilizando una ecuación de regresión calculada entre fragmentos de tamaño conocido (50 bp DNA ladder, Fermentas USA) y su distancia de migración en el gel.
Diversidad genética: la diversidad genética de cada población y grupos de poblaciones fue cuantificada por el número de alelos por locus (A), heterocigosis observada (Ho), heterocigosis esperada (He) y heterocigosis esperada de Nei bajo el equilibrio Hardy-Weinberg (1978), para cada locus y el promedio para todos los loci. Para todas las poblaciones se determinó la diversidad genética promedio. Para las frecuencias de los alelos se aplicó la prueba de homogeneidad a través de todas las poblaciones para cada locus. Estos análisis se elaboraron con el programa POPGENE 1.32 (Yeh et al. 1999). También, se realizó la prueba exacta de Fisher con los valores imparciales de P a través del método de la cadena de Markov (Guo y Thompson 1992), para determinar si los resultados se ajustaban a las predicciones del equilibrio Hardy-Weinberg. Para este análisis se utilizó el programa TFPGA 1.3 (Miller 1997).
Aquellas poblaciones en que se recolectó un bajo número de individuos, no se incluyeron en los análisis anteriores, pero si fueron incluidas en la comparación de frecuencias de alelos para cada grupo. Esto se hizo para corroborar si el alelo más común en las poblaciones con muestras iguales o superiores a 17 individuos es el mismo que las poblaciones en que se recolectó un número reducido de individuos (menor a 10 individuos).
Estructura de poblaciones: para estudiar la estructura de poblaciones se utilizaron solamente aquellas poblaciones con al menos 17 individuos. Por lo tanto, el grupo de Maracaibo se descartó por presentar baja cantidad de muestras. La estructura poblacional fue analizada utilizando los coeficientes F de Wright, según el modelo de alelos infinitos (Weir y Cockerham 1984). También se calculó el flujo genético (Nm) para cada grupo de poblaciones. Este análisis se hizo con el programa POPGENE 1.32 (Yeh et al. 1999).
Relaciones de parentesco: para analizar las relaciones de parentesco entre las poblaciones silvestres y cultivadas de pejibaye se estimó la distancia genética Dm (Neis minimum genetic distance; Nei 1973) basado en las frecuencias alélicas de cuatro loci de micro-satélites. La matriz generada de la distancia Dm entre las poblaciones fue representada utilizando el método del vecino más cercano ó "Neighbor-Joining" (Saitou y Nei 1987). La robustez de cada rama en el dendograma fue probada realizando 100 reiteraciones de muestreo utilizando la técnica de Bootstrapping. Este análisis fue realizado con el programa POPULATIONS 1.2.28 (Langella 2002). El dendograma generado fue visualizado con el programa TREEVIEW (Page 1996). Además, este dendograma fue modificado al determinar el taxon más distante después del análisis de agrupamiento. En este análisis se utilizó como el taxon más distante las tres poblaciones silvestres de la región de Maracaibo. Asimismo, en este análisis no se incluyo la población de Yurimaguas debido a su origen híbrido e intensa selección por tallos sin espinas que ha sufrido, lo cual podría dar falsos agrupamientos a nivel molecular.
Resultados
Diversidad genética: todos los loci resultaron polimórficos y se identificaron 50 alelos en total. El número de alelos observados por locus fue de 10 (Bg51), 12 (Bg06 y Bg17) y 16(Bg63) (Promedio=12.5±2.5). El nivel de variación fue alto, entre 18 a 35 alelos por población (Fig. 1). También se observó que la mayoría de los alelos se encuentran presentes en más de una población. La población de Tembe presenta en el locus Bg6 los alelos 156 y 228 como privados y raros; y en el locus Bg17 los alelos 219 y 221 como privados. Por otra parte, uno de los dos individuos de la población de Chontilla presenta un alelo privado, el 143 en el locus Bg51 (Cuadro 3). Las poblaciones de Azuero y Yurimaguas presentan el más alto número de alelos (35 y 33 respectivamente); mientras que las poblaciones de Tucurrique y Guatuso presentan el menor número de alelos (18 para ambas) (Fig. 1).
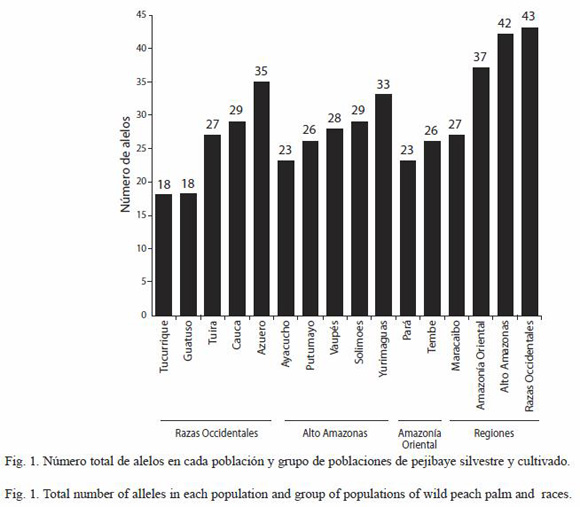
Las poblaciones del grupo Occidental presentan el mayor número de alelos (43), mientras que el grupo de la cuenca de Maracaibo el menor número (27) (Fig. 1). A pesar de que en las poblaciones de Maracaibo se analizaron pocas muestras, el número de alelos observados es alto. Por otra parte, dentro de los diferentes grupos de poblaciones se comparte un ámbito semejante en el tamaño de los alelos más comunes en los cuatro loci (Cuadro 3).
Poblaciones occidentales: el análisis genético indica que las poblaciones del subgrupo de poblaciones Occidentales presentan en el locus Bg6 un ámbito del tamaño de los alelos más comunes, entre los alelos 182 y 201 (Azuero, 182; Guatuso y Chontilla, 185; Tuira y Tucurrique, 192; Cauca, 201). En el locus Bg17 el ámbito se encuentra entre los alelos 230 y 233 (Tuira y Chontilla, 230; Azuero, Guatuso y Cauca, 232; y Tucurrique, 233). En el locus Bg63 el alelo más común es el 135 para las poblaciones de Azuero, Guatuso, Tucurrique y Cauca, y es el segundo más común para la población de Tuira; sin embargo, las poblaciones de Tuira y Chontilla presentan los alelos 119 y 178 respectivamente como los más comunes. En el locus Bg51, todas las poblaciones divergen respecto a su alelo más común. En general en las Poblaciones Occidentales, el alelo más común del locus Bg6 es el 192; en el locus Bg17 el 232; y en el locus Bg63 es el alelo 135 (Cuadro 3).
Alto Amazonas: para la región del Alto Amazonas los datos revelan que en el locus Bg6 el alelo más común es el 172 para las poblaciones de B. dahlgreniana, Vaupés, Yurimaguas, Solimões y Puerto Ayacucho; en cambio la población de Putumayo presenta el alelo 182 y 185 como el primero y segundo más común. En el locus Bg17 el alelo más común es el 234 para las poblaciones de Putumayo, Vaupés, y Puerto Ayacucho; aunque los alelos más comunes para las poblaciones de B. dahlgreniana, Yurimaguas y Solimões fueron el 225, 230 y 232 respectivamente. En el locus Bg63 el alelo más común para las poblaciones de B. dahlgreniana y Putumayo es el alelo 129; para las poblaciones de Solimões y Puerto Ayacucho es el alelo 143; para las poblaciones de vaupés y Yurimaguas los alelos más comunes fueron el 135 y 119 respectivamente. En el locus Bg51, el ámbito de los alelos más comunes está entre los alelos 192 y 195; aunque el alelo más común de la población de Putumayo es el 162. Para este subgrupo del Alto Amazonas en general, se observa que en el locus Bg6 el alelo más común es el 172; en el locus Bg17 es el 234; y en el locus Bg63 es el 143 (Cuadro 3).
Amazonía Oriental: en la región de la Amazonía Oriental se observa que en el locus Bg6 el alelo más común es el 177 para todas las poblaciones (Tembe, Pará y Acre). En el locus Bg17 los alelos más comunes para Tembe y Pará son el 221 y 222 respectivamente; sin embargo para Acre es el alelo 232. En el locus Bg63 el ámbito de los alelos más comunes está entre los alelos 146 y 157 ( Pará y Acre, 146; Tembe, 153; y Acre, 157 ). En el locus Bg51, el ámbito de los alelos más comunes se encuentra entre los alelos 162 y 169. En este subgrupo de la Amazonía Oriental, para todas las poblaciones en general se observa que el locus Bg6 presenta el alelo 177 como el más común; en el locus Bg17 es el alelo 222; y en el locus Bg63 es el alelo 146 (Cuadro 3).
Maracaibo: para las poblaciones de Maracaibo los alelos más frecuentes en el locus Bg6 presentan un ámbito entre los alelos 177 y 182 (B. macana var veragua y B. caribaea, 177; B. macana var arapuey, 182). En el locus Bg17 el alelo más común para las poblaciones de B. macana var veragua y B. macana var arapuey fue el alelo 230, pero también el alelo 224 fue el más común en la población de B. macana var arapuey; en cambio en la población de B. caribaea el alelo más común fue el 237. En el locus Bg63 el alelo más común para las poblaciones de B. macana var arapuey y B. caribaea fue el 159; sin embargo en la población de B. macana var veragua fueron los alelos 141 y 157. En el locus Bg51, el alelo más común es el 186, además en la población de B. caribaea también se observa el alelo 149 como el más común. En general para el grupo de Maracaibo, se observa que en el locus Bg6, el alelo más común es el 177; en el locus Bg17 el alelo más común es el 230; y en el locus Bg63 el más común es el alelo 159 (Cuadro 3).
También, se realizó una prueba de homogeneidad de frecuencias alélicas entre las diferentes poblaciones. Este análisis revela una heterogeneidad altamente significativa en la frecuencia de los alelos entre las poblaciones para los cuatro loci analizados (p<0.0001).
La heterocigosis promedio observada en todas las poblaciones de pejibaye se encuentran entre 0.36 y 0.74. Las poblaciones de Pará y Tucurrique presentaron el más bajo nivel de heterocigosis promedio observado (0.36); mientras que la población de Puerto Ayacucho presentó el más alto (0.74) (Cuadro 4). El ámbito de heterocigosis observado varía según el locus, en el locus Bg6 se encuentra entre 0.20 y 0.65, en el locus Bg17 entre 0.30 y 0.76, en el locus Bg63 entre 0.20 y 0.65, y en el locus Bg51 entre 0.15 y 0.82.
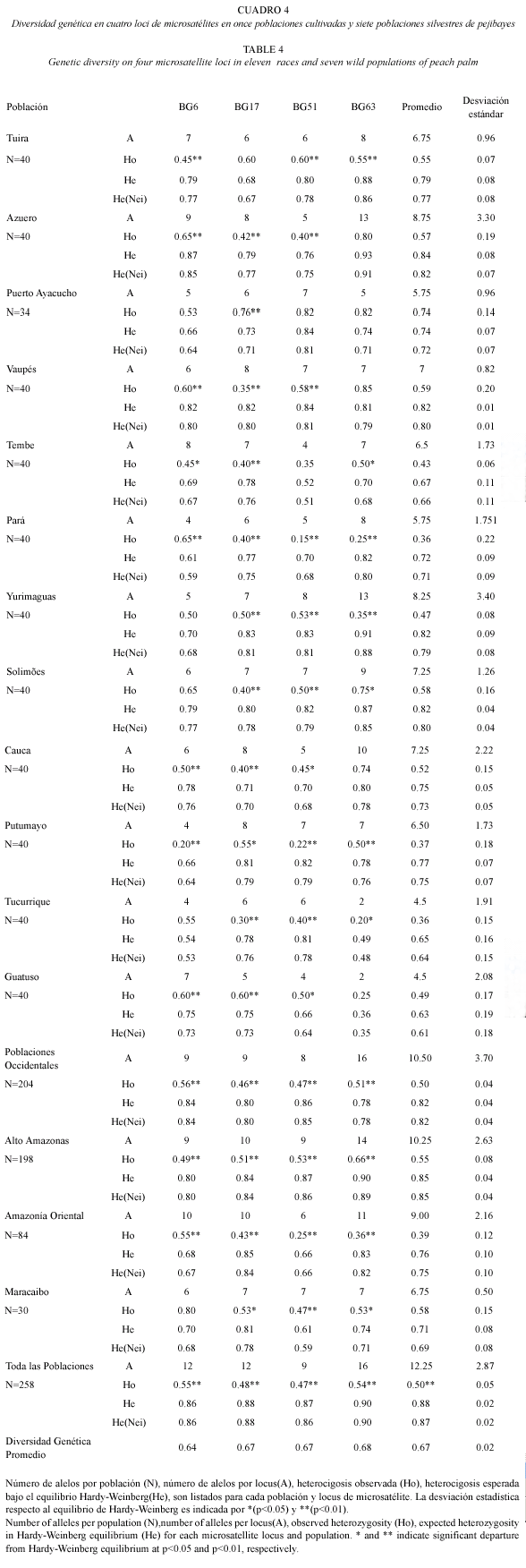
La diversidad genética promedio para todos los loci fue de 0.67. En los cuatro loci no se observó diferencia significativa entre los valores de diversidad genética promedio. El locus Bg6 presenta una diversidad genética promedio de 0.64 ligeramente menor que aquella de los loci Bg17 y Bg51 la cual es de 0.67, y del locus Bg63 la cual es 0.68.
La prueba exacta de Fisher mostró que de las 48 comparaciones realizadas por locus en cada población, 36 presentaban una desviación significativa con respecto al equilibrio Hardy-Weinberg (p<0.05; Cuadro 4). Todas las poblaciones de pejibaye cultivado y silvestre presentan por lo menos un locus con desviación significativa. En la mayoría de los subgrupos de poblaciones de pejibaye, se observó una desviación del equilibrio Hardy-Weinberg para todos los loci, excepto en el subgrupo de Maracaibo para el locus Bg6.
Estructura de poblaciones: para todas las poblaciones de pejibaye, el coeficiente de endogamia promedio dentro de las poblaciones (FIS) de todos los loci es de 0.31 (Cuadro 5). Por otra parte, los grupos y subgrupos de pejibaye dentro de las poblaciones presentan un ámbito de endogamia entre 0.29 y 0.42. Las Poblaciones Occidentales y el Alto Amazonas presentan los valores más bajos de endogamia (0.30 y 0.29 respectivamente), mientras que el Amazonía Oriental presenta el mayor valor de endogamia (0.42).
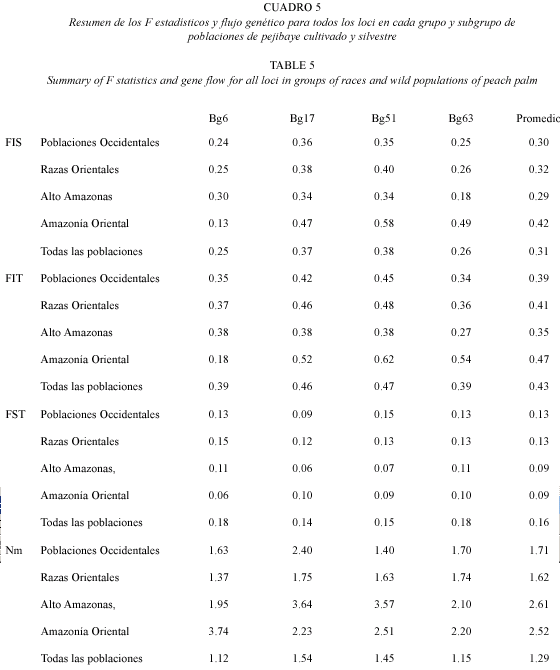
En este análisis se obtuvo, para todas las poblaciones de pejibaye, un valor de 0.43 en el coeficiente total de endogamia (FIT) de todos los loci (Cuadro 5). Entre los subgrupos de poblaciones de pejibaye el que presentó mayor valor de endogamia total fue el de la Amazonía Oriental (0.47); mientras que el subgrupo del Alto Amazonas fue el más bajo (0.35).
Entre todas las poblaciones de pejibaye, se observó un FST promedio de 0.16 de todos los loci (Cuadro 5). Los grupos de poblaciones de Razas Orientales y Poblaciones Occidentales presentaron los mayores valores de FST promedio para todos los loci (0.13); sin embargo, al dividir el primero en dos subgrupos: Alto Amazonas y Amazonía Oriental, se obtuvo el valor de FST más bajo para ambos (0.09).
El flujo genético promedio de todos los loci entre todas las poblaciones de pejibaye fue de 1.29 (Cuadro 5). El subgrupo de poblaciones que presentó el mayor flujo genético fue el Alto Amazonas (2.61), mientras que reunidas todas las poblaciones del Alto Amazonas y Amazonía Oriental en Razas Orientales como un solo grupo fue menor (1.62).
Análisis de parentesco: las distancias genéticas Dm entre las poblaciones de pejibaye cultivado y silvestre, están representadas en el dendograma construido con el método del vecino más cercano (Fig. 2). Se observa una distribución de tres grupos principales, con el último grupo dividido en dos subgrupos. Dentro de cada grupo, las poblaciones son vecinas dentro de una extensa pero definida región geográfica. Los valores de reiteraciones (Bootstrap values) en el dendograma fueron bajos (=35 % de reiteraciones). Las ramas principales del dendograma que separan cada grupo geográfico también presentaron valores bajos (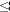 35 % de reiteraciones), mientras que algunos de los nodos intermedios y terminales entre dos poblaciones fueron mayores a 35 % de reiteraciones. En este dendograma, las poblaciones que presentaron el más alto soporte de valor de reiteraciones fueron Pará y Acre (86 %), y Tucurrique y Guatuso (82 %). Por otra parte, se observa que todas las poblaciones silvestres y cultivadas de pejibaye se agruparon con los grupos geográficos esperados.
35 % de reiteraciones), mientras que algunos de los nodos intermedios y terminales entre dos poblaciones fueron mayores a 35 % de reiteraciones. En este dendograma, las poblaciones que presentaron el más alto soporte de valor de reiteraciones fueron Pará y Acre (86 %), y Tucurrique y Guatuso (82 %). Por otra parte, se observa que todas las poblaciones silvestres y cultivadas de pejibaye se agruparon con los grupos geográficos esperados.
Discusión
Diversidad genética: existe una alta diversidad genética dentro y entre las poblaciones de pejibaye. Esta diversidad está asociada a su antigüedad y amplia distribución que incluye una gran variedad de hábitats y al aislamiento entre poblaciones debido a barreras físicas. Estos factores permiten la divergencia y la evolución independiente de las poblaciones. Por último, la influencia del hombre a través del proceso de domesticación y selección ha contribuido a la aparición de una cantidad considerable de razas, las cuales contribuyen al incremento de la variabilidad fenotípica.
Las poblaciones de pejibaye cultivado y silvestre se agrupan principalmente por compartir el alelo más común o alelos de tamaños semejantes. A pesar de que en la mayoría de las poblaciones silvestres se recolectaron pocas muestras, es frecuente que predomine en ellas el alelo más común de su grupo. Este agrupamiento concuerda con la ubicación geográfica y similitudes morfológicas de las familias raciales de pejibayes cultivados propuesto por Mora-Urpí (1993).
Las poblaciones de pejibaye presentan una diversidad genética promedio similar a otras especies de cocoides cultivados de amplia distribución geográfica. Perera et al. (2000), estudiaron la diversidad genética de Cocos nucifera L. utilizando ocho loci de microsatélites y encontraron también una diversidad genética promedio alta de 0.589 la cual es ligeramente menor a la observada en pejibaye (0.67). Por otra parte, Billote et al. (2001) estudiaron la diversidad genética de Elaeis guineensis y E. oleifera utilizando 21 loci de microsatélites. En la especie E. guineensis, se estimó una diversidad genética promedio de 0.68 para 21 microsatélites; mientras que E. oleifera presentó una diversidad genética promedio de 0.63, ambas medidas fueron similares a la diversidad genética promedio del pejibaye. Estos cultivos e incluyendo el pejibaye, presentan una alta diversidad genética debido a su amplia distribución geográfica y a la selección llevada a cabo por el hombre. La diversidad genética observada en el pejibaye es similar a la reportada para estas tres especies.
La diversidad genética promedio descrita en este estudio fue mucho mayor que la descrita por otros autores utilizando diferentes marcadores (Rodrigues et al. 2004, Adin et al. 2004). Rodrigues et al. (2004) encontraron una diversidad genética promedio de 0.31 utilizando 113 marcadores RAPDs, en 220 plantas de siete morfo-razas de pejibaye (Tuira, Utilis, Guatuso, Putumayo, Solimões, Pampa Hermosa sin. Yurimaguas, B. dahlgreniana var benjamín constant y B. dahlgreniana var acre). Adin y sus colaboradores (2004) evaluaron la diversidad genética de las poblaciones de pejibaye cultivado de los ríos Paranapura y Cuiparillo de Perú, cerca de Yurimaguas. Además incluyeron como un grupo aparte las poblaciones de Putumayo, Solimões y Pará. En las poblaciones de Perú la diversidad genética promedio fue de 0.248 y para las poblaciones de Putumayo, Solimões y Pará fue de 0.237 utilizando 203 marcadores AFLPs . Sin embargo, los mayores niveles de variación reportados en el estudio presente, concuerdan con lo esperado en la utilización de microsatélites por tener estos una alta tasa de mutación (Goldstein y Pollock 1997, Estoup y Cornuet 1999).
Nuestros datos revelan que la diversidad alélica de las poblaciones silvestres es alta. La población silvestre de Azuero presentó el mayor número de alelos (35) de todas las poblaciones estudiadas y una alta heterocigosis observada (0.57). Por otra parte, las tres poblaciones de la región de Maracaibo en conjunto, también presentaron un alto número de alelos (27) y valor de heterocigosis (0.58); aunque sólo se recolectaron pocos individuos de ellas (15). Estos resultados coinciden con lo reportado por Couvreur et al. (2005), que observaron una alta heterocogosis en dos poblaciones silvestres de Chontilla (0.58 y 0.56 respectivamente). Las poblaciones de la Amazonía Oriental, Tembe (c.f., B. insignis) y Pará, aparentemente han estado aisladas de las demás poblaciones del Alto Amazonas. En la población de Tembe, la presencia de 4 alelos privados (156 y 228, Bg6; 219 y 221, Bg17), evidencia su aislamiento. Por otra parte, la población de Pará presenta la más baja heterocigosis observada promedio de todas las poblaciones (0.36), lo cual sugiere un alto nivel de endogamia dentro de la población.
La población de Puerto Ayacucho, cuenca del río Orinoco, podría ser el resultado de una reciente hibridación. Esta población presenta un número de alelos obtenidos no muy alto (23), pero la mayor heterocigosis (0.74). Esto sugiere que tiene un origen híbrido. Sin embargo, los frutos de esta población son morfológicamente muy homogéneos (J. Mora-Urpí, obs. pers.). Basado en los datos obtenidos y en los antecedentes, es difícil proponer una hipótesis sobre el origen de la población de Puerto Ayacucho, pero es muy probable que exista una estrecha relación con vaupés e Inírida, ya que éstas son las dos poblaciones conocidas más cercanas geográficamente a la población de Puerto Ayacucho. Esta hibridación sería una de las más recientes ya que el proceso de selección del cultivo no ha tenido suficiente tiempo para disminuir su heterocigosis.
La población de Vaupés tiene una alta variabilidad a nivel molecular; sin embargo, morfológicamente es bastante homogénea. Esta población presenta altos valores de número de alelos observados (28) y heterocigosis promedio observada (0.59). En contradicción, es una de las más seleccionadas por el mayor tamaño de frutos (>70 g) y su semilla tiene una forma particular que no está presente en las otras poblaciónes (Mattos 1992, Mora-Urpí et al. 1993). Por otra parte, la heterocigosis observada del locus Bg17 es una de las más bajas (0.35). Esto parece ser el resultado de la selección hecha por el hombre, la cual pudo haber ocurrido a partir de un grupo bastante heterogéneo ó bien asociado a múltiples introgresiones de otras razas morfológicamente similares, como las razas hermanas de Putumayo, Caquetá e Inírida (Mora-Urpí et al. 1993).
La población del río Solimões ó curso medio del Amazonas es un híbrido. Esta población presenta valores altos de número de alelos observados (29) y heterocigosis observada (0.58) en los cuatro loci. Adin et al. (2004) reportaron que dos poblaciones locales de la raza Solimões mostraron una alta diversidad genética estimada utilizando AFLPs. De manera similar, Rodrigues et al. (2004) también reportaron una alta heterocigosis estimada para ésta población utilizando RAPDs. Por otra parte, Rodrigues et al. (2004) proponen que esta raza es el resultado de la hibridación de las morfo-razas de Putumayo y Pará. Otra alternativa, sería que la diversidad presente en el río de Solimões fuera el resultado de múltiples hibridaciones de diferentes razas de pejibaye que se encuentran en contacto a través de los tributarios del río Amazonas.
En la población de Putumayo se evidencia la intensa selección hecha por el hombre para obtener frutos de gran tamaño y ausencia de espinas (Mora-Urpí 1993, Mora-Urpí et al. 1993). En los loci Bg6 y Bg51 se presentaron heterocigosis de las más bajas observadas de todos los loci de todas las poblaciones (0.20 y 0.22 respectivamente). Estos datos indican que la selección hecha por el hombre afectó fuertemente estos dos loci, posiblemente por ligamiento más estrecho a genes que expresan las características sometidas a selección.
La población de Yurimaguas ha sido considerada por Mora-Urpí (1993) de origen híbrido por su diversidad morfológica y esterilidad masculina. Esta población ocupa el segundo lugar por el alto número de alelos obtenidos (33) para los cuatro loci. Pero por otra parte, no presentó la alta heterocigosis promedio observada (0.47) que se esperaría de un híbrido. Sin embargo, esta población ha sido sometida a una alta presión de selección por los indígenas para obtener tallos sin espinas, razón por la cual es apreciada en la actualidad.
La población de Cauca también presenta una alta diversidad. Esta población muestra un alto número de alelos observados (29) y uno de los más altos valores de heterocigosis observado (0.52). Esta raza podría situarse en un sitio de confluencia de razas derivadas básicamente de Chinamato ó var chichagui (sensu Karsten). A la vez, es un sitio intermedio donde podría ocurrir flujo genético de la raza Tuira desde el Norte, y de la población silvestre de Chontilla desde el Sur (Mora-Urpí 1993).
Las poblaciones de Guatuso y Tucurrique pueden resultar de una intensa selección hecha por los grupos indígenas de Costa Rica. Estas poblaciones presentan el más bajo número de alelos observados de todas las poblaciones y una baja frecuencia de heterocigosis. En el locus Bg63 de estas poblaciones se presentaron únicamente los alelos 135 y 163, indicando que estas poblaciones se originan de un mismo ancestro.
La población de Tuira presenta la más alta heterocigosis de todas las poblaciones occidentales cultivadas (0.55). El sitio en que se ubica esta población podría tener un flujo genético de la población cultivada de Cauca, y las poblaciones silvestres de Chinamato ó var chichagui y Darién; esta última traslapa su distribución geográfica con la población cultivada de Tuira. A su vez, podría haber existido un flujo genético entre la población de Tuira y la silvestre de Azuero, ya que estas poblaciones comparten el alelo 178 del locus Bg63.
En resumen los datos de diversidad genética indican que las poblaciones cultivadas de Putumayo, Vaupés, Tucurrique y Guatuso parecen haber sufrido una intensa selección. Además, estos resultados muestran la posible existencia de las poblaciones híbridas: Azuero, Tuira, Cauca, vaupés, Puerto Ayacucho, Yurimaguas y Solimões, como consecuencia del intercambio de semillas entre los pobladores del neotrópico e introgresiones de poblaciones simpátricas silvestres y cultivadas.
Estructura de poblaciones: las poblaciones del Alto Amazonas tienen un origen diferente a las poblaciones de la Amazonía Oriental. La diferenciación genética entre las poblaciones (FST) de las Razas Orientales (0.13) en conjunto, es mayor que la presente en sus subgrupos separados: Alto Amazonas (0.09) y Amazonía Oriental (0.09).
La dispersión de pejibayes también sucede en gran medida a través de los ríos. Esto se evidencia en los subgrupos de pejibaye del Alto Amazonas y Amazonía Oriental donde muestran la presencia de un mayor flujo genético que en el grupo de las Poblaciones Occidentales (Cuadro 5). Esto puede estar asociado a la compleja historia geológica del río Amazonas y sus tributarios responsables de la dispersión del pejibaye. Por otra parte, las Poblaciones Occidentales se encuentran aisladas de la Orientales por la infranqueable cordillera de Los Andes y no se encuentran interconectadas sus poblaciones por una red de ríos como las demás razas y poblaciones silvestres.
Las Poblaciones Occidentales difieren de las Razas Orientales. Al evaluar en conjunto las Poblaciones Occidentales reunidas con las Razas Orientales, el nivel de diferenciación genética (FST) fue de 0.16, en tanto que los valores observados dentro de los grupos de Poblaciones Occidentales y Razas Orientales fueron de 0.13 para ambos. Normalmente una diferenciación genética entre poblaciones (FST) superior a 0.15 es considerada como una indicación de significante diferenciación entre ellas (Frankham et al. 2002).
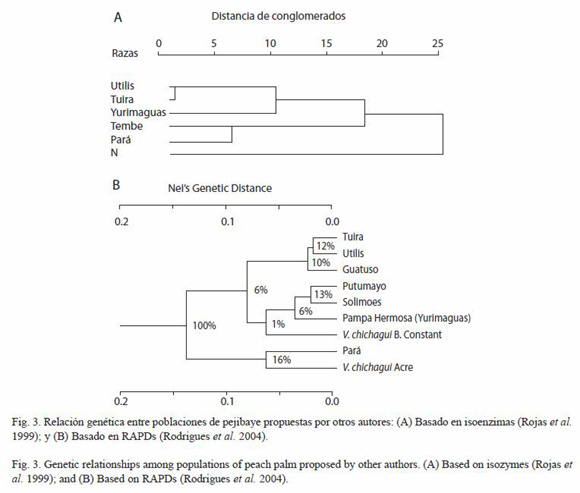
Relaciones de parentesco: en este estudio, los resultados son similares al de otras investigaciones que utilizaron otros marcadores (Rojas et al. 1999, Rodrigues et al. 2004) (Fig. 3). Nuestro análisis basado en distancia genética Dm, revela que existen tres grupos de poblaciones silvestres y razas de pejibaye (Fig. 2). Además uno de estos grupos se divide en dos subgrupos.
Las poblaciones silvestres de pejibaye del área del Gran Maracaibo se utilizaron como el taxa más distante, debido a sus características morfológicas, aislamiento geográfico e historia geológica. Ferreira (1999) estudió la filogenia morfológica y anatómica de varias especies del género Bactris, y ubicó a Bactris macana (Macana Norte para Ferreira) como la población de pejibaye silvestre más primitiva. El mismo autor propone que la población de Acre (Macana Sur para Ferreira) es la más cercana a los pejibayes cultivados. Por otra parte, Zúñiga (2004) realizó una revisión de la historia geológica de la cuenca de Maracaibo y propone que las poblaciones de B. caribaea y B. macana son el resultado de una migración de pejibayes silvestres ocurrida cuando el río Amazonas vertía sus aguas en la cuenca de Maracaibo, lo cual ocurrió durante el Mioceno temprano (aproximadamente hace 23-16 millones de años). Además, Zúñiga (2004) señala que en la cuenca de Maracaibo pareciera que no existieron pejibayes cultivados hasta su reciente introducción por el hombre. Los resultados de la presente investigación indican una estrecha relación entre las variedades Arapuey y veragua de B. macana (67 % de reiteraciones) y estas dos variedades se agrupan junto con B. caribaea para formar el grupo de Maracaibo (Fig. 2).
Las poblaciones cultivadas de Pará y Tembe (c.f., B. insignis), y la población silvestre de Acre (B. dahlgreniana sensu Clement), presentan un parentesco ancestral común (Fig. 2). La población de Acre se agrupa estrechamente con la raza de Pará (86 % de reiteraciones), y juntas se agrupan con la población de Tembe (60 % de reiteraciones). Basado en las características morfológicas y distribución geográfica, Mora-Urpí (1993) argumenta que las razas de Tembe y Pará comparten el mismo ancestro y propone que fueron domesticadas a partir de la población silvestre de B. insignis. En otra investigación, Rojas et al. (1999) utilizaron diez loci isoenzimáticos y se obtuvo un dendograma que mostraba una estrecha relación fenética entre las razas de Pará y Tembe. Además, Rodrigues et al. (2004) también obtienen la agrupación de Acre con la población de Pará. Esto parece indicar que esta extensa región aquí denominada Amazonía Oriental, comprende un centro de domesticación independiente del pejibaye, en donde no se practicó una selección muy estricta a juzgar por el pequeño tamaño de los pejibayes cultivados en ella.
Las poblaciones del Alto Amazonas se agrupan estrechamente (Fig. 2). En el dendograma se observa que se agrupan la población silvestre de B. dahlgreniana (sensu Glassman) con la población cultivada de Vaupés; y la población de Puerto Ayacucho con la población de Solimões; y todas juntas como un grupo monofilético. Por otra parte, la población de Putumayo se ubica como un grupo hermano(Sister group) de las demás poblaciones del Alto Amazonas, sin embargo el arreglo de esta rama en el dendograma no es significativa según el análisis de reiteraciones. Mora-Urpí (1993) afirma que las razas Vaupés e Inírida, por su proximidad geográfica y gran similitud morfológica, son obviamente razas hermanas, y a la vez pueden agruparse junto con la raza Putumayo, incluyendo la población del río Caquetá. Además considera que todas ellas podrían ser derivadas de la población silvestre "Capu" (Mora-Urpí 1993) y representa una segunda gran área –aquí llamada Alto Amazonas- en donde tuvo lugar un segundo centro de domesticación del cultivo y en donde la selección se practicó intensamente. En la investigación de Rodrigues et al. (2004) utilizando RAPDs, las poblaciones de Putumayo, Solimões, Pampa Hermosa (sin. Yurimaguas) y B. dahlgreniana la población silvestre de Benjamín Constant (B. dahlgreniana var benjamín constant; sensu Rodrigues et al. 2004), se reunieron dentro de un mismo grupo.
Las poblaciones de Guatuso y Tucurrique tienen una estrecha relación, mostrando un origen común. Mattos (1992) reportó una asociación fenotípica positiva entre las poblaciones de Tuira (Panamá), Guápiles (Costa Rica) y San Isidro de El General (Costa Rica). Estas dos últimas poblaciones no son las mismas que se utilizaron para este estudio con micro-satélites, pero tienen estrecha relación morfológica y cercanía geográfica con las razas de Guatuso y Tucurrique, y juntas se agrupan bajo el nombre de Utilis (Mora-Urpí 1999). Esta relación también fue descrita por Rojas et al. (1999), donde se mostró una estrecha relación fenética de las razas Utilis-Guápiles (Costa Rica) y Tuira-Darién (Panamá). Es posible que las poblaciones de pejibaye de Costa Rica se originaran de la población silvestre de Darién ó de una muy semejante genéticamente, de la cual desciende también la raza Tuira de Panamá (Rojas et al. 1999). Sin embargo, en esta investigación no se observa en el dendograma una relación directa entre Guatuso y Tucurrique con Tuira, sino más cercanas a Cauca; este arreglo en el dendograma se podría deber a la estrecha relación morfológica y geográfica entre Tuira y Cauca.
De acuerdo a los datos aquí obtenidos, las poblaciones de Cauca y Tuira muestran una relación polifilética derivadas de un origen en común (Fig. 2). Mora-Urpí (1993) propuso que la raza cultivada de Cauca era derivada de la población silvestre del valle del Cauca denominada popularmente Chinamato (B. gasipaes var chichagui sensu Karsten). Una explicación alternativa para esta estrecha relación podría ser un alto flujo genético entre estas razas debido a su cercanía geográfica. Para comprobar estás hipótesis es necesario hacer nuevas investigaciones que incluyan las poblaciones silvestres de Chinamato y Darién.
Asimismo, los resultados muestran que las poblaciones de Tuira y Chontilla tienen una relación monofilética. Por otra parte, Chontilla y Tuira también presentan una gran similitud morfológica (J. Mora-Urpí, obs. pers.), sin embargo la poca cantidad de muestras de la población de Chontilla podría estar dando una apreciación equivocada de la filogenia.
Las Poblaciones Occidentales comparten un origen en común, según el análisis de parentesco realizado en este trabajo (Fig. 2). Asimismo, otros investigadores reportaron que las Poblaciones Occidentales Tuira, Tucurrique y Guatuso se agruparon juntas (Rodrigues et al. 2004). La población silvestre de Azuero se agrupa como el más lejano (Sister group) de las demás poblaciones Occidentales. De este resultado se infiere que las poblaciones cultivadas occidentales se domesticaron independientemente de las amazónicas a partir de poblaciones silvestres nativas, constituyendo una tercera área o "centro" de domesticación. Sin embargo, estas conclusiones no pueden considerarse definitivas hasta poder incluir más muestras de Chontilla; Chinamato, Darién, Azuero y Upala.
La inclusión de híbridos en este tipo de estudio de poblaciones podría generar conclusiones equivocadas como resultado de su condición híbrida. En está investigación, el análisis de parentesco agrupó fuera del grupo geográfico esperado a dos poblaciones híbridas: Puerto Ayacucho (Orinoco) y Solimões (curso medio del amazonas) (Fig. 2). La estrecha relación obtenida entre estas poblaciones podría deberse a un efecto que provoca falsos agrupamientos por sus orígenes híbridos. Lo esperado geográficamente, es que Solimões se agrupe más cercano con Putumayo, como fue observado por Rodrígues et al. 2004, Sousa et al. 2001 y Clement et al. 2002; y Puerto Ayacucho con la población de vaupés (J. Mora-Urpí, obs. pers.). En esta investigación, los resultados de diversidad genética molecular indican que la población de Solimões es un híbrido, y los trabajos realizados por Adin et al. (2004) y Rodrigues et al. (2004) confirman que la población de Solimões tiene un origen híbrido.
La población de Yurimaguas fue excluida del dendograma debido a que se agrupó fuera de su grupo geográfico esperado con las Razas Occidentales (datos no mostrados). Al incluir una población híbrida e intensamente seleccionada por sus tallos sin espinas podría dar falsas agrupaciones. Esto ha sido ilustrado en un estudio de filogenia molecular en el género Solanum, donde los resultados en el análisis de agrupamiento, ubican a los clones híbridos de papa en subgrupos que no corresponden al linaje de cada híbrido (Sun et al. 2003). Por lo tanto, al utilizar poblaciones de origen híbrido complejo, como se presume que es el caso de Yurimaguas, en una investigación de filogenia molecular con marcadores de origen nuclear podrían surgir falsos agrupamientos; mientras que al utilizar marcadores de origen mitocondrial o de cloroplasto evitarían estos errores.
A priori, se esperaba que las poblaciones silvestres de Maracaibo tuvieran una mayor relación filogenética con las poblaciones del Alto Amazonas, por la historia geológica de este río. Sin embargo, los resultados ubican a las poblaciones silvestres de Maracaibo como un grupo aparte de los demás (Fig. 2). Una revisión de la historia geológica y climatológica de la cuenca del Amazonas, cordillera de Los Andes y Centroamérica, es importante para comprender las condiciones que han determinado la distribución de los pejibayes.
Prehistoria y el protopejibaye: el género Bactris, y con este el subgrupo de los pejibayes ó Guilielma, es endémico del Neotrópico, lo cual indica que se originó en el continente suramericano posteriormente a su separación del continente africano. Luego, la historia evolutiva de los pejibayes está íntimamente relacionada con la historia geológica de la cuenca del Amazonas y sus tributarios, la emergencia de las cordilleras de Los Andes y finalmente la formación de Centro América.
El río Amazonas originalmente desembocaba en el Océano Pacífico a la altura de Guayaquil en Ecuador (Hoorn et al. 1995, Fig. 4). Se considera que los ríos han sido históricamente importantes dispersores de semillas, complementados por otros agentes. Se asume aquí, que el Amazonas y sus afluentes distribuyeron el pejibaye a través de su recorrido, llevándolo hasta el occidente de Ecuador (Mioceno Medio, hace 16-10 millones de años), de donde se deriva la población silvestre actual de "Chontilla". Esta población muestra -como es de esperarse- una relación genética con las Poblaciones Occidentales. Luego, estos pejibayes continúan su migración al norte hasta eventualmente alcanzar la costa del Caribe de Nicaragua, dando origen a diferentes poblaciones silvestres (Chinamato, Chontilla, Darién, Azuero, Upala, y eventualmente a varias razas) a través de ese inmenso territorio, constituyendo el complejo de pejibayes "Occidentales". Por otra parte, los pejibayes que permanecen en la cuenca amazónica constituyen el complejo de pejibayes "Orientales"(Mora-Urpí 1993).
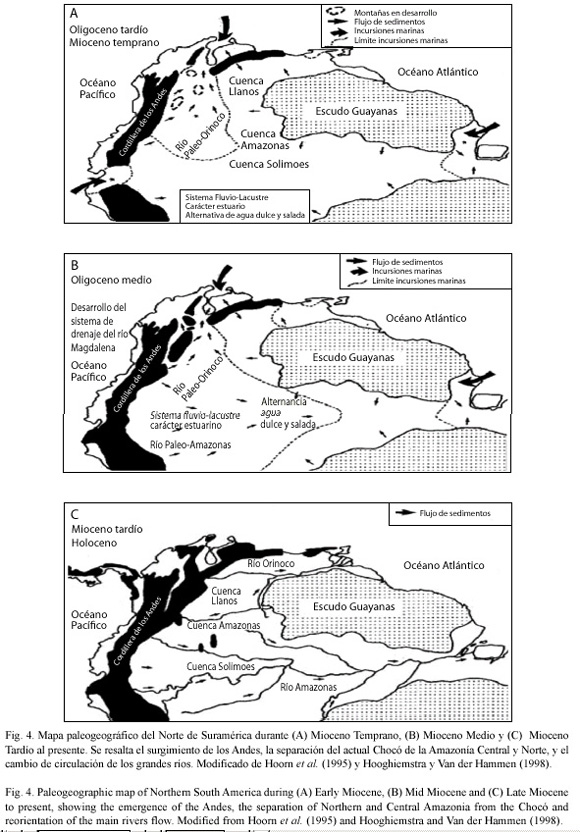
Al elevarse paulatinamente la cordillera de Los Andes y obstruir la salida del Amazonas al Pacífico, lo obliga a buscar una salida alternativa. Esto lo hace formando, con su ruta, un arco hacia el norte para desembocar en la entonces bahía de Maracaibo (Hoorn et al. 1995). Para ello sigue la ruta de lo que eventualmente, al levantarse la cordillera de Los Andes Orientales (Mioceno tardío hace 7-6 millones de años) y cerrarle de nuevo esta salida al Amazonas, se convertiría en el río Magdalena en su trecho final, el cual continuaría desaguando en el Mar Caribe. De nuevo, el curso prehistórico del Amazonas habría contribuido a dispersar los pejibayes Orientales por esa ruta. En este estudio sólo se utilizaron algunas poblaciones silvestres de la región de Maracaibo, pero relacionando los resultados con esta historia geológica, es de esperar que las poblaciones silvestres y razas de la cuenca del Magdalena muestren afinidad genética con las de Maracaibo.
La desembocadura del Amazonas en el Caribe durante el Mioceno tardío podría representar una segunda puerta para que el pejibaye migrara al occidente de Los Andes, pero de acuerdo con los resultados expuestos en este estudio eso no ocurrió. Posiblemente la región del Caribe de Colombia, históricamente de clima seco (Hernández-Camacho et al. 1992), representó una barrera infranqueable para el pejibaye. Por otra parte, también se considera que el antiguo Río Orinoco desembocaba en la cuenca de Maracaibo durante el Mioceno temprano hasta finalizar el Mioceno Medio; y en este último periodo cambio su curso a la salida actual por la región de los Llanos de Venezuela (Díaz de Gamero 1996), lo cual también pudo llevar pejibayes silvestres a esta región. Silva & Clement (2005) describen un pejibaye silvestre en la región del río Xingú, situado al oriente del río Madeira, con un fruto de peso similar a B. caribaea (0.45 g y 0.50 g respectivamente), y de igual manera adaptado a condiciones de trópico seco y suelos pobres. Estas evidencias podrían apoyar la teoría de la existencia de un protopejibaye que migró hasta la cuenca de Maracaibo.
Los resultados indican que el grupo de la Amazonía Oriental ha estado aislado del grupo Alto Amazonas por un largo período, ya que los resultados los muestra genéticamente distantes. El territorio que ocupa, abarca la cuenca del río Madeira con sus tributarios. Los ríos Mamoré y Beni son el área en donde se encuentra B. insignis y el río Acre afluente del río Purus, es el territorio de las poblaciones aquí llamadas Acre. Incluye además la cuenca inferior del Amazonas propiamente dicho, en donde se cultiva la raza Pará, genéticamente muy cercana a la raza Tembe de Bolivia (Mora-Urpí 1993, Rojas et al. 1999 Fig. 3A). La lejanía genética entre los grupos del Alto Amazonas y Amazonía Oriental induce a preguntarse cual pudo ser la barrera que los ha mantenido aislados uno de otro por millones de años. No se ve una respuesta clara y solo se puede aventurar una conjetura, y esta es, que ambas regiones han pertenecido a cuencas de drenaje separadas, ya que el Alto Amazonas drenó hacia el Océano Pacífico a partir de un punto cercano a Manaus, y el Madeira debe haber drenado en esa época en la intrusión del Océano Atlántico de lo que es hoy el sector inferior del Amazonas (Bigarella y Ferreira 1985).
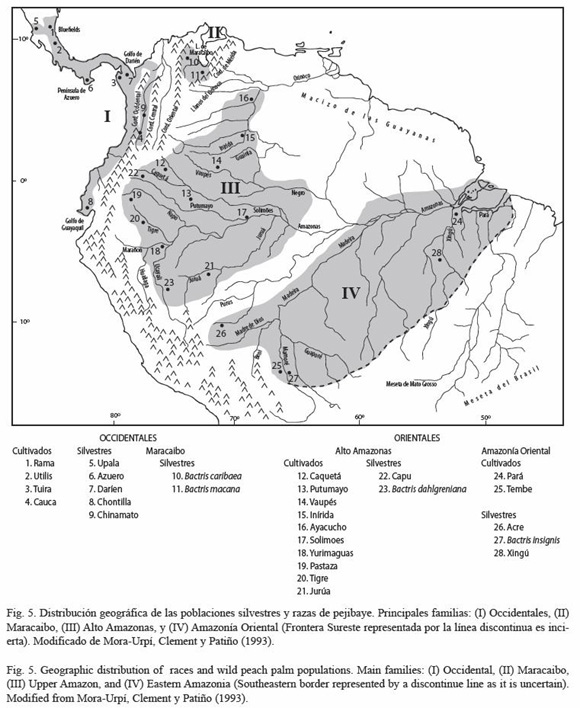
Los otros ríos afluentes del Amazonas situados al oriente del Madeira posiblemente posean poblaciones silvestres genéticamente relacionadas dentro del grupo Amazonía Oriental. En este caso, el pejibaye silvestre descrito por Silva y Clement (2005) en las riveras del río Xingú, podría tener afinidad cercana con este grupo, pero hasta que se realicen los análisis genéticos se podrá confirmar esta hipótesis. En consecuencia, la aparición de esta población agrega incertidumbre sobre los límites de la frontera Sureste del área de distribución del grupo Amazonía Oriental (Fig. 5).
Las poblaciones de pejibaye silvestre no pudieron ingresar a Centroamérica sino hasta finales del Plioceno (hace 3.1 millones de años). Durante el Plioceno tardío, época anterior al Pleistoceno existían dos corredores oceánicos que comunicaban el océano Pacífico y el Mar Caribe. Uno de ellos era el corredor del canal de Panamá. El otro era el corredor de Atrato que comunica las cuencas de los ríos Chucunaque y Atrato, Panamá. Estos corredores se cerraron hace 3.1 millones de años (Duque-Caro 1990), permitiendo entonces el ingreso del pejibaye en Centroamérica.
Las poblaciones de pejibayes silvestres se aislaron durante las glaciaciones que tuvieron lugar en el pleistoceno (entre 2.5 millones de años y 10 000 años). Durante dos millones de años, se alternaron en el neotrópico condiciones frías y secas (presencia de glaciares en las altas montañas) con otras húmedas y menos frías (Colinvaux 1997). Según los datos geológicos hubo 4 glaciaciones bien marcadas y la última empezó hace 22 mil años y finalizó hace 11 mil años (Hernández-Camacho et al. 1992). Se estima que durante la última glaciación se produjo una reducción del 40 % de las precipitaciones promedio en esta zona (Hooghiemstra y van der Hammen 1998). En este periodo las poblaciones de pejibaye silvestre solo pudieron sobrevivir en los refugios húmedos propuestos por varios autores (Haffer 1969, AbSáber 1977, Prance 1985).
Las glaciaciones, posiblemente no afectaron gravemente las poblaciones de pejibaye silvestre en Panamá. Según el registro fósil de polen y paleolimnológico en los lagos, el Valle y la Yeguada, indican que hubo suficiente precipitaciones sobre la vertiente Pacífica de Panamá (Bush y Colinvaux 1990). Por lo tanto, el pejibaye silvestre Azuero que hoy día se encuentra en la península del mismo nombre en Panamá, debe ser una colonización antigua.
De igual manera, el pejibaye silvestre pudo haber migrado hasta Costa Rica y la costa Caribe de Nicaragua durante el Pleistoceno. Algunos autores creen que la cobertura boscosa de Centroamérica no se vio mayormente interrumpida durante las sequías glaciares del Pleistoceno (Bush et al. 1992). Sin embargo su presencia natural en Centro América sólo había sido reportada por Conzemius (1932) en la costa Caribe de Nicaragua, y numerosas referencias orales de su presencia en Costa Rica, tanto en la vertiente del Pacífico como del Caribe, no han sido verificadas, excepto por una población de pejibaye silvestre en la región de Upala, situada en el norte del país al sur del lago de Nicaragua (Mora-Urpí y Arroyo 1996).
Cabe tener presente que no todas las poblaciones silvestres han sido descubiertas, y tampoco se conoce a ciencia cierta la cantidad de razas presentes en los cuatro grupos (Occidentales, Alto Amazonas, Amazonía Oriental y Maracaibo). Pero es muy posible que aquellas que se describan en el futuro, dentro cada uno de esos territorios, muestren afinidad genética con el grupo correspondiente.
En cuanto a las razas ó variedades primitivas resulta claro que responden a patrones genéticos correspondientes a poblaciones silvestres particulares de la región en que están ubicadas. Por lo tanto, se puede decir con un considerable grado de certeza que obedecen a procesos independientes de domesticación dentro de cada grupo. Esto queda ilustrado cuando se observa que las poblaciones silvestres de B. insignis y las plantaciones de la raza Tembe en Bolivia son morfológicamente casi idénticas, indicando que esta raza no ha sido muy mejorada por selección humana. Una situación semejante se observa en Ecuador con los pejibayes cultivados y silvestres de Chontilla, y así como también sucede lo mismo con Azuero en Panamá. En el caso de Chontilla, Couvreur et al. (2005) reportó una estrecha proximidad genética entre las poblaciones cultivadas y silvestres. Sin embargo, los resultados fueron interpretados como múltiples introgressiones entre poblaciones silvestres nativas y cultivadas introducidas (originarias de la Amazonía para Couvreur et al. 2005), sin tomar en cuenta que dichas poblaciones cultivadas eran nativas y existían mucho antes que las primeras introducciones de material genético cultivado de la Amazonía (J. Mora-Urpí, obs. pers.).
Domesticación del cultivo: el origen del pejibaye cultivado es el resultado de la domesticación independiente de distintas poblaciones silvestres del trópico húmedo americano (Mora-Urpí 1993). Las relaciones genéticas que se observan en este trabajo muestran múltiples orígenes de las poblaciones cultivadas de pejibaye. Las razones para interpretar los resultados en ese sentido resultan obvias al observar las relaciones de parentesco. Entre los puntos sobresalientes se señalan los siguientes: 1. las poblaciones silvestres no se agrupan juntas; 2. cada población silvestre lo hace separadamente con un grupo de razas definidas geográficamente, así la población silvestre de Acre y las razas de Tembe y Pará forman el grupo del Amazonía Oriental; así también lo hace B. dahlgreniana sensu Glassman (al cual habría que agregar Capu) que se asocia con las razas del grupo del Alto Amazonas (Putumayo, Vaupés, Solimões y Puerto Ayacucho); Chontilla y Azuero (a los cuales habría que agregar Chinamato, Darién y Upala) se asocian con Tuira, Cauca y Utilis (Guatuso y Tucurrique) para constituir el grupo Occidental; 3. y por último el grupo Maracaibo (B. caribaea y B. macana), que muestra una relación distante con las razas incluidas en este trabajo, y requiere de un estudio de las poblaciones silvestres y cultivadas de la cuenca del Magdalena para establecer si existe relación con ellas. En general estos resultados contradicen la propuesta de que existió un único centro de domesticación para el pejibaye.
Rodrigues et al. (2004) deducen que el cultivo de pejibaye tuvo su origen en el suroeste del Amazonas, con dos rutas de migración: una hacia el oriente y otra hacia el oeste hasta América Central. Las razones para creer diferente son las siguientes: 1. la raza cultivada de Pará se agrupa con la población silvestre de Acre (sin. var chichagui-acre sensu Henderson 2000), aparte y distante de todas las demás poblaciones de pejibaye(Fig. 3b ); 2. la población silvestre de Benjamín Constant (var chichagui-benjamín constant, sensu Henderson 2000) aparece en el agrupamiento como probable ancestro de las razas cultivadas del Alto Amazonas (Putumayo, Solimões y Pampa Hermosa sin.Yurimaguas), lo cual es correcto; 3. las poblaciones silvestres Benjamín Constant y Acre se sitúan en grupos separados, indicando que son bastante diferentes; y 4. las poblaciones cultivadas Amazónicas y Occidentales tampoco se agrupan juntas, como se esperaría de un único origen de las poblaciones cultivadas.
En resumen, aquí se propone que el origen del pejibaye cultivado es el resultado de la domesticación independiente de varias poblaciones silvestres nativas del trópico húmedo americano (Mora-Urpí 1993). Su área de distribución abarca desde Bolivia hasta Nicaragua con al menos tres centros de domesticación. Según los datos obtenidos, el centro de origen de las poblaciones cultivadas habita de manera independiente en tres regiones: Occidente, Alto Amazonas y Amazonía Oriental (Fig. 5).
Agradecimientos
Agradecemos a Carlos Arroyo, Antonio Bogantes, Rigoberto Pizarro y Carlos Garita, de la Estación Experimental los Diamantes (Guápiles, Costa Rica) por su colaboración durante el trabajo de campo. A Gabriel Aguilar, Luis Castro, Julieta Fernández y Gloriana Álvarez por suministrar ayuda en el trabajo de laboratorio. A C.R. Clement por la revisión del manuscrito y sus valiosas sugerencias. Al Centro Agronómico Tropical de Investigación y Enseñanza (CATIE), por brindarnos algunas accesiones de su Banco de Germoplasma. Este trabajo fue financiado por la vicerrectoría de Investigación de la Universidad de Costa Rica y fue posible gracias a donaciones de equipo de la Fundación Internacional para las Ciencias (IFS), la Fundación CR-USA y el Instituto Internacional de Recursos Genéticos de Plantas (IPGRI).
Resumen
Se evaluó la diversidad genética en cuatro microsatélites de ADN de pejibaye (Bactris gasipaes Kunth) para relacionarlos con su evolución y domesticación. Se analizaron 258 muestras procedentes de siete poblaciones silvestres y once razas cultivadas. Todos los loci eran polimórficos y se identificaron 50 alelos en total. La diversidad genética fue alta (0.67). Todas las poblaciones reunidas obtuvieron una alta diferenciación genética (Fst=0.16), pero cuando se separaron en poblaciones occidentales y orientales fue menor (Fst=0.13 para ambas). El flujo genético presente en las poblaciones occidentales fue mayor (Nm=1.71) que en las orientales (Nm=1.62). Por otra parte, se encontró que las razas de Putumayo, Yurimaguas, Vaupés, Tucurrique, y Guatuso aparentemente han sido sometida a una intensa selección humana. Además, la existencia de poblaciones híbridas es el resultado del intercambio entre pueblos del neotrópico e introgresiones con poblaciones silvestres y cultivadas. Se estimó la distancia genética Dm para generar un dendograma por el método del vecino más cercano. Definimos tres grupos de poblaciones: Maracaibo (B. caribaea, B. macana var veragua y B. macana var arapuey), Amazonía Oriental (Tembe, Pará y Acre) y el grupo compuesto por dos subgrupos, Occidental (Azuero, Chontilla, Tuira, Cauca, Tucurrique y Guatuso) y Alto Amazonas (B. dahlgreniana, Puerto Ayacucho, Solimões, Vaupés y Putumayo). La relación genética coincide con la hipótesis de que la palmera del pejibaye ha sido domesticada independientemente por lo menos en tres regiones.
Palabras clave: Bactris gasipaes, pejibaye, diversidad genética, relaciones filogenéticas, domesticación cultivo, microsatélites, razas, poblaciones silvestres.
Received 18-IX-2006. Corrected 07-V-2007. Accepted 27-VII-2007.
Referencias
AbSáber, A.N. 1977. Espacos ocupados pela expansão dos climas secos na América do Sul, por ocasito dos períodes glaciais cuaternarios. Paleoclimas 3: 1-19. [ Links ]
Adin, S., J.C. Weber, C. Sotelo Montes, H. Vidaurre, B. Vosman & M.J.M. Smulders. 2004. Genetic differentiation and trade among populations of peach palm (B. gasipaes Kunth) in the Peruvian Amazon–implications for genetic resource management. Theor. Appl. Genet. 108: 1564-1573. [ Links ]
Bigarella, J.J. & A.M.M. Ferreira. 1985. Amazonian Geology and the Pleistocene and the Cenozoic Environments and Paleoclimates, p. 49-71. In AMAZONIA. Pergamon, Oxford, Inglaterra. [ Links ]
Billote, N., A.M. Risterucci, E. Barcelos, J.L. Noyer, P. Amblard & F.C. Baurens. 2001. Development, characterisation, and across-taxa utility of oil palm (Elaeis guineensis Jacq.) microsatellite markers. Genome 44: 413-425. [ Links ]
Bush, M.B. & P.A. Colinvaux.1990. A pollen record of a complete glacial cycle from lowland Panama. J. Veg. Sci. 1: 105-118. [ Links ]
Bush, M.B., D.R. Piperno, P.A. Colinvaux, L. Krissek, P.E. de Oliveira, M.C. Miller & W.E. Rowe. 1992. A 14 300-yr paleoecological profile of a lowland tropical lake in Panamá. Ecol. Monogr. 62: 251-276. [ Links ]
Clement, C.R. & J. Mora-Urpí. 1982. The pejibaye palm (Bactris gasipaes H:B.K.) come of age. Principes 26: 150-152. [ Links ]
Clement, C.R. 1986. Descriptores mínimos para el pejibaye (Bactris gasipaes H.B.K.) y sus implicaciones filo-
genéticas. Tesis de Maestría, Universidad de Costa Rica, San José, Costa Rica.
Clement, C.R., J. Aguiar, D.B. Arkcoll, J. Firmino & R. Leandro. 1989. Pupunha brava (Bactris dahlgreniana Glassman): progenitora da pupunha (Bactris gasipaes H.B.K.)?. Bol. Mus. Para Emilio Goeldi, Bot. 5: 39-55. [ Links ]
Clement, C.R., N.R. Sousa, D.P. Rodrigues, S. Astolfi-Filho, Y. Núñez, V. Torres & F.Gallego. Use of AFLPs to distinguish landraces of pejibaye (Bactris gasipaes) in Brazilian Amazonía. Sci. Agric. 59: 749-753. [ Links ]
Cole, D., T. White, P.K.R. Nair. 2007. Maintaining genetic resources of peach palm (Bactris gasipaes Kunth): The role of seed migration and swidden-fallow management in northeastern Peru. Genet. Resour. Crop. Ev. 54: 189–204. [ Links ]
Colinvaux, P.A. 1997. The ice-age Amazon and the problem of diversity. NWO Huygens Lecture, The Netherlands Organization for Scientific Research, La Haya, Holanda. [ Links ]
Couvreur, T.L.P, N. Billote, A.M. Risterucci, C. Lara, Y. Vigouroux, B. Ludeña, J.L. Pham & J.C. Pintaud. 2005. Close genetic proximity between cultivated and wild Bactris gasipaes Kunth revealed by microsatellite markers in Western Ecuador. Gen. Res. Crop. Evol. 00: 1-13. [ Links ]
Conzemius, E. 1932. Ethnographic survey of the Miskito and Sumo Indians of Honduras and Nicaragua. Smithsonian Institution, Bureau of American Ethnology, Bulletin 106, Washington D.C., EEUU. [ Links ]
Corrales, F. & J. Mora-Urpí. 1990. Sobre el proto-pejibaye en Costa Rica. Serie Técnica Pejibaye (Guilielma), Boletín Informativo, Universidad de Costa Rica, San José, Costa Rica 2: 1-11. [ Links ]
Díaz de Gamero, M. 1996. The changing course of the Orinoco River during the Neogene: a review. Palaeogeogr. Palaeocl. 123: 385-402. [ Links ]
Duque-Caro, H. 1990. Neogene stratigraphy, paleoceanography, and paleobiology in northwest South America and the evolution of the Panamá Seaway. Palaeogeogr. Palaeocl. 77: 203-234. [ Links ]
Estoup, A. & J. Cornuet. 1999. Microsatellite evolution: inferences from population data, p. 49-65. In D. Goldstein & C. Schlötterer (eds). Microsatellites Evolution and Applications, Oxford, Inglaterra. [ Links ]
Ferreira, E. 1999. The Phylogeny of Pupunha (Bactris gasipaes Kunth, Palmae) and Allied Species, p. 225-236. In A. Henderson & F. Borchsenius (eds.). Evolution, variation, and Classification of Palms. Memoirs of The New York Botanical Garden. The New York Botanical Garden, Bronx, Nueva York, EEUU. [ Links ]
Ferrero, L. 1981. Costa Rica Precolombina. Costa Rica, San José, Costa Rica. [ Links ]
Frankham, R., J.D. Ballou & D.A. Briscou. 2002. Introduction to Conservation Genetics. Cambridge, Reino Unido. [ Links ]
Font Quer, P. 1963. Diccionario de Botánica. Labor, Barcelona, España. [ Links ]
Goldstein, D.B. & D.D. Pollock. 1997. Lauching microsatellites: A review of mutation processes and methods of phylogenetic inference. J. Hered. 88: 335-342. [ Links ]
Guo, S.W. & E.A. Thompson. 1992. Performing the exact test of Hardy-Weinberg proportion for multiple alleles. Biometrics 48: 361-372. [ Links ]
Haffer, J. 1969. Speciation in Amazonían forest birds. Science 165: 131-137. [ Links ]
Henderson, A. 2000. Bactris (Palmae). Flora Neotrop. Monog. 79: 1-181. [ Links ]
Hernández-Camacho, J., T. Walschburger, R. Ortiz & A. Hurtado. 1992. Origen y Distribución de la biota Suramericana y Colombiana, p. 3-24. In G. Halffter (ed.). La Diversidad Biológica de Iberoamérica. Acta Zool. Mex. (n.s.). Volumen especial.CYTED-D, Programa Iberoamericano de Ciencia y Tecnología para el Desarrollo, México D.F., México. [ Links ]
Hooghiemstra, H. & T. van der Hammen. 1998. Neogene and Quarternary development of the neotropical rain forest: the forest refugia hypothesis, and a literature overview. Earth Sci. Rev. 44: 147-183. [ Links ]
Hoorn, C., J. Guerrero, G.A. Sarmiento & M.A. Lorente. 1995. Andean tectonics as a cause for changing drainage patterns in Miocene northern South America. Geology 23: 237-240. [ Links ]
Huber, J. 1904. A origem da pupunha. Bol. Mus. Para Emilio Goeldi, Bot. 4: 474-475. [ Links ]
Lodhi, M.A., G.N. Ye, N.F. Weeden & B.I. Reisch. 1994. A simple and efficient method for DNA extraction from grapevine cultivars and Vitis species. Plant. Mol. Biol. Rep. 12: 6-13. [ Links ]
Martínez, A.K., E. Gaitán-Solís, M.C. Duque, R. Bernal & J. Thome. 2002. Primer Note: Microsatellite loci in Bactris gasipaes (Arecaceae): their isolation and characterization. Mol. Ecol. Notes 2: 408-410. [ Links ]
Mattos, L. 1992. Diferenciación Taxonómica de Diez Razas de Pejibaye Cultivado (Bactris (Guilielma) Gasipaes Kunth) y su relación con otras Especies de Bactris. Magister Scientiae, Universidad de Costa Rica, San José, Costa Rica. [ Links ]
Mora-Urpí, J. 1992. Pejibaye (Bactris gasipaes), p. 209-219. In J.E. Hernández & J. León (eds.). Cultivos marginados otra perspective de 1492. Producción y Protección vegetal. Organización de las Naciones Unidas para la Agricultura y la Alimentación, Colección FAO, Roma, Italia. [ Links ]
Mora-Urpí, J. 1993. Diversidad Genética en Pejibaye: II. Origen y Evolución, p. 21-29. In J. Mora-Urpí, L.T. Szott, M. Murillo & V.M. Patiño (eds.). IV Congreso Internacional sobre Biología, Agronomía e Industrialización del Pijuayo, Iquitos, Perú. Universidad de Costa Rica, San José, Costa Rica. [ Links ]
Mora-Urpí, J. 1999. Origen y Domesticación, p. 17-24. In J. Mora-Urpí & E. Gainza (eds.). Palmito de pejibaye (Bactris gasipaes Kunth): su cultivo e industrialización. Universidad de Costa Rica, San José, Costa Rica. [ Links ]
Mora-Urpí, J. & C. Arroyo. 1996. Sobre origen y diversidad en pejibaye. Serie Técnica Pejibaye (Guilielma), Boletín Informativo, Universidad de Costa Rica, San José, Costa Rica 5: 18-25. [ Links ]
Mora-Urpí, J., C. Clement & V. Patiño. 1993. Diversidad Genética en Pejibaye: I. Razas e Híbridos, p. 11-20. In J. Mora-Urpí, L.T. Szott, M. Murillo & v.M. Patiño (eds.). IV Congreso Internacional sobre Biología, Agronomía e Industrialización del Pijuayo, Iquitos, Perú. Universidad de Costa Rica, San José, Costa Rica. [ Links ]
Mora-Urpí, J. & E. Solís. 1980. Polinización en Bactris gasipaes H.B.K. Palmae. Rev. Biol. Trop. 28: 153-174. [ Links ]
Mora-Urpí, J., J. Weber & C.R. Clement. 1997. Peach Palm Bactris gasipaes Kunth. Promoting the conservation and use of underutilized and neglected crops. 20. Institute of Plant Genetics and Crop Plant Research-IPK, Gatersleben/International Plant Genetic Resources Institute-IPGRI, Roma, Italia. [ Links ]
Nei, M. 1973. The theory and estimation of genetic distance, p. 45-54. In N.E. Morton (ed.). Genetic Structure of Population. Hawai, Honolulu, EEUU. [ Links ]
Page, R.D.M. 1996. Treeview: An application to display phylogenetic trees on personal computers. Comput. Appl. Biosci. 12: 357-358. [ Links ]
Patiño, V.M. 1963. Plantas cultivadas y animales domésticos en América Equinoccial. Tomo I Frutales. Imprenta Departamental, Cali, Colombia. [ Links ]
Patiño, V.M. 1989. Comportamiento de las plantas nativas colombianas bajo cultivo: situación actual del cultivo chontaduro. Rev. Acad. Colomb. Cienc. 17: 259-264. [ Links ]
Pellizzaro, S.M. 1978. La celebración de Uwi. Publicación de los Museos del Banco Central de Ecuador, Quito, Ecuador. [ Links ]
Perera, L., J.R. Russell, J. Provan & W. Powell. 2000. Use of microsatellite DNA markers to investigate the level of genetic diversity and population genetic structure of coconut (Cocos nucifera L.). Genome 43: 15-21. [ Links ]
Prance, G.T. 1982. The pejibaye, Guilielma gasipaes (H.B.K.) Bailey and papaya, Carica papaya L. In D. Stone (ed.). Pre-Columbian plant migration. Paper Peabody Mus. Arch. Etno 76: 87-104. [ Links ]
Prance, G.T. 1985. The Changing Forests, p. 146-165. In AMAZONIA. Pergamon, Oxford, Inglaterra. [ Links ]
Rodrigues, D.P., C.R. Clement & S. Astolfi-filho. 2001. Analise das morfo-raças primitivas de Pupunha (Bactris gasipaes Kunth) mantidas no banco ativo de germoplasma de Pupunha com Marcadores Moleculares RAPIDs. Dissertaçäo de Maestrado, Dept. de Biología Celular, Univ. Brasilia, Brasil. [ Links ]
Rodrigues, D.P., S. Astolfi-filho & C.R. Clement. 2004. Molecular marker-mediated validation of morphologically defined landraces of Pejibaye (Bactris gasipaes) and their phylogenetic relationships. Genet. Resour. Crop. Ev. 51: 871–882. [ Links ]
Rojas, S., P. Ramírez & J. Mora-Urpí. 1999. Polimorfismo isoenzimático en cuatro razas y un híbrido de Bactris gasipaes (Palmaea). Rev. Biol. Trop. 47: 755-761. [ Links ]
Saitou, N. & M. Nei. 1987. The neighbor-joining method: A new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 14: 406-425. [ Links ]
Silva, J.B. & C.R. Clement. 2005. Wild pejibaye (Bactris gasipaes Kunth var. chichagui) in Southeastern Amazonía. Acta. Bot. Bras. 19: 281-284. [ Links ]
Sousa, N.R., D.P. Rodrigues, C.R. Clement, E.O. Nagao & S. Astolfi-Filho. 2001. Discriminação de raças primitivas de pupunha (Bactris gasipaes) na Amazônia brasileira por meio de marcadores moleculares (RAPDS). Acta Amazonica 31: 539-545. [ Links ]
Sun, G., G. Wang-Pruski, M. Mayich & H. Jong. 2003. RAPD and pedigree-based genetic diversity estimates in cultivated diploid potato hybrids. Theor. Appl. Genet.107: 110-115. [ Links ]
Vavilov, N.I. 1951. The origin, variation, immunity and breeding of cultivated plants. The Ronald, Nueva York, EEUU. [ Links ]
Weir, B.S. & C.C. Cockerman.1984. Estimating F-statistics for the analysis of population structure. Evolution 38: 1358-1370. [ Links ]
Yeh, F.C., R. Yang & T. Boyle. 1999. POPGENE, version 1.32. Microsoft Window-Based Freeware for Population Genetic Analysis. University of Alberta, Edmonton, Canada. [ Links ]
Zúñiga, A. 2004. Geografía y Ecología de los pejibayes silvestres de la cuenca del lago de Maracaibo-Venezuela. Memoria de la Práctica dirigida para optar por el título de Licenciatura en Geografía, Universidad de Costa Rica, San José, Costa Rica. [ Links ]
Referencias de Internet
Langella, O. 2002. Population 1.2.28. Logiciel de génétique des populations. Laboratoire Populations, génétique et évolution, CNRS UPR 9034, Gif-sur-Yvette. (Descargada: 1 noviembre del 2004, http://www.cnrs-gif.fr/pge/). [ Links ]
Miller, M. 1997.Tools for population genetic analysis (TFPGA) 1.3: A windows program for analysis of allozyme and molecular population genetic data. (Descargada: 10 noviembre del 2004, http://herb.bio.nau.edu/~miller/tfpga.htm). [ Links ]














