Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista de Biología Tropical
On-line version ISSN 0034-7744Print version ISSN 0034-7744
Rev. biol. trop vol.55 n.3-4 San José Sep./Dec. 2007
Caracterización de la diversidad genética en el pez Brycon henni (Characiformes: Characidae) en Colombia central por medio de marcadores RAPD
Hermes Pineda Santis1,2, Lucy Arboleda Chacón2, Amparo Echeverry Echavarria2, Silvio Urcuqui Inchima3, Diego Pareja Molina1, Martha Olivera Ángel1 & Juan Builes Gómez4
1 Grupo de Fisiología y Biotecnología de la Reproducción, Facultad de Ciencias Agrarias, Universidad de Antioquia, AA 1226, Medellín, Colombia; herpis@gmail.com
2 Grupo de Investigación en Acuicultura, Facultad de Ciencias Agrarias, Politécnico Colombiano Jaime Isaza Cadavid, Of. 124, Medellín, Colombia; larboleda@elpoli.edu.co
3 Inmunovirología, Sede de Investigación Universitaria, Universidad de Antioquia, AA 1226, Medellín, Colombia; surcuqui@virologia.udea.edu.co
4 Laboratorio Genes Ltda., CC Monterrey Oficina 612, Medellín, Colombia; genforense@epm.net.co
Abstract: Characterization of the genetic diversity of the fish Brycon henni (Characiformes: Characidae) in central Colombia with RAPD markers. Knowledge on the genetic diversity of wild fish species is essential for conservation and appropriate management of individuals in repopulation programs. In Colombia, Brycon henni has been reported in the Magdalena and Cauca river basins, but the population and range have diminished as a consequence of anthropic activities. In this study, the Random Amplified Polymorphic DNA (RAPD) was used to estimate the actual genetic structure in this species. For the purpose, six sample sites located in the department of Antioquia (Central Chain Mountains of Colombia) were used. Thirty five primers (87.5 %), out of forty used, yielded 1 466 reliable and consistent fragments; 417 were considered as unique fragments able to discriminate among the Magdalena (Humarada-1 and Humarada-2) and Cauca (Piedras, La Clara y Guaracú) river basins samples, suggesting that each is a discrete unit. This diversity suggests that anthropic effects of over fishing, dam building, deforestation and water pollution, have contributed to the isolation of these fish groups on the high mountains. Brycon moorei and Colossoma macropomum, as an interspecific control groups, were placed out of the B. henni general group, confirming their taxonomic classification through morphologic data. The RAPD technique was useful to know the genetic diversity and to discriminate among B. henni populations from different geographic origins, as a basis for an appropriate plan of repopulation, conservation and wildlife management. Rev. Biol. Trop. 55 (3-4): 1025-1035. Epub 2007 December, 28.
Key words: Conservation, fish, genetic diversity, RAPD, restoration, Brycon henni.
La identificación y caracterización genética de grupos de peces localmente adaptados y diferenciados, es fundamental para preservar la variación genotípica en poblaciones naturales, así como para detectar, en algunos casos oculta, la diferenciación adaptativa en respuesta a la heterogeneidad ambiental a pequeña escala (Carvalho y Hauser 1995). Colombia es considerado como uno de los países megadiversos, porque en una extensión menor al 1 % de la superficie continental mundial, contiene el 10 % de la diversidad biológica del planeta, incluyendo una alta variedad de especies de peces marinos y continentales (Beltrán Turriago y Villaneda-Jiménez 2000, Instituto Alexander von Humboldt 2000). Durante la practica de la pesca tradicional en los ríos colombianos, un gran número de especies de peces son capturados indiscriminadamente sin tener en cuenta el impacto genético en las poblaciones; lo que ha conllevado a una fuerte disminución en un gran número de animales de especies tales como: Prochilodus magdalenae, Pseudoplatystoma fasciatum, Pseudoplatystoma tigrinum, Brycon moorei, de las principales cuencas hidrográficas (INPA 2001). Para contrarrestar esta erosión íctica, el gobierno nacional planea recuperar algunas de las zonas de pesca mas devastadas mediante la repoblación. Si bien se conocen aspectos relevantes sobre la reproducción, alimentación y cultivo de algunas de estas especies (Beltrán-Turriago y Villaneda-Jimenez 2000), poco o nada se conoce sobre su composición genética, lo cual es indispensable para una adecuada intervención en cuencas o microcuencas.
Brycon henni (Eigenmann 1913), orden Characiformes, familia Characidae, subfamilia Bryconinae, comúnmente conocida como sabaleta, es una de las especies migratorias más importantes de los pequeños ríos que tienen su origen en la cordillera Central de Colombia y atraviesan las zonas cafeteras del país. Su distribución geográfica cubre los pequeños y grandes ríos que descienden hacia los valles que forma el río Magdalena, por la margen derecha hacia el centro-oriente del territorio nacional, y el río Cauca, por la margen izquierda hacia el centro-occidente del país (Dahl 1971, Mojica 1999), donde se localizan los departamentos de Antioquia, Cauca, Caldas y Valle. El buen gusto y calidad de su carne, así como su comportamiento de intensa lucha ante la captura con anzuelo en la pesca deportiva, la han convertido en una de las especies mas apetecidas de dicha región. Sin embargo, actualmente B. henni, entre otras especies de este género, se encuentra en riesgo de extinción ya que esta sometida a una considerable presión, debido a la sobre pesca, construcción de represas que obstruyen los canales, contaminación de las fuentes de agua y deforestación de bosques (Ceccarelli y Senhorini 1996). Por tal razón, esta especie es considerada como un buen prototipo para iniciar un programa de repoblación de pequeños ríos aledaños a las zonas cafeteras del departamento de Antioquia (Colombia), en donde hace muchos años era su hábitat natural. Sin embargo, se debe tener en cuenta que se desconoce la diversidad genética de esta especie, así como el impacto ecológico de la actividad humana, aunque se esta profundizando en otros aspectos tales como; reproducción, caracterización seminal, nutrición y cultivo.
La técnica de Polimorfismo de ADN Amplificados al Azar (RAPD), es una herramienta útil para determinar la diversidad genética entre poblaciones (Koh et al. 1999, Barman et al. 2002, Barfai et al. 2003). Esta técnica consiste en la amplificación de pequeños fragmentos de ADN por la Reacción en Cadena de la Polimerasa (PCR), distribuidos a lo largo del genoma, utilizando un único iniciador de secuencia arbitraria (Welsh y McClelland 1990, Williams et al. 1990). Es una técnica que se utiliza con mucha frecuencia porque ofrece muchas ventajas: no se necesita tener un conocimiento previo de las secuencias en el genoma a analizar, el iniciador utilizado se puede hibridar al azar en cualquier región del cromosoma, permite asegura una buena interpretación de la diversidad genética al usar un número alto de iniciadores, y finalmente porque permite obtener un gran número de fragmentos con poca cantidad de ADN, además de ser un método simple que no necesita equipos sofisticados (Palumbi 1996, Bardakci 2001). El interés de este estudio fue determinar, la diversidad genética en B. henni,a través de los fragmentos amplificados de ADN dominantes, obtenidos mediante la técnica de RAPD en diferentes hábitats naturales (ríos) del departamento de Antioquia.
Materiales y métodos
Se utilizaron diez animales de B. henni de cada uno de los seis sitios de muestreo; como grupos de control inter específico se utilizaron diez animales de B. moorei (río Magdalena) y diez de Colossoma macropomum (río Meta), los cuales se identificaron por sus características morfológicas, siguiendo la taxonomía descrita por Dahl (1971) y Gery (1977). Especimenes testigos de B. henni se encuentran en el Instituto Alexander von Humboldt, Claustro de San Agustín, Villa de Leyva, Boyacá, Colombia (Referencia: IavHP 4304 y IavHP 4355). La relación de especies y las coordenadas geográficas de los sitios de muestreo se describen en el Cuadro 1.

El ADN genómico se extrajo siguiendo el protocolo descrito por Miller et al. 1988, con las siguientes modificaciones: de cada animal se tomó 1 ml de sangre en tubos que contenían 5 ml de tampón de lisis TNES (10 mM Tris-HCl pH 8.0, 0.3 M NaCl, 10 mM EDTA pH 8.0, 1 % SDS) (Estoup et al. 1993), de allí posteriormente se tomó 30 µl de la mezcla y se adicionó 500 µl de tampón TNES y 20 µl de proteinasa K (20 mg/ml). La solución se incubó en baño maría por 24 horas a 56 °C; se adicionó 100 µl de 5 M NaCl, seguido de vortex por 20 s y se centrifugó a 14 000 rpm durante 5 min. Por cada volumen del sobrenadante se adicionó nueve volúmenes de isopropanol al 100 %, se mezcló por inversión y se incubó durante dos horas a –20 °C; se centrifugó a 14 000 rpm durante 5 min. El ADN precipitado se lavó con 300 µl de etanol 70 %, se centrífugo nuevamente y una vez eliminado el sobrenadante, se dejó secar a temperatura ambiente. Finalmente, se resuspendió en 70 µl de agua ultra pura y la calidad del ADN se determinó en un gel de agarosa 1 %; la cuantificación se realizó en un espectrofotómetro (Perkin Elmer., USA). Para evaluar la diversidad genética existente en B. henni para cada zona, se tomaron 20 µl de ADN (30 ng/µl) de cada uno de los animales (diez por sitio de muestreo), y hacer una mezcla única de 200 µl que represente el acervo genético total. Se realizó la mezcla con el propósito de agrupar la información por cada zona de muestreo y así poder obtener un fragmento particular nítido que asegure una gran cantidad de fragmentos polimorficos (Michelmore et al. 1991). El mismo procedimiento se siguió para las especies B. moorei y C. macropomum, que sirvieron como grupos externos.
Para la obtención de fragmentos de RAPD se utilizaron los estuches, OPA y OPB (Operon Technologies., USA), que contienen 20 iniciadores cada uno, con diez nucleótidos por iniciador y un contenido de GC igual al 60 % (Cuadro 2). La amplificación por PCR del ADN se realizó en un termociclador PTC 100 (MJ Research., USA), siguiendo el rango de temperatura descrito por Yu y Pauls (1992): un ciclo de 94 °C por 4 min, seguido de 35 ciclos; así: 94 °C por 35 s, 36 °C por 35 s y 72 °C por 1 min. La reacción se realizó en 25 µl que contenía: 2 µl de ADN matriz (60 ng), 10 pmol del iniciador de RAPD especifico, 1 unidad de Taq ADN polimerasa (Promega., USA), 0.2 mM de cada dNTP´s (Promega., USA), 2 mM de MgCl (Promega., USA) y 2.5 µl de tampón Taq 10X. Un control negativo, con todos los reactivos excepto el ADN matriz, fue utilizado en todas las amplificaciones. Las condiciones de reacción se establecieron luego de 16 repeticiones, que corresponde al 5 % del total de reacciones (320), con resultados reproducibles, es decir, siempre se obtuvo el mismo patrón de fragmentos; para ello se tuvo en cuenta la calidad del ADN, concentración del iniciador, temperatura de hibridación y el número de fragmentos de ADN visibles en geles de poliacrilamida. Los productos de PCR amplificados con el mismo iniciador para cada uno de los sitios de muestreo, se colocaron a migrar en un gel de poliacrilamida al 4 %. El tamaño de cada uno de los fragmentos de ADN obtenido, se determinó con un marcador de peso molecular de ADN; pGEM DNA (Promega., USA) (Sambrook et al. 1989), la visualización de los fragmentos obtenidos en la PCR se hizo mediante tinción con plata (Promega., USA), siguiendo el protocolo descrito por Dinesh et al. 1993 (Fig. 1). Solo se consideró para el análisis los fragmentos intensos y reproducibles entre 222 y 2 645 pb, obtenidos con el mismo iniciador.
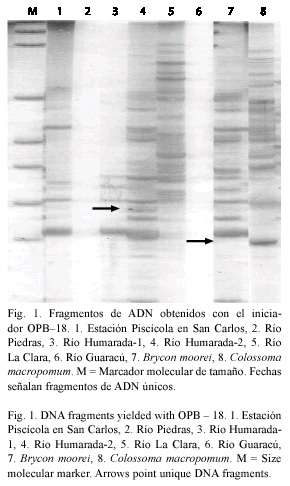
Para la interpretación de los datos, se asignó con el número uno (presencia) ó cero (ausencia) del fragmento, comparado con el resultado obtenido para los animales de los otros sitios de muestreo corridos en el mismo gel, con estos resultados se realizó una matriz de comparación para calcular la Distancia Genética (DG). De los cuarenta iniciadores de RAPD, los que no produjeron fragmentos contundentes fueron OPB-02, OPB-03, OPB-09, OPB-16 y OPB-19.
Los valores de DG se calcularon mediante la fórmula de Nei y Li 1979:
DGij = 1- [2nij /(ni + nj)]
Donde nij es el número total de fragmentos compartidos por dos grupos, ni y nj son el número total de fragmentos en los grupos i y j, respectivamente. El nodo del árbol (Efron 1979, Felsenstein 1985) del dendrograma Neighbor Joining (NJ) (Saitou y Nei 1987), fue obtenido mediante 10 000 réplicas realizado con el programa de computador PAUP* 4.0 (Swofford 1996).
Resultados
Los 35 iniciadores de RAPD (87.5 %), que permitieron obtener un resultado positivo, produjeron 1 466 fragmentos amplificados, reproducibles y consistentes. De éstos 964 correspondieron a B. henni, procedente de los seis sitios de muestreo: Estación Piscícola en San Carlos, río Piedras, río Humarada-1, Humarada-2, río La Clara, río Guaracú; 238 y 264 fragmentos fueron observados en B. moorei (río Magdalena) y C. macropomum (río Meta), respectivamente (Cuadro 3). Estos resultados muestran la existencia de un alto número de fragmentos amplificados para estas especies. Para B. henni, dependiendo del sitio de muestreo, se observó una alta diferencia en el número de fragmentos totales. Así por ejemplo, el mayor número de fragmentos (211) se encontró en el río Humarada-1, y el menor número (19) se encontró en los animales del río Guaracú. Sin embargo, entre los sitios de muestreo (Estación Piscícola en San Carlos, río Piedras, río Humarada-1 y Humarada-2), no hubo mucha diferencia (Cuadro 3). El número promedio de fragmentos por iniciador en B. henni osciló entre 0.5 y 6.0; pero los mayores promedios (6.8 y 7.5) se observaron en B. moorei y C. Macropomum respectivamente, utilizados como grupos de comparación inter específico (controles) (Cuadro 3). Se presentaron 417 fragmentos únicos, de los cuales 153 corresponden a B. henni, y el mayor número (54) corresponde a la muestra procedente de la Estación piscícola en San Carlos y el menor número de fragmentos (2) para la muestra del río Guaracú (Cuadro 3). En B. moorei y C. macropomum, se detectaron 120 y 144 fragmentos únicos respectivamente. El mayor porcentaje de fragmentos únicos (25.8 %) para B. henni se observó en la muestra de la Estación piscícola en San Carlos, y menor (9.5 %) en la muestra de Humarada-1 (Cuadro 3). Los mayores porcentajes se observaron en los grupos de comparación inter específico B. moorei y C. macropomum, 50.4 % y 54.5 % respectivamente (Cuadro 3).
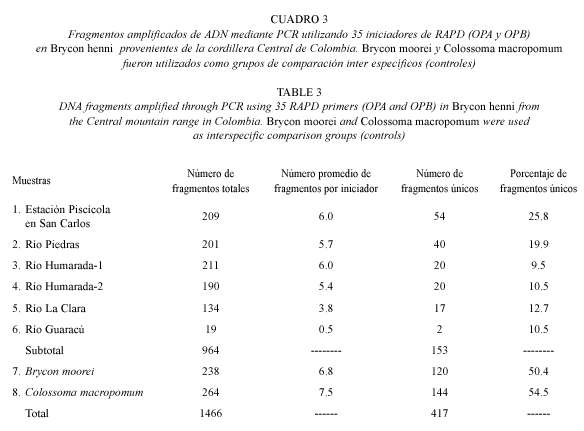
En el estuche OPA todos los iniciadores presentaron fragmentos amplificados de ADN únicos, mientras que en OPB cinco de ellos no produjeron fragmentos (OPB-02, OPB-03, OPB-9, OPB-16 y OPB-19) (Cuadro 4). Las especies B. moorei y C. macropomum presentaron el mayor número de fragmentos únicos, difiriendo de los obtenidos en B. henni, los cuales estuvieron distribuidos al azar (Cuadro 4) .
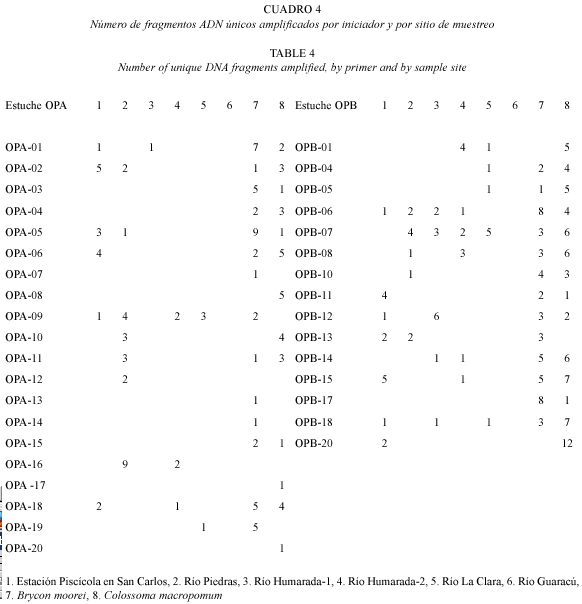
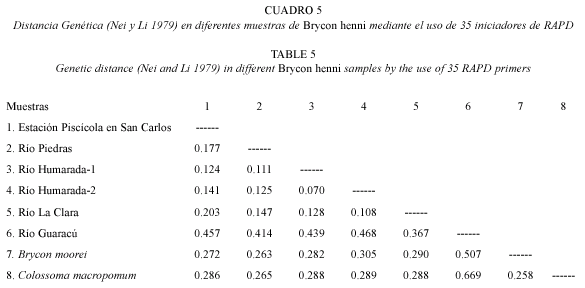
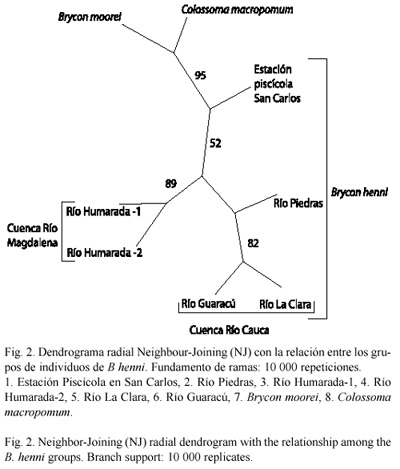
En B. henni, la menor distancia genética observada fue 0.070 (Río Humarada-1 y Río Humarada-2) y la mayor fue 0.468 (río Humarada-2 y río Guaracú) (Cuadro 5). Considerando los controles de comparación inter específicos, la menor distancia observada fue 0.258 (B. moorei y C. macropomum) y la mayor fue 0.669 (río Guaracú y C. macropomum) (Cuadro 5). El dendrograma radial NJ presentó una alta confiabilidad (95), en el nodo del árbol que discriminó entre la especie B. henni y los utilizados como control inter específico (Fig. 2). Igualmente, se discriminó entre animales de diferente origen geográfico, esto es, grupos de animales provenientes de la cuenca del río Magdalena, diferente a los provenientes de la cuenca del río Cauca. Un poco más distante se encontró la Estación piscícola en San Carlos (Fig. 2). Los iniciadores que discriminaron entre las diferentes especies fueron OPA-08, OPA-19, OPB-13 y OPB-20.
Discusión
El número de fragmentos totales obtenido con 35 iniciadores fue importante y constituye un acercamiento de tipo genético para estas especies de la familia Characidae, para la cual se han hecho muy pocos estudios en Colombia. En otras especies de peces, utilizando 60 iniciadores (Guo et al. 2001) ó 40 (Elo et al. 1997, Liu et al. 1998, Kawamura et al. 2001), se describió un menor número de fragmentos. Los iniciadores utilizados en el presente trabajo permitieron discriminar entre grupos de animales de la misma especie, procedente de dos cuencas diferentes (i.e. cuenca del río Magdalena y cuenca del río Cauca), entre especies del mismo género (i.e. B. moorei y B. henni) y entre otras especies (i.e. Brycon spp. y Colossoma spp.) (Fig. 2), sugiriendo un alto poder discriminatorio de la técnica.
La estación piscícola en San Carlos, reúne condiciones relacionadas con la intervención del hombre; posiblemente un proceso de cruzamientos indiscriminados entre animales de procedencia desconocida (J Cardona, com. pers.), que podría explicar el resultado obtenido en este sitio de muestreo. En efecto, esta estación piscícola surgió como proyecto piloto que tenia como objetivo la recuperación íctica de pequeños ríos de la zona oriental del departamento de Antioquia, trasladando animales de B. henni de ríos con mayor abundancia a otros menos abundantes. Desafortunadamente, no se tuvo en cuenta las precauciones necesarias para la correcta identificación y manejo de los especimenes en cautiverio, lo que posiblemente ocasionó un proceso reproductivo no controlado, lo cual se podría ver reflejado en el alto número de fragmentos de ADN únicos obtenido en los animales estudiados, que no refleja una unidad genética discreta, esto es, un grupo de animales aislados por barreras geográficas que comparten un componente genético común (Gyllensten 1985, Ward et al. 1994, Carvalho y Hauser 1995).
Los otros cinco sitios de muestreo estudiados, reflejaron la unidad genética discreta esperada en peces aislados por barreras geográficas, tal como, la cordillera Central de Colombia, cuyo pico mas alto en la región de muestreo tiene alrededor de 2 500 msnm. El río Humarada, afluente del río Magdalena, estableció en el dendrograma un solo grupo con una alta con fiabilidad (89) de los dos sitios de muestreo considerados en este trabajo, sugiriendo un componente genético relacionado (Fig. 1). Los ríos Piedras, La Clara y Guaracú, son tributarios del río Cauca; el grupo de animales provenientes del río Piedras presentó una gran cantidad de fragmentos únicos, lo cual se podría asociar con el aislamiento a que ha sido sometido por la construcción de la represa del río Piedras en el año 2 000. Esto ha hecho que se mantenga en cautiverio un gran número de animales, ocasionando un aumento de los fragmentos únicos, pero conservando las características propias de su grupo genético original. El río La Clara y las dos muestras del río Humarada, conservan su perfil genético propio, si se tiene en cuenta el número de fragmentos únicos observados (Cuadro 3), sugiriendo que estas muestras de animales forman grupos discretos diferenciables. Los animales de esta especie tienen poca intervención del hombre, por lo que se consideraría como fuente natural de recurso genético. Con relación a los animales del río Guaracú, se sugiere un estudio mas a fondo del componente genético, ya que presentó pocos fragmentos, convirtiéndolo en el grupo menos diverso entre la especie de B. henni, posiblemente debido, a un efecto de deriva genética, lo que ocasionó la distorsión de los resultados esperados. La región en la cual se localiza este río, tiene antecedentes de deforestación y construcción de obras civiles, lo que posiblemente ha conducido a una dispersión de los animales. Muchas de las poblaciones de B. henni a lo largo de las dos cuencas han disminuido sus animales, o se han desplazado a las zonas de altas montañas, donde los nacimientos de los pequeños ríos les sirven de refugio, creando así grupos aislados, pero les asegura su reproducción y supervivencia, evitando la contaminación de aguas, la sobre pesca y la deforestación.
Con base en los resultados, consideramos que la inadecuada intervención del hombre mediante la captura indiscriminada de animales y la construcción de represas, afectó la composición genética en B. henni en algunas zonas donde se llevó a cabo el muestreo. Es necesario ampliar los estudios sobre la distribución genética real de esta especie en las cuencas de los ríos Cauca y Magdalena, y confirmar su clasificación taxonómica respecto a otras especies de la subfamilia Bryconinae a la luz de la sistemática molecular. En este estudio, el bajo número de animales utilizados de cada sitio de muestreo, no proporcionó una sobre estimación de las distancias genéticas obtenidas, ya que la confiabilidad de los resultados tiene un idóneo número de réplicas (10 000), que confirma la estabilidad relativa de cada agrupación, sugiriendo una topología ajustada al fenómeno que se observó en el medio natural. El bajo número de fragmentos únicos en B. henni mostró la homogeneidad de los grupos analizados, aun así, existen fragmentos que hacen la diferencia, lo anterior contrasta con lo observado con las especies B. moorei y C. macropomum las cuales contienen muchos fragmentos que separan a estas especies de peces. La diversificación inter especifica confirmó la relación taxonómica entre las dos especies, B. moorei y la B. henni, realizada por Dalh (1971) y Gery (1977) mediante características morfométricas y revisada posteriormente por Howes (1982).
En este trabajo, la técnica de RAPD fue apropiada para evaluar el componente genético de B. henni, localizada en el departamento de Antioquia, zona central de Colombia. El perfil de fragmentos discriminó entre animales provenientes de dos cuencas diferentes, río Magdalena y río Cauca; y a aquellas que han sufrido un efecto antrópico. Los grupos de animales se comportan como grupos discretos lo cual debe ser tenido en cuenta para futuros programas de repoblación.
Agradecimientos
Agradecemos a las personas e instituciones que hicieron posible este proyecto, el cual tuvo el apoyo económico de COLCIENCIAS (contrato 1115-09-10859), La Universidad de Antioquia y el Laboratorio Genes Ltda.
Resumen
El conocimiento sobre la diversidad genética de especies nativas de peces, es esencial para la conservación y manejo apropiado de animales en los programas de repoblación. Brycon henni ha sido reportada en las cuencas de los ríos Magdalena y Cauca; actualmente, la especie ha disminuido su número de animales y reducido su distribución geográfica como consecuencia de los efectos antrópicos. Por lo tanto, es necesario conocer el componente genético de los reducidos grupos de animales en los riachuelos, para iniciar algunos programas de repoblación. En este estudio el Polimorfismo de ADN Amplificado al Azar (RAPD), fue utilizado para estimar el componente genético actual en esta especie. Para este propósito; se evaluaron seis sitios de muestreo localizados en el departamento de Antioquia, cordillera Central de Colombia. De cuarenta iniciadores utilizados, treinta y cinco de ellos (87.5 %) produjeron 1 466 fragmentos reproducibles y consistentes; 417 fueron considerados como fragmentos únicos, que permitieron discriminar entre las muestras de las cuencas de los ríos Magdalena (Humarada-1 y Humarada-2) y Cauca (Piedras, La Clara y Guaracú), sugiriendo que cada una es una unidad discreta. Esta diversidad en los resultados, según el sitio de muestreo y por las características de cada uno de ellos, posiblemente sugiere, que los efectos antrópicos como presión por pesca, construcción de embalses, deforestación y contaminación del agua, han contribuido al aislamiento de estos grupos de peces en las zonas de alta montaña. Brycon moorei y Colossoma macropomum, como grupos de control inter especifico, se ubicaron fuera del grupo general de B. henni, confirmando su clasificación taxonómica mediante datos morfológicos. La técnica de RAPD fue útil para conocer la diversidad genética y discriminar entre poblaciones de B. henni de diferente origen geográfico, ello permitiría realizar un plan apropiado de conservación y manejo en medio natural.
Palabras clave: Conservación, pez, diversidad genética, RAPD, restauración, Brycon henni.
Referencias
Bardakci, R. 2001. Random Amplified Polymorphic DNA (RAPD) markers. Turk J Biol. 25:185-196. [ Links ]
Barfai, R., S. Egedi, G.H. Yue, B. Kovacs, B. Urbanyi, G. Tamas, L. Horvath & L. Orban. 2003. Genetic analysis of two commom carp broodstocks by RAPD and microsatellite markers. Aquaculture 219:157-167. [ Links ]
Barman, H.K., A. Barat, B.M. Yadav, S. Banerjee, P.K. Meher, P.V. Reddy & R.K. Jana. 2002. Genetic variation between four species of Indian major carps as revealed by random amplified polymorphic DNA assay. Aquaculture 2002: 1-9. [ Links ]
Beltrán-Turriago, C.S. & A.A. Villaneda-Jimenez. 2000. Perfil de la pesca y la acuicultura en Colombia. Instituto Nacional de Pesca y Acuicultura (INPA), Bogotá, Colombia. [ Links ]
Carvalho, G.R. & L. Hauser. 1995. Molecular genetics and the stock concept in fisheries, p. 55-79. In G.R. Carvalho & T.J. Pitcher (eds.) Molecular genetics in fisheries. Chapman & Hall, Nueva York, EEUU. [ Links ]
Ceccarelli, O.S. & J.A. Senhorini. 1996. Brycon: viabilização da produção de alevinos. Panorama da aqüicultura Maio/Juhno, 10-11. São Paulo, Brasil. [ Links ]
Dahl G. 1971. Los peces del norte de Colombia. Litografía Arco. Bogotá, Colombia. 520 p. [ Links ]
Dinesh, K.R., T.M. Lim, K.L. Chua, W.K. Chan & V.P.E. Phang. 1993. RAPD analysis: an efficient method of ADN fingerprinting in fishes. Zoolog. Sc. 10: 849-854. [ Links ]
Efron, B. 1979. Bootstrap methods: another look at the jackknife. Ann. Statist. 7: 1-26. [ Links ]
Elo, K., S. Ivanoff, J.A. Vuorinen & J. Piironen. 1997. Inheritance of RAPD markers and detection of interspecific hybridization with brown trout and Atlantic salmon. Aquaculture 152: 55-65. [ Links ]
Estoup, A., P. Presa, F. Kreig, D. Vaiman & R. Guyomard. 1993. (CT)n and (GT)n microsatellite: a new class of genetic markers for Brown trout (Salmo trutta L.). Heredity 7: 1 029-1 039. [ Links ]
Felsenstein, J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution 39: 783-791. [ Links ]
Gery, J. 1977. Characoids of the world. P. Publications, Nueva Jersey, EEUU. [ Links ]
Guo, H.R., S.C. Zhang, S.L. Tong & J.H. Xiang. 2001. Analysis of three marine fish cell lines by RAPD assay. In vitro Cell Dev. Biol. 37: 430-433. [ Links ]
Gyllensten, U. 1985. The genetic structure of fish: differences in the intraspecific distribution of biochemical genetic variation between marine, anadromous and freshwater species. J. Fish Biol. 26: 691-699. [ Links ]
Howes, G. 1982. Review of the genus Brycon (Teleostei, Characoidei). Bull. Br. Mus. Zool. 43:1-47. [ Links ]
INPA (Instituto Nacional de Pesca y Acuicultura). 2001. Boletín Estadístico Pesquero. Bogotá, Colombia. [ Links ]
Instituto Alexander von Humboldt. 2000. Colombia mega diversa: cinco años explorando la riqueza de un país biodiverso. Instituto de Investigación de Recursos Biológicos Alexander von Humboldt. Villa de Leyva. Boyacá, Colombia. [ Links ]
Kawamura, K., T. Ueda, R. Arai, Y. Nagata, K. Saitoh, H. Ohtaka & Y. Kanoh. 2001. Genetic introgression by the rose bitterling, Rhodeus ocellatus ocellatus, into the Japanese rose bitterling, R. O. kurumeus (Teleostei: Cyprinidae). Zoolog. Sc. 18: 1027-1039. [ Links ]
Koh, T.L., G. Khoo, L.Q. Fan & V.P.E. Phang. 1999. Genetic diversity among wild forms and cultivated varieties of Discus (Symphysodon spp.) as revealed by Random Amplified Polymorphic DNA (RAPD) fingerprinting. Aquaculture 173: 485-497. [ Links ]
Liu, Z., P. Li, B.J. Argue & R.A. Dunham. 1998. Inheritance of RAPD markers in channel catfish (Ictalurus punctatus), blue catfish (I. furcatus), and their F1, F2 and backcross hybrids. Animal Gen. 29: 58-62. [ Links ]
Michelmore, R.W., I. Paran & R.V. Kesseli. 1991. Identification of markers linked to disease-resistance genes by bulked segregant analysis: A rapid method to detect markers in specific genomic regions by using segregating populations. Proc Natl Acad Sci. 88: 9 828-9 832. [ Links ]
Miller, S.A., D.D. Dykes & H.F. Polesky. 1988. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Res. 16: 1215. [ Links ]
Mojica, J.I. 1999. Lista preliminar de especies dulceacuícolas de Colombia. Rev. Acad. Col. Cienc. Suplemento especial 23: 547-566. [ Links ]
Nei, M. & W.H. Li. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proc. Natl. Acad. Sci. 76: 5 269-5 273. [ Links ]
Palumbi, S.R. 1996. Nucleic Acids II: The Polymerase Chain Reaction, p. 205-247. In D.M. Hillis, C. Moritz & B.K. Mable (eds). Molecular systematics. Sinauer Ass. Sunderland, Massachussetts, EEUU. [ Links ]
Saitou, N. & M. Nei. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 4: 406-425. [ Links ]
Sambrook, J., E.F. Fritsch & T. Maniatis. 1989. Molecular cloning: A Laboratory Manual. Cold Spring Harbor, Nueva York, EEUU. [ Links ]
Swofford, D.L. 1996. PAUP: Phylogenetic Analysis Using Parsimony (and other methods), version 4.0. Sinauer, Sunderland, Massachussetts, EEUU. [ Links ]
Ward, R.D., M. Woodwark & O.F. Skibinski. 1994. A comparison of genetic diversity levels in marine, freshwater and anadromous fishes. J. Fish Biol. 44: 213-232. [ Links ]
Welsh, J. & M. McClelland. 1990. Fingerprinting genomes using PCR with arbitrary primers. Nucleic Acids Res. 18: 7 213-7 218. [ Links ]
Williams, J.G.K., M.K. Kubelik, K.J. Licak, J.A. Rafalski & S.V. Tingey. 1990. DNA polymorphism amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res 18: 6 531-6 535. [ Links ]
Yu, K. & K.P. Pauls. 1992. Optimization of the PCR program for RAPD analysis. Nucl Acids Res 20: 2 606. [ Links ]














