Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista de Biología Tropical
On-line version ISSN 0034-7744Print version ISSN 0034-7744
Rev. biol. trop vol.47 n.4 San José Dec. 1999
Bactris gasipaes(Palmae)
Sonia Rojas-Vargas 1 Pilar Ramírez 1 2 y Jorge Mora-Urpí1
Received 25- IX-1998. Corrected 12-IV-1999. Accepted 16-IV-1999.
Abstract
The study of genetic diversity in peach palm (Bactris gasipaes K.) is important for the breeding work on this palm and to corroborate the hypotheses on its origins. For that purpose it is necessary to use alternative techniques to complement the morphological studies traditionally made. One of the techniques that responds to that need is isozyme electrophoresis. The isozymes are biochemical markers of importance in the study of genetic variability in plants of economic importance, because they are the primary products of genetic expression. This work is an electrophoretic analysis on gels of polyacrilamide to study phenetic relations using twelve isozymes on four races and an spontaneous hybrid of peach palm. The biological material used came from the germplasm bank from Los Diamantes Experimental Station, Guápiles-Costa Rica. Four races and a hybrid were selected from that collection. They represented different procedences with morphological differences as well as having particular geographical distribution patterns; they were: Tembé-Chapare (Bolivia-Bo), Pará -Belem (Brasil-Bra), Utilis Guápiles (Costa Rica-CR), Tuira-Darién (Panamá-Pa) and Yurimaguas (Perú-Pe). Five trees of each race and the Yurimaguas hybrid were studied. Different tissues were tried: leaf, adventitious roots, spines, masculine flowers and ripe fruits. An standard native and discontinued method was established on a polyacrilamide matrix for the analysis of twelve isozymes: (PRX, EST, ACP, DIA, G6PDH, ME, MDH, GOT, ADH, PGI, PGM and SOD). The amount of tissue used was 1. 5 g, with 20 min clarification time at 15000 gravity value and a temperature of 4°C. The protocol proposed by Baaziz & Saaidi (1988) was followed with a modification of concentration of the phosphate buffer solution from 0,005 M to 0. 1M with a 50 to 200 volts gradient. The root, flower and spine tissue were difficult to homogenize because of their high fiber content. The fruit has too much oil and starch, which distort the banding. For those reasons they were discarted. The leaves were selected because they are easy to homogenize and are available all year round. The standardized electrophoretic method allowed the effective use of ten of the twelve isozymes in the leaf tissue. Those ten isozymes showed phenetic polymorphism and are considered useful polymorphic markers for genetic and phenetic diversity studies in Bactris gasipaes. A discriminant marker for the Utilis-Guápiles race was found with the PRX isozyme, which shows an additional locus only in material from that procedence. The conglomerate statistical analysis showed that the races and hybrid studies can be placed in three groups: one is formed by Par and Tembé, the second one by Utilis and Tuira, and the hybrid Yurimaguas is a class by itself and although it seems a bit closer to Utilis-Tuira, it is suspected that has its origin from unknown parents. These results fit previous morphological interpretations. A genetic analysis was not made because of the small sample size, but methodological procedures were established for future research projects using larger populations to study peach palm isozyme electrophoretic diversity.
Key words
Pejibaye, Bactris gasipaes, Peach palm, isozyme, biodiversity, polymorphism, electrophoresis.
El pejibaye (Bactris gasipaesKunth) es una palmera nativa del bosque tropical húmedo americano, que crece entre los paralelos 16° N y 17° S y posee un alto potencial económico (Mora-Urpí 1991, Mora-Urpí et al. 1997). Existen varias hipótesis sobre el origen y domesticación de esta planta, una sugiere que el origen de la planta tuvo lugar en la región ubicada a ambos lados de la cordillera de los Andes y sur de Centro América, y que la domesticación ocurrió independientemente en varias localidades a lo largo de este territorio a partir de diferentes poblaciones silvestres (Mora-Urpí 1991). La otra hipótesis sugiere que el pejibaye fue domesticado una sola vez en algún lugar del Alto Amazonas (Clement 1995).
A nivel morfológico, existe una gran diversidad entre las poblaciones de pejibayes silvestres y las cultivadas. Mora-Urpí (1991) considera que esta variabilidad se debe al origen polifilético del pejibaye cultivado y cuyos cultivares resultantes han participado en hibridaciones posteriores.
El estudio de la diversidad genética del pejibaye es de suma importancia para el mejoramiento integral de esta palmera y para comprobar las hipótesis sobre el origen del cultivo. Es necesario emplear técnicas que faciliten su análisis genético y que complementen los estudios morfológicos tradicionalmente utilizados, una de las técnicas que responden a esta necesidad es la electroforesis de isoenzimas. Las isoenzimas son marcadores bioquímicos de gran importancia en el estudio de variabilidad genética de muchas plantas de interés económico porque son el producto primario de la expresión génica (Crawford 1989).
El objetivo de esta investigación es establecer un sistema de análisis electroforótico de isoenzimas en geles de poliacrilamida, que permita determinar el grado de polimorfismo y las relaciones fenéticas de cuatro razas y un híbrido de pejibaye.
Materiales y Métodos
Material vegetal: Se usó material del banco de germoplasma ubicado en la Estación Experimental "Los Diamantes", Guápiles, Costa Rica. De esa colección fueron seleccionadas cuatro razas y un híbrido de pejibaye de diferentes procedencias (Cuadro 1) y que presentan variaci–n morfológica entre sí: Tembé-Chapare (Bolivia-Bo), Pará-Belem (Brasil-Bra), Utilis-Guápiles (Costa Rica-CR), Tuira-Darién (Panamá-Pa) y el híbrido Yurimaguas (Perú-Pe). Se estudiaron cinco árboles por cada raza e híbrido, de cada uno se evaluaron tejidos de hoja, raíz adventicia, espina, flores masculinas y fruto maduro.
Procendencia y ubicación en el banco de germoplasma de los materiales vegetativos de pejibaye empleados en este estudio
| | Introducción | | | | |
| Mesocarpa | Yurimaguas | | | | 2 |
| Mesocarpa | Yurimaguas | | | | 2 |
| Mesocarpa | Yurimaguas | | | | 2 |
| Mesocarpa | Yurimaguas | | | | 5 |
| Mesocarpa | Yurimaguas | | | | 9 |
| Microcarpa | Pará-Belem | | | | 5 |
| Microcarpa | Pará-Belem | | | | 5 |
| Microcarpa | Pará-Belem | | | | 1 |
| Microcarpa | Pará-Belem | | | | 6 |
| Microcarpa | Pará-Belem | | | | 9 |
| Microcarpa | Tembé-Chapare | | | | 9 |
| Microcarpa | Tembé-Chapare | | | | 6 |
| Microcarpa | Tembé-Chapare | | | | 5 |
| Microcarpa | Tembé-Chapare | | | | 8 |
| Microcarpa | Tembé-Chapare | | | | 2 |
| Microcarpa | Tuira-Darién | | | | 1 |
| Microcarpa | Tuira-Darién | | | | 5 |
| Microcarpa | Tuira-Darién | | | | 2 |
| Microcarpa | Tuira-Darién | | | | 2 |
| Microcarpa | Tuira-Darién | | | | 8 |
| Mesocarpa | Utilis-Guápiles | | | | 2 |
| Mesocarpa | Utilis-Guápiles | | | | 1 |
| Mesocarpa | Utilis-Guápiles | | | | 3 |
| Mesocarpa | Utilis-Guápiles | | | | 1 |
| Mesocarpa | Utilis-Guápiles | | | | 6 |
Tratamiento de las muestras: Se tomaron muestras de 2 g, 1. 5g, 1g y 0. 5g de cada tejido, las que se maceraron en morteros y pistilos de cerámica preenfriados, a las que se les añadieron 3 ml de buffer de extracción (Baaziz y Saaidi 1988). Los extractos fueron centrifugados a 4°C por 5, 10, 15, 20, 25 min. en las siguientes gravedades: 10 000, 15 000, 20 000 y 25 000. El sobrenadante fue recobrado y almacenado a -70°C hasta el momento de su análisis.
Análisis electroforético y evaluación de zimogramas: Se utilizó el método nativo y discontínuo sobre una matriz de separación de poliacrilamida (Hames y Rickwood 1987).
Se emplearon los procedimientos de revelado descritos por Vallejos (1983) y Harris y Hopkinson (1976). Las isoenzimas evaluadas fueron: peroxidasa (PRX), esterasa (EST), fosfatasa ácida (ACP), diaforasa (DIA), fosfoglucoisomerasa (PGI), glucosa-6-fosfato deshidrogenasa (G6PDH), enzima málica (ME), malato deshidrogenasa (MDH), glutamato oxalato transaminasa (GOT), alcohol deshidrogenasa (ADH), superóxido dismutasa (SOD), fosfoglucomutasa (PGM).
La evaluación de los zimogramas o fenotipos se basó en que cada banda horizontal probablemente corresponde a un locus y que la posición de cada una equivale a un alelo (Wendel y Weeden 1989, Bennanceur et al. 1991). Los loci se denominaron con letras de A hasta Z y los alelos se catalogaron de o hasta n según Ghesquiere (1987) y Sánchez (1988).
Mediante un análisis de conglomerados se establecieron las relaciones de similitud de fenotipos electroforéticos por isoenzima entre el híbrido y las cuatro razas estudiadas.
Resultados
Material vegetal y tratamiento de las muestras: El proceso de homogeneización de los tejidos de raíz adventicia y flores masculinas resultó arduo y difícil por la dureza de los tejidos, y del todo imposible, con el tejido de espinas, debido a la naturaleza altamente fibrosa de los tejidos de pejibaye. Sin embargo, el tejido de hoja y fruto se homogeneizaron más fácilmente porque contiene menos fibras. El tejido de fruto se descartó debido a que su alto contenido de grasa y almidón distorsionaron el patrón de bandas y el sistema de electroforesis se recalentó, probablemente alterando la estructura de las proteínas. De los cinco tejidos estudiados, el tejido de hoja mostró los mejores resultados en los procesos de homogeneización, extracción y análisis enzimáticos, y reveló los mejores patrones de bandas. Por todas estas razones se eligió para el análisis electroforético de las doce isoenzimas.
Se encontró que la cantidad óptima de tejido fue 1. 5 g, con un tiempo de clarificación de 20 min. a una gravedad de 15 000, y a una temperatura de 4°C.
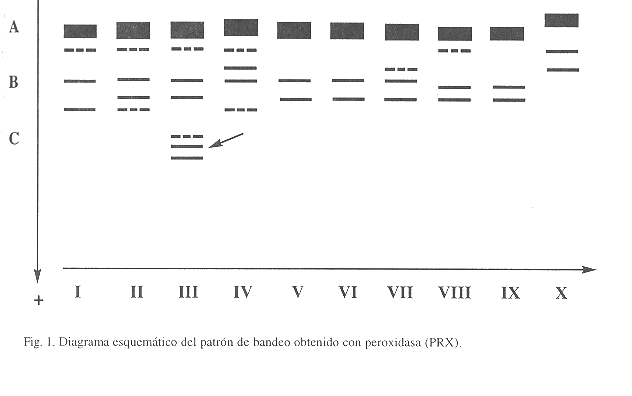
Análisis electroforético y evaluación de zimogramas: El método de electroforesis utilizado en este estudio permitió la visualización de diez de las doce isoenzimas evaluadas; las enzimas ADH y GOT no mostraron actividad enzimática en estas condiciones. La resolución de los zimogramas varió con cada enzima y los mejores patrones de bandas se obtuvieron con PRX, G6PDH, PGI y PGM. Las isoenzimas fueron variables en mayor o menor grado. DIA es la isoenzima con mayor nmero de fenotipos por gel (14) y PGM (3) la que presentó menor número (Cuadro 2). Las muestras de la raza Utilis-Guápiles (CR) analizadas con la isoenzima PRX mostraron un locus adicional, locus C del fenotipo III (Fig. 1).
Actividad, resolución y análisis de los zimogramas de las doce isoenzimas ensayadas con tejido de hojas de pejibaye
| Enzima | | | bandas | | por gel |
| PRX | | | | 3(A,B,C) | |
| EST | | | | 4(A,B,C,D) | |
| ACP | | | | 2 (A,B) | |
| MEPC | | | | 2 (A,B) | |
| DIA | | | | 3 (A,B,C) | |
| MDH | | | | 1 (A) | |
| G6PDH | | | | 2 (A,B) | |
| PG1 | | | | 2 (A,B) | |
| SOD | | | | 3 (A,B,C) | |
| PGM | | | | 1 (A) | |
| GOT* | | | | - | |
| ADH* | | | | - | |
Resolución: +++ = Muy buena + = Regular -+ = Pobre - = Ninguna
* = Se debe mejorar el método de electroforésis
Pc = Patrones de bandas complejos
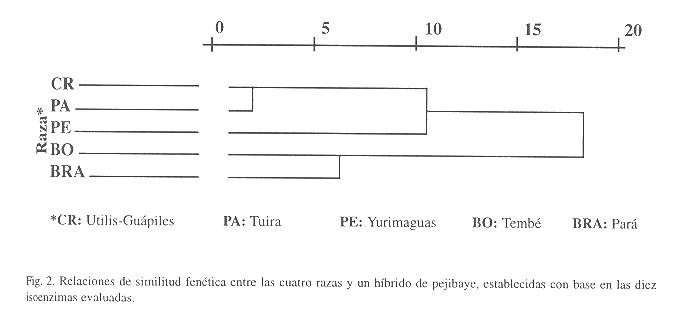
Mediante el análisis estadístico de conglomerados se encontró que las razas y el híbrido estudiados se aglomeran en tres grupos y que cada uno comparte similitudes fenotípicas entre sí. El grupo uno está compuesto por las razas de Pará-Belem (Br) y Tembé-Chapare (Bo); el grupo dos lo conforman las razas de Utilis-Guápiles (CR) y Tuira-Darién (Pa), en tanto, el híbrido Yurimaguas (Pe) se ubica en una posición disímil, ligeramente más cercana a Utilis-Guápiles (CR) y Tuira-Darién (Pa) (Fig. 2).
Discusión
Las diez isoenzimas estandarizadas y analizadas en este estudio representan marcadores polimórficos útiles para futuros estudios de variabilidad genética y fenética en Bactris gasipaes. Debido a que la muestra usada en esta investigación fue pequeña, no se hizo un análisis genético, pero se sientan las bases metodológicas para otras investigaciones que busquen evaluar poblaciones más grandes y conocer la enorme variabilidad genética del pejibaye a través de la electroforesis de isoenzimas.
Es importante mencionar que los tejidos de pejibaye son difíciles de homogeneizar por los altos contenidos de fibras. La hoja es el tejido más adecuado porque se homogeneizó fácilmente, produjo zimogramas claros, es un tejido disponible durante todo el año, es fácil de recolectar y permite evaluar varios sistemas enzimáticos.
Se observó variación de fenotipos electroforéticos entre las plantas de diferentes procedencias; es altamente probable que este polimorfismo fenético sea el producto de la gran variabilidad genética que posee esta especie, y que se manifiesta a través de sus características morfológicas. (Mora-Urpí 1991).
Otras palmeras como P. datilifera y E. oleifera también muestran un alto polimorfismo, que se evidencia por una amplia diversidad morfológica (Torres y Tisserat 1980, Ghesquiere 1987, Bennanceur et al. 1991). Este alto polimorfismo podría obedecer a su reproducción alógama, ser producto de adaptaciones a diversas condiciones ambientales o a la coexistencia de razas genéticamente diferentes (Bennanceur et al. 1991). En el pejibaye cultivado esta diversidad podría deberse a su origen polifilético (domesticación de varias especies hermanas), hibridación, introgresión, reproducción alógama, selección, deriva genética y mutación (Mora-Urpí 1991). Sin embargo, Clement (1995) encontró poca diversidad dentro de las poblaciones que estudió con isoenzimas, lo atribuye a una base genética reducida.
La peroxidasa reveló un locus nico con las muestras de la raza de Utilis-Guápiles (CR), el cual podría usarse como un marcador discriminatorio para esta raza. Es posible que sea producto de una mutación simple, aunque es necesario estudiar un mayor nmero de individuos para corroborar tal hipótesis.
El análisis de conglomerados mostró una estrecha relación fenética entre las razas Pará-Belem (Br) y Tembé-Chapare (Bo), posiblemente porque ambas descienden de Bactris insignis; mientras que las razas Utilis-Guápiles (CR) y Tuira-Darién (Pa) también se agrupan fenotípicamente aún cuando la morfología entre estas razas es algo disímil. Mora-Urpí (1991) comenta que esta ltima relación se debe a que estas razas posiblemente comparten ancestros. En otras plantas se han encontrado similitudes isozímicas entre razas geográficas, taxonómicamente reconocidas como subespecies o variedades (Doebley 1989).
El análisis de similitud fenotípica que combina todos los zimogramas isoenzimáticos muestra a Yurimaguas separado del resto de razas y aunque ligeramente más cercano al grupo Utilis-Tuira, por lo que constituye una clase por sí sola, cuyos padres aún no han sido identificados.
Agradecimientos
Agradecenos al equipo de trabajo del Proyecto de Mejoramiento Genético de Pejibaye en Guápiles por su colaboración en la recolección de muestras. A Joe Tohme y a la Unidad de Biotecnología del Centro Internacional de Agricultura Tropical (CIAT) por su colaboración en la implementación del sistema de electroforesis e interpretación de datos. A Ramiro Barrantes y su equipo de trabajo (INISA) por su colaboración. A Daniel Crawford (Ohio State University) por su asesora en la evaluación de zimogramas y a Harry Hernández por la asesoría en el análisis estadístico. A Charles Clement por sus sugerencias y aportes.
Resumen
Se estandarizó un sistema de electroforesis de isoenzimas con tejido de hojas que permitió la identificación del polimorfismo fenético entre cuatro razas y un híbrido de pejibaye Bactris gasipaes provenientes de Brasil, Perú, Bolivia, Panamá y Costa Rica. Las isoenzimas ensayadas fueron: PRX, EST, ACP, ME, DIA, MDH, G6PDH, PGI, SOD, PGM, ADH, GOT, de estas solamente las dos ltimas no mostraron actividad. El resto de isoenzimas mostraron polimorfismo fenético en diferentes grados, por esto se consideran marcadores polimórficos potenciales para estudios de variabilidad genética en pejibaye. Se encontró un locus único en el zimograma de la enzima PRX en las muestras de Utilis-Guápiles (CR), el cual podría usarse como un marcador discriminatorio para esta raza. Se establecieron relaciones de similitud isoenzimática entre las razas Utilis-Guápiles (CR) y Tuira-Darién (Pa); Tembé-Chapare (Bo) y Pará-Belem (Bra) respectivamente, mientras que el híbrido Yurimaguas (Pe) se ubicó aparte y ligeramente más cercano a Utilis y Tuira, por esto se considera que posiblemente desciende de padres aún no identificados.
Referencias
Baaziz, M. & M. Saaidi. 1988. Preliminary identification of date palm cultivars by esterase isoenzymes and Benannceur, M, C. Lanaud, M. Chevalier & N. Bounaga. 1991. Genetic diversity of the date palm (Phoenix dactylifera L) from Algeria revealed by enzyme markers. Plant Breeding 107: 56-69. [ Links ]
Clement, C, R. 1995. Pejibaye, Bactris gasipaes (Palmae). In J. Smartt & N. W. Simmonds (eds). Evolution of crops. Longman, Londres, p. 383-388. [ Links ]
Crawford, D. J. 1989. Enzyme electrophoresis and plant systematics, p 146-165. In. Soltis, D. E. & P. S. Soltis. Isozymes in plant biology. Dioscorides, Portland, Oregon, 268p. [ Links ]
Doebley, J. 1989. Isozymic evidence and the evolution of crop plants, p 165-87. In. D. E. Soltis & P. S. Soltis. Isozymes in plant biology. Dioscorides, Oregon, 268p. [ Links ]
Ghesquiere, M. 1987. Polimorphisme enzymatique chez Elaeis oleifera H. B. K. Analyse des populations du Bassin amazonien. Oleagineux 42: 145-153. [ Links ]
Hames, B. D. & D. Rickwood 1987. Gel electrophoresis in human genetics. North Holland, Amsterdan. p1-15. [ Links ]
Harris, H. & D. A. Hopkinson. 1976. Handbook of enzyme electrophoresis in human genetics. North Holland, Amsterdam. 115 p. [ Links ]
Mora-Urpí J, J. C. Weber & C. R. Clement. 1997. Peach palm. Bactris gasipaes Kunth. Promoting the conservation and use of under utilized and negleted crops. IPGRI, Roma, 83 p. [ Links ]
Mora-Urpí J. 1991. Diversidad genética en pejibaye (Bactris (guilielma) gasipaes Kunth): Origen y domesticación. Cuarto Simposio Internacional sobre Biología, Agronomía e Industrialización del Pijuayo. Universidad de Costa Rica, San José. p21-29. [ Links ]
Sánchez, I. 1988. Etude de la variabilite genetique de Terminalia superba. Engler & Diels foretier d' Afrique Tropicale. These. Université Paris VII, Paris 128p. [ Links ]
Torres, A. M. & B. Tisserat. 1980. Leaf isozyme as genetic markers in date palms. Amer. J. Bot 67: 162-167. [ Links ]
Vallejos, C. E. 1983. Enzyme activity staining. p 482-507. In, S. D. Tankley & T. J. Orton (eds.). Isozymes in plant breeding. Part A.. Elsevier Science, Amsterdam. [ Links ]
Wendel. F. J. & N. F. Weeden. 1989. Visualization and interpretation of plant isozymes. In: Soltis, D. E. & P. S. Soltis. Isozymes in plant biology. Dioscorides. Portland, Oregon. 259 p. [ Links ]
1 Escuela de Biología, Universidad de Costa Rica, 2060 San José, Costa Rica.
2 Centro de Investigación en Biología Celular y Molecular, Universidad de Costa Rica, 2060, San José,
Costa Rica.