Introducción
La acuicultura, a nivel mundial, ha crecido a lo largo de los últimos 60 años, y el cultivo de tilapia se ha convertido en una actividad acuícola de gran importancia comercial (Fitzsimmons, 2013). La producción mundial de tilapia ha aumentado aproximadamente 4.7 % anualmente desde el año 2000. Para el 2016, se reportó una producción de 5 897 652 t, donde los principales países productores son China, Egipto, Indonesia, Brasil, Filipinas y Tailandia, entre otros (FAO, 2015). En Guatemala, la industria acuícola en el 2017 produjo alrededor de 9 753 TM de tilapia, evidenciando un crecimiento del 3.9 % anual desde el año 2010 (FAO, 2018).
El aumento de la producción acuícola ha sido acompañado por el desarrollo de problemas ecológicos y patológicos, este último es el de mayor preocupación en el sector acuícola. La alta densidad de siembra, manejo deficiente de los peces, fluctuaciones en los factores ambientales, mala calidad de agua y una mala nutrición contribuyen a que los organismos se encuentren inmunológicamente suprimidos, susceptibles a infecciones bacterianas, por patógenos facultativos u oportunistas.
Las bacterias de mayor frecuencia en los sistemas acuícolas se clasifican como bacterias Gram negativas; estas, en gran parte, ocasionan el síndrome de la septicemia hemorrágica bacteriana, y como bacterias Gram positivas, en su mayoría causan infecciones granulomatosas.
Dentro de las bacterias más predominantes a nivel mundial se pueden encontrar: Aeromonas hydrophila, A. liquifaciens, A. punctata, A. sobria, Edwardsiella tarda, Flexibacter columnaris, Pseudomonas aeruginosa, P. fluorescens, Streptococcus iniae y S. agalactiae (Mzula, et al., 2020; Chitmanat, et al., 2016; Plumb & Hanson, 2010).
En los sistemas de producción de tilapia en Guatemala se reportan mortalidades por patógenos bacterianos entre 20 % - 30 %; y en algunos casos extremos se llega a obtener hasta 90 % de mortalidad. Las familias bacterianas de mayor presencia son: Aeromonadaceae, Pseudomonadaceae, Pasteurellaceae, Aerococcaceae, Comamonadaceae, Streptococcaceae, Alteromonadaceae y Enterobacteriaceae (Marroquín-Mora & García-Pérez, 2015).
La prevención y el control de las enfermedades se han convertido en la prioridad para la sostenibilidad de esta industria acuícola. En la praxis tradicional, para evitar mortalidades, se utilizan medicamentos de uso veterinario de amplio espectro, suministrados en el medio de cultivo y a través del alimento, con el fin de controlar y prevenir la infección de diversos patógenos bacterianos. A nivel global se utilizan diversos antibióticos, los más comunes oxitetraciclina, sulfadiazina y florfenicol, los cuales han sido reportados en los países de mayor producción, y con menor frecuencia: enrofloxacina, eritromicina, sulfametoxazol, sulfadimidina y trimetoprim, entre otros (Mo et al., 2017). El uso indiscriminado de los agentes antimicrobianos (antibióticos) provenientes de la acuicultura resulta en la contaminación de agua y sedimento, promueve una presión selectiva y crea reservorios de bacterias resistentes a los medicamentos (Espinosa, & Bermúdez, 2012).
Desafortunadamente, la información disponible sobre los diversos patógenos bacterianos presentes en los cultivos de tilapia en Guatemala es muy limitada. Con el fin de generar directrices de uso responsable de los antibióticos, tampoco se cuenta con datos de sensibilidad de las bacterias a los antimicrobianos utilizados en la industria en el país.
Por lo tanto, el presente estudio tiene como finalidad identificar los patógenos bacterianos de mayor prevalencia y determinar su resistencia antimicrobiana a los antibióticos disponibles para la industria acuícola en Guatemala.
Metodología
Aislamiento e identificación de las bacterias patógenas
Durante los años 2018-2019 se realizaron muestreos por conveniencia en diez centros de producción de tilapia. Las granjas visitadas se encuentran en los departamentos de: Escuintla, Santa Rosa, Retalhuleu, Zacapa, Izabal y Petén. Las visitas fueron programadas únicamente cuando se presumía presencia de brotes infecciosos. Se evaluaron organismos de Oreochromis spp., con un rango de peso de 50 a 1 000 g. En cada unidad de producción se seleccionaron diez organismos con signología clínica de enfermedad bacteriana. Los organismos seleccionados fueron sacrificados, según el Código Sanitario para los Animales Acuáticos (Artículo 7.4.6, Literal 1), de la Organización Mundial de Sanidad Animal (2013).
Se registró la signología macroscópica externa e interna por cada organismo evaluado, con el fin de determinar la frecuencia de signos clínicos y el agente etiológico causante de la enfermedad. Se realizó una tabla de frecuencia para determinar las siguientes signologías: melanización corporal (MC), exoftalmia uni / bilateral (EX), áreas hiperémicas (AH), ulcera cutánea (UC), licuefacción focalizada del músculo (LFM), palidez de branquias (PB); congestión branquial (CB), ascitis (AS), congestión en estómago (CE); congestión y hemorragia intestinal (CHI), pericarditis (PR), esplenomegalia (ES), granulomas en bazo (GB), centros melanomacrófagicos (CM), vasculitis en hígado (VH), hígado friable (HF), palidez en hígado (PH) e hiperemia renal (HR), por cada lote de organismos evaluados. Luego se clasificó la frecuencia de signos externos e internos como: Sin presencia de signos clínicos (- / 0 %), poca frecuencia de signos clínicos (+ / 1 - 33 % frecuencia), frecuencia media o intermedia de signos clínicos (++ / 34 - 66 % frecuencia), y alta frecuencia de signos clínicos (+++ / 67 - 100 % frecuencia).
Posteriormente, se realizó la desinfección superficial de los organismos con etanol 70 %, y se procedió a realizar la necropsia para tomar muestras bacteriológicas de fluido sanguíneo, úlceras y otras áreas hemorrágicas, riñón anterior, bazo, meninges, hígado y corazón. Las muestras fueron sembradas inicialmente en agar tripticasa soja enriquecido con sangre de carnero al 5 % e incubadas por un período de 24 horas a temperatura de 28°C ± 1˚C. Al finalizar el periodo de incubación, se seleccionaron las colonias bacterianas predominantes, las cuales fueron identificadas a nivel de especie, a través de pruebas bioquímica API20E, API 20NE y API STREP (bioMérieux S.A). Las bacterias identificadas fueron conservadas bajo el protocolo descrito por Liao & Shollenberger (2003), en solución salina tamponada con fosfato (PBS) 1X.
Actividad antibacteriana con antibióticos de uso comercial en acuicultura de Guatemala
La susceptibilidad a los antibióticos fue determinada por el método de difusión de disco con los antibióticos de mayor uso en acuicultura en Guatemala: oxitetraciclina (40 µg), florfenicol (40 µg), enrofloxacina (40 µg) y fosfomicina (40 µg), de acuerdo con la metodología establecida por Alderman & Smith (2001). Se realizaron cultivos puros por 18 h con las bacterias patógenas previamente aisladas, las cuales fueron suspendidas en PBS 1X, hasta alcanzar una concentración bacteriana de 1 - 2 x 108 UFC/mL. Posteriormente, se inoculó 100 µL de cada una de las suspensiones bacterianas preparadas y se esparció, de forma uniforme, en placas de agar Mueller-Hinton, la siembra fue por triplicado, y se dejó reposar por 5 min a temperatura ambiente. Finalmente, se colocaron sensidiscos de los antibióticos, con una distancia aproximada de 2 cm, entre cada uno. Las placas fueron incubadas durante un periodo de 24 horas a una temperatura de 28 ± 1˚C. Pasado el tiempo de incubación se midió el área de inhibición y las bacterias fueron clasificadas como resistentes (R) o sensibles (S), de acuerdo con el antibiótico utilizado.
Análisis de datos
Se realizó una estadística descriptiva para determinar la frecuencia y detección de las diversas bacterias patógenas aisladas. Se hizo un análisis de correspondencia para explorar la relación entre las variables "Bacterias patógenas aisladas" y "Signología clínica frecuente". El análisis estadístico se llevó a cabo con el programa estadístico Past V.3 (Hammer, Harper & Ryan, 2004).
Resultados
Se evaluaron un total de 80 peces con signología clínica de enfermedad bacteriana. En total se aislaron 16 bacterias patógenas, de las cuales 15 aislamientos resultaron bacterias Gram-negativa (G-) y solo un aislamiento Gram-positiva (G+). Las bacterias más representativas fueron Aeromonas sobria (6 aislamientos: 38 %), seguido por Aeromonas hydrophila (4 aislamientos: 26 %), para el resto de las bacterias solamente se aisló una cepa correspondiente al 6 % poblacional (Tabla 1).
Tabla 1: Signología macroscópica externa e interna más frecuente, en los aislamientos bacterianos
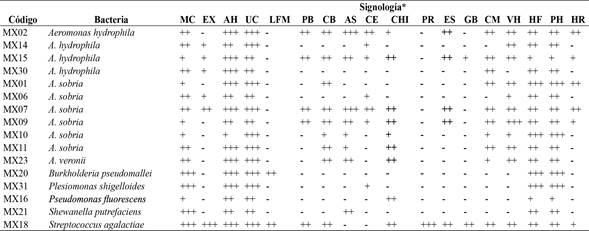
Nota: *Signología: melanización corporal (MC); exoftalmia uni / bilateral (EX); áreas hiperémicas (AH); ulcera cutánea (UC), licuefacción focalizada del músculo (LFM); palidez de branquias (PB); congestión branquial (CB); ascitis (AS); congestión en estómago (CE); congestión y hemorragia intestinal (CHI); pericarditis (PR); esplenomegalia (ES); granulomas en bazo (GB); centros melanomacrófagicos (CM); vasculitis en hígado (VH); hígado friable (HF); palidez en hígado (PH); hiperemia renal (HR); sin presencia (- : 0%), poco frecuente ( + : 1 -33% frecuencia), medianamente frecuente (++ : 34 - 66 % frecuencia), muy frecuente (+++ : 67 - 100 % frecuencia).
Las principales patologías externas observadas en los peces enfermos fueron úlceras cutáneas con áreas hiperémicas en diversas partes del cuerpo, seguido de melanización corporal total, ascitis y congestión branquial (Figura 1). Mientras que las patologías internas macroscópicas más comunes fueron: hígado friable y pálido, vasculitis en hígado, congestión y hemorragia intestinal y centros melanomacrófagicos en bazo (Figura 2).

Nota: Fuente propia de la investigación.
Figura 1 Principales patologías externas: Úlcera cutánea con áreas hiperémicas en diversas partes del cuerpo.

Nota: Fuente propia de la investigación.
Figura 2 Principales patologías internas: Hígado friable y decolorado, vasculitis en hígado, congestión y hemorragia intestinal.
En general se observa que los géneros Aeromonas, Plesiomonas, Pseudomonas y Shewanella presentan una estrecha relación con la signología de úlceras cutáneas, áreas hiperémicas, melanización corporal, hígado pálido y friable. Estos géneros corresponden al grupo catalogado como septicemia hemorrágica bacteriana (SHB). Mientras que el género Streptococcus se relaciona más a los signos de pericarditis, granulomas en bazo, licuefacción focalizada del músculo y exoftalmia; esta bacteria corresponde al grupo de bacterias granulomatosas (Gráfica 1).
Para todos los géneros de bacterias evaluados, la resistencia fue mayor a la oxitetraciclina (25 %) y fosfomicina (25 %). Todas las bacterias fueron sensibles al florfenicol y la enrofloxacina (Tabla 2). No se observó múltiple resistencia a los antibióticos en las bacterias aisladas. Las bacterias que presentaron resistencia a la oxitetraciclina fueron A. sobria (3 aislamientos) y A. hydrophila (1 aislamiento) y en el caso de la fosfomicina fueron A. hydrophila, P. fluorescens, S. putrefaciens y B. pseudomallei.
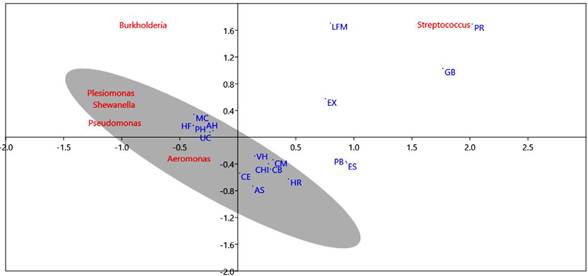
Grafica 1 Análisis de correspondencia entre las bacterias patógenas aisladas y la signología externa e interna más común. *Signología: melanización corporal (MC); exoftalmia uni / bilateral (EX); áreas hiperémicas (AH); úlcera cutánea (UC), licuefacción focalizada del músculo (LFM); palidez de branquias (PB); congestión branquial (CB); ascitis (AS); congestión en estómago (CE); congestión y hemorragia intestinal (CHI); pericarditis (PR); esplenomegalia (ES); granulomas en bazo (GB); centros melanomacrófagicos (CM); vasculitis en hígado (VH); hígado friable (HF); palidez en hígado (PH); hiperemia renal (HR). Fuente propia de la investigación.
Tabla 2: Susceptibilidad bacteriana a los antibióticos de mayor uso en acuicultura en Guatemala
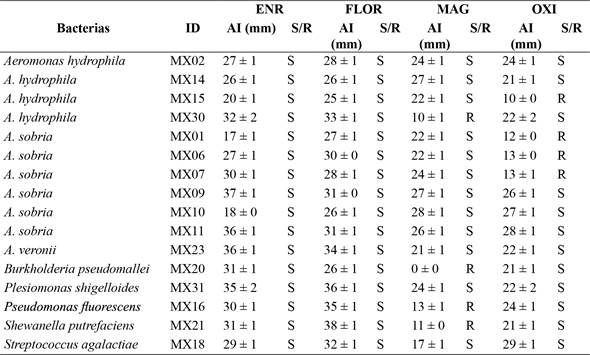
Nota: AI: área de inhibición (mm); ENR: enrofloxacina (40 µg); FLOR: florfenicol (40 µg); MAG: fosfomicina (40 µg); OXI: oxitetraciclina (40 µg); S: sensible; R: resistente. Fuente propia de la investigación.
Discusión
En Guatemala, durante 2018, la acuicultura alcanzó una producción de 26 360 TM (FAO, 2018), dominada por el cultivo de camarón con un 62 %, seguida por el cultivo de tilapia (Oreochromis spp) con un 37 %. El crecimiento exponencial de la producción de tilapia ha provocado la intensificación de los sistemas de cultivo, lo cual ha conllevado a la aparición de brotes bacterianos nuevos o patógenos bacterianos existentes con resistencia a la gama de antibióticos disponibles en el mercado.
Actualmente no existen reportes sobre los diversos agentes bacterianos que afectan la industria de la tilapia en Guatemala. Este es el primer estudio que recopila la información de los patógenos bacterianos más recurrentes en el periodo 2018-2019. Se detectaron 16 bacterias patógenas en la tilapia, donde el género más frecuente es Aeromonas, seguido por Plesiomonas, Pseudomonas, Burkholderia, Shewanella y Streptococcus. Todos los géneros son considerados como limitantes en la salud acuícola, pues contribuyen a las pérdidas económicas a nivel mundial (Dong et al., 2017; Figueiredo et al., 2012; Shuxia & Levin, 2010; Sierralta-Chichizola, Mayta-Huatuco, & León-Quispe, 2016; Zhigang et al., 2015). Las afecciones detectadas fueron atribuidas a exposiciones prolongadas a estrés provocado por manejo deficiente y factores ambientales desfavorables. De acuerdo con Akinbowale, Peng & Barton (2006), estos factores predisponen a los peces a infecciones bacterianas.
El género Aeromonas presentó la mayor distribución y frecuencia en los sistemas de cultivos evaluados. La signología clínica observada concuerda con lo descrito por Ashiru et al., (2011); Austin & Austin (2012); Noga (2010); Shayo, Mwita & Hosea (2012), quienes también observaron lesiones ulcerativas, vascularización, ruptura de aletas, necrosis en el área opercular y base craneal. A. hydrophila y A. sobria fueron las especies bacterianas aisladas con mayor frecuencia en las granjas evaluadas, por lo que se estima que son bacterias con un alto potencial para causar un impacto negativo en los cultivos de tilapia a nivel nacional, ya que son bacterias oportunistas e inmunosupresores (Raj et al., 2019). En países como Malasia, Egipto y Pakistán se reporta el aislamiento de las mismas bacterias en sistemas de producción de tilapia, lo cual provoca impactos negativos (Dong et al., 2017; Li & Cai, 2011; Shayo et al., 2012).
Por otra parte, en B. pseudomallei, P. shigelloides, P. fluorescens, S. putrefaciens y S. agalactiae, se observó menor frecuencia de aislamiento. Los peces infectados con estas bacterias presentaron diversos signos, incluidos: lesiones ulcerativas, vascularización, ascitis, hiperemia renal, atrofia hepática, esplenomegalia, pericarditis con necrosis multifocal. Estos signos clínicos coinciden con la sintomatología descrita por varios estudios (Abdel-Tawwab et al., 2010; Anshary et al., 2014; Austin & Austin, 2012; Buller, 2004; Hernández, Figueroa, & Iregui, 2009; Sierralta-Chichizola et al., 2016; Shayo et al., 2012; Zhigang et al., 2015). Los problemas asociados con estas bacterias se vuelven serios cuando la homeostasis se rompe dentro del sistema de cultivo, principalmente con S. agalactiae, considerada un patógeno que produce una enfermedad crónica (Aisyhah et al., 2014; Anshary et al., 2014; Noga, 2010).
No se encontraron bacterias como Flavobacterium columnare, Vibrio spp, Francisella spp, Lactococcus garvieae, entre otras bacterias, como lo reportan varias investigaciones (Dong et al., 2015; Meyburgh, Bragg & Boucher, 2017), lo cual se atribuye al efecto de la temporalidad, sistema de cultivo, manejo, especies, entre otros factores que ayudan a modificar la diversidad de la comunidad bacteriana externa presente.
La resistencia bacteriana en acuicultura se reporta desde la década de los 80 (Brown 1989) y avanza con rapidez, debido al uso indiscriminado de los antibióticos y a la reducción de sustancias permitidas. En Guatemala solo existen dos antibióticos registrados para el uso en la producción de tilapia: oxitetraciclina y florfenicol. El número reducido de opciones para tratar las enfermedades bacterianas permite un abuso en su uso, ya que en la producción cada vez utilizan mayor cantidad para tratar organismos enfermos. Además, son utilizados como preventivos y modulan las poblaciones bacterianas de los sistemas de producción.
Los resultados del presente estudio demuestran que A. sobria y A. hydrophila presentaron resistencia a oxitetraciclina, coincidiendo con lo reportado por otros estudios en diversos países (Igbinosa et al., 2017; Monteiro, et al., 2016). Por lo tanto, se infiere que la resistencia del género Aeromonas spp a oxitetraciclina se evidencia en el uso intensivo del antibiótico para el manejo y control de enfermedades bacterianas.
Cabe resaltar que el género Aeromonas posee genes de resistencia móviles como plásmidos, transposones e integrones, los cuales facilitan su rápida transferencia entre bacterias de los genes de resistencia para antibióticos de uso comercial (Jacobs & Chenia, 2007; Stratev & Odeyemi, 2016).
Por otra parte, antibióticos que no están permitidos para la producción de tilapia, pero sí para producción de camarón como la fosfomicina, también muestran resistencia a las bacterias A. hydrophila, P. fluorescens, S. putrefaciens y B. pseudomallei. Estos datos evidencian el uso excesivo de los antibióticos en la acuicultura en general de Guatemala, por lo tanto, sugieren la necesidad de regulaciones por parte del gobierno para evitar el incremento de la resistencia bacteriana frente a los métodos tradicionales de control, o bien de crear nuevas alternativas para el control bacteriano.
Conclusión
Las enfermedades bacterianas son importantes para el cultivo de tilapia, debido a que pueden generar pérdidas de hasta el 100 % de la población en cultivo. En el presente estudio se muestra un primer análisis sobre la diversidad de los patógenos bacterianos, su relación con los signos clínicos, así como la sensibilidad de los antibióticos más utilizados en la industria acuícola en Guatemala.
Se pudo determinar que las unidades de producción presentan a Aeromonas como el grupo bacteriano más diverso y frecuente, seguido por Burkholderia, Plesiomonas, Pseudomonas, Shewanella, y Streptococcus. Los organismos evidenciaron en su mayoría, ulceras cutáneas, áreas hiperémicas, melanización corporal, hígado pálido y friable, vasculitis en hígado, congestión y hemorragia intestinal. De las bacterias evaluadas, la resistencia mayor fue obtenida con la oxitetraciclina (25 %) y fosfomicina (25 %), mientras que todas las cepas bacterianas fueron sensibles a florfenicol y enrofloxacina. En Guatemala solo se encuentran registrados legalmente los antibióticos oxitetraciclina y florfenicol para el uso en los cultivos de tilapia, por lo tanto, en el manejo de las enfermedades bacterianas se utilizan altas dosis de oxitetraciclina y florfenicol, lo cual evidencia que se está promoviendo la resistencia al antibiótico oxitetraciclina, primordialmente.
La resistencia a oxitetraciclina es una amenaza cada vez más grave para la industria acuícola, la cual requiere de acciones inmediatas. En los cultivos de tilapia en Guatemala se deben adoptar protocolos adecuados para el control y manejo de las enfermedades bacterianas, reduciendo el uso de los antibióticos, y promoviendo el uso adecuado de estos mismos. Deben establecerse estrategias adecuadas para mitigar la aparición y propagación de la resistencia a los antibióticos, a través de enfoques biológicos alternativos para la producción acuícola sostenible. El uso de plantas medicinales o sus extractos puede considerarse como estrategias respetuosas con el medio ambiente y con los organismos de cultivo, que consigue ayudar a prevenir y a controlar enfermedades de origen bacteriano, a fortalecer el sistema inmunológico de los organismos de cultivo y a favorecer las condiciones de la microbiota del ambiente acuático.
Aunque el uso de plantas medicinales ha mostrado resultados prometedores en otros países, se necesitan estudios adicionales, particularmente con el recurso vegetal disponible en Guatemala, para formular estrategias sobre la base de su composición química y mecanismo de acción.















