Introducción
La apicultura (crianza y manejo de abejas melíferas) es una actividad que se realiza en casi todo el mundo y que tiene una gran importancia ecológica y socioeconómica para la humanidad. De la apicultura se pueden obtener diferentes productos como: miel, polen, propóleos, jalea real, cera, veneno, entre otros. Pero aún más importante, las abejas polinizan una gran variedad de especies de plantas, incluyendo los árboles de los bosques y cultivos agrícolas (1). Se estima que cerca del 30% del alimento consumido por la población mundial es derivado de cultivos polinizados por abejas (2). Por tanto, la conservación de estos insectos es vital para la productividad de los sistemas agrícolas y la dinámica de los ecosistemas (3).
Las abejas A. mellifera son afectadas por distintos agentes etiológicos como virus, bacterias, hongos y parásitos. Se han descrito más de 35 enfermedades asociadas a esta especie de abeja y la mayoría ocasiona daños severos en la apicultura a nivel mundial (4). En Costa Rica, según (5), las enfermedades de mayor prevalencia en las colmenas de abejas melíferas son Loque Europeo (bacteria que afecta a las larvas), Varroosis (parasitosis causada por el ácaro Varroa destructor) y Nosemiasis o Nosemosis (provocada por el microsporidio Nosema spp.). La Nosemiasis fue descrita por primera vez en el año 1985 (6) y reportada como problema en el 2009 cuando se descubrieron elevados niveles de infección (7).
La Nosemiasis es considerada una de las enfermedades de mayor impacto económico a nivel mundial (8). Esta enfermedad puede ser causada por dos especies de microsporidios, Nosema apis y Nosema ceranae (Microspora, Nosematidae), los cuales forman esporas e infectan las células epiteliales del intestino de las abejas adultas y esto se ha traducido en daños cuantiosos a las colmenas (9, 10, 11). Estas esporas son ingeridas por las obreras jóvenes al realizar actividades de limpieza en panales contaminados, la reina se infecta a través de la jalea real de abejas nodrizas enfermas y los zánganos se contagian cuando reciben alimento contaminado de las obreras. Cuando las esporas son ingeridas por la abeja, pasan rápidamente al intestino, donde se multiplican de forma muy acelerada (8) provocando degeneración del tejido digestivo, desnutrición, comportamientos desorientados y muerte prematura (12, 13).
Una fuerte infección por Nosemiasis provoca disminución o no producción de jalea real, reducción en la postura de huevos; esto claramente disminuye la población de la colmena y por ende su pérdida (8). Según todo lo expuesto, esta enfermedad repercute de manera negativa sobre la diversidad de las especies vegetales, la productividad de los cultivos y la producción de miel y derivados (14). Por lo tanto, resulta de vital importancia contar con una herramienta moderna, eficiente, confiable y económica que permita diagnosticar el nivel de infección presente en una colmena.
Muestreo y captura de fotografías
Identificación del agente
Según la Organización Mundial de Sanidad Animal (OIE), en algunos casos muy severos las abejas muestran signos claros de infección (8, 13, 15). Sin embargo, la mayoría de las colonias no los muestran, ni siquiera cuando el nivel de infección es suficiente como para causar pérdidas significativas en la producción de miel y en la eficiencia de la polinización (16).
Por lo anterior, sólo se puede realizar un diagnóstico exacto de Nosemiasis mediante el examen microscópico del abdomen o ventrículo de la abeja adulta, ya sea por medios moleculares o por microscopía electrónica de transmisión (17).
Los exámenes microscópicos (a 400 aumentos) de los contenidos abdominales de las abejas afectadas revelarán la presencia de las esporas ovales de Nosema spp., que miden aproximadamente 5-7 × 3-4 μm, con un contorno oscuro. Nosema ceranae es ligeramente más pequeña, pero es difícil distinguir entre especies empleando la microscopía óptica, sobre todo porque pueden ocurrir infecciones mixtas (18).
Pruebas de laboratorio para el diagnóstico de Nosemiasis
Para determinar la presencia de Nosemiasis, se debe obtener una muestra de abejas de la entrada (piquera) de la colmena, lo cual garantiza abejas de edad avanzada en las que hay una mayor probabilidad de determinar la infección (9, 17). Existen varios métodos de laboratorio que son utilizados actualmente para el conteo de esporas y la determinación del nivel de infección por Nosemiasis de la colmena. En este artículo se exponen el método de Cantwell y Hemocitómetro pues fueron los utilizados en la etapa de captura y etiquetado de muestras (16, 18). Es importante mencionar que también es muy utilizado el método de reacción en cadena de la polimerasa (PCR), el cual consiste en pruebas de ADN para discriminar las esporas, más este no fue considerado debido a los objetivos y alcance de este artículo (puede verse (19, 20) para ampliar).
Método de Cantwell
En (21) se describe el método de Cantwell. El proceso seguido en este proyecto fue tomar 30 abejas adultas por muestra y colocarlas sobre un papel absorbente. Luego, se separaron los abdómenes de las abejas, los cuales fueron colocados en un mortero donde se maceraron. Se añadió 1.0 ml de agua destilada por cada abdomen, es decir 30 ml en total. El macerado se agitó por un minuto para homogenizar, luego se colocó una gota de la suspensión en un portaobjetos. Al final de este proceso los abdómenes macerados estuvieron listos para la toma de fotografías a través del microscopio a un aumento de 40x. En total se sometieron a este proceso un total de 75 muestras, lo que equivale a 2250 abejas adultas.
Método del Hemocitómetro
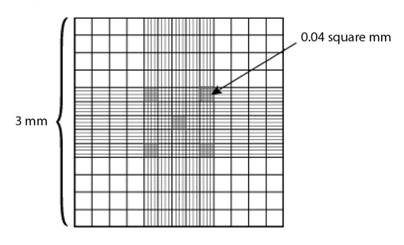
La segunda etapa consistió en contar el número de esporas en la muestra con el propósito de cuantificar el nivel de infección mediante el método del hemocitómetro. El líquido sobrenadante de la muestra se agitó de manera mecánica con una micropipeta para homogenizarlo, se tomó una alícuota de 10 µl, se colocó en el hemocitómetro por dos minutos con el fin de que las esporas sedimentaran. Según este método deben contarse todas las esporas enmarcadas por líneas dobles, incluyendo en el conteo a todas las que toquen las líneas dobles del lado izquierdo y superiores de cada bloque, pero no las que toquen las líneas dobles inferiores y las del lado derecho del bloque. Esto se hace únicamente en cinco de los veinticinco bloques centrales (ver figura 1), específicamente los cuatro bloques de las esquinas y el central.
Según (21), para determinar el nivel de infección de Nosemiasis en una colmena debe utilizarse la siguiente fórmula:
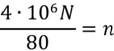
donde N es el número total de esporas contadas en los cinco bloques y n es el número de esporas por abeja que se contrasta en la tabla Jaycox (cuadro 1).
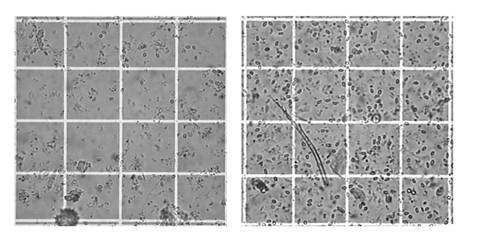
El conteo se registró con la toma de fotografías y la creación de carpetas con las fotos de los bloques utilizados, cada carpeta se identificó con una etiqueta con el nivel de infección diagnosticado por los expertos. En el muestreo total de 75 carpetas se tiene una colección de 375 fotografías.
Cuadro 1 Tabla de Jaycox para determinar el nivel de infección (NI) de la Nosemiasis en las abejas.
| NI | n |
| Muy leve | 104 - 106 |
| Leve | 106 - 5x106 |
| Moderado | 5x106- 107 |
| Semifuerte | 107 - 2x107 |
| Fuerte | Superior a 2x107 |
Fuente: Molina et al. 1990
Es imprescindible indicar que este procedimiento resultó laborioso, lento y demanda experticia de la persona que observa la muestra (por la apariencia de las esporas, estas pueden confundirse con levaduras, esporas fúngicas, cuerpos grasos o quistes de Malpighamoeba mellificae (Amebiasis) entre otras estructuras).
Sistema automático de cuantificación de infección
Con el objetivo de diagnosticar de manera confiable y eficiente, el nivel de infección por Nosemiasis en una colmena a partir de fotografías, se decidió replicar el proceso realizado por los expertos. Es decir, a partir de las fotos de los cinco bloques se realizó el conteo, luego se utilizó la tabla Jaycox para discriminar el nivel de infección. El proceso completo se dividió en tres etapas: toma de muestras y fotos, segmentación de las imágenes e implementación y ajuste de descriptores para el conteo de las formas que corresponden a esporas. Para validar los resultados se utilizaron imágenes independientes de las usadas en el entrenamiento y se contrastó la decisión del algoritmo con la previamente hecha por los expertos.
Segmentación de imagen
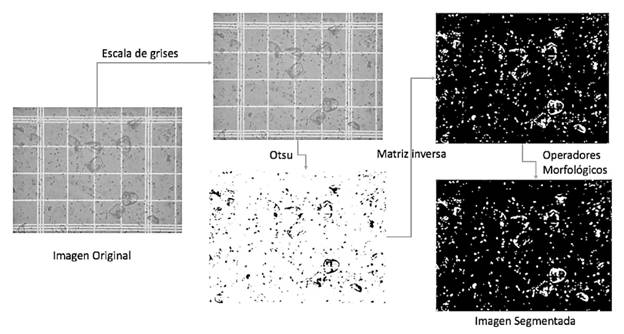
Las técnicas de segmentación de imágenes referidas a umbralización, histogramas, uso de color, binarización entre otras, son ampliamente tratadas en muchas investigaciones, por ejemplo (22, 23, 24, 25, 26). La figura 2 muestra dos fotografías típicas obtenidas del microscopio, en ellas se nota con claridad que hay mucho ruido (elementos presentes en la foto que no corresponden a esporas de Nosema), además, por la similitud de las esporas con el fondo se concluyó que el color no ets un buen discriminador.
El primer paso consistió en binarizar la imagen, para esto se utilizaron herramientas bastante conocidas (umbralización por Otsu, operadores morfológicos y detección de bordes) (23, 24, 25, 26, 27). Se tomó la decisión de ajustar el valor del umbral dado por Otsu de modo que fuese más sensible a valores oscuros, esto porque las esporas apenas se distinguen del color del fondo. Se utilizó la fórmula 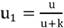 donde u es el valor dado por Otsu, u1 es el valor ajustado y k una constante obtenida de forma empírica.
donde u es el valor dado por Otsu, u1 es el valor ajustado y k una constante obtenida de forma empírica.
Es importante destacar que el ajuste realizado tiene como resultado la presencia de una mayor cantidad de formas que no corresponden a las esporas. Esto se consideró necesario porque en los ensayos donde se utilizó el valor umbral sugerido por Otsu directamente, una gran cantidad de esporas no se detectaron pues eran consideradas como fondo de la imagen. Se decidió utilizar elementos estructurales de tamaño grande en los procesos de dilatación con el objetivo de rescatar las esporas que por similitud con el fondo se detectaron como objetos pequeños.
Al final del proceso de segmentación se tiene una imagen binarizada donde las esporas de Nosema (y una cantidad enorme de ruido) se han identificado como regiones en color blanco. La siguiente tarea consistió en discriminar cuales de esas regiones blancas corresponde a esporas para cuantificar el nivel de infección.
Uso de descriptores para conteo de esporas
En esta etapa se implementaron tres descriptores del objeto espora: el tamaño, la forma elíptica y la circularidad. Es importante acotar que los descriptores fueron implementados de modo que fuesen invariantes a la escala y rotación de las imágenes.

Figura 4 a) Ilustración del cálculo de área por conteo de píxeles. b) Detección de bordes y cálculo de la longitud del lado del cuadrado.
Tamaño. El tamaño de las esporas fue considerado el principal descriptor puesto que las fotografías presentaron una cantidad importante de otros objetos mucho más grandes que estas. Aunque la discriminación por tamaño da la impresión de ser simple, en visión por computador no lo es. Existen dos cuestiones realmente importantes a considerar; el tamaño de la espora respecto de la imagen depende de la escala de esta y la forma en cómo se mide el tamaño en una imagen puesto que no hay sistemas de medidas tradicionales en ellas.
Se decidió utilizar el área (cantidad de píxeles) de las regiones en blanco como referencia de su tamaño (ver figura 4 a), sin embargo, aún se tenía el problema de la dependencia de la escala de imagen.
En (27) se hace referencia a los rangos del tamaño de los radios de las esporas de Nosema (5-7 x 3-4µm) y como puede apreciarse en la figura 1, también es conocida el área de cada cuadrado del hemocitómetro (0.04 mm2). Considerando los valores mínimos y máximos de la espora (28) y el hecho que esta tiene forma elipsoidal, se concluye entonces que el área de una espora oscila entre 15π µm2 y 28π µm2. Además, estos datos permitieron acotar el área de las esporas entre 0.015 l2 y 0.44 l2, donde l es el lado de uno de los cuadrados pequeños del hemocitómetro.
Con el uso de detección de bordes y puntos esquinas se calculó el tamaño, en píxeles, del lado de un cuadrado (es la distancia euclideana desde dos puntos esquina, como se aprecia en figura 4b), y puesto que la relación entre el área de las esporas y el lado del cuadrado debía mantenerse, se logró establecer un rango invariante a la escala para el tamaño de las esporas.
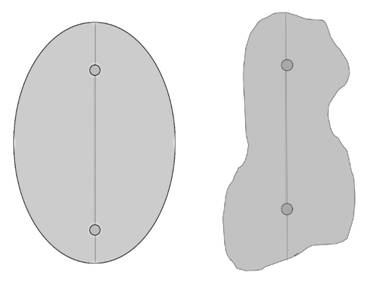
Excentricidad. Los referentes más exitosos en la detección de esporas de Nosema u otras formas similares (24, 25, 27, 29) tienen como principal referencia la forma de una elipse para describir los objetos de interés. Específicamente en (30), los autores se olvidan de cualquier otro objetivo y se dedican a contar elipses en las fotografías, esto bajo la premisa que las esporas tienen esa forma.
A pesar de la importante cantidad de referencias en las que se sugiere que las esporas de Nosema son elipsoidales, el proceso de segmentación que se había aplicado no permitió asumir este hecho tal cual. Es decir, las esporas ciertamente tienen forma elíptica (esto se utilizó en el cálculo de su área), pero al utilizar un umbral en el proceso de binarización y luego utilizar operadores morfológicos generó, en muchos casos, que las esporas se deformaran (ver figura 5). En el proceso se decidió sacrificar la forma en aras de una mejor detección; para los objetivos de la investigación es preferible detectar una zona “espora deforme” que tener una colección incompleta de elipses perfectas.
Dado este panorama no se abandonó la idea de utilizar la similitud de las esporas con las elipses, sino que se incorporó el uso de la excentricidad de la región detectada como descriptor de espora. Este descriptor es invariante a la escala y a la rotación, además permite incluir regiones con formas similares a elipses. Durante el proceso de experimentación se determinaron dos cotas para las cuales una región podría considerarse espora; fuera de ese rango la región detectada se desecha por ser muy redonda o bien muy alargada.
Compacidad. Con la implementación del tamaño y la excentricidad como descriptores de las regiones que serían contadas como esporas, solo resta incluir un descriptor que descarte formas un tanto cuadradas o con presencia de contornos irregulares. Se incluyó para esto el Factor de Compacidad (FC) o circularidad, el cual consiste en calcular un cociente entre el área y el cuadrado del perímetro (23, 24, 25, 31). Además, el FC se dividió por 4π de modo que la circularidad de un círculo fuese 1 y de esta forma tener una cota para discriminar regiones.
Para no viciar el efecto que tendría el uso del FC, en la etapa de preprocesado de la imagen, se utilizó un elemento estructural en forma de diamante, con esto tanto la erosión como la dilatación de las regiones se suavizaron. Para el cálculo del perímetro se contaron los pixeles de cada región que tuviese un vecino de color negro (ver figura 6), además, para calcular FC se utiliza información sobre el área que se tiene disponible.
Implementación del sistema para la cuantificación de la infección
Una vez que se tienen la totalidad de imágenes, las técnicas de segmentación identificadas y programadas y los descriptores debidamente ajustados e implementados, se procedió a la creación del sistema que identificaría el nivel de infección de la colmena. En la figura 7 puede apreciarse el esquema completo del sistema.
En una carpeta se dispone de las cinco fotografías que han de ser procesadas, estas corresponden a las mismas cuadrículas que se usan en el conteo manual. Cada imagen debe ser binarizada (Otsu, operadores morfológicos, ajuste de parámetros), luego esta imagen llena de regiones blancas pasa a una segunda etapa donde cada región debe ser caracterizada como espora de Nosema o no. En esta etapa primero se discriminan por tamaño, luego según el valor de su excentricidad y por último deben cumplir con las cotas de circularidad. Las regiones que cumplan con los tres requisitos son consideradas esporas (se pintaron de verde para poder visualizarlas fácilmente).
Con el conteo realizado en cada una de las cinco fotografías se procede a comparar con la tabla Jaycox y finalmente se hace el diagnóstico del nivel de infección.
Resultados y discusión
Como se ha mencionado anteriormente el proceso de validación se realizó con imágenes independientes a las utilizadas en la fase de entrenamiento. Cada grupo de fotos fue procesado y diagnosticado con el sistema automático, los resultados que se obtuvieron se contrastaron con las etiquetas (muy leve, leve, moderado, semifuerte, fuerte) que los expertos habían asignado de previo a cada grupo. De esta forma si el sistema acertaba la etiqueta del grupo se consideraba el ensayo como exitoso, de no ser así se consideraba un error del sistema. Puntualmente se logró una cuantificación de infección exitosa del 84%, esto significa que solamente en el 16% de los casos se asignó un nivel de infección que no correspondía de forma directa con lo que los expertos diagnosticaron.
Cuadro 2 Proporción de asignaciones automáticas exitosas según nivel de infección.
| Muy leve | Leve | Moderado | Semifuerte | Fuerte | |
| Muy leve | 0.8 | 0.2 | 0 | 0 | 0 |
| Leve | 0 | 1 | 0 | 0 | 0 |
| Moderado | 0 | 0.18 | 0.79 | 0.03 | 0 |
| Semifuerte | 0 | 0.12 | 0.08 | 0.8 | 0 |
| Fuerte | 0 | 0 | 0 | 0.6 | 0.4 |
Fuente: Elaboración propia.
El cálculo directo de los aciertos parece sugerir que el margen de error es muy alto, sin embargo, si se revisa el cuadro 2 se notará que la diagonal principal de la matriz y sus vecinos, contienen casi la totalidad de la muestra. Por ejemplo, en la tabla se indica que todos los casos etiquetados como moderados, el 79% se identificó como tal; mientras que 18% se ubicó en un nivel inferior (leve) y solo el 3% en el nivel superior semifuerte. Los niveles más alejados no tuvieron ninguna asignación. Este comportamiento indica que los índices de infección sugeridos por el sistema son exactos o al menos son cercanos a los etiquetados por los expertos.
Conclusiones y recomendaciones
La infección por Nosemiasis es una de las principales causas de la pérdida de colmenas o la disminución de su productividad. Existen métodos de laboratorio confiables para diagnosticar el nivel de infección y así poder tomar medias para mitigar los daños a la colmena, sin embargo, estos son lentos, caros y demandan la presencia de un experto.
Con el uso de herramientas tecnológicas, algoritmos para procesamiento de imágenes y la caracterización de las esporas de Nosema se ha logrado implementar un sistema robusto de diagnóstico de infección automático. Los descriptores tamaños, excentricidad y circularidad funcionaron de buena forma en la discriminación de objetos considerados esporas.
El porcentaje de éxito del sistema se ha considerado alto (84%) con relación a los consultados en las referencias, principalmente por la cantidad de fotografías utilizadas en el proceso de validación y por la exigencia puesta en la identificación de las esporas. En (30) y (31) se mencionan porcentajes más altos, sin embargo, en uno de ellos se utilizó únicamente 12 imágenes (con esta cantidad da la impresión de que solo se trata de un proceso de entrenamiento, de ser así un alto porcentaje es engañoso) y el otro utiliza imágenes donde las esporas están resaltadas del fondo y esto es un requisito muy alto para las fotografías.
Con la revisión detallada del algoritmo se ha concluido, además, que la incorporación de un filtrado en la etapa de preprocesado que permita resaltar las esporas mejoraría el proceso de detección y por tanto el conteo y diagnóstico de infección.