Descripción del género Brucella
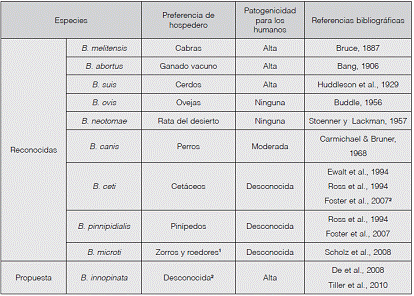
El género Brucella está constituido por bacterias cocobacilares, Gram negativas, aerobias, no mótiles, no esporuladas y no encapsuladas (Corbel y Brinley-Morgan, 1984). Filogenéticamente, se clasifican en la subdivisión a2 de las Proteobacterias (Moreno et al., 1990). Las Alfaproteobacterias agrupan patógenos de plantas y mamíferos, así como simbiontes y organismos de vida libre. Brucella se comporta como un patógeno zoonótico intracelular facultativo (Ficht, 2010). En el Cuadro 1 se describen las nueve especies reconocidas porel Subcomité Internacional de Taxonomía de Brucella. Existe una especie adicional que fue propuesta en 2008 pero no ha sido aceptada (Banai y Corbel, 2010).
El genoma de la mayoría de las brucelas posee dos cromosomas circulares, uno de de 2.1 Mb (cromosoma 1) y otro de 1.2 Mb (cromosoma 2). Entre ambos codifican aproximadamente 3500 genes (Michaux et al., 1993).
Características de la brucelosis
Las bacterias del género Brucella causan brucelosis, enfermedad endémica en países de Centroamérica y Suramérica, la cuenca mediterránea, el Norte y el Este de África, Medio Oriente y Asia (Corbel, 2006). La Organización Mundial de la Salud (OMS) considera la brucelosis como una de las siete zoonosis más extendidas a nivel mundial y menos priorizada por los sistemas de salud (http://www.who.int/zoonoses/neglected_zoonotic_diseases/en/index.html).
B. abortus presenta tropismo por el sistema reproductor del ganado vacuno. En los machos, la infección puede ocasionar epididimitis, orquitis o atrofia testicular; mientras que las hembras infectadas desarrollan abortos, retención de placenta, infertilidad y disminución en la producción de leche (Songer y Post, 2005). En placentas de rumiantes, la replicación bacteriana intracelular destruye los trofoblastos y las bacterias se diseminan a los tejidos y fluidos cercanos. La transmisión de animal a animal se da generalmente por ingestión de material contaminado después del aborto, ya que las bacterias son eliminadas a través del feto, las membranas fetales y las secreciones uterinas (Corbel, 2006). De esta manera, se reanuda el ciclo de infección, replicación intracelular y muerte de la célula hospedera (Anderson et al., 1986a; Anderson et al., 1986b).
En humanos, B. abortus causa una infección caracterizada por síntomas inespecíficos como malestar, anorexia y postración. En ausencia de tratamiento, estos síntomas pueden persistir por semanas o meses. Algunos signos reportados son fiebre intermitente, engrosamiento del hígado, bazo o nódulos linfáticos (Corbel, 2006). La diseminación y multiplicación de la bacteria en los nódulos linfáticos, bazo, hígado, médula ósea y órganos sexuales ocurre, principal pero no exclusivamente, por medio de los macrófagos (Ko y Splitter, 2003). La infección puede tornarse crónica y presentar complicaciones osteoarticulares, gastrointestinales, hepatobiliares, pulmonares, genitourinarias, cardiovasculares y neurológicas, entre otras consecuencias potencialmente mortales (Corbel, 2006). Las vías de transmisión al ser humano incluyen la ingestión de productos lácteos no pasteurizados, el contacto directo con animales infectados y la inhalación de aerosoles bacterianos (Godfroid et al., 2005).
En América Central, las pérdidas económicas por brucelosis ascienden a US$25 millones por año. Esta cifra resulta significativa si se considera que los países centroamericanos cuentan con bajos recursos y que la ganadería es una de las diez principales fuentes de ingresos de la región (Moreno, 2002). Además, el costo del tratamiento en humanos implica una inversión alta por parte de los sistemas de salud, ya que consiste en una terapia combinada de varios antibióticos durante periodos que van desde tres hasta ocho semanas (Akova et al., 1993; Ariza et al., 1985; FAO & WHO, 1986).
Patogénesis
En una gran parte de los patógenos bacterianos conocidos se han descrito factores de virulencia considerados clásicos, por ejemplo, cápsulas, fimbrias y exotoxinas. Sin embargo, la mayoría de estos factores no se han encontrado en Brucella. La virulencia de esta bacteria pareciera estar determinada por su habilidad para internalizarse, sobrevivir y replicarse dentro de fagocitos profesionales y no profesionales (Moreno y Moriyón, 2002; Moreno y Gorvel, 2004).
Brucella emplea distintas estrategias para invadir a sus células hospederas. En macrófagos, la cadena O del lipopolisacárido (LPS) liso de las bacterias no opsonizadas interactúa con moléculas receptoras ubicadas en la membrana de la célula hospedera, como ManR, entre otros. Luego es ingerida por medio de las balsas lipídicas. Generalmente ingresa a la célula hospedera mediante un mecanismo de fagocitosis tipo "zipper", el cual se caracteriza por la inducción de rearreglos moderados en la membrana y el citoesqueleto de la célula hospedera, aunque no se da un reclutamiento extensivo de actina (Porte et al., 2003; Moreno y Gorvel, 2004). Los macrófagos también pueden ingerir brucelas opsonizadas a través de receptores de Fc, de complemento o de fibronectina (Moreno y Gorvel, 2004). En células intestinales M y epiteliales, también se ha constatado el ingreso de Brucella por medio del mecanismo tipo "zipper". En células HeLa, Brucella se une a receptores desconocidos en la membrana y penetra por fagocitosis con reclutamiento moderado de actina y activación de GTPasas pequeñas como Cdc42, Rac y Rho. La bacteria se une en mayor cantidad y se internaliza de manera más eficiente cuando las células HeLa se tratan con el factor citotóxico necrotizante (CNF), el cual induce la formación de pliegues y fibras de estrés en la membrana de la célula hospedera (Guzmán-Verri et al., 2001).
En general, los fagocitos profesionales logran internalizar un alto número de bacterias debido a su naturaleza fagocítica (Celli et al., 2003). Sin embargo, la eficiencia de internalización en fagocitos no profesionales suele ser baja, lo que sugiere que no todas las células son permisivas, o bien que no todas las bacterias son capaces de unirse a fagocitos no profesionales (Sola-Landa et al., 1998). En macrófagos, la mayoría de las bacterias ingeridas son dirigidas a los fagolisosomas y muy pocas logran alcanzar el retículo endoplásmico, que es su nicho replicativo. Por el contrario, en células epiteliales, la mayoría de las bacterias ingeridas son dirigidas al retículo endoplásmico y en menor proporción a los lisosomas (Moreno y Gorvel, 2004).
Según el modelo propuesto por Celli et al. (2003), una vez que Brucella se internaliza en los macrófagos, escapa de la vía endocítica y se localiza en un compartimento no replicativo denominado BCV (Brucella-containing vacuole), el cual interactúa brevemente con los lisosomas, pero escapa de ellos y se transforma en una vacuola con pH ácido. Luego, interactúa de manera prolongada con el retículo endoplásmico e inclusive se fusiona de forma limitada con esta organela, lo que conlleva a la adquisición de algunas de sus propiedades como, por ejemplo, el pH neutro. Así, la BCV se transforma en una organela derivada del retículo endoplásmico que permite la replicación bacteriana.
Tanto en macrófagos como en células epiteliales, Brucella inhibe o retrasa la fusión de fagosomas y lisosomas (Ko y Splitter, 2003). Una vez que la bacteria alcanza su nicho replicativo, se encuentra protegida de antibióticos y factores bactericidas del hospedero como el complemento y los anticuerpos, lo cual facilita el establecimiento de una infección crónica (Roop et al., 2009). Luego de la replicación de la bacteria, la BCV se convierte en un compartimento con características autofágicas abreviado como aBCV. La formación de la aBCV requiere algunas proteínas que están implicadas en el inicio de la autofagia, pero es independiente de proteínas asociadas a la elongación. La aBCV es necesaria para completar el ciclo intracelular de Brucella y para la diseminación de la bacteria entre célula y célula, lo que demuestra que esta bacteria es capaz de apropiarse selectivamente de los complejos de iniciación de la autofagia para tomar control de la célula hospedera y promover la infección (Starr et al., 2012). Brucella también logra reconocer algunas señales de estrés (acídico o nutricional) a nivel intracelular para regular su propia expresión génica e incluso modificar algunas funciones de los fagocitos profesionales. Por ejemplo, puede inducir resistencia a la apoptosis en macrófagos, así como inhibir la capacidad de presentación de antígenos de las células dendríticas (Roop et al., 2009).
Determinantes bacterianos asociados a la patogenicidad
En patógenos Gram negativos, los componentes de la membrana externa pueden contribuir al establecimiento de la infección, pero también pueden convertirse en blancos para su neutralización por parte del hospedero (Cloeckaert et al., 2002). En cambio, el lipopolisacárido (LPS) de Brucella presenta particularidades que contribuyen a su supervivencia intracelular, algunas de ellas ancestrales como, por ejemplo, su baja actividad biológica (no induce signos de sepsis, fase de respuesta aguda, reclutamiento de neutrófilos ni producción significativa de citoquinas), y otras intrínsecas, como su resistencia a péptidos catiónicos bactericidas producidos por el hospedero. El LPS de Brucella tampoco activa la vía alterna del complemento (Moreno y Moriyón, 2002; Ko y Splitter, 2003; Roop et al., 2009).
Brucella presenta además una reducción en los patrones moleculares asociados a patógenos (PAMPs), los cuales normalmente se ubican en la envoltura celular y son reconocidos por los mecanismos antimicrobianos de la inmunidad innata del hospedero (Janeway et al., 2002; Lapaque et al., 2006; Moreno et al., 1981; Rasool et al., 1992; Barquero-Calvo et al., 2007).
Las bacterias del género Brucella presentan gran heterogeneidad en los componentes y en la topología de su membrana externa, lo cual se traduce en propiedades antigénicas y funcionales diferentes que influyen en la interacción con el hospedero. Las proteínas de membrana externa (OMP) de Brucella se clasifican en tres grupos de acuerdo con su peso molecular. El grupo 1 está constituido por OMP menos abundantes con pesos moleculares de 88-94 kDa. El grupo 2 consiste de porinas de 36-38 kDa, mientras que en el grupo 3 se incluyeron inicialmente proteínas de 25-27 kDa y posteriormente se incluyeron otras proteínas de 31-34 kDa. En B. abortus se encontró que las OMP de 25-27 kDa y las de 31-34 kDa están codificadas por los genes omp25 y omp31, respectivamente, por lo que estas OMP se designaron Omp25 y Omp31. La secuenciación de genomas de las diferentes especies de Brucella ha revelado que existe una familia de siete OMP constituida por Omp31b, Omp25b, Omp25c, Omp25d, Omp22, Omp25 y Omp31. En conjunto, esta familia se conoce como Omp25/Omp31 y se ha visto que sus miembros se encuentran fuertemente unidos al LPS en la membrana externa. La familia Omp25/Omp31 posee proteínas ortólogas en otras alfaproteobacterias como Rhizobium, Bartonella y Agrobacterium. Algunas de estas OMP ortólogas están implicadas en la interacción hospedero-patógeno (Cloeckaert et al., 2002).
Algunos patógenos Gram negativos presentan en su envoltura celular complejos multiproteicos conocidos como sistemas de secreción tipo IV (T4SS), que están implicados en la traslocación de efectores de virulencia a la célula hospedera (Backert y Meyer, 2006). En Brucella se ha descrito el T4SS VirB (Delrue et al., 2001) que está codificado por un operón de 12 genes. Los mutantes para el T4SS VirB no pueden controlar las interacciones de la BCV con el retículo endoplásmico y permanecen en compartimentos inmaduros que eventualmente se fusionan con los lisosomas. Por lo tanto, el T4SS VirB es esencial para el establecimiento de un nicho intracelular seguro que posibilite la supervivencia a largo plazo de la bacteria (Celli et al., 2003). Según Sieira y colaboradores (2004), la expresión del operón virB depende de la acidificación de la BCV y alcanza su máximo nivel a las 5 h post-infección pero luego es rápidamente apagada. de Jong y colaboradores (2008) describieron dos efectores traslocados por el T4SS VirB, denominados VceA y VceC, pero se desconoce su función. Marchesini y colaboradores (2011), por su parte, confirmaron por inmunofluorescencia el transporte de la proteína BPE123 a través del T4SS VirB. Esta es una proteína de 17 kDa altamente conservada en las especies de Brucella con genoma secuenciado. Además, presenta homología con proteínas descritas en otras alfaproteobacterias filogenéticamente relacionadas como Ochrobactrum y Bartonella. Sin embargo, BPE123 no es esencial para la virulencia de B. abortus. Los autores especulan que podría haber una redundancia en la función de los diferentes efectores secretados a través del T4SS VirB. Finalmente, de Barsy y colaboradores (2011) identificaron otro efector designado como RicA que es traslocado a través del T4SS VirB y que recluta la GTPasa Rab2 del hospedero hacia la vacuola que contiene a la bacteria durante el tráfico intracelular de Brucella. Recientemente, Dóhmer y colaboradores (2014) identificaron otro efector codificado en el locus Bab1_1492 y denominado SepA (Secreted Effector Protein A), el cual participa en etapas tempranas de la infección. Sin embargo, tampoco es esencial para la virulencia en ratones.
Regulación de la expresión de genes implicados en la virulencia
Generalmente, las bacterias están expuestas a condiciones ambientales cambiantes y para poder adaptarse deben alterar sus patrones de expresión génica. Los genes bacterianos están organizados como operones. Estos son un conjunto de genes que se encuentran bajo el control de un mismo promotor, por lo que su transcripción puede ser apagada o encendida de manera simultánea para dar una respuesta rápida a los cambios ambientales (Ralston, 2008). Los procariotas emplean distintos mecanismos para regular la expresión de sus genes. A nivel transcripcional destacan, por ejemplo, los sistemas de transducción de señales de dos componentes (TCS) y el quorum sensing (QS), entre otros.
En general, los TCS comprenden una proteína integral de membrana con actividad histidina quinasa, que constituye el componente sensor, y una proteína citoplasmática separada conocida como componente regulador. La proteína sensora tiene dos dominios, uno extracelular que reconoce señales externas, y otro citoplasmático, que transduce la señal al interior celular. Cuando una señal es reconocida por el dominio extracelular del componente sensor, se produce un cambio conformacional en el mismo, lo que cual desencadena una autofosforilación del dominio citoplasmático en un residuo conservado de histidina. El sensor fosforilado interactúa con su proteína contraparte reguladora, estimulando la fosforilación de residuos de aspartato en su dominio receptor. Este, una vez fosforilado, puede unirse al ADN mediante su dominio efector regulando la transcripción de genes blanco (Dale y Park, 2004; López-Goñi et al., 2004). Se estima que en Brucella podrían existir alrededor de 15 TCS (Lavín et al., 2010).
El TCS BvrRS está constituido por BvrS y BvrR. La primera es una proteína sensora de 601 aminoácidos que está asociada a la membrana interna. Su extremo amino-terminal tiene dos dominios transmembrana y una región periplásmica, que son importantes para el reconocimiento de señales ambientales. Su extremo carboxilo terminal tiene un dominio citoplasmático de dimerización y autofosforilación, así como un dominio con actividad ATPasa. BvrS tiene secuencias altamente conservadas que son típicas de las proteínas sensoras de los TCS. La proteína BvrR, por su parte, contiene 237 aminoácidos y pertenece a la familia de reguladores génicos OmpR. Tiene un dominio regulador conservado en el extremo amino terminal y un dominio efector en el extremo carboxilo. En el dominio regulador hay presencia de residuos de aspartato y lisina altamente conservados que interactúan con grupos fosfato. El dominio efector tiene un motivo de unión a ADN (Sola-Landa et al., 1998; López-Goñi et al., 2002).
Sola-Landa y colaboradores (1998) realizaron una mutagénesis por transposición insertando el Tn5 en el genoma de B. abortus y posteriormente seleccionando mutantes sensibles a péptidos catiónicos que afectan la envoltura celular, como la polimixina B. Solo obtuvieron dos mutantes con estas características, uno para bvrR (bvrR::Tn5) y otro para bvrS (bvrS::Tn5). En ambos mutantes, el Tn5 interrumpió el marco de lectura de cada gen. Los dos mutantes han sido utilizados en estudios previos sobre el papel del TCS BvrR/BvrS en la virulencia de B. abortus, determinándose que ambos, en comparación con la cepa silvestre, aparte de ser sensibles a péptidos bactericidas, tienen mayor permeabilidad a surfactantes, invasión celular reducida, tráfico intracelular deficiente y ausencia de virulencia en ratones (Guzmán-Verri et al., 2002; Sola-Landa et al., 1998). Los mutantes bvrR::Jn5 y bvrS::Tn5 no presentan la proteína Omp25 (Guzmán-Verri et al., 2002) y expresan bajos niveles de las proteínas VirB1, VirB5, VirB8 y VirB9 (Martínez-Núñez et al., 2010). Un estudio de proteómica por espectrometría de masas con fragmentos de membrana externa liberados espontáneamente y recuperados a partir de sobrenadantes de cultivos bacterianos en fase de crecimiento exponencial, señaló que los mutantes por transposición expresan 167 proteínas de forma diferencial en comparación con la cepa silvestre y con la cepa bvrR::Jn5 complementada con un plásmido que expresa BvrR. 25 de las proteínas diferencialmente expresadas corresponden a OMP. La expresión de aproximadamente la mitad de las OMP disminuyó, mientras que la otra mitad aumentó. Entre las OMP disminuidas se encuentran cinco proteínas de la familia Omp3 (que incluye Omp25), mientras que la expresión de cinco lipoproteínas se incrementó. También hubo una expresión diferencial de 60 proteínas periplásmicas, de las cuales el 80% aumentó su expresión en al menos uno de los dos mutantes por transposición. Las proteínas periplásmicas son importantes para la captura y transporte de nutrientes, por lo que se cree que el incremento en las mismas está relacionado con una respuesta de los mutantes ante un posible estrés nutricional debido a la falta de algunas OMP (Lamontagne et al., 2007). Por otra parte, un análisis de transcriptómica reveló que el mutante bvrR::Tn5 presenta, en comparación con la cepa silvestre, una expresión diferencial en 127 genes, entre ellos genes para OMP, lipoproteínas, biosíntesis de LPS y ácidos grasos, respuesta a estrés, flagelos, sistemas de transporte, metabolismo del carbono y desnitrificación, así como otros reguladores transcripcionales (Viadas et al., 2010). Estos datos, junto con los de proteómica, sugieren que el TCS BvrR/BvrS es un regulador global muy importante para la transición entre la vida extra e intracelular.
Cabe mencionar que el TCS BvrR/BvrS está altamente conservado en el género Brucella y en otras alfaproteobacterias filogenéticamente relacionadas. BvrR tiene 76-86% de similitud con BatR de Bartonella bacilliformis y con ChvI presente en Mesorhizobium loti, Sinorhizobium meliloti y Agrobacterium tumefaciens. BvrS, por su parte, tiene 59-68% de similitud con BatS de B. bacilliformis, ChvG de A. tumefaciens y ExoS de M. loti y S. meliloti (Cheng & Walker, 1998). En la bacteria fitopatógena A. tumefaciens, el TCS ChvI/ChvG es esencial para la virulencia. Los mutantes en este sistema no inducen la formación de tumores en el tejido vegetal, son sensibles a detergentes, antibióticos y pH ácido y presentan alteraciones en la permeabilidad de su envoltura externa (Charles y Nester, 1993; Mantis y Winans, 1993). En el endosimbionte de plantas S. meliloti, el TCS ChvI/ExoS está implicado en el control de la interacción entre la bacteria y la célula hospedera, la producción de succinoglicano, la movilidad flagelar, la formación de biofilms, la utilización de nutrientes y la simbiosis (Chen et al., 2008).
El TCS bvrR/bvRS afecta positivamente la transcripción de un regulador génico de tipo QS similar a LuxR denominado VjbR (Vacuolar Jacking Brucella Regulator) (Martínez-Núñez et al., 2010). El QS es un mecanismo de comunicación celular para sincronizar la expresión génica en una población bacteriana. Consiste en la liberación al medio externo de pequeñas moléculas autoinductoras. Conforme aumenta la densidad poblacional, se incrementa la concentración extracelular del autoinductor hasta alcanzar un umbral determinado. En este punto la molécula señal es reconocida por receptores y, como consecuencia, se activan o reprimen factores de transcripción citoplasmáticos que regulan la expresión génica de forma coordinada en la población bacteriana (Bassler, 1999).
Los mutantes en vjbR presentan un fenotipo atenuado en cultivos celulares (macrófagos y células HeLa) y en ratones infectados experimentalmente (Delrue et al., 2005). Además, presentan cambios en la superficie celular (Uzureau et al., 2007). En B. melitensis, Vjbr regula la expresión de algunas OMP implicadas en la virulencia (Uzureau et al., 2010; de Jong et al., 2008). En B. abortus, VjbR activa directamente la transcripción del T4SS VirB mediante unión al promotor del operón virB y a la región intergénica entre virBI y virB2. También activa la transcripción de vceA y vceB que codifican para dos efectores del T4SS VirB (de Jong et al., 2008). Por último, está implicado en la regulación de genes flagelares (Léonard et al., 2007).
Además de VjbR, en Brucella se ha reportado otro regulador de QS, denominado BlxR (Brucella LuxR-like Regulator) (BlxR también es conocido como BabR) (Rambow-Larsen et al., 2008). VjbR activa la transcripción de blxR y, por el contrario, BlxR reprime la expresión de vjbR. Además existen 27 genes blanco regulados por ambos mecanismos de QS. Para el 55% de ellos, los dos reguladores actúan de forma opuesta. Según la etapa de la infección, pareciera existir una interregulación entre VjbR y BlxR (Rambow-Larsen et al., 2008; Uzureau et al., 2010). Se cree que VjbR actúa al inicio de la infección, posibilitando la adaptación temprana a las condiciones de estrés que predominan en la BCV, disminuyendo el metabolismo básico (para evitar la replicación bacteriana mientras la vacuola madura) y promoviendo el ensamblaje del T4SS, que es importante para alcanzar el nicho replicativo. BlxR, por su parte, actúa en etapas posteriores de la infección para reactivar el metabolismo y permitir la replicación (Uzureau et al., 2010). Lo anterior sugiere que, aunque la función de ambos reguladores no es redundante, los dos participan en una vía en común que regula la expresión de procesos importantes para la vida intracelular de la bacteria (Rambow-Larsen et al., 2008).
En la regulación del operón virB también participan otros factores de transcripción como IHF (Integration Host Factor), HutC (Histidine UTilization pathway) y MdrA (MarR-like sodium deoxicholate-responsive activator). Los dos primeros se unen al promotor del operón virB, aumentando la expresión del mismo (Arocena et al., 2010; Sieira et al., 2010). Sieira y colaboradores (2012) mencionan que IHF es una proteína asociada al nucleoide y HutC es un regulador de la familia GntR que relaciona la expresión del operón virB con el catabolismo de histidina. Por otro lado, MdrA es un regulador de la familia de factores de transcripción MarR, que regula genes de virulencia, genes implicados en el catabolismo de compuestos aromáticos y genes de respuesta a estrés (Wilkinson y Grove, 2006). MdrA se une a dos sitios diferentes en el promotor del operón virB y regula su expresión de acuerdo con la fase de crecimiento. La función de MdrA es redundante con respecto a la de HutC. Además, compite con IHF y HutC por la unión al promotor del operón virB. Como es común en la familia de reguladores MarR, la presencia de un ligando disocia al regulador de su sitio de unión. En el caso de MdrA, el ligando identificado es el deoxicolato de sodio, lo que indica que in vivo MdrA podría responder a señales ambientales (Sieira et al., 2010).
Conclusión
Más que de factores discretos, la virulencia de B. abortus depende de complejas redes de regulación génica que afectan principalmente la envoltura celular, así como el metabolismo. Algunos mecanismos de regulación génica de la virulencia son redundantes, mientras que otros son complementarios ya que actúan en diferentes etapas de la infección. El TCS BvrR/ BvrS actúa como un regulador global que afecta la expresión de una gran variedad de genes y factores de transcripción importantes para la transición entre la vida extra e intracelular. Lo mismo pareciera ocurrir en otras alfaproteobacterias que poseen sistemas ortólogos al TCS BvrR/BvrS como A. tumefaciens y S. meliloti. Por este motivo, el estudio detallado del TCS BvrR/BvrS podría considerarse como un modelo para comprender mejor los fenómenos de parasitismo y endosimbiosis de las alfaproteobacterias. En el ITCR se continúa investigando sobre este tema, en conjunto con el Programa de Investigación en Enfermedades Tropicales (PIET) la Escuela de Medicina de la Universidad Nacional y el Centro de Investigación en Enfermedades Tropicales (CIET) de la Facultad de Microbiología de la Universidad de Costa Rica. Se espera que en un futuro el conocimiento generado pueda contribuir a contar con mejores herramientas terapéuticas y preventivas.