Introducción
Las sociedades humanas son impulsadas por la información en forma de datos. Las disciplinas científicas, incluidas la agronomía, la biología, la ecología, la microbiología, la química y la medicina, se benefician del rápido crecimiento de tecnologías que producen y manejan cantidades masivas de datos. Esto permite la toma de decisiones más precisas y acertadas acorde a la realidad que cada sociedad vive.
Un ejemplo claro de esto es la medicina personalizada o de precisión que permite realizar diagnósticos basados en la información propia de cada paciente, como la genética. Esto facilita la prescripción de tratamientos médicos hechos a la medida de la condición propia de cada paciente. También se pueden integrar diferentes niveles moleculares a estudios GWAS (Genome Wide Association Studies) para comprender mejor las enfermedades humanas, o incluso asistirse de estrategias como inteligencia artificial para describir padecimientos con modelos descriptivos y predictivos. En el futuro, podría ser posible modificar genes defectuosos con técnicas como CRISPR-Cas9 (Razzouk, 2018), o estrategias basadas en ARN (ácido ribonucleico) de interferencia.
Otro ejemplo actual ilustrado por la pandemia del SARS-CoV-2, es la relevancia de la vigilancia genómica global de patógenos, la cual permite una respuesta oportuna para el control de la diseminación de la enfermedad, o quizás incluso prevenirla por completo (Gardy & Loman, 2017). Adicionalmente, la información generada de la secuenciación del genoma del SARS-CoV-2, permitió un diseño rápido y dirigido de vacunas sin precedentes (Kyriakidis et al., 2021).
En el tema de bioprospección, con las tecnologías de secuenciación y análisis de datos se pueden describir comunidades enteras de microorganismos. De estas secuencias se pueden identificar los genes de esos organismos para encontrar moléculas con potencial aplicación biotecnológica y por ende económico (Coutinho et al., 2018).
Costa Rica tiene ejemplos relevantes de contribuciones y aplicaciones de la bioinformática. Entre ellas están el retrato de la riqueza de la biodiversidad nacional, la estructura genética de la población humana según las genealogías registradas en el país, y las detalladas caracterizaciones fenotípicas de venenos de serpientes (Campos-Sánchez et al., 2013; CBOL Plant Working Group, 2009; Lomonte et al., 2014; Lomonte & Calvete, 2017; Segura-Wang et al., 2010; Suárez-Esquivel et al., 2017a). La participación en esfuerzos globales como el proyecto “International Barcoding of Life - iBOL” y la secuenciación del genoma completo (WGS) de familias con esquizofrenia o trastorno bipolar también reflejan contribuciones importantes en el avance de la generación y la investigación basada en cantidades masivas de datos de Costa Rica (Glahn et al., 2019; Shokralla et al., 2015).
Durante los últimos diez años, el país ha registrado una serie de eventos que reflejan el aumento de interés y capacidad de realizar investigación en el campo. Uno de ellos es la instalación de seis instrumentos Illumina de secuenciación en instituciones tanto públicas como privadas. En recursos de computación, existe al menos un clúster computacional moderno con libre acceso para investigación (Kabré del Colaboratorio Nacional de Computación Avanzada - CNCA). En el área académica, se creó en el 2011 el Programa de Maestría en Bioinformática y Biología de Sistemas (MBBS) de la Universidad de Costa Rica, se han organizado múltiples cursos internacionales entre ellos el de Genómica y Vigilancia Epidemiológica de Patógenos Bacterianos en Costa Rica desde el 2013. Además, se han organizado sesiones de Bioinformática y Biología de Sistemas como parte de las conferencias internacionales IWOBI 2018 (IEEE International Work Conference on Bioinspired Intelligence), CARLA 2019 (Latin America High Performance Computing Conference) e IWOBI 2020. A nivel nacional, el Consejo Técnico en Bioinformática Clínica (CTBC) del Ministerio de Salud ha organizado dos Jornadas en Bioinformática Clínica (2019 y 2020) con participación profesional y estudiantil.
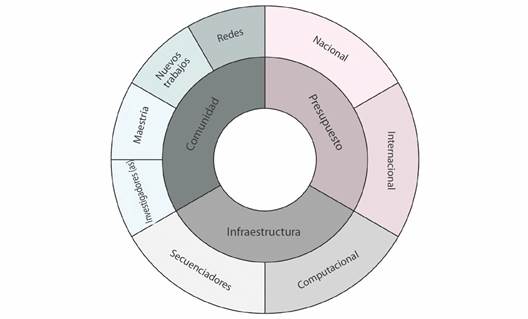
En este trabajo se recopila y presenta evidencia de la evolución de la bioinformática en Costa Rica desde el 2010. Para esto se usaron datos de la literatura científica, se realizaron búsquedas en bases de datos de secuencias y páginas en línea, y entrevistas a profesionales y docentes relacionados con el campo a nivel nacional. Los datos indican un avance importante en esta disciplina. Además, con este trasfondo, se identificaron debilidades y fortalezas para proponer acciones a futuro. Los autores sugieren acciones desde la perspectiva científica para impulsar el crecimiento de la investigación en genómica y bioinformática con potencial impacto social y económico. Esto dependerá de la capacidad de fortalecer la comunidad, de conseguir fondos de investigación y del desarrollo de una infraestructura de investigación bien articulada (Fig. 1).
Materiales y métodos
El objetivo principal de esta revisión es evaluar el progreso de la investigación y el ambiente nacional con respecto a la bioinformática en Costa Rica en los últimos once años. Para esto se cuantificaron aspectos como el aumento en el número de publicaciones, número de secuencias depositadas en bases de datos y número de personas capacitadas. Además, se midieron los avances en términos de acceso a fondos de financiamiento e infraestructura computacional, colaboración en redes científicas y la legislación que regula el acceso y análisis de datos genómicos. Todas las búsquedas se realizaron del 15-23 de abril del 2021, abarcando los años 2010 hasta el 23 de abril del 2021. En resumen, el proceso de recolecta de información se dividió en tres etapas: búsqueda en bases de datos de artículos científicos, búsqueda en bases de datos de secuencias de nucleótidos y recolecta de información directamente de páginas web o personas ligadas a las actividades relacionadas con la bioinformática.
Búsqueda en bases de datos de artículos científicos: Se realizaron búsquedas en tres bases de datos: PubMed, Web of Science y IEEEXplore. Las dos primeras difieren en su formato de búsqueda, así como en el contenido de las bases de datos, aunque hay un traslape que no se cuantificó. IEEEXplore contiene artículos tipo “proceedings” de conferencias que no están presentes en PubMed o Web of Science.
La búsqueda en PubMed se hizo de tres maneras, buscando el campo “title”, “title/abstract” o “allfields”. Este es un ejemplo del formato title: (((((((((((((((((((bioinformatic[Title]) OR (systems biology[Title])) OR (computational biology[Title])) OR (genome[Title])) OR (genomic[Title])) OR (transcriptomic[Title])) OR (transcriptome[Title])) OR (rna seq[Title])) OR (RNA-Seq[Title])) OR (microbiome[Title])) OR (microbiota[Title])) OR (metagenome[Title])) OR (metagenomic[Title])) OR (proteomic[Title])) OR (proteome[Title])) OR (microarray[Title])) OR (exome[Title])) OR (NGS[Title])) AND (Costa Rica[Affiliation])) AND ((“2010”[Date - Publication] : “3000”[Date - Publication])). Los resultados se exportaron en formato csv y se extrajo únicamente la fecha de publicación.
La búsqueda en Web of Science se realizó de tres formas, buscando el campo “title”, “topic” y “allfields”. Un ejemplo de búsqueda es el siguiente: TITLE: (bioinformatics) OR TITLE: (systems biology) OR TITLE: (computational biology) OR TITLE: (genome) OR TITLE: (genomic) OR TITLE: (transcriptomic) OR TITLE: (transcriptome) OR TITLE: (rnaseq) OR TITLE: (RNA-Seq) OR TITLE: (microbiome) OR TITLE: (microbiota) OR TITLE: (metagenome) OR TITLE: (metagenomic) OR TITLE: (proteomic) OR TITLE: (proteome) OR TITLE: (microarray) OR TITLE: (exome) OR TITLE: (NGS). Los resultados fueron luego filtrados en Refine results con la palabra Costa Rica y por organizaciones (ORGANIZATIONS-ENHANCED: (UNIVERSIDAD COSTA RICA OR UNIVERSIDAD NACIONAL COSTA RICA OR HOSP NACL NINOS DR CARLOS SAENZ HERRERA OR CATIE CENTRO AGRONOMICO TROPICAL DE INVESTIGACION Y ENSENANZA OR FDN INCIENSA OR UNIVERSIDAD ESTATAL A DISTANCIA UNED, INST TECNOL DE COSTA RICA). Los resultados se exportaron con la opción “other file formats” usando la opción “Record content = Full record” y “File format = plain text”. De estos archivos se extrajo únicamente la fecha de publicación (identificado como PY).
En IEEEXplore la búsqueda incluyó dos palabras bioinformatics AND Costa Rica para todos los años que hubiera datos disponibles.
Adicionalmente, se realizó una nube de palabras con la herramienta en línea wordclouds (https://www.wordclouds.com/) usando los títulos de las publicaciones identificadas en la búsqueda de PubMed por título y resumen. Todas las palabras de los títulos se cambiaron a minúsculas para hacer el conteo.
Búsqueda en bases de datos de secuencias de nucleótidos: Para esta revisión se hicieron búsquedas en dos bases de datos: ENA (European Nucleotide Archive) y SRA (Sequence Read Archive). Cada base de datos tiene campos de búsqueda distintos y alberga tanto datos compartidos (redundantes) como únicos, así que no son completamente comparables.
Se realizó la búsqueda limitando la fuente geográfica a 19 países Latinoamericanos, uno cada vez (Argentina, Belice, Bolivia, Brasil, Chile, Colombia, Costa Rica, Cuba, Ecuador, El Salvador, Guatemala, Honduras, México, Nicaragua, Panamá, Paraguay, Perú, Uruguay y Venezuela). Las búsquedas se enfocaron en datos generados por cualquier tecnología de secuenciación de lecturas cortas y largas.
Se usó la interfaz web del ENA bajo la opción búsqueda avanzada y el tipo de datos “Raw reads”. La búsqueda tuvo el siguiente formato (reemplazando el nombre de país): (country = “Costa Rica”) AND (instrument_platform = “ABI_SOLID” OR instrument_platform = “BGISEQ” OR instrument_platform = “CAPILLARY” OR instrument_platform = “COMPLETE_GENOMICS” OR instrument_platform = “DNBSEQ” OR instrument_platform = “HELICOS” OR instrument_platform = “ILLUMINA” OR instrument_platform = “ION_TORRENT” OR instrument_platform = “LS454” OR instrument_platform = “OXFORD_NANOPORE” OR instrument_platform = “PACBIO_SMRT”). Los resultados se exportaron como un archivo TSV (columnas separadas por tabs) y se extrajo la información relevante con scripts de Unix. Los campos de búsqueda fueron: “first_created”, “instrument_platform”, y en el caso de Costa Rica también se extrajo el nombre de la institución que sometió las secuencias (“center_name”).
Una búsqueda similar se realizó en el SRA del NCBI, sin embargo, el formato de la búsqueda fue el siguiente: (Costa Rica) AND (“2010”[Publication Date]: “3000”[Publication Date]). Se repitió la búsqueda con el resto de 18 países mencionados anteriormente. Los datos se exportaron en formato csv con las opciones “file” y “format RunInfo”. Se usaron scripts para extraer los datos de “release date”, “center name” y “platform”.
Para ambas bases de datos, los datos se normalizaron por tamaño de la población usando el dato del 2020 según la página web Worldometers (https://www.worldometers.info/world-population/population-by-country/, accesada el 21 de abril del 2021).
Búsqueda de datos en páginas web o directamente con personas: Otra información que se presenta en este trabajo fue recabada de representantes de organizaciones, incluyendo la dirección de la Maestría en Bioinformática y Biología de Sistemas de la UCR (19 de abril 2021), CNCA (30 abril 2021), colaboradores del proyecto CABANA en Costa Rica (25 de abril 2021), colaboradores de las redes RedBioSea, RedBioAplicada y Rigatrop. También se obtuvo información de profesores de la UCR, UNA e ITCR relacionados con la enseñanza de la bioinformática en sus instituciones.
Se recabó información de páginas web de las redes SOIBio y BIOCANET, y de publicaciones de estas redes.
Resultados
Producto de la búsqueda y análisis de información de bases de datos de publicaciones y secuencias, páginas web de redes y entrevistas, se observa en general: un incremento en la producción científica y el entrenamiento de profesionales, el impacto positivo de la colaboración en redes, el acceso a infraestructura computacional gratuita y avances en la legislación para regular la investigación. También se evidenció un desestímulo en los últimos años en el financiamiento nacional para la ciencia en general, que podría afectar a la bioinformática directamente.
Producción científica: En la Fig. 2 se muestra el aumento en la producción científica a partir del 2014 en Costa Rica, según los datos recolectados en PubMed y Web of Science con 18 palabras clave. Esta tendencia creciente después del año 2014 se observa también en el número de registros en la base de datos ENA (Fig. 3), así como en todas las publicaciones científicas de Costa Rica registradas en la base de datos HIPATIA del Programa Estado de la Nación (https://hipatia.cr/dashboard/publicaciones-cientificas). Especialmente, en el 2020 se muestra un crecimiento acelerado de publicaciones, quizás siendo un preámbulo para un 2021 aún mayor. Entre ambas bases de datos hay un traslape de publicaciones que no se cuantificó, sin embargo, es evidente que PubMed captura mejor las búsquedas inclusive solo usando el título y resumen de los artículos. Esta realidad supera los resultados brindados por de las Rivas y colaboradores (Rivas et al., 2017), en un análisis de Latinoamérica enfocado en publicaciones científicas buscadas en Web of Science con tres palabras clave únicamente: bioinformatic, computational biology o biological database.
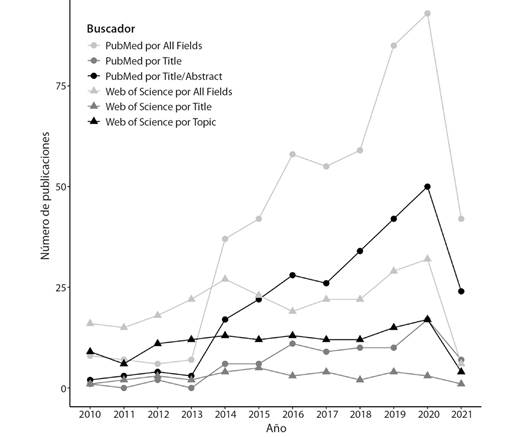
Fig. 2 Número de publicaciones científicas relacionadas a bioinformática desde el 2010 identificadas utilizando diversas palabras clave en las bases de datos PubMed y Web of Science. En estas publicaciones al menos un autor tiene afiliación de alguna institución de Costa Rica. Los datos del 2021 son los registrados hasta el 23 de abril. Fig. 2. Number of scientific publications related to bioinformatics since 2010 identified using various keywords in PubMed and Web of Science databases. In these publications, at least one author is affiliated with an institution in Costa Rica. Data for 2021 was obtained before April 23rd.

Fig. 3 Nube de palabras de 255 títulos de publicaciones identificadas en PubMed. Fig. 3. Word cloud of 255 publication titles identified in PubMed.
En la base de datos de IEEEXplore se encontraron adicionalmente 22 publicaciones de conferencias que no se encuentran reportadas en PubMed o Web of Science. Estas publicaciones corresponden a los años 2014 (1), 2016 (4), 2017 (1), 2018 (12), 2019 (3) y 2020 (1). En el 2018 se desarrolló la conferencia IWOBI en Costa Rica, por lo que hubo una importante participación de la comunidad nacional (ver sección Redes nacionales e internacionales).
En la representación en nube de las palabras de los 255 títulos de publicaciones identificadas en Pubmed se observan los temas más relevantes (Fig. 3). Entre ellos es clave la presencia de los estudios en venenos de serpientes; el estudio de bacterias de los géneros Brucella, Clostridium y Pseudomonas; estudios en cáncer, enfermedad bipolar y asma; y aplicaciones en genética, genómica, microbiomas y proteómica.
Por otra parte, en las bases de datos de secuencias se muestra un comportamiento similar a las publicaciones, con una tendencia al incremento a partir del 2015 y los primeros registros en el 2011 (Fig. 4). Estos registros son un reflejo de las actividades de investigación nacional, así como de colaboraciones internacionales en proyectos de investigación. En estas bases de datos es posible que haya secuencias no publicadas en artículos científicos, por lo que es importante rescatar esta información.
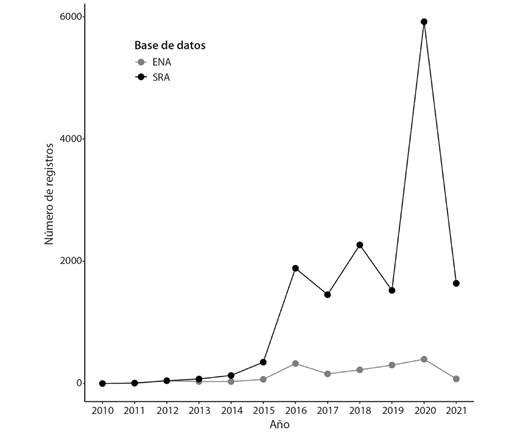
Fig. 4 Número de registros de Costa Rica en las bases de datos ENA y SRA buscados en el periodo 2010 hasta abril 2021. Los datos corresponden a secuencias provenientes de muestras de Costa Rica, pero solo una fracción fueron depositadas por instituciones nacionales. Fig. 4. Number of records for Costa Rica in the ENA and SRA databases for the period 2010 until April 2021. The sequences come from Costa Rican samples, but only a fraction was submitted by national institutions.
En la base de datos ENA se muestra un total 1 662 registros de secuencias provenientes de Costa Rica, sin embargo, esto no significa que la institución que depositó los datos sea costarricense. En detalle, del total de secuencias solo 43 fueron depositadas por la Universidad Nacional de Costa Rica y 4 por la Universidad de Costa Rica, es decir un 2.8 %. En una búsqueda similar realizada en el SRA se encontraron 15 297 registros desde el 2011 (Fig. 4). En este caso solo un 2.4 % de las secuencias fueron depositadas por instituciones costarricenses y corresponden a: 306 UCR, 45 UNA, cinco del CENIBIOT, nueve de una investigadora del ITCR y una del SENASA.
En el SRA, el mayor número de registros en este periodo 2010-2021 corresponde a genomas humanos con 6 590 records (43 %), principalmente del proyecto PRJNA295247 (6 580 genomas). En segundo lugar, para el mismo período, se depositaron 1 350 metagenomas humanos, de los cuales 1 345 son del proyecto PRJNA623584. Estos mismos dos proyectos son los responsables del aumento observado en el 2020 (5 919 records, Fig. 4) con un total de 3 395 registros, más otros tres proyectos asociados a biodiversidad ambiental (PRJEB42019 con 314 registros, PRJNA623020 con 582 registros y PRJNA530637 con 640 registros).
En comparación con otros 18 países latinoamericanos, la contribución de Costa Rica al registro público de datos de secuencias de nucleótidos es comparable a la de Colombia y Chile en el SRA (Fig. 5A) cuando se usan números absolutos, y es comparable a Nicaragua y Bolivia según el ENA (Fig. 5A). Sin embargo, cuando se normaliza por millón de habitantes en cada país (per cápita, tamaño de la población al 2021), se observa que la contribución de Costa Rica es mayor que todos los países excepto Belice (Fig. 5B) y comparable a la contribución de Panamá.
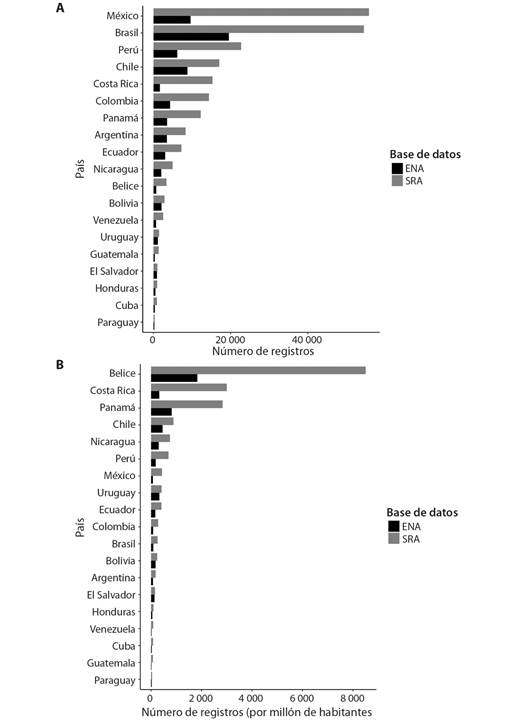
Fig. 5 Número de registros en las bases de datos ENA y SRA buscados por país y año (2010-2021). A. Clasificación de los países de América Latina según el número de registros totales encontrados en las bases de datos. B. Número de registros normalizados per cápita (por millón de habitantes en el 2020). Fig. 5. Number of records in the ENA and SRA databases searched by country and year (2010-2021). A. Classification of Latin American countries according to the number of total records found in the databases. B. Number of normalized records per capita (per million inhabitants for the year 2020).
Educación y entrenamiento: De los datos recolectados en el presente estudio, se observa que en los últimos cinco años se han dado múltiples eventos positivos en el área de formación profesional, incluyendo la participación de estudiantes en programas nacionales de maestría y pregrado, y la formación local de profesionales por medio de cursos presenciales y virtuales en bioinformática.
En el 2011, la Maestría en Bioinformática y Biología de Sistemas (MBBS) fue creada con el objetivo de proporcionar a la comunidad nacional recurso humano capacitado en bioinformática, particularmente en aplicaciones relacionadas a la salud humana. Desde entonces, 45 estudiantes han sido admitidos, de los cuales 12 abandonaron sus estudios de posgrado por motivos personales o porque el posgrado no satisfizo sus expectativas en algunos cursos. De los 33 restantes sólo cuatro se han graduado y el resto están realizando tesis o llevando cursos (comunicación personal de la directora del programa). La respuesta a por qué la tasa de graduación es tan baja (12 %) requiere un análisis más profundo. Sin embargo, la dirección de la maestría menciona varias circunstancias que les comunicaron los estudiantes. Una de ellas es que, al ser una maestría autofinanciada, los estudiantes deben trabajar para pagar sus estudios, lo que limita su dedicación a la investigación y retrasa su avance. Otra razón es la dificultad de encontrar tutor y tema de tesis, especialmente si son estudiantes ajenos al ambiente de la universidad.
Otro cambio positivo en los últimos cinco años es la oferta de clases de bioinformática básica para estudiantes de pregrado. Estas clases son ofrecidas por las Escuelas de Biología de la Universidad Nacional (UNA) y la Universidad de Costa Rica (UCR), la Facultad de Microbiología de la UCR y el programa de Ingeniería Biotecnológica del Instituto Tecnológico de Costa Rica (ITCR). Un producto indirecto de estas actividades es la creación de la edición Costa Rica del Grupo Regional de Estudiantes de la Sociedad Internacional de Biología Computacional (ISCB-CR) en el 2018 (Shome et al., 2019). Esta formación básica en bioinformática tendrá implicaciones importantes para las nuevas generaciones de científicos que combinan experimentos de laboratorio y biología computacional en sus primeras etapas.
Las oportunidades de capacitación están aumentando tanto en la demanda como en la variedad de temas en comparación con informes anteriores (Orozco et al., 2013). En los últimos cinco años se impartieron más de 12 talleres presenciales en la UCR, capacitando a más de 150 personas en temas como ensamblaje y anotación de genomas, análisis de genes 16S con Mothur (Schloss et al., 2009) y QIIME (Caporaso et al., 2010), Python, R, GATK (McKenna et al., 2010) y COPASI (Hoops et al., 2006). El CNCA, por su parte, tiene un portafolio de capacitación que incluye siete cursos basados en Python, R, visualización, machine learning, programación y análisis de datos (https://cnca.cenat.ac.cr/en/#intro). Otro ejemplo de gran impacto, es el curso internacional sobre Genómica y Vigilancia Epidemiológica de Patógenos Bacterianos que se imparte anualmente desde el 2013. Este curso está patrocinado por el Wellcome Trust Advanced Courses en colaboración con socios de Costa Rica, América Latina y el Reino Unido. Su objetivo es proporcionar capacitación en las habilidades necesarias para generar e interpretar datos NGS (Secuenciación de nueva generación) en un entorno de salud pública de América Latina. Hasta el momento, se han capacitado alrededor de 130 participantes del sector académico y de salud pública de América Latina. Esta oportunidad también ha ayudado a los investigadores costarricenses a avanzar en su trabajo colaborativo, lo que resultó en múltiples publicaciones (Alvarez et al., 2020; Baker et al., 2017; Chinen et al., 2016; Quesada-Gómez et al., 2015; Ramírez-Vargas et al., 2017; Suárez-Esquivel et al., 2017a; Suárez-Esquivel et al., 2017b; Suárez-Esquivel et al., 2020).
La sociedad IEEE en Medicina y Biología (EMB) organizó varias actividades de formación y un congreso en Biocomputación - el IWOBI 2018 (http://iwobi.ulpgc.es/2018/). En este evento se brindó la oportunidad a los estudiantes nacionales (10 estudiantes de la MBBS) para presentar sus resultados de investigación en un evento internacional, de lo cual también se generaron doce publicaciones documentadas en IEEEXplore.
Los cursos en línea también son una alternativa, como ejemplo están los recursos de Train-online ofrecidos por EMBL-EBI (Laboratorio Europeo de Biología Molecular - Instituto Europeo de Bioinformática), los cursos de FutureLearn de Wellcome Genome Campus y Sanger Institute, cursos de Coursera o Harvard, y más recientemente los tutoriales de eLearning del proyecto CABANA ofrecidos en inglés y español para la comunidad Latinoamericana (https://cabana.online/elearning).
Redes nacionales e internacionales: Durante los últimos siete años se crearon varias redes dentro de instituciones académicas (por ejemplo, Bio-SEA, Rigatrop, RedBioAplicada, RICC) para promover la formación de estudiantes y profesionales, así como brindar oportunidades de colaboración. Como ejemplo, la RedBioAplicada de la UCR se enfoca en la investigación y educación en bioinformática y bioestadística. Actualmente con 118 miembros (profesionales y estudiantes), coordinó cinco acciones formativas con colaboradores nacionales e internacionales, y continúa promoviendo oportunidades de formación. Por otra parte, estudiantes de diversas instituciones organizaron la edición ISCB-CR, abriendo puertas de interacción con una de las sociedades de biología computacional más grandes del mundo (Shome et al., 2019). Las consecuencias de esta interacción pueden ser relevantes para las nuevas generaciones en Costa Rica.
En el 2018 (La Gaceta Alcance N.36), el Ministerio de Salud conformó el Consejo Técnico en Bioinformática Clínica (CTBC) con miembros de universidades, hospitales e instituciones nacionales (Ministerio de Salud, 2021). Este es el único esfuerzo a nivel gubernamental para promover el uso de la bioinformática en la salud. Parte de sus objetivos son promover el desarrollo tecnológico y la innovación en salud; fomentar la investigación, la actividad académica y la formación profesional; propiciar el desarrollo de aplicaciones genómicas, entre otras. Entre sus actividades más representativas se reporta la organización de las Jornadas en Bioinformática Clínica en los años 2019 y 2020, agrupando a múltiples profesionales y estudiantes reflejando el trabajo en este tema en el país.
A nivel internacional, Costa Rica ha formado parte de colaboraciones con la región Latinoamericana en grupos como la Sociedad Iberoamérica de Bioinformática (SoIBio) (de las Rivas et al., 2017) (http://www.soibio.org/es) y la Red Centroamericana de Bioinformática y Biocomputación Molecular (BioCANET) (https://www.biocanet.org/). En el 2017, entró en el consorcio CABANA (Capacitación en Bioinformática para América Latina, http://www.cabana.online/), para fortalecer la capacidad de investigación en bioinformática en toda América Latina, en tres áreas de desafío: enfermedades contagiosas, protección de la biodiversidad y producción sostenible de alimentos.
CABANA está financiado por el Fondo de Investigación de Desafíos Globales (GCRF, parte del Presupuesto de Ayuda del Reino Unido). Costa Rica, como parte del consorcio, está aprovechando esta oportunidad con la formación de profesores e investigadores a través de pasantías de investigación, programas de formación para entrenadores y talleres. Al menos, una profesora pasó seis meses en EMBL-EBI (2018-2019) desarrollando cursos de capacitación en línea (e-Learning), y tres profesores han participado en el curso Train-the-trainer para diseñar e impartir capacitación en bioinformática en América Latina. Otros dos profesores y un estudiante de maestría participaron en capacitaciones internacionales en bioinformática en México, Chile y Colombia. Actualmente, se están desarrollando proyectos regionales relacionados con la genómica de la enfermedad de la roya en café, detección de diversidad genética en café y la genómica del SARS-CoV-2 en Latinoamérica, en los cuales Costa Rica es un colaborador activo. Todo esto contribuirá al crecimiento de la comunidad investigadora de América Latina y Costa Rica.
Financiamiento para investigación: En el lado positivo, el número de las oportunidades de financiamiento aumentaron en el país hasta el 2019, así como el tamaño del aporte. Por ejemplo, el Ministerio de Ciencia, Tecnología y Telecomunicaciones de Costa Rica (MICITT) y el Consejo de Investigaciones Científicas (CONICIT) han realizado convocatorias anuales para proyectos de investigación en diversos campos otorgando fondos desde US$18-80k. La UCR creó fondos mayores (US$12.5-50k) para períodos más largos de ejecución que permitieron la inclusión de proyectos que involucran NGS y análisis de datos desde el 2017. La Universidad Nacional (UNA) desde hace más de 15 años ha otorgado fondos de investigación a través del programa Fondos para el Desarrollo Institucional Académico, otorgando actualmente hasta US$70k para proyectos a ejecutar en un período de dos años.
En el lado negativo, desde el 2020 se ha dado una reducción del presupuesto de las universidades públicas (Castro, 2020) y del MICITT (MICITT, 2020), lo que tendrá un impacto directo en la generación de ciencia y por tanto en las investigaciones que apliquen la bioinformática. Estas medidas responden al problema de la crisis fiscal y mecanismos del gobierno para reducir gastos. Adicionalmente, la situación de la pandemia por el SARS-CoV-2 vino a agravar la situación actual.
En contraste, las oportunidades de financiación internacional son más y de aporte mayor. Existen fondos disponibles del NIH, ICGEB, EMBO, BBSRC, GCRF, NSF, Cancer UK, Horizon Europe, la fundación NVIDIA, Microsoft, Bill y Melinda Gates, entre otros. En muchos de estos casos la colaboración internacional es esencial, así como demostrar experiencia en la propuesta que se plantea.
Infraestructura para la investigación: Dos aspectos importantes de la investigación bioinformática son: i) la infraestructura donde se generan y almacenan los datos y ii) la infraestructura para el análisis de datos. En el 2015, se compró el primer instrumento MiSeq en el país con fines académicos; éste se encuentra en el CIBCM-UCR. Actualmente, hay siete secuenciadores Illumina instalados en el país en diversas instituciones para la vigilancia epidemiológica, investigación, diagnóstico genético y análisis forense. Estas instituciones son: CIBCM-UCR, CIHATA-UCR, SENASA, INCIENSA, Poder Judicial, Hospital Nacional de Niños y el Laboratorio de Docencia en Cáncer-UCR (acá está el único NextSeq). A pesar de tener una infraestructura abundante con respecto al tamaño de la población costarricense, estos equipos en su mayoría están subutilizados.
Los recursos computacionales son limitados pero eficientes y gratuitos. El CNCA es el principal laboratorio computacional financiado por el CONARE (Consejo Nacional de Rectores) que brinda servicios de computación a investigadores de todas las universidades públicas del país. Su clúster computacional llamado Kabre, tiene 46 nodos (32 de simulación, 2 de bioinformática, 6 de aprendizaje máquina y 6 de ciencias de datos). El RAM de los mismos está entre 16-1 024 GB. Los dos nodos de bioinformática tienen 36 núcleos (2 hilos por núcleo) de 3 GHz y 1 024 GB de RAM (https://kabre.cenat.ac.cr/userguide/). Aproximadamente, 195 de 427 usuarios trabajan en aplicaciones bioinformáticas (dato del coordinador del clúster). Kabre, con todos sus recursos, sigue siendo inadecuado para algunos programas que requieren una infraestructura y requisitos de memoria particulares, por ejemplo, Galaxy, SMRT y COPASI. Otros recursos computacionales alojados en la UCR incluyen al menos siete clústeres en funcionamiento, sin embargo, la mayoría de ellos solo son utilizados por uno o un pequeño grupo de investigadores, y el uso es en general muy bajo.
La computación en la nube es otra alternativa que incluye servicios de Amazon AWS, Microsoft Azure y Google Cloud, entre otros. Sin embargo, no es una opción prioritaria por la comunidad de investigadores, debido a limitaciones presupuestarias, falta de conocimiento y/o experiencia para su implementación.
Regulación ética: La bioinformática se vincula directamente con aspectos éticos principalmente desde dos perspectivas: derechos de autor y la gestión de datos biológicos. En Costa Rica, ambos aspectos están incorporados como parte de diferentes leyes, que a su vez responden a iniciativas globales para asegurar las mejores prácticas éticas.
Respecto a derechos de autor en bioinformática, algunos autores separan las regulaciones en función de tres dimensiones: (i) las secuencias de moléculas, (ii) las bases de datos y (iii) el software y el hardware (McBride, 2002; Shilpa, 2018). En nuestro país, la Ley N° 6 683 sobre Derechos de Autor y Derechos Conexos es la que regula estos aspectos, en un marco que aplica los derechos patrimoniales y morales a obras del campo literario, científico y artístico, que incluiría indirectamente la producción en bioinformática.
Respecto a la gestión de datos biológicos (recolección o generación, análisis, interpretación y uso), éstos son un eje fundamental en bioinformática. La evolución que ha tenido la bioinformática no sería posible sin las estrategias para proveer la gestión de datos masivos (Elissa et al., 2020), y por tanto, las regulaciones del uso de datos, en este caso biológicos, son también aplicables a la bioinformática. A nivel global existe un escenario ético amplio de discusión y regulación que involucra diferentes niveles de protección de los datos, los individuos y los estudios científicos. Esta protección se enmarca en la Declaración Universal de Derechos Humanos, la Declaración Universal sobre el Genoma Humano y los Derechos Humanos, la Declaración Universal de los Derechos del Animal, entre otros. En Costa Rica, la protección de los datos personales y datos genómicos se ejerce bajo las leyes nacionales Ley N° 5 395 General de Salud, Ley N° 8 968 de Protección de la Persona Frente al Tratamiento de sus Datos Personales, y el Reglamento del Registro de Datos de Perfiles de ADN para Identificación Humana, entre otras leyes y reglamentos conexos. Estos mismos aplican para la investigación con seres humanos, junto con la regulación específica dada por la Ley N° 9 234 Reguladora de Investigación Biomédica, y otros acuerdos internacionales. Finalmente, estudios científicos en animales y recursos de la biodiversidad están regulados bajo la Ley N° 7 451 Ley de Bienestar de los Animales y la Ley N° 7 788 de Biodiversidad, respectivamente, entre otras regulaciones.
Así, la gestión de datos biológicos para su uso en bioinformática implica una serie de consideraciones que indirectamente han sido ya incluidas en la regulación actual en el manejo de la información de las personas, el uso de animales y recursos de la biodiversidad en Costa Rica. Sin embargo, algunas de estas leyes y reglamentos responden a una generalidad que no siempre es aplicable al caso de la bioinformática, siendo una situación que se presenta en el resto del mundo (Elissa et al., 2020).
En 2020 Costa Rica ha iniciado su participación en el “Genomic Data Policy Framework and Ethical Tensions”, que busca resaltar la necesidad de políticas y legislación en el tema de la genómica con consideraciones éticas para los responsables políticos, los líderes empresariales, investigadores, pacientes y otros al tomar acciones que afecten o involucren la recolección y el uso de datos genéticos y genómicos humanos para investigación y uso clínico masivos (Elissa et al., 2020). Quizás el papel del CTBC del Ministerio de Salud será relevante en este tema en los años venideros.
Discusión
Este trabajo recaba evidencia de algunos componentes para medir el avance de la bioinformática en Costa Rica, que van desde la visión de la comunidad científica, la infraestructura disponible y el acceso al financiamiento (Fig. 1). Estos mismos componentes son referencia para definir acciones que impulsen el fortalecimiento de esta disciplina.
La búsqueda de publicaciones científicas y secuencias en bases de datos arrojó resultados que indican que la contribución del país ha ido en aumento a lo largo de los años. La razón de este aumento requiere un análisis detallado que integre información de las fuentes de financiamiento y colaboraciones en los artículos. Esto permitiría determinar si los fondos nacionales o internacionales han impulsado este progreso. Lo que sí fue evidente es que los organismos más estudiados en términos de secuencias depositadas son los humanos con 6 590 genomas y 1 350 metagenomas depositados (51.9 % del total), y un 10 % de metagenomas ambientales diversos, según el SRA. Sería importante medir el impacto de estas investigaciones en el ambiente nacional como un ejemplo a futuro.
Al poner en perspectiva la contribución en número de secuencias de Costa Rica con la región Latinoamericana, es evidente la actividad científica que se está realizando. Sin embargo, al evaluar las instituciones que depositan las secuencias, más de un 97 % corresponde a instituciones internacionales. La explicación de esto requiere un análisis más profundo, para determinar la razón por la cual información de organismos costarricenses aparece depositada por instituciones internacionales restando visibilidad a la comunidad científica nacional.
Del análisis de los títulos de las publicaciones científicas se pueden identificar las áreas de impacto de estas contribuciones. En este caso fue evidente que los venenos de serpientes, Clostridium, Pseudomonas, Brucella, asma, enfermedad bipolar, entre otros, predominaron en la nube de palabras. Sin embargo, un análisis que integre esta información con los nombres de los autores nacionales, los colaboradores y las fuentes de financiamiento enriquecería la discusión al permitir determinar el impacto agregado para la comunidad nacional (trabajo en progreso). Otro aspecto que se podría derivar del análisis de las publicaciones es el número de profesionales trabajando en bioinformática en el país, que actualmente es superior a los 40 (conteo informal realizado en este trabajo).
En el tema de educación y comunidad, hace 13 años, Moreno y colaboradores (Moreno et al., 2008) presentaron un ensayo sobre la contribución científica de Costa Rica a la biología computacional tomando como ejemplo tres institutos de investigación de renombre nacional: el Instituto Clodomiro Picado, el INBio - Instituto Nacional de Biodiversidad, cerrado en 2015) y el PIET - Programa de Investigación en Enfermedades Tropicales. En aquel entonces, las carreras científicas en las universidades públicas ignoraban los cursos computacionales, lo que obstaculizaba el interés de las nuevas generaciones por la biología computacional. Adicionalmente, se mencionaba el efecto de la “fuga de cerebros”, donde profesionales egresados en el extranjero no regresaban al país debido a las limitadas y menos competitivas oportunidades laborales. Sin embargo, este efecto se desmitificó en un ensayo de Moreno (2014) y en el análisis actual del Programa Estado de la Nación reportados en la base de datos HIPATIA (https://hipatia.cr/dashboard/diaspora-cientifica), que involucra un análisis de todas las áreas de la ciencia, tecnología e innovación.
En este trabajo se muestra un contraste con esta perspectiva luego de más de 10 años, siendo dos logros positivos el incluir cursos de bioinformática a nivel de pregrado y la creación de la MBBS. Sin embargo, no se cuantificó el regreso de profesionales luego de concluir sus estudios en el exterior que podría ser un dato relevante para la comunidad en bioinformática. Este dato se podría obtener en parte de la base de datos Ticotal de talento costarricense estudiando o trabajando en el exterior (http://ticotal.cr/conozca-acerca-de-ticotal.html#) y de la base de datos HIPATIA (https://hipatia.cr/dashboard/diaspora-cientifica).
Adicionalmente, algo que sí permanece es la falta de oportunidades laborales especializadas en análisis de datos biológicos. Esto en parte, dificulta la adopción rápida de esta ciencia en instituciones que la podrían estar aplicando (por ejemplo, el sistema de salud nacional).
Otra evidencia del avance de la bioinformática es la creación de redes nacionales y la integración de Costa Rica a redes internacionales. El impacto se ha observado en la coordinación de cursos de capacitación y desarrollo de colaboraciones para investigación. Esto reduce la anterior conceptualización de la bioinformática como una herramienta que era usada como parte de un tema médico-biológico central (el cual sigue siendo vigente en muchos casos), pero que también puede ser desarrollada como una entidad central en investigación. Esto es, una visión de investigación en “bioinformática pura”.
En el tema de infraestructura se evidenció el incremento de la capacidad de secuenciación nacional. Si bien esto podría representar un gran avance y una gran oportunidad, el impacto de esta iniciativa se ve contrarrestado por tres situaciones. Una es la falta de alternativas comerciales a los kits de preparación de bibliotecas de Illumina en el país. Aunque en el 2020 se empezó a ofertar la química Collibri (Thermo Fisher). En segundo lugar, está el alto costo de los reactivos de secuenciación, encarecidos en gran manera (~25 %) debido a los costos de importación. En tercer lugar, la carga de secuenciación es reducida lo que hace que el costo de cada muestra sea mayor, como consecuencia esto reduce la competitividad respecto a otras empresas que brindan servicios de secuenciación (ej. Psomagen o Novogene). Todo esto conduce a una producción de datos ineficiente, que es una de las razones por la que muchos de los instrumentos Illumina en Costa Rica están subutilizados (datos no mostrados). Muchos científicos prefieren enviar sus muestras al extranjero para secuenciar, resolviendo el problema del tiempo y los costos. A pesar de estas limitaciones, algunos investigadores aún secuencian en las instalaciones locales para tener control sobre la contaminación, con fines didácticos, e incluso para estimular la adopción de estas tecnologías en el país (datos personales). Este tema requiere también un análisis más detallado para determinar las oportunidades a futuro que brinda tener tanta capacidad de secuenciación en el país.
La capacidad computacional nacional para análisis de datos si bien es gratuita y suficiente para la mayoría de aplicaciones, no es una limitante para el desarrollo de la bioinformática. Existen múltiples recursos en la nube y en redes como RedCLARA/SCALAC (Sistema de Cómputo Avanzado para América Latina y el Caribe, https://scalac.redclara.net/index.php/es/) de las cuales Costa Rica forma parte. Sin embargo, se debe considerar que en algunos casos estos recursos externos requieren financiamiento adicional, recurso humano dedicado y habilidades que se deben desarrollar. Esta es una dificultad que se analizará en otro estudio.
Finalmente, entre los aspectos que mostraron avance está la regulación ética para investigación biomédica y el manejo de datos sensibles. Este aspecto es fundamental para la implementación de tecnologías aplicadas a la salud humana como lo son la secuenciación de genomas o exomas de pacientes. Sin embargo, la legislación en cuanto a manejo de material genético no humano, llámese muestras ambientales, animales, plantas, con potencial bioeconómico aún son muy complejas de resolver en el ambiente nacional.
Uno de los aspectos preocupantes analizados en este trabajo es la financiación nacional, pero este no es un asunto nuevo pues fue presentado por Moreno et al. (Moreno et al., 2008). En Costa Rica solo el 0.46 % del PIB se destina a actividades de investigación y desarrollo, como se reveló en el 2017 (Mora, 2017). Además, los efectos de la pandemia y la crisis fiscal vienen a agravar el aspecto financiero. Aunque es bien sabido que la ciencia acelera el desarrollo económico de una nación muchos gobiernos no priorizan en este campo (Scheneegans et al., 2021). A pesar de estas situaciones circunstanciales, se recomienda a futuro ajustar el uso de los fondos para que incluyan componentes esenciales para la investigación en bioinformática y biología computacional y para la transición posterior, a decir:
La compra de equipos computacionales o pago de servicios en la nube, además de la compra de reactivos o servicios.
La contratación de analistas de datos bioinformáticos. Que bien podría ser un estudiante que está haciendo su tesis de maestría.
Diseñar una estrategia para retener al analista de datos (comúnmente un estudiante) en la universidad o institución.
Desarrollar fondos de becas para candidatos de maestría/doctorado a tiempo completo para que estudien en el país. Esto reduciría el problema del estudiante o colaborador de dividir su tiempo entre investigación, trabajo y responsabilidades personales. Y al mismo tiempo aportaría al desarrollo de la comunidad de expertos.
Evaluar el efecto del cambio de colones a dólares, que tiende al alza. Esto tiene un impacto en la adquisición de insumos de investigación que se pagan principalmente en dólares.
Otra oportunidad siempre presente es obtener financiamiento internacional, lo que requiere equipos sólidos, interdisciplinarios, con apoyo administrativo a nivel institucional para que el proceso sea eficiente. Esto ha sido logrado por pocos grupos de investigación en el país.
Considerando el panorama presentado en este trabajo, visualizamos un mayor desarrollo de la bioinformática en Costa Rica en los próximos 10 años. Para promover este desarrollo, proponemos cinco acciones directas resumidas en la Fig. 1.
Primero, se necesita estimular la consolidación de grupos de investigación en el país que utilicen la bioinformática como herramienta para responder las preguntas científicas relevantes en el contexto nacional o internacional. Esta consolidación podría estimularse dentro de la academia, incluidos los estudiantes de la MBBS, exbecarios que regresan al país, promoviendo la conexión con la industria y otras instituciones gubernamentales. En este sentido, es necesaria una estrategia a nivel CONARE que permita ubicar a los exbecarios en grupos afines a su disciplina, evitando la duplicidad de oferta académica en el país y contribuyendo a la generación de grupos colaborativos cada vez mayores, mejor organizados y multidisciplinarios. Actualmente, los grupos son escasos, pero a medida que la comunidad de investigadores crece, también aumentan los intereses comunes y las colaboraciones duraderas.
En segundo lugar, es claro que hoy en día los fondos para investigación son adjudicados prioritariamente a grupos o redes de investigación, más allá de individuos o instituciones. La consolidación de una red nacional o comunidad que interactúa a distintos niveles epistémicos aumentaría las probabilidades de obtener recursos para apoyar proyectos de bioinformática, incluyendo la contratación de estudiantes graduados y/o investigadores postdoctorales colaborando en proyectos extendidos.
En tercer lugar, a medida que se aprueben las solicitudes por fondos, algunas políticas y protocolos de administración dentro de las universidades, se debe coadyuvar para cambiar y mejorar el sistema de contratación de recurso humano, incluyendo puestos como asistentes de investigación en bioinformática o analistas de datos, pagados por entes financiadores externos.
Cuarto, la integración activa con sociedades internacionales como ISCB, SoIBio o BioCANET, permitirá potenciar los recursos humanos y promover la investigación nacional.
Una consecuencia directa será un aumento en el número de publicaciones, como quinta acción. Este aumento de publicaciones expondrá la ciencia costarricense que podrá atraer más financiamiento internacional. En conjunto, esto tendrá un impacto positivo en la economía y en la sociedad en general.
La bioinformática en Costa Rica ha experimentado una evolución significativa a lo largo de más de 10 años. Esto se observó en el aumento progresivo anual de publicaciones, la cantidad de personas capacitadas en el país, la creación de la MBBS y de diversas redes, el acceso a infraestructura computacional y el desarrollo de un marco bioético que regula la investigación en genómica. Todos estos eventos positivos han tenido un impacto en la investigación. El efecto se ha ampliado al trabajo de laboratorios gubernamentales que han adoptado tecnologías NGS para abordar casos forenses, en la detección de patógenos y para vigilancia epidemiológica.
Esta evolución se ha dado en parte por las colaboraciones en redes nacionales e internacionales, el trabajo de personal capacitado para liderar investigación en bioinformática, y el acceso a fondos de financiamiento sustanciales para realizar los procesos de secuenciación masiva. En algunos casos la evolución se ha dado por la necesidad imperante de aplicar estas tecnologías para la sociedad, como es el caso de la vigilancia epidemiológica genómica.
Debido a que este progreso recae principalmente en las personas capacitadas en bioinformática, es necesario seguir brindando oportunidades a las nuevas generaciones. Sin embargo, también se deben generar nuevos empleos dentro del país, incluidas oportunidades como puestos de posgrado, investigador postdoctoral, analista de datos o asistente de investigación financiados con subvenciones nacionales o internacionales. En consecuencia, estos esfuerzos tendrán un efecto en el tamaño, la solidez y la experiencia de la comunidad, que a su vez tendrá un mayor potencial para obtener financiamiento de investigación. Nuestra experiencia representa una oportunidad para apoyar a la región centroamericana con capacitaciones y colaboraciones.
Entre los esfuerzos actuales en bioinformática se incluyen los estudios del microbioma infantil y la transcriptómica de la enfermedad periodontal con fondos administrados por la Universidad de Costa Rica; la secuenciación completa del genoma de ~180 miembros de familias que viven en Costa Rica diagnosticadas con esquizofrenia o trastorno bipolar (Carmiol et al., 2014; Glahn et al., 2019); la contribución a las bases de datos internacionales de genomas de patógenos circulantes locales como parte de las redes PULSE NET, RELAVRA y OPS; y más recientemente, el estudio de la genómica del SARS-CoV-2 (Molina-Mora et al., 2021) y la contribución a bases de datos mundiales como Nextstrain (https://nextstrain.org/ncov/gisaid/global).
A futuro, se pueden formular proyectos de resecuenciación más grandes de importancia para la población humana similares al UK10K (Kaye et al., 2014). Costa Rica es una de las pocas poblaciones humanas con un registro genealógico detallado desde el siglo XVI (Meléndez-Obando, 2004), y en la región se han localizado temas de gran interés (por ejemplo, la zona azul de longevidad en Nicoya (Rosero-Bixby et al., 2013). Costa Rica tiene aproximadamente más del 5 % de la biodiversidad mundial que puede protegerse con el uso de información genómica. Otros proyectos relevantes para el país podrían ser proyectos financiados por la misma población similares a los proyectos microbioma intestinal estadounidense o británico (McDonald et al., 2018), una forma de ciencia ciudadana donde las personas brindan voluntariamente muestras y apoyan financieramente la investigación, contribuyendo en última instancia a la comprensión de la salud humana. La aplicación de la medicina de precisión podría ser guiada siguiendo la experiencia de proyectos pioneros como Genomics England del Departamento de Salud del Reino Unido entre otros (Stark et al., 2019).
Para que este tipo de proyectos se desarrollen, primero deben ocurrir otros cambios políticos y administrativos en Costa Rica. Dentro de diez años, esperamos que estos cambios y esfuerzos permitan desarrollar una comunidad científica sólida donde la investigación brinde oportunidades para mejorar la vida, proteger el medio ambiente y estimular la economía.
Declaración de ética: los autores declaran que todos están de acuerdo con esta publicación y que han hecho aportes que justifican su autoría; que no hay conflicto de interés de ningún tipo; y que han cumplido con todos los requisitos y procedimientos éticos y legales pertinentes. Todas las fuentes de financiamiento se detallan plena y claramente en la sección de agradecimientos. El respectivo documento legal firmado se encuentra en los archivos de la revista.












 uBio
uBio