Introducción
Costa Rica es reconocida por su impresionante riqueza biológica (Obando-Acuña, 2002; MINAE - SINAC - CANAGEBIO - FONAFIFO, 2018). Sin embargo, mientras la biodiversidad terrestre ha sido por lo general bien documentada, el conocimiento de la contraparte marina es incompleto, habiéndose centrado mayoritariamente en el estudio de las tortugas marinas (Alvarado, Herrera, Corrales, Asch, & Paaby, 2011; Santidrián-Tomillo et al., 2017). Una recopilación de toda la información existente hasta la fecha sobre la biodiversidad marina del país se recoge en la monografía de Wehrtmann y Cortés (2009), la cual constituye una importante base de partida para cualquier investigación que se desarrolle en este ámbito.
Los equinodermos son uno de los principales componentes de la macrofauna. En la mayor parte de los ecosistemas bentónicos forman parte importante de las cadenas tróficas del océano y ocupan los más diversos roles funcionales (Hughes, Brunner, Cook, Kelly, & Wilson, 2012). A pesar de ello, su conocimiento es todavía muy incipiente en la Región Panámica (Alvarado, Solís-Marín, & Ahearn, 2010; Alvarado, Barraza, & Sancho-Mejías, 2013) y particularmente deficiente en el caso de las ofiuras. Igualmente, son muy pocas las publicaciones dedicadas a los equinodermos de la vertiente pacífica de Costa Rica (Alvarado & Fernández, 2005; Alvarado & Chiriboga, 2008; Alvarado & Cortés, 2009; Azofeifa et al., 2017), ninguna referida específicamente a los Ophiuroidea (Alvarado & Fernández, 2005). Los datos aportados sobre este grupo por Alvarado et al. (2017) proceden de la exhaustiva revisión de los ejemplares depositados en distintos museos y en diversas bases de datos.
Sorprendentemente, a pesar de su importancia ecológica como reductores de la eutrofización (Giraldi et al., 2017) y bioindicadores de la calidad del agua (Hughes, 1998) entre otros, y de constituir la clase de equinodermos más diversa, el número de publicaciones sobre este grupo es muy inferior con respecto a las clases Asteroidea (estrellas de mar) o Echinoidea (erizos). Lo mismo sucede respecto a la información sobre secuencias nucleotídicas disponible en GenBank. Sin duda, ello se debe a la dificultad en la identificación de las especies. De hecho, la taxonomía a nivel específico de los Ophiuroidea sigue siendo en la actualidad muy deficiente e inestable. Por lo general, la descripción de nuevas especies en el pasado se ha basado en escasos caracteres, principalmente de las estructuras esqueléticas externas, aunque en los últimos años se ha abordado una redefinición de los distintos caracteres morfológicos y discutido su utilidad taxonómica (Martynov, 2010; Thuy & Stöhr, 2016). Por otro lado, la mayor parte de las diagnosis de las especies se basa en las fases adultas, mientras que las fases juveniles solo se han descrito en apenas medio centenar de especies, a pesar de que los caracteres de los juveniles difieren considerablemente de los de los adultos (Stöhr, 2005).
La clase Ophiuroidea comprende alrededor de 2 100 especies reconocidas como válidas (Stöhr, O’Hara, & Thuy, 2012) repartidas en 33 familias actuales de acuerdo con la reciente clasificación propuesta por O’Hara, Stöhr, Hugall, Thuy y Martynov (2018), aunque se considera que todavía quedan por describir algunos centenares de ellas. En cualquier caso, esta clasificación se encuentra actualmente en fase de total reestructuración (Thuy & Stöhr, 2016; O’Hara, Hugall, Thuy, Stöhr, & Martynov, 2017; O´Hara et al., 2018). Son muy pocos los estudios moleculares realizados centrados en niveles taxonómicos inferiores, algunos de los cuales muestran la existencia de conjuntos de especies crípticas (e.g. O’Hara, Byrne, & Cisternas, 2004; Stöhr, Boissin, & Chenuil, 2009; Naughton, O’Hara, Appleton, & Cisternas, 2014; Boissin, Egea, Féral, & Chenuil, 2015) que requieren de una adecuada diferenciación y descripción morfológica.
El presente estudio se ha centrado en los Ophiuroidea del litoral del Parque Nacional Marino las Baulas (PNMLB). La caracterización ecológica de su sector marino aportada por el proyecto BIOMARCC-SINAC-GIZ (2015) de apoyo al Sistema Nacional de Áreas de Conservación (SINAC), constituye la única aportación al conocimiento de la biodiversidad marina de este Parque. En este proyecto se destaca la alta diversidad de equinodermos y, entre ellos, se mencionan dos especies de ofiuras: Ophiocoma aethiops y Ophiocoma alexandri, ahora considerada como Ophiocomella alexandri (O’Hara et al. 2018). Asimismo, Alvarado y Fernández (2005) señalaron que las ofiuras eran el grupo de equinodermos más abundante en el Parque Nacional Marino Ballena (Pacífico, Costa Rica) y señalaron que estas dos últimas especies eran las más frecuentes.
Los objetivos concretos que se persiguen con este estudio son: i) caracterizar morfológica y genéticamente las especies de ofiuras de la franja intermareal del PNMLB, ii) establecer la posición filogenética de las especies recolectadas mediante las secuencias del marcador mitocondrial citocromo c oxidasa subunidad I (COI) y iii) testar la utilidad de dicho marcador a nivel de género y especie.
Materiales y métodos
Área de estudio: El muestreo se realizó en el PNMLB, ubicado en la provincia de Guanacaste, en el Pacífico Norte de Costa Rica. Este Parque se extiende aproximadamente 22 km sobre el litoral marino hasta una cota batimétrica de 40 m, abarcando las playas Punta del Morro, Carbón, Ventanas, Grande, Tamarindo y Langosta, con manglares en Tamarindo y Ventanas. Incluye bosques, playas, algunos acantilados, zonas intermareales rocosas, esteros y manglares. La totalidad del parque cubre una superficie de 26 788 ha, de la cual el 97 % (26 056.58 ha) es área marina (BIOMARCC-SINAC-GIZ 2015). La conservación de este parque nacional marino es importante, dado que figura como un lugar de anidamiento de la tortuga baula o laúd (Dermochelys coriacea), especialmente en Playa Grande (Nordmoe et al., 2004; BIOMARCC-SINAC-GIZ, 2015). La zona intermareal donde se desarrolló el muestreo en este trabajo está influenciada por mareas semidiurnas con un rango de amplitud de hasta unos tres metros (Lizano, 2006) y se divide en dos subsistemas: la zona intermareal rocosa y la zona intermareal arenosa (BIOMARCC-SINAC-GIZ, 2015).
Toma de muestras: El periodo de recolecta se extendió entre enero y abril de 2018 en la zona intermareal. El muestreo se llevó a cabo durante la marea baja, abarcando los diferentes hábitats presentes (sustrato rocoso, arenoso y arrecife muerto). Se realizó una identificación preliminar de los ejemplares observados y se establecieron 12 morfoespecies, etiquetadas como Opae, OpA, OpB, OpC, OpD, OpE, OpF, OpG, OpH, OpI, OpJ y OpK, según se iban encontrando. Se extrajeron fragmentos de brazo de cada ofiura, así como individuos completos de cada morfotipo considerado. El fragmento de brazo cortado no fue mayor de la mitad de éste, asegurando así la supervivencia de los ejemplares de estudio. Se recolectaron finalmente muestras de 214 ejemplares en total (Apéndice 1), de los cuales 15 fueron individuos completos. Empezando por el área más septentrional del parque, en Punta del Morro se tomaron 17 muestras, en Playa Carbón y Playa Grande, 33, en Playa Ventanas, 33. En Playa Tamarindo se hizo una distinción entre dos zonas para la recolecta, debido a que una de ellas tiene gran aglomeración de turistas por encontrarse en área hotelera (denominada en este estudio Playa Tamarindo Ciudad). En esta playa se recolectaron muestras de 37 ejemplares, y en la zona más alejada, sin aglomeración (denominada en este estudio simplemente como Playa Tamarindo), 26. Por último, en Playa Langosta, ubicada más al sur del PNMLB, se obtuvieron 35 muestras. Así, el muestreo se desarrolló en siete puntos del parque, para abarcar toda su línea de costa. De cada ofiura se tomó una fotografía, tanto dorsal como ventral, antes de su liberación al mar o de fijar los individuos completos, tomando además las coordenadas de cada punto de colecta (Apéndice 2). Las muestras se fijaron al momento en etanol absoluto. Todos los datos de la recolecta se almacenaron en la base de datos EPICOLLECT. Las muestras se depositaron en el Museo Nacional de Ciencias Naturales de Madrid (MNCN-CSIC) y se conservan en etanol absoluto a 5 ºC.
Identificación morfológica: Para las identificaciones morfológicas de los individuos se utilizó un microscopio estereoscópico LEICA MZ16 A, con toma de fotografías de los principales caracteres diagnósticos, y las claves taxonómicas y descripciones existentes (principalmente de Clark, 1948; Boolootian & Leighton, 1966; Brusca, 1980; Granja-Fernández et al., 2014; Hendler, 1996; McClendon, 1909; Nielsen, 1932 y Ziesenhenne, 1940, entre otros). Se atendió a los caracteres diagnósticos tradicionales (ver Hendler, 1996) redefinidos y actualizados por Stöhr et al. (2012): disco dorsal, escudos radiales, placas primarias, placas dorsales, placas accesorias y placas laterales de los brazos, espinas del brazo, disco ventral, hendiduras bursátiles, mandíbulas, papilas orales, dientes, escudo oral, escudos adorales, placas ventrales de los brazos y escamas tentaculares. Para la nomenclatura específica se han seguido los criterios de Stöhr, S., O’Hara, T., & Thuy, B. (2020) y para su clasificación la propuesta reciente de O’Hara et al. (2018).
Análisis moleculares: A partir de segmentos de los brazos se obtuvo el tejido para la extracción del ADN mediante el kit de extracción BioSprint 15 DNA Blood Kit (Qiagen) de acuerdo con su protocolo, con una extensión del tiempo de homogeneización con proteinasa K de alrededor de 18 horas y utilización de ARNasa. Se amplificaron fragmentos del gen mitocondrial citocromo c oxidasa subunidad I (COI). Los cebadores utilizados fueron CO1a-H (5’-TGT ATA RGC GTC TGG RTA GTC-3’), CO1f-L (5’- CCT GCA GGA GGR GGA GAY CC-3’) modificados de Palumbi et al. (2002) y COI-Of-Rint (5’-TGA ATA ACG ACG TGG CAT TC-3’) diseñado en este estudio. La pareja de cebadores CO1f-L /CO1a-H genera secuencias de 649 pares de bases (pb), mientras que la pareja CO1f-L /COI-Of-Rint genera secuencias de 629 pb, en ambos casos tras eliminar la región de los cebadores. Cada amplificación se llevó a cabo en un volumen de 50 µl con 1 o 2 µl de ADN diluido de 20 a 50 veces, con 5 µl de tampón 10x con MgCl2, 1 µl de dNTPs 40 mM, 1 µl de cada cebador 10 µM y 0.3 µl de Taq polimerasa de 5 U/µl. Las condiciones de las PCR fueron las siguientes: 2 min a 94 ºC, seguido por un ciclo de desnaturalización de 45 s a 94 ºC, anillamiento de 45 s a 45 ºC, elongación de 1 min a 72 ºC y un post-ciclo de 10 min a 72 ºC. Los ciclos de desnaturalización, anillamiento y elongación se repitieron 40 veces. Los fragmentos de ADN amplificados se purificaron cortando y filtrando las regiones de las bandas amplificadas tras su migración en un gel de agarosa al 1.5 % en TA (TRIS-acético, pH = 8) y se enviaron para su secuenciación a Secugen S. L. (Madrid). Mediante el programa Sequencher 4.1.4 se editaron las secuencias obtenidas.
Análisis filogenéticos: En primer lugar, para comprobar que el gen COI es filogenéticamente informativo para este grupo, se realizó un test de saturación a partir de los datos de divergencia (% entre cada par de individuos y las transiciones y transversiones en cada posición del codón obtenidos con PAUP* (Swofford, 2001). Se creó una tabla en Excel con esos datos y se generaron los gráficos para los cambios de cada posición del codón a fin de comprobar si existe saturación del marcador.
Para los análisis filogenéticos se añadieron 115 secuencias obtenidas en GenBank (Apéndice 3) a las 209 generadas en este trabajo, obteniendo así una matriz de 324 secuencias. Se fijaron como grupo externo los asteroideos Achantaster planci y Patiria miniata. Las relaciones fueron deducidas por inferencia bayesiana utilizando MrBayes v3.2.1 (Ronquist & Huelsenbeck, 2003). Se analizaron cuatro cadenas de Markov-Monte Carlo (MCMC) para estimar las probabilidades posteriores en dos carreras en paralelo durante 150 millones de generaciones con un comienzo aleatorio y teniendo en cuenta las tres posiciones de los codones. Se extrajeron los parámetros y árboles cada 15 000 generaciones para obtener el árbol consenso (MCC, “maximum clade credibility”) que se editó en FigTree (Rambaut, 2016) (Apéndice 4). Por último, con el software MEGA X se calcularon las distancias genéticas intra e interespecíficas.
Delimitación de especies: Se generaron seis matrices, agrupando en cada matriz individuos del mismo género o géneros próximos, siempre formando un grupo monofilético. Con jModeltest se obtuvieron los modelos evolutivos que mejor se ajustaron a los datos.
Para la delimitación de las especies se utilizó el método bGMYC (“Generalized Mixed Yule Coalescent”) en un marco bayesiano. Para llevarlo a cabo se necesita un árbol ultramétrico, que aquí fue obtenido con el programa BEAST (Drummond & Rambaut, 2007), utilizando un reloj molecular relajado (“uncorrelected relaxed clock”) con un proceso de especiación de muestreo incompleto (“speciation: birth-death incomplete sampling”), comenzando con un árbol aleatorio e incluyendo el modelo obtenido con jModeltest. El análisis para cada matriz se corrió un número de réplicas suficiente para que las dos cadenas de análisis hubieran convergido. En todos los casos fue de 100 millones de generaciones, salvo para Ophionereis, donde el número de réplicas de 50 millones fue suficiente. También se comprobó la estabilidad de los parámetros obtenidos y si su muestreo fue suficiente mediante el programa Tracer (Rambaut, Suchard, Xie, & Drummond, 2014). Se obtuvo el árbol MCC con TreeAnnotator (Drummond& Rambaut, 2007). Finalmente, la delimitación de especies mediante bGMYC se llevó a cabo en R (Reid & Carstens, 2012). Para evitar la sobre- y subestimación de especies, se consideró una probabilidad de co-especificidad de 0.5, la cual representa la media posterior del análisis (Reid & Carstens, 2012). Los parámetros utilizados para cada matriz, tasa de cambio en la coalescencia (ph1 y ph2), tasa de cambio en la especiación (pc1 y pc2), el número de especies potenciales (t1 y t2), los puntos de comienzo (start) y la escala (scale) se especifican en la Tabla 1.
TABLA 1 TABLE 1 Parámetros utilizados para cada grupo en bGMYC (“Generalized Mixed Yule Coalescent”) Parameters used for each group in bGMYC (“Generalized Mixed Yule Coalescent”)
| Grupo | ph1 | ph2 | pc1 | pc2 | t1 | t2 | start | scale |
| Ophionereis | 0 | 5 | 0 | 4 | 1 | 8 | (2,2,6) | (1,1,0.3) |
| Ophiothrix | 0 | 1 | 0 | 1 | 1 | 25 | (1,1,13) | (1,1,0.3) |
| Ophiocoma | 0 | 5 | 0 | 1.5 | 1 | 10 | (3,0.6,5) | (3,5,0.3) |
| Amphiuridae | 0 | 5 | 0 | 4 | 1 | 8 | (2,2,6) | (1,1,0.3) |
| Ophioderma | 0 | 6 | 0 | 20 | 1 | 10 | (2,2,5) | (1,1,0.3) |
| Ophiactis | 0 | 2 | 0 | 2.5 | 1 | 20 | (1,1,16) | (1,1,0.3) |
ph1, ph2: tasa de cambio en la coalescencia. pc1, pc2: tasa de cambio en la especiación. Start: puntos de comienzo. Scale:escala.
ph1, ph2: coalescent rate. pc1, pc2: speciaton rate. Start: start points, Scale.
Resultados
Análisis filogenéticos: En el tratamiento bayesiano la desviación entre las dos carreras fue menor de 0.01 y con Tracer se comprobó que los valores de ESS (“effective simple size”) fueran mayores de 200, por lo que se pudo confirmar la convergencia de ambas carreras del análisis y un muestreo suficiente de los distintos parámetros.
En el filograma obtenido por MrBayes (colapsado en la Fig. 1 y detallado en el Apéndice 4) se observa que en un clado con apoyo de 1 de probabilidad posterior (PP) se situaron OpH y OpJ, con ramas muy cortas entre sí, junto con individuos del género Ophioderma. Los individuos OpF se relacionaron con los dos ejemplares de Ophiocomella alexandri con un apoyo de PP también máximo. Los denominados como Opae y OpC se posicionaron junto a Ophiocoma aethiops dentro del mismo clado con una PP de 0.93. Los etiquetados como OpD, OpA_039 y OpB formaron un grupo monofilético de alto apoyo (PP= 1) como grupo hermano de Ophionereisreticulata y Ophionereis annulata (PP= 0.97). Las muestras OpA y OpE se agruparon monofiléticamente con la máxima probabilidad asociados como grupo hermano con dos individuos del género Ophiophragmus, salvo OpA_193, más alejado de los anteriores y formando una politomía con el grupo anterior, Microphiopholis gracillima y Amphipholis linopneusti. En este pequeño clado, OpA_004 se posicionó como grupo hermano del resto (OpA_008, OpA_124, OpA_156 y OpE_040). Los individuos OpK formaron un grupo monofilético (PP = 1) con especies del género Ophiactis, dos con O. simplex, una con O. rubropoda y otra sin definir a nivel de especie. Por último, tanto los OpG como OpI se situaron junto a individuos del género Ophiothrix, los primeros junto a Ophiothrix spiculata (PP = 1) y el segundo junto a Ophiothrix lineata (PP = 0.96).
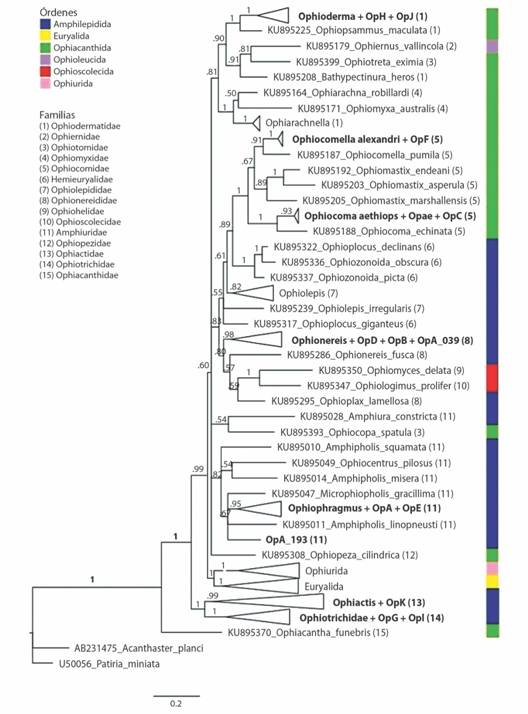
Fig. 1 Fig. 1 Árbol colapsado resultante de la inferencia bayesiana basado en el gen COI. En la leyenda se indican los distintos órdenes y familias representados (el árbol detallado se muestra en el Apéndice 4). Collapsed tree resulting from bayesian inference based on the COI gen. The different orders and families are indicated in the legend (the detailed tree is shown in Appendix 4).
Como visión general, esta filogenia no resultó acorde con las categorías taxonómicas superiores, mostró un gran número de politomías a varios niveles y presentó muchas ramas con valores de probabilidad muy bajos. Los tests de saturación indicaron una cierta saturación en divergencias superiores al 15 - 20 %.
Los altos valores de divergencia encontrados entre integrantes del mismo género y entre las distintas familias pudieron ser los responsables de escasa definición en las relaciones basales del grupo. De hecho, la divergencia media intraespecífica fue de 1.5 %, y la media interespecífica dentro del mismo género de 20.5 %. La divergencia entre familias osciló en un rango que fue del 19 al 28 %. OpJ y OpH divergieron en un 0.31 %. En el linaje de Ophioderma longicauda la mayor distancia fue de 7.51 %. La asociación de OpB y OpD_112 presentó una distancia entre ellas de 0.46 %, más alejadas con el resto de las OpD, con una divergencia de OpD_083 de 5.4 % y 8.6 %, respectivamente. Ophionereis annulata se distanció de las OpD con alrededor del 10 % de divergencia. En el grupo de OpA (salvo OpA_193) y OpE, resultó una distancia mayor entre OpA_004 y el resto: de media se encontró a una distancia de las demás de 1.9 % y el resto entre sí de 1.1 %. Por último, la divergencia dentro del linaje del género Ophiactis osciló entre 0.86 y 1.72 %.
Delimitación de especies: En el género Ophioderma, OpH_139 y OpJ_194 resultaron agrupadas como una misma especie y sin asociación con ninguna de las obtenidas del GenBank. Las tres secuencias de Ophioderma longicauda obtenidas de GenBank se diferenciaron en dos grupos, separando O. longicauda (KU895218) de las otras dos. En el grupo en el que había representantes de Ophiocomaaethiops, se añadieron las muestras designadas como Opae y OpC como una misma especie, como ocurrió para los dos individuos de Ophiocomella alexandri y las OpF. Todas las OpD junto con OpA_039 formaron un grupo, excepto OpD_112, que formó otro junto con OpB. Todas las OpA se agruparon en una misma especie con OpE, exceptuando a OpA_193. Los dos individuos de Ophiactis simplex, Ophiactis sp., Ophiactis rubropoda, OpK_209, OpK_210 y OpK_210 se consideraron como una misma especie. La probabilidad de que O. rubropoda fuera una especie diferente a las anteriores fue de un 2 %. Ophiothrix spiculata se asoció a las OpG. Por último, OpI conformó un linaje propio. Así, el método bGMYC de delimitación de especies, previa a la identificación morfológica, dio como resultado 10 especies en el PNMLB: OpH + OpJ = Ophioderma sp., Opae + OpC = Ophiocoma aethiops, OpF = Ophiocomella alexandri, OpD + OpA_039 = Ophionereis sp.1, OpD_112 + OpB = Ophionereis sp.2, OpA + OpE = Amphiuridae indet 1, OpA_193 = Amphiuridae indet 2, OpK = Ophiactis sp., OpG = Ophiothrix spiculata, OpI = Ophiothrix rudis.
Identificación morfológica de las especies: La información aportada por los análisis filogenéticos obtenidos y la delimitación de especies, distinguen 10 especies. Añadiendo la identificación morfológica se llega a que las 12 morfoespecies identificadas preliminarmente durante la recolecta representan 11 taxones de rango específico repartidos en dos órdenes (Ophiacanthida y Amphilepidida), seis familias (Ophiocomidae, Ophiodermatidae, Ophionereididae, Ophiotrichidae, Amphiuridae y Ophiactidae) y ocho géneros (Ophiocoma, Ophiocomella, Ophioderma, Ophionereis, Ophiotrix, Microphiopholis, Ophiophragmus y Ophiactis) representados en la Tabla 2.
TABLA 2 TABLE 2 Especies identificadas en el PNMLB y su clasificación taxonómica según las evidencias morfológicas y moleculares (se indica también las claves de morfoespecie correspondientes a cada una) Identified species in the PNMLB and their taxonomic classification according to morphological and molecular evidence (morphospecies keys are also indicated)
| Orden | Familia | Género | Especie | Etiquetas |
| Amphilepidida | Amphiuridae | Microphiopholis | puntarenae | OpA_193 |
| platydisca | OpA_004 | |||
| Ophiophragmus | aff. stellatus | OpA, OpE | ||
| Ophiactidae | Ophiactis | simplex | OpK | |
| Ophionereididae | Ophionereis | aff. annulata 1 | OpD, OpA_039 | |
| aff. annulata 2 | OpD_112, OpB | |||
| Ophiotrichidae | Ophiothrix | spiculata | OpG | |
| rudis | OpI | |||
| Ophiacanthida | Ophiocomidae | Ophiocoma | aethiops | Opae, OpC |
| Ophiocomella | alexandri | OpF | ||
| Ophiodermatidae | Ophioderma | aff. panamensis | OpH, OpJ |
En el Apéndice 5 para cada especie se aportan fotografías del disco y de los brazos (por sus caras oral y aboral), las sinonimias (en su caso), observaciones pertinentes sobre los ejemplares revisados, datos de distribución geográfica y citas en Costa Rica, así como los datos disponibles sobre hábitats. En lo referente al “material estudiado” se indica el ejemplar completo que se utilizó para la descripción morfológica.
Aproximación a la riqueza y distribución en PNMLB: En el Apéndice 6 se especifica el número de ejemplares recolectados de cada especie en cada localidad. En resumen, la diversidad de especies encontradas de ofiuras varió según la localidad. Las playas con mayor diversidad fueron Playa Tamarindo Ciudad, con siete especies, y Playa Langosta, con seis. Le siguen en diversidad Playa Ventanas y Playa Grande con cinco especies, y Playa Carbón y Playa Tamarindo, con cuatro. El menor número de especies observado fue en Punta del Morro con tres. La especie más abundante en el Parque resultó ser Ophiocoma aethiops, seguida de Ophiocomella alexandri, Ophionereis aff. annulata 1 y Ophiothrix spiculata. El resto de las especies se encontraron muy poco representadas. La mayor abundancia de O. aethiops se observó en Playa Langosta, donde los individuos formaban aglomeraciones masivas.
En cuanto a los hábitats, O. aethiops y Ophiophragmus aff. stellatus fueron las únicas especies encontradas en los tres sustratos disponibles. En general, el sustrato predominante fue el rocoso (47.20 %), seguido del arenoso (39.72 %) y el arrecife muerto (13.08 %). En el Apéndice 7 se muestran los porcentajes de individuos recolectados en cada sustrato.
Discusión
Se aporta el registro de ofiuras observadas en el Parque Nacional Marino las Baulas y se asignan los ejemplares encontrados a once especies, de las que nueve constituyen nuevos aportes para el Parque: Ophionereis aff. annulata 1, Ophionereis aff. annulata 2,Microphiopholis platydisca, Microphiopholis puntarenae, Ophiophragmus aff. stellatus, Ophiothrix (Ophiothrix) rudis, Ophiothrix (Ophiothrix) spiculata, Ophioderma aff. panamensis y Ophiactis simplex,y dos, Ophiocoma aethiops y Ophiocomella alexandri, que ya se habían documentado previamente (BIOMARCC-SINAC-GIZ, 2015). Asimismo, se aportan 209 nuevas secuencias del marcador COI para que puedan ser de utilidad en futuras investigaciones.
Debido a que el objetivo del estudio era principalmente cualitativo, los datos de distribución y abundancias deben tomarse como una aproximación. La disponibilidad de los diferentes sustratos no es equilibrada en las distintas localidades. Además, el menor número de especies presente en arrecife muerto (13.08 %) puede responder a la dificultad de muestreo en ese sustrato, caracterizado por presentar multitud de cavidades y túneles internos cuyo único acceso sería la destrucción del propio hábitat. Por el contrario, el arrecife muerto parece ofrecer un gran número de nichos y superficie que ocupar. Asimismo, la mayor abundancia de individuos recolectados en el sustrato rocoso puede deberse a la facilidad de localizarlos en ese medio levantando las rocas sueltas.
Según los resultados obtenidos, el PNMLB puede considerarse con una alta diversidad de ofiuras si se compara con otras localidades del Pacífico de Costa Rica donde se han realizado estudios similares sobre los equinodermos. Alvarado & Fernández (2005) citaron seis especies de ofiuras en el Parque Nacional Marino Ballena, Alvarado & Chiriboga (2008) encontraron seis especies en la Isla del Coco, y Azofeifa-Solano et al. (2017) citan dos especies en el sistema estuarino de Térraba-Sierpe. Además, hay que tener en cuenta que en este estudio solo se han obtenido ejemplares del intermareal, sin haberse muestreado estuarios, manglares, ni mayores profundidades, por lo que el número de especies presentes se considera subestimado.
Los resultados obtenidos sugieren cierta cautela en su interpretación, ya que se han observado algunas diferencias entre las identificaciones derivadas de los métodos moleculares y las morfológicas. De no ser por el uso adicional de herramientas moleculares, la morfología de los ejemplares juveniles hubiera podido inducir a errores y considerarse como especies distintas. Es el caso de OpJ (Apéndice 5, Fig. A.5.12), que gracias a la delimitación molecular se ha resuelto como un juvenil de OpH (Apéndice 5, Fig. A.5.11), con una distancia genética de 0.31 %, ya que los caracteres morfológicos, como escudos orales y placas de los brazos sin desarrollar, y la coloración son muy diferentes a los que presenta el adulto. Ambos se determinaron como Ophioderma aff. panamensis, pues las características morfológicas del ejemplar estudiado no se ajustan completamente a la descripción de esta especie (presentando este individuo los escudos radiales cubiertos por granulación, carente en los de O. panamensis). Por este motivo, podría tratarse de una nueva especie, pero es necesario disponer de individuos de O. panamensis para ampliar los análisis tanto morfológicos como moleculares. Otros casos de individuos juveniles que se han podido emparejar con sus adultos son OpC: juveniles de Ophiocoma aethiops (Opae), OpA_039: juvenil de Ophionereis aff. annulata 1 (OpD) y OpB_009: juvenil de Ophionereis aff. annulata 2 (OpD_112).
Por otro lado, la delimitación molecular, al contrario que en los anteriores casos, separa como diferentes especies individuos morfológicamente similares y que se habían identificado previamente como la misma. Por ejemplo, el ejemplar OpD_112 resultó con una divergencia del 8.6 % con respecto al resto de OpD y OpA_039 sí se agrupó con el resto de las OpD. Asimismo, al ejemplar OpB se diferenció de los OpD por presentar un tamaño relativo muy pequeño y con diferencias en la coloración, aunque los caracteres morfológicos son similares. Este ejemplar presentó una divergencia con respecto al resto del 5.4 %. Sin embargo, OpD_112 y OpB solo divergen el 0.46 % la una de la otra, constituyendo así, una misma especie, siendo OpB un juvenil. De este modo, el linaje formado por las OpD y OpA_039 y el formado por OpD_112 y OpB podrían constituir nuevas especies y un caso de especies crípticas (Ophionereis aff. annulata 1 y Ophionereis aff. annulata 2, respectivamente). En ambos casos se dejó como affinis ya que los análisis filogenéticos y de delimitación de especies nos dicen que no pertenecen a la misma especie que la secuencia de Ophionereis annulata disponible en GenBank (con una divergencia del 10 % aproximadamente) y los caracteres morfológicos son similares a la descripción de O. annulata salvo que los individuos de este estudio no presentan una placa primaria central evidente. El trabajo del que se extrajo esa secuencia de O. annulata (Hugall, O’Hara, Hunjan, Nilsen, & Moussalli, 2015) no aporta datos sobre su identificación, por lo que podría no ser acertada. Para llegar a una conclusión definitiva es necesario disponer de individuos de O. annulata y realizar los pertinentes análisis morfológicos y moleculares.
La existencia de especies crípticas puede explicarse por tres factores: caracterización morfológica insuficiente, especiación reciente o “cladogénesis estática” (existencia de aislamiento genético que no se traduce en una divergencia morfológica) (Bickford et al.,2007). En la clase Ophiuroidea se han detectado varios complejos de especies crípticas (O’Hara et al., 2004; Stöhr et al., 2009; Naughton, et al., 2014; Boissin et al., 2015; Weber, Stöhr, & Chenuil, 2017). Para ello las herramientas moleculares se muestran muy útiles para detectar estos casos, así como para identificar a los individuos juveniles (Bribiesca-Contreras, Solís-Marín, Laguarda-Figueras, & Zaldívar-Riverón, 2013).
Un linaje conocido como un complejo de especies es el de Ophioderma longicauda (Weber et al., 2017) lo que explica que de las tres secuencias que incluimos en este estudio para esa especie, el análisis dGMYC las separa en dos especies, con una divergencia de 7.51 %, un valor muy elevado para tratarse de un mismo taxón. Por otro lado, en el linaje de Ophiactis simplex (OpK) hay un rango de distancia genética que va del 0.86 al 1.54 % y también está considerado como un complejo de especies que se encuentra en revisión (Granja-Fernández, comunicación personal, 2018). Ophiactis rubropoda se encuadra dentro de este linaje junto con O. simplex, y ambas forman parte de un grupo único por presentar hemoglobina en su sistema vascular junto con Ophiactis virens y Hemipholis elongata (Christensen, Christensen, & Weisrock, 2008). La discriminación morfológica entre O. simplex y O. rubropoda es difícil, pero esta última se distribuye en el Caribe (Pomory, 2007), mientras que la primera habita en el Pacífico (Granja-Fernández et al., 2014). La distancia genética del COI entre ellas es pequeña (la que hay entre O. rubropoda y OpK_210 es de 1.72 %) con respecto a lo que ocurre en otros taxones separados por el Istmo de Panamá (Christensen et al., 2008), cerrado hace unos 3.5 millones de años (De Gracia, O’Dea, Rodríguez, & D’Croz, 2012). Por ejemplo, especies hermanas de camarón separados por el istmo presentaron una divergencia de aproximadamente el 5 % (Christensen et al., 2008). Una hipótesis plausible sería que la divergencia es intraespecífica, con cambios en el fenotipo, y que su distribución en ambos lados del istmo se produjo por acción humana más reciente, como ocurrió con Ophiothela mirabilis, una especie nativa del Pacífico que invadió el Océano Atlántico en las últimas décadas por la probable asociación a los cascos de los barcos (Barboza, Mattos, & Paiva, 2015). Finalmente, no existen dudas de que las OpG corresponden a Ophiothrix (Ophiothrix) spiculata y que el único individuo OpI pertenece a Ophiothrix (Ophiothrix) rudis; resultados robustos por obtenerse tanto molecular como morfológicamente.
También en nuestros resultados se da el caso contrario: ejemplares que difieren morfológicamente pero que no difieren en el marcador molecular utilizado. Por ejemplo, sucede con Microphiopholis platydisca (OpA_004), que se diferenció de Ophiophragmus aff. stellatus (OpA y OpE) atendiendo a su morfología y, sin embargo, el filograma y el método de delimitación de especies apenas muestran diferencias entre ellas. La distancia genética entre Microphiopholis platydisca y el resto es de 1.9 %. Además, se incluye formando un grupo monofilético con Ophiophragmus filograneus y Ophiophragmus wurdermani. En cuanto a Microphiopholis puntarenae, los análisis moleculares la sitúan formando una politomía junto con los géneros Amphipholis y Ophiophragmus y queda muy alejado de Microphiopholis platydisca. Estos resultados muestran que estos géneros no están bien resueltos y, por lo tanto, para poder llegar a una conclusión, es necesario hacer un análisis morfológico-molecular de más especímenes y quizá el uso de otros marcadores moleculares. Ophiophragmus aff. stellatus responde por completo a la descripción de Ziesenhenne (1940) salvo que la papila oral media no presenta forma de espina y por ello se dejó como affinis. Para poder saber si se trata de una especie nueva se necesita el estudio de individuos de O. stellatus.
En lo que se refiere a los taxones de rango superior, el árbol filogenético obtenido apoya la monofilia de los órdenes Ophioscolecida, Euryalida, Ophiurida y el superorden Euryophiurida, relación que ya encontró Martinov (2010) basándose en caracteres de las articulaciones de las espinas de los brazos. Por otro lado, la relación obtenida como grupos hermanos de las familias Ophiactidae y Ophiotrichidae está apoyada fuertemente por los trabajos anteriores de Smith, Paterson y Lafay (1995) y O’Hara, Hugall, Thuy y Moussalli (2014). Sin embargo, por lo general las familias se encuentran mejor resueltas como monofiléticas en anteriores trabajos (Hunter, Brown, Alexander, Kroeger, & Rose, 2016), basados en un gen mitocondrial (ARNr 16S) y un gen nuclear (ARNr 18S). Es importante señalar que los taxones aquí estudiados constituyen una representación incompleta y se precisa de una gran cantidad de secuencias para descifrar relaciones ancestrales (Hunter et al., 2016). Además, en la prueba de saturación se observa que alrededor del 15 % de divergencia el marcador comienza a saturarse. La distancia genética media intraespecífica (1.5 %) y la interespecífica dentro del mismo género (20.5 %) son del mismo orden de magnitud que las encontradas en equinodermos en general por Hoareau y Boissin (2010) (1.3 y 20.3 %, respectivamente). Las divergencias entre familias en este trabajo se encuentran en un rango del 19 - 28 %, por lo que el marcador utilizado pierde calidad informativa a esos niveles y superiores. La inclusión de taxones bastante diferenciados en este análisis podría ser la explicación de estos resultados e indicarían una subvaloración con respecto a las divergencias reseñadas.
Recientemente, se están considerando nuevos caracteres morfológicos internos como diagnósticos, sobre todo en niveles taxonómicos superiores, al tiempo que los estudios moleculares se van ampliando con el uso de varios marcadores, transcriptomas y bases de datos masivos (O’Hara, Hugall, Thuy, Stöhr, & Martynov, 2017).
En cualquier caso, este trabajo pretende ser una contribución al conocimiento de las ofiuras de la Región Pacífica Panameña sin pretensiones filogenéticas. En total, combinando los caracteres morfológicos y genéticos, se han identificado 11 especies, cuatro de las cuales podrían ser nuevas, nueve constituyen nuevos registros en el PNMLB y una de las especies se ha encontrado por primera vez en Costa Rica (Ophiophragmus aff. stellatus). Se aporta un nuevo inventario de especies marinas del PNMLB, mediante la caracterización morfológica y genética de las ofiuras, con una aproximación a su abundancia y distribución. Con el seguimiento de las comunidades de ofiuras se puede contribuir a evaluar la salud del ecosistema intermareal, que sufre intensas actividades propias del desarrollo costero de la ciudad de Tamarindo dentro del Parque. Conocer el estado de la biodiversidad de una zona es siempre importante para llevar a cabo actuaciones adecuadas de conservación. Por otro lado, se aportan observaciones morfológicas e imágenes como contribución a la taxonomía de este grupo de equinodermos, se analizan algunos aspectos filogenéticos, y la utilidad del marcador COI a distintos niveles taxonómicos y se provee de secuencias de ADN mitocondrial, que pueden llegar a contribuir a una gran gama de estudios como análisis filogenéticos, biogeográficos y evolutivos.
Declaración de ética: los autores declaran que todos están de acuerdo con esta publicación y que han hecho aportes que justifican su autoría; que no hay conflicto de interés de ningún tipo; y que han cumplido con todos los requisitos y procedimientos éticos y legales pertinentes. Todas las fuentes de financiamiento se detallan plena y claramente en la sección de agradecimientos. El respectivo documento legal firmado se encuentra en los archivos de la revista.












 uBio
uBio 

