Introducción
El uso generalizado de plaguicidas en la agricultura como método unilateral de manejo de problemas fitosanitarios de los cultivos ha ocasionado serias afectaciones en la salud de personas aplicadoras, sus familias y comunidades enteras, además de contaminación y deterioro de los ecosistemas (Tudi, et al., 2021; Aktar, Sengupta & Chowdhury, 2009; WHO, 1990).
En particular el suelo ha sido seriamente dañado por las actividades derivadas de la agricultura convencional, siendo la contaminación por plaguicidas uno de los tantos problemas (además de erosión, desertificación, salinización, acidificación, y agotamiento) que es necesario atender para recuperar los suelos.
Una tecnología propuesta para eliminar plaguicidas del suelo con éxito es la biorremediación, la cual emplea organismos vivos como bacterias, hongos, algas y plantas (Hussain, et al., 2009; Verma, 2021). La característica de los microorganismos de poseer una gran versatilidad metabólica es aprovechada en la práctica de la biorremediación, al utilizar (degradar) diversas fuentes de carbono y energía para su crecimiento y reproducción, incluidas aquellas de naturaleza química compleja como los plaguicidas (Johnsen, Jacobsen & Torsvik, 2001).
Mediante la biorremediación, las moléculas de plaguicidas son descompuestas en moléculas de menor peso y eventualmente, oxidadas vía aeróbica hasta su mineralización, dando como resultado dióxido de carbono y agua (Tang, Ji & Hou, 2017). La biorremediación cuenta con ventajas respecto a otras formas de descontaminación convencionales, ya que es una tecnología económica, con resultados satisfactorios y amigable con el ambiente debido al mantenimiento y enriquecimiento de la diversidad edáfica (Vidali, 2001; Chibueze, Blaise & Chijioke, 2016).
De acuerdo con investigaciones realizadas por el Instituto Regional de Estudios en Sustancias Tóxicas (IRET), dos de los ingredientes activos (i. a.) de plaguicidas de mayor uso en Costa Rica y cuya presencia ha sido evidenciada en diversas matrices ambientales, incluyendo el suelo, son clorotalonil y clorpirifos (Ramírez-Muñoz, et al., 2014; Bravo-Durán, et al., 2015; Bravo, et al., 2013), ambos han sido clasificados como altamente peligrosos (PAN, 2021).
El clorotalonil es el segundo fungicida de amplio espectro más utilizado en la agricultura (Wang, et al., 2011; Betancourt-Portela, et al., 2018), este puede permanecer en el suelo por periodos de hasta un año si se aplica de manera repetitiva (Singh, Walkey y Wright, et al., 2002; Motonaga, et al., 1996).
Este fungicida es sumamente tóxico para aves, peces e invertebrados acuáticos, y en humanos causa afectaciones de salud como dermatitis, problemas gastrointestinales, irritaciones severas de los ojos y la piel (Caux, et al., 1996; Draper, et al., 2003). Además, es considerado como un agente causante de cáncer (Cox, 1997).
Por otro lado, el clorpirifos es un insecticida de amplio espectro utilizado alrededor del mundo (Wang, et al., 2019; Akbar & Sultan, 2016), tiene una persistencia en el suelo de entre 10 y 120 días (Nandhini, Harshiny & Gummadi, 2021); y a pesar de que es considerado como un plaguicida moderadamente tóxico, es capaz de causar afecciones a la salud como sensibilidad a la luz, taquicardia, e incluso incrementa el riesgo a desarrollar diversos tipos de cáncer (Nandhini, Harshiny & Gummadi, 2021).
La presencia de plaguicidas como clorotalonil y clorpirifos en el suelo podría interferir en procesos edáficos vitales para su funcionamiento, como la fragmentación de la materia orgánica y mineralización del nitrógeno, ya que se les atribuye efectos como la reducción de las poblaciones y diversidad de micro- y macroorganismos y su actividad enzimática (Sigler & Turco, 2002; Menon, et al., 2004).
No obstante, existen innumerables informes de especies bacterianas capaces de tolerar y degradar este tipo de plaguicidas; por ejemplo, Enterobacter cloacae TUAH1 (Tang, et al., 2017) y Stenotrophomonas acidaminiphila BJ1 (Zhang, et al., 2019) pueden metabolizar clorotalonil, mientras que otras, como Pseudomonas aeruginosa, o Bacillus cereus (Lakshmi, Kumar & Khanna, 2008) y Alcaligenes faecalis (Yang, et al., 2005) pueden degradar clorpirifos.
Debido a que la base de las estrategias de biorremediación consiste en el uso de microorganismos principalmente, existe gran interés por aislar microorganismos para utilizarse en la descontaminación de suelos agrícolas, con el objetivo de contribuir a regenerar agroecosistemas. Por esta razón, el presente trabajo tuvo como objetivo el aislamiento, identificación y caracterización de cepas bacterianas con potencial de utilización del fungicida clorotalonil y del insecticida clorpirifos como fuente de carbono, para su eventual uso en la biorremediación de suelos agrícolas contaminados con plaguicidas.
Metodología
Aislamiento de bacterias con potencial de degradación de clorotalonil y clorpirifos
El medio mínimo mineral (MSM, por sus siglas en inglés) fue utilizado para el aislamiento de las cepas bacterianas a partir de muestras de suelo; este consiste en 0,5 g KH2PO4, 1,2 g Na2HPO4, 0,5 g NH4NO3, 0,2 g MgSO4·7H2O, 5 mg FeSO4·7H2O, y 10 mL de solución de elementos traza (50 mg/L
Ca(OH)2, 30 mg/L H3BO3, 10 mg/L ZnSO4·7H2O, 5 mg/L MnSO4·H2O, 5 mg/L CuSO4·5H2O y 5 mg/L Na2MoO4·2H2O), todo lo anterior, disuelto en un litro de agua destilada (Harada, et al., 2010). Además, se utilizó el medio Luria-Bertani (LB) (25 g/L) para el mantenimiento de las cepas y el crecimiento bacteriano previo a la extracción de ADN.
Las bacterias con potencial de degradación de clorotalonil y clorpirifos fueron aisladas de suelo. Estas muestras se recolectaron en zonas aledañas a fincas productoras de fresa en Fraijanes, Alajuela (10.157317266584888, -84.2198366899621), y en zonas aledañas a fincas productoras de papaya en Parrita, Puntarenas (9.558563030501617,-84.35910780384678).
Para el aislamiento se utilizó la metodología indicada en Zhang, et al., (2014), para lo cual se pesó suelo fresco (un gramo), colocado en matraces de 250 mL y adicionado con 100 mL de MSM suplementado con 20 mg/L de uno de los plaguicidas; clorotalonil o clorpirifos (grado analítico; Dr. Ehrenstorfer, Sigma-Aldrich).
La concentración seleccionada representa aproximadamente, el doble de la dosis comercial promedio recomendada para una aplicación de clorotalonil y más de 10 veces la dosis comercial promedio recomendada para una aplicación de clorpirifos. Los matraces se incubaron en agitador orbital (MRC) a 120 rpm y 30 °C durante siete días.
Pasado este tiempo se tomaron alícuotas de cinco mL de cada uno de los cultivos y se inocularon dos matraces Erlenmeyer conteniendo 100 mL de medio MSM con 20 mg/L de cada uno de los plaguicidas (un matraz por plaguicida), los cuales fueron incubados nuevamente, en las condiciones anteriormente descritas.
Al final del periodo de incubación, se realizaron diluciones decimales de cada uno de los cultivos, para lo cual se tomaron alícuotas de 0,1 mL que se depositaron en placas Petri conteniendo medio LB sólido, las alícuotas se dispersaron con ayuda de un asa de Drigalski. Las placas inoculadas se incubaron a 30 °C durante dos días, posteriormente, las colonias que se observaron fueron purificadas realizando resiembras en medio LB sólido.
Caracterización morfológica y fisiológica
Las cepas aisladas fueron caracterizadas e identificadas preliminarmente, lo cual consistió en determinar su morfología colonial y microscópica, y caracterización fisiológica mediante determinación del perfil bioquímico con el equipo VITEK® 2 GN, que incluyó 48 pruebas diferentes; por ejemplo, actividad de una variedad de arilamidasas, hidrolasas, descarboxilasas, glucosidasas, acidificación del medio por metabolismo de diversas fuentes de carbono, además de fosfatasa, ureasa y otras.
Con base en el perfil bioquímico obtenido, el VITEK® 2 GN reportó una identificación de las bacterias aisladas. Adicionalmente, se realizó una prueba antibiograma para obtener información de perfiles de resistencia a 13 antibióticos diferentes: ampicilina, piperacilina, ceftiofur, amicacina, tobramicina, marbofloxacina, nitrofurantoína, cefpodoxima, imipenem, gentamicina, enrofloxacina, tetraciclina y cloranfenicol y dos mezclas; trimetoprim + sulfa y amoxicilina + ácido clavulánico.
Además, se realizaron cinéticas de crecimiento de las bacterias en medio MSM suplementado con alguno de los dos plaguicidas (clorotalonil o clorpirifos). La caracterización de las cepas se realizó en el laboratorio de Ecotoxicología del IRET, excepto por el perfil bioquímico y antibiograma, los cuales fueron determinados en el laboratorio de Bacteriología de la Escuela de Medicina Veterinaria de la Universidad Nacional (UNA).
La cinética de crecimiento de las bacterias se ejecutó con base en la metodología descrita por Zhang, et al. (2014), para lo cual se prepararon suspensiones en solución salina estéril (80 % p/v) de cada una de las cepas aisladas, utilizando como referencia el estándar de turbidez de Mc Farland 0,5.
Un mL de cada una de las suspensiones ajustadas fue inoculado en matraces Erlenmeyer que contenían 150 mL de MSM suplementado con 20 mg/L de clorotalonil o clorpirifos. Los matraces inoculados se incubaron en agitación orbital (120 rpm) a 30 °C durante 16 días. El crecimiento bacteriano fue determinado mediante medición de la densidad óptica (absorbancia) diariamente en espectrofotómetro (Thermo Fisher Scientific) a una longitud de onda de 600 nm.
Caracterización molecular
La caracterización molecular de las cepas aisladas se realizó en el Laboratorio de Análisis Genómico (LAGen) de la Escuela de Ciencias Biológicas, UNA. Las bacterias se inocularon en cinco mL de caldo LB y se incubaron a 30 °C durante 24 horas para obtener un botón de células suficiente para la extracción de ADN, y para esta se utilizó el protocolo estándar del kit DNeasy Blood & Tissue para células bacterianas (QIAGEN®).
La identificación molecular se realizó mediante la amplificación del gen ADNr 16S utilizando los cebadores universales 27F (5-AGAGTTTGATCMTGGCTCAG-3) y 1492R (5-TACCTTGTTCGACTT-3), los cuales generan un amplicón de 1400 a 1500 pares de bases (pb).
La mezcla de reacción de PCR consistió en 12,5 μL de Dream Taq PCR Master Mix 2X (Thermo Scientific), 0,25 μL de cada cebador (0.1 μM), 10 μL de agua libre de nucleasas y 2 μL de ADN, para un volumen final de 25 μL. El perfil térmico consistió en una desnaturalización inicial de cinco min. a 95 °C, seguido de 36 ciclos de 95 °C por 45 s, 57°C por un min. y una extensión de 72 °C por un min., con una extensión final de 72 °C por cinco min.
Con el fin de obtener la identidad molecular de las bacterias, se secuenciaron los productos PCR mediante química de Sanger en dos direcciones en un analizador genético ABI 3500 (Applied Biosystems, USA). Las secuencias genéticas obtenidas se editaron manualmente en el programa Geneious versión prime 2019 (Biomatters).
La identidad taxonómica preliminar en el nivel de género se realizó con el programa BLASTn (Chen, et al., 2015), mediante parámetros por defecto y disponible en National Center for Biotechnology Information (NCBI, http://www.ncbi.nlm.nih. gov/blast). Posteriormente, se realizó un alineamiento múltiple de las secuencias CP, CT1 y CT2 con 12 secuencias obtenidas del genbank (https://www.ncbi.nlm.nih.gov/ genbank) y utilizando el programa MAFFT v7.308 (Katoh & Standley, 2013) mediante el método iterativo de refinamiento G-INS-i (200PAM/k= 2). Se procedió a realizar una curación del alineamiento con el programa en línea Gblocks (http://phylogeny.lirmm. fr/phylo_cgi/one_task.cgi?task_type=gblocks, Castresana, 2000), obteniendo bloques informativos de 1273 pb.
El mejor modelo de sustitución nucleotídica (TN+F+I) para generar la topología filogenética se obtuvo con el programa ModelFinder (Kalyaanamoorthy, et al., 2017). Seguidamente, con la plataforma en línea IQ-TREE (http://iqtree.cibiv.univie. ac.at) (Nguyen, et al., 2015; Trifinopoulos, et al., 2016) se obtuvieron las inferencias topológicas de posicionamiento taxonómico por máxima verosimilitud.
Los parámetros empleados para la inferencia filogenética fueron el uso de heterogeneidad de tasa libre y un soporte de las ramas mediante un test de rama única (SH-aLRT) con 10000 réplicas y un cálculo de soporte de los nodos derivado del método UFboot con 10000 permutaciones (Minh, et al., 2013). Finalmente, los árboles obtenidos se visualizaron y editaron en el programa FigTree v1.4 (Rambaut, 2009).
Análisis y resultados
Aislamiento, caracterización morfológica y fisiológica e identificación preliminar
Se aislaron dos cepas bacterianas capaces de crecer en MSM suplementado con clorotalonil (como única fuente de carbono), denominadas CT1 y CT2, y una cepa bacteriana capaz de crecer en medio MSM suplementado con clorpirifos (única fuente de carbono), denominada CP. Las cepas CT1 y CT2 se aislaron de suelo proveniente de una zona aledaña a fincas productoras de fresa en Fraijanes, Alajuela, mientras que la cepa CP se aisló de suelo proveniente de una zona aledaña a fincas productoras de papaya en Parrita, Puntarenas. Las tres cepas bacterianas formaron colonias circulares, brillantes, convexas y con bordes enteros. La CT1 y CT2 formaron colonias blanquecinas, y la CP colonias color crema. En el nivel celular, las tres cepas bacterianas eran bacilos Gram negativo.
Por otro lado, las tres cepas bacterianas mostraron perfiles bioquímicos diferentes, lo que se tradujo en la utilización variable de diversas fuentes de carbono debido a la producción de enzimas (tabla 1). La caracterización fisiológica producida por el equipo VITEK® 2 GN es de naturaleza cualitativa, ya que indica la presencia (+) o ausencia (-) de las actividades enzimáticas evaluadas, pero no su magnitud.
Esta información permitió realizar una identificación preliminar de las cepas, indicando las siguientes identificaciones: CT1 = Enterobacter cloacae, CT2 = Ochrobactrum anthropi y CP = Pseudomonas aeruginosa. La identidad de las dos primeras cepas fue reportada con un 99 % de probabilidad, mientras que la tercera se identificó con un 95 % de probabilidad.
De las 48 pruebas realizadas por el VITEK® 2 GN, solo cinco fueron positivas para las tres cepas, que son: actividad de arilamidasas de los aminoácidos prolina y tirosina (ProA y TyrA), uso de citrato (CIT), y la alcalinización del medio por el metabolismo de L-lactato (ILATk) y succinato (SUCT) (tabla1).
Tabla 1 Perfil fisiológico de las tres cepas bacterianas aisladas, a partir de suelo, mediante cultivo de enriquecimiento, adicionado con los plaguicidas clorotalonil o clorpirifos. CP: cepa aislada de medio adicionado con clorpirifos = Pseudomonas aeruginosa; CT1 y CT2: cepas aisladas de medio adicionado con clorotalonil = Enterobacter cloacae y Ochrobactrum anthropi, respectivamente.
| Prueba bioquímica | Cepa | ||
|---|---|---|---|
| CP | CT1 | CT2 | |
| P.aeruginosa | E. cloacae | O. anthropi | |
| Ala-Phe-Pro-arilamidasa (APPA) | - | - | - |
| Glutamil arilamidasa pNA (AGLTp) | - | - | - |
| Beta-alanina arilamidasa pNA (BAIap) | + | - | + |
| L-prolina arilamidasa (ProA) | + | + | + |
| Tirosina arilamidasa (TyrA) | + | + | + |
| Glicina arilamidasa (GlyA) | - | + | + |
| Glu-Gly-Arg-arilamidasa (GGAA) | - | - | - |
| L-pirrolidonil-arilamidasa (PyrA) | - | - | + |
| Beta-glucosidasa (BGLU) | - | - | - |
| Beta-galactosidasa (BGAL) | - | + | - |
| Beta-n-acetil-glucosaminidasa (BNAG) | - | + | - |
| Beta-glucoronidasa (BGUR) | - | - | - |
| Beta-N-acetil-galactosaminidasa (NAGA) | - | - | - |
| Alfa-glucosidasa (AGLU) | - | - | + |
| Alfa-galactosidasa (AGAL) | - | + | - |
| Gamma-glutamil-transferasa (GGT) | + | + | - |
| Ornitina descarboxilasa (ODC) | - | + | - |
| Lisina descarboxilasa (LDC) | - | - | - |
| Descarboxilasa base (0DEC) | - | + | - |
| Beta-xilosidasa (BXYL) | - | + | - |
| Ureasa (URE) | - | - | + |
| Fosfatasa (PHOS) | - | + | |
| Lipasa (LIP) | - | - | - |
| D-maltosa (dMAL) | + | + | - |
| D-manitol (dMAN) | - | + | - |
| D-manosa (dMNE) | + | + | - |
| D-glucosa (dGLU) | + | + | - |
| D-cellobiosa (dCEL) | - | + | - |
| D-sorbitol (dSOR) | - | + | - |
| D-tagatosa (dTAG) | - | - | + |
| D-trehalosa (dTRE) | - | + | - |
| L-arabitol (IARL) | - | - | - |
| Adonitol (ADO) | - | - | + |
| Palatinosa (PLE) | - | + | - |
| Sacarosa/sucrosa (SAC) | - | + | - |
| Citrato (CIT) | + | + | + |
| Malonato (MNT) | - | + | - |
| Cumarato (CMT) | - | - | - |
| Fermentación/ glucosa (OFF) | - | + | - |
| 5-keto-d-gluconato (5KG) | - | - | - |
| L-histidina asimilación (IHISa) | - | - | - |
| L-malato asimilación (IMLTa) | - | - | - |
| L-lactato asimilación (ILATa) | - | - | - |
| L-lactato alcalinización (ILATk) | + | + | + |
| Succinato alcalinización (SUCT) | + | + | + |
| O/129 resistencia (O129R) | - | + | - |
| Ellman (ELLM) | - | - | + |
| H2S (producción H2S) | - | - | - |
Nota: Fuente de elaboración propia con información reportada por el equipo VITEK® 2 GN del Laboratorio de Bacteriología de la Escuela de Medicina Veterinaria de la Universidad Nacional
Catorce de las 48 pruebas fueron negativas para las tres cepas (APPA, AGLTp, GGAA, BGLU, BGUR, NAGA, LDC, LIP, IARL, 5KG, IHISa, IMLTa, ILATa y H2S) (tabla 1). Únicamente una prueba, la acti vidad de la glicina arilamidasa (GlyA) fue positiva para CT1 y CT2, mientras que la beta-alanina arilamidasa (BAIap) fue la única actividad enzimática común a CP y CT2.
Inicialmente, se planteó la hipótesis de que CT1 y CT2 realizarían un mayor número de actividades enzimáticas comunes; sin embargo, los resultados evidenciaron lo contrario, ya que CTI y CP presentaron el a de la Universidad Nacional.
mayor número de actividades enzimáticas comunes, las que incluyen: la gamma-glutamil-transferasa (GGT), y la acidificación del medio por catabolismo de los carbohidratos maltosa (dMAL), manosa (dMNE) y glucosa (dGLU) (tabla 1).
Las actividades enzimáticas probadas en el presente trabajo no necesariamente están directamente relacionadas con la degradación de plaguicidas. Existen reportes que indican que las tres familias enzimáticas más importantes en la degradación de dichas sustancias son las esterasas, glutatión S-transferasas (GSTs) y sistema citocromo P450 (Bass & Field, 2011), las cuales son enzimas específicas que no fueron evaluadas en este trabajo.
No obstante, Trigo, et al. (2009), mencionan que el metabolismo central de la red global de biodegradación incluye una gran variedad de transferasas, isomerasas, hidrolasas y ligasas, posiblemente algunas de las cuales fueron determinadas por el VITEK® 2 GN; por ejemplo, Gamma-glutamil-transferasa (GGT), Beta-glucosidasa (BGLU), Beta-galactosidasa (BGAL), Alfa-glucosidasa (AGLU), ureasa (URE), entre otras.
Adicionalmente, las tres cepas bacterianas presentaron perfiles de resistencia variables a los antibióticos evaluados en el antibiograma realizado por el VITEK® 2 GN. La cepa CTI fue resistente a un antibiótico y una mezcla, mientras que CT2 y CP presentaron resistencia a entre ocho y nueve antibióticos, ya sea individuales o mezclas (tabla 2).
Los resultados muestran que el fenómeno de resistencia a antibióticos se está presentando en bacterias edáficas, muy probablemente en respuesta a la creciente exposición a antibióticos usados en agricultura, medicina veterinaria y humana (Cycoń, Mrozik & Piotrowska-Seget, 2019). En el suelo la transferencia horizontal de material genético (plásmidos) entre bacterias de diferentes especies está ampliamente documentada (Sun, et al., 2019).
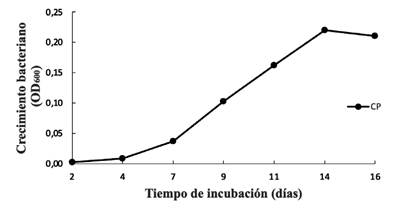
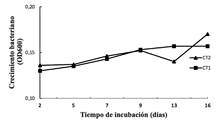
Las cinéticas de crecimiento mostraron comportamientos distintos de acuerdo con el plaguicida utilizado; clorotalonil o clorpirifos (figuras 1 y 2). En general, las cinéticas de crecimiento presentaron un comportamiento
Tabla 2 Lista de antibióticos a los cuales las bacterias aisladas de suelo mediante cultivo de enriquecimiento adicionado con los plaguicidas clorotalonil o clorpirifos presentaron resistencia (R) o sensibilidad (S). CP: cepa aislada de medio adicionado con clorpirifos= Pseudomonas aeruginosa; CT1 y CT2: cepas aisladas de medio adicionado con clorotalonil = Enterobacter cloacae y Ochrobactrum anthropi respectivamente.
| Antibiótico | Cepa | ||
|---|---|---|---|
| CP | CT1 | CT2 | |
| P. aeruginosa | E. cloacae | O. anthropi | |
| Ampicilina | R | S | R |
| Piperacilina | S | S | R |
| Ceftiofur | R | S | R |
| Amicacina | S | S | S |
| Tobramicina | S | S | S |
| Marbofloxacina | S | S | S |
| Nitrofurantoína | R | S | R |
| CefpodoximaS | S | R | S |
| Imipenem | S | S | R |
| Gentamicina | S | S | S |
| Enrofloxacina | S | S | S |
| Tetraciclina | R | S | R |
| Cloranfenicol | R | S | R |
| Trimetoprim + sulfa | R | S | S |
| Amoxicilina + Ácido clavulánico | R | R | R |
Nota: Bacteriología de la Escuela de Medicina Veterinaria de Fuente de elaboración propia con información emitida por el equipo VITEK® 2 GN del Laboratorio de la Universidad Nacional.
esperado; el cual se caracteriza por una fase exponencial seguida de una fase estacionaria. Sin embargo, en el caso de las cepas CT1 y CT2, la etapa exponencial estuvo caracterizada por una pendiente poco pronunciada.
El incremento en la densidad óptica evidenció cierto crecimiento de ambas cepas en medio MSM suplementado con clorotalonil; sin embargo, este crecimiento fue escaso, lo cual indica que las células bacterianas estuvieron bajo estrés, muy probablemente nutricional, debido a dificultades para utilizar la fuente de carbono proporcionada por el clorotalonil (figura 1).
La utilización de clorotalonil como fuente de carbono y energía está vinculada a la gran versatilidad de los microorganismos para utilizar fuentes de carbono diversas. Existen reportes de bacterias con capacidad de utilizar clorotalonil como fuente única de carbono; por ejemplo, Flavobacterium sp. NL0-1 y Ochrobactrum lupini TP-D1 (Katayama, et al., 1991; Shi, et al., 2011).
Por otro lado, también ha sido reportado que este fungicida es pobremente utilizado (y degradado) por los microorganismos cuando se encuentra como fuente única de carbono (Wang, Liang & Li, 2011; Katayama, et al., 1991). En este caso, el uso o degradación de clorotalonil es más eficiente mediante cometabolismo; es decir, cuando la degradación del fungicida ocurre a expensas de otras fuentes de carbono que se encuentran presentes en el medio de manera simultánea al plaguicida.
La función de la fuente adicional de carbono es apoyar el crecimiento microbiano (Motonaga, Takagi & Matumoto, 1996). Por lo tanto, se recomienda realizar ensayos adicionales para verificar si las bacterias aisladas en este estudio logran utilizar clorotalonil de manera más eficiente mediante cometabolismo, aspecto que deberá considerarse también para el diseño de una estrategia de biorremediación de suelos contaminados por dicho fungicida.
La cinética de crecimiento de la cepa CP, inoculada en medio MSM suplementado con clorpirifos como fuente de carbono figura 2), muestra una curva de crecimiento con un aumento exponencial en la densidad óptica del día 7 al 14, indicando el uso del carbono presente en las moléculas de clorpirifos. En este caso, la posibilidad de que el plaguicida pueda ser removido del suelo de manera eficiente por la bacteria se incrementa, facilitando la incorporación de esta bacteria en una estrategia de biorremediación de suelos contaminados con clorpirifos.
Nota: Fuente de elaboración propia
Figura 1 Cinética de crecimiento de las cepas CT1 y CT2 en medio MSM suplementado con el fungicida clorotalonil (20 mg/L) como fuente única de carbono.
Es importante mencionar que las cepas CT1 y CP formaron flóculos en el medio MSM, esto puede dificultar técnicamente la medición de la densidad óptica; no obstante, esta característica puede ser de utilidad en la biorremediación de suelos, ya que se mejora la interacción entre células y con las sustancias químicas al aumentarse la superficie de contacto favoreciendo la degradación del contaminante (Mau, Vega & Sánchez, 2011).
Caracterización molecular
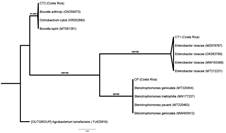
La secuencia obtenida de la amplificación parcial del gen ADNr 16S para la cepa CT1, se agrupó en la topología de posicionamiento taxonómico con secuencias de la especie Enterobacter cloacae(figura 3). Por otro lado, la cepa CT2 se relaciona filogenéticamente con Brucella
Nota: Fuente de elaboración propia
Figura 3 Topología de posicionamiento taxonómico por máxima verosimilitud (ML) de las secuencias parciales del gen ADNr 16S de este estudio, con secuencias obtenidas del genbank (el número de accesión se muestra entre paréntesis). En las ramas, los números son porcentajes que se muestran como soporte de test de rama única (% SH-aLRT)/soporte bootstrap ultra-rápido (% UFboot). El grupo externo de esta topología es A. tumefaciens.
anthropi, recientemente conocida como Ochrobactrum anthropi, con B. lupini, recientemente conocida como O. lupini y con O. cytisi (figura 3), resultados que coinciden en el nivel de género con la identificación obtenida mediante el perfil fisiológico de las cepas por la plataforma VITEK® 2 GN.
Para la cepa CP se mostró una relación taxonómica con la agrupación de Stenotrophomonas sp. (figura 3). Este hallazgo coincide parcialmente, con la identificación realizada mediante determinación del perfil fisiológico, ya que S. maltophilia es equivalente a Pseudomonas hibiscicola.
Usualmente, S. maltophilia es confundida con P. aeruginosa debido a que presentan muchas características similares (Spierer, Miller & O'Brien, 2018; Lee et al., 2012); por ejemplo, comparten aspectos estructurales de la fimbria, lo cual está implicado en procesos de adhesión y producción de biofilms en la naturaleza (Ryan, et al. 2009).
Los altos valores porcentuales de soporte de las ramas (entre 91 y 100), indican que los tres agrupamientos son robustos, demostrando que las cepas evaluadas con potencial biotecnológico de degradación de plaguicidas: CT1 (clorotalonil), CT2 (clorotalonil) y CP (clorpirifos) se localizan taxonómicamente, en los géneros Enterobacter, Ochrobactrum y Stenotrophomonas, respectivamente.
Conclusiones
Se aisló una cepa bacteriana con la capacidad de utilizar clorpirifos como fuente de carbono y energía, la cual, de acuerdo con los análisis moleculares, fue identificada como Stenotrophomonas sp. Los resultados de la cinética de crecimiento indican que es viable incorporar esta bacteria dentro de una potencial estrategia de biorremediación de suelos contaminados con clorpirifos, ya que esta molécula puede ser utilizada de manera eficiente por la bacteria.
Por otro lado, se aislaron dos cepas bacterianas con potencial de degradación de clorotalonil, ya que mostraron tolerancia a dicha sustancia y capacidad parcial de utilizar este fungicida como fuente de carbono y energía para su crecimiento. Las cepas fueron identificadas como Enterobacter cloacae y Ochrobactrum sp.
Se sugiere realizar ensayos para determinar si el proceso de cometabolismo contribuye a incrementar la eficiencia de degradación de clorotalonil por parte de E. cloacae y Ochrobactrum sp. y de esta manera incorporar estos microorganismos en una estrategia de biorremediación de suelos contaminados con clorotalonil.
Si bien existen reportes sobre la capacidad de estas especies bacterianas de utilizar los plaguicidas clorotalonil y clorpirifos contribuyendo a su remoción en el suelo, es necesario realizar un llamado de atención sobre el régimen de uso de estos dos plaguicidas, los cuales se utilizan intensivamente en la agricultura costarricense.
Por otro lado, es necesario considerar el uso de antibióticos, ya sea en la agricultura, ganadería, medicina humana y veterinaria, ya que existe información creciente sobre la selección de cepas resistentes y multirresistentes a los antibióticos de mayor uso. Este aspecto es crítico para una estrategia de biorremediación de suelos contaminados con plaguicidas, ya que, a pesar de ser una opción, la diseminación de microorganismos edáficos resistentes constituye un posible riesgo para la salud pública global.
Financiamiento
La presente investigación fue financiada por el Fondo Institucional de Desarrollo Académico (FIDA) de la Universidad Nacional de Costa Rica, proyecto ''Estrategia integral para disminuir el uso de plaguicidas y mejorar la calidad ambiental en Zarcero, Alajuela''código SIA 0496-16.
Agradecimiento
A las estudiantes Johanna Ramírez Ruíz, Violeta Oliva Mercado y Stephanny Sánchez Vargas por su contribución al trabajo desarrollado con las cepas bacterianas en el marco del curso de Biotecnología Aplicada del programa de Bachillerato en Biología con énfasis en Biotecnología de la Escuela de Ciencias Biológicas, UNA.
Declaración de la contribución de los autores
Todos los autores afirmamos que se leyó y aprobó la versión final de este artículo. El porcentaje total de contribución para la conceptualización, preparación y corrección de este artículo fue el siguiente: J.E.G 35 %, R.U.C 20 %, C.S.B. 20 % y M.O.A. 25 %.