Introducción
Los insectos necrófagos y en especial los Dípteros, son de gran interés al ser los principales responsables de la reducción de materia orgánica en descomposición, incluidos los restos cadavéricos1. Tienen una asociación directa con la materia orgánica en putrefacción, el estudio de la entomofauna necrófaga es fundamental para la aplicación de la Entomología Forense2. Esta disciplina forense es la encargada de examinar la fauna entomológica, con el objetivo de extraer información, para precisar el tiempo, lugar y modo en el que ha tenido lugar un suceso3.
En Bolivia, se han realizado escasas investigaciones sobre la sucesión de la entomofauna cadavérica, siendo el biomodelo más utilizado el cerdo doméstico (Sus scrofa)4,5,6,7 Los estudios realizados tuvieron el propósito de conocer a los insectos presentes en cadáveres en sus diferentes etapas de descomposición, los de mayor importancia fueron los Dípteros de las familias Calliplioridae, Muscidae y Sarcophagidae, pero no se logró identificar las especies en diferentes etapas de su desarrollo, huevos, larvas, puparium y adultos8 . Para relacionar las distintas fases de descomposición, así como estimar el tiempo de muerte (Intervalo Post - Mortem-IPM), conlleva a la cría de larvas en el laboratorio, recolectar pupas, lo que se traduce a un gasto de tiempo y recursos, debido a que es una labor compleja8 y que además requiere del conocimiento taxonómico del especialista, confiando a menudo en el reconocimiento de pequeñas variaciones en las características fenotípicas9 . No obstante las semejanzas morfológicas de las diferentes especies, especialmente en estadios inmaduros, dificulta su identificación y esto ocasiona a generar interés por el desarrollo de nuevas alternativas, que ayuden a resolver estos problemas taxonómicos de manera exacta y certera.
En este sentido, se ha desarrollado técnicas en biología molecular que facilitan la determinación de especies empleando el material genético de los mismos. El estudio del sistema de Códigos de Barras, en insectos, se desarrolla en el análisis del ADNmt que comprende alguna región de los genes que codifican el COI o COII9. La obtención de secuencias de ADNmt han sido a partir del Gen COI, este gen es propuesta como Barcode estándar para la mayoría de grupos de animales, es una región de 648 pares de base del gen mitocondrial Citocromo C Oxidasa Sub Unidad 1 (COI), cuya efectividad ha sido demostrada en varios trabajos de identificación de aves, peces, mariposas, moscas, entre otros10,11
El gen COI, es de amplio uso para identificar especies de insectos problema8 en cualquier estadio inmaduro, así como con un simple fragmento de insecto que, en la mayoría de los casos no es tomado en cuenta debido a la dificultad de identificar claves dicotómicas certeras. La identificación taxonómica molecular genética de los insectos necrófagos, permite clasificar al insecto en cualquier estadio del ciclo de vida y a partir de cualquier fragmento o estructura o tejido del mismo en un periodo de tiempo. Además de poder clasificarlos a nivel de especies y subespecies.
El objetivo del presente estudio fue de realizar la identificación molecular rápida de la entomofauna cadavérica en diferentes biomodelos para su posterior aplicación en investigaciones criminales.
Es así que el presente estudio examina a la ciencia entomológica desde un punto de vista de la biología molecular moderna como un método complementario en datación de muerte y otros, tratando de contribuir a las instituciones jurídicas del Estado Boliviano, Ministerio Público y Policía Boliviana, a la resolución rápida de la investigación forense evitando de esta manera, la retardación de justicia de cualquier caso.
Materiales y métodos
Se tomó muestras de huevos, larvas en diferentes estadios y adultos a partir de los biomodelos (S. scrofa, Cerdo domestico), muestras proporcionadas por la Dra. Castillo y el Tte. Bernal, con un intervalo postmordem no mayor a 24 hrs. (Fig. 1), a los cuales se realizó una disección del insecto adulto de cabezas con un peso estimado de 0,17g.
La extracción del material genético se realizó con el sistema Wizard® Genomic DNA Purification Kit (Promega) de acuerdo al protocolo de Miller12. Para la amplificación de Barcode se empleó la pareja de cebadores universales LCO1490 y HCO2198 descritos por Hebert13 que permiten la amplificación de un fragmento de 709 pb. Para la reacción de PCR se realizó empleando el kit comercial GoTaq® Colorless Master Mix (Promega). Los productos amplificados fueron sometidos a una purificación alcohólica, realizándose posteriormente el PCR desbalanceado empleando el kit BigDye® Terminator v3.1 Cycle Sequencing Kit (Applied Biosystems). Este producto fue secuenciado en un Analizador Genético ABI 3500 (Applied Biosystems) de acuerdo a las especificaciones del fabricante.
Las secuencias obtenidas fueron analizadas con el programa Sequencing Analysis v.6.0. (Applied Biosystems). Las secuencias se alinearon con el programa Mega 5.1. Las secuencias parciales y completas del gen citocromo oxidasa I (COI) y relacionadas a las especies fueron descargadas del GenBank14 y del Boldsystems v.3.0.15
Cabe mencionar que los árboles filogenéticos fueron construidos a partir de la plataforma bioinformática del Blast Tree View, generados con los repositorios de los datos del Genbank.
Resultados
El análisis de secuencias incluyó 600 pares de bases correspondientes a la región 3´del gen COI, de donde la identidad de las secuencias se verificó por comparación con la secuencia de referencia de Ianius mosca, cuya secuencia se encuentra depositada en GenBank con número de acceso MF130285.1. En total se obtuvieron diez secuencias. Se presenta la clasificación taxonómica molecular de 5 muestras de insectos adultos, observándose en la muestra CG-M3-ENT una probabilidad de correspondencia de especie (P.C.) del 100%, la muestra CG-M5-ENT presenta una P.C. de especie de 99,8% y la muestra CG-M6-ENT tiene una P.C. de especie de 94%. (Tabla 1)
Tabla 1 Resultados de los algoritmos de las bases de datos mundiales BoldSystems y GenBank, Estadio Adulto. NDB: No Determinado en Bases de Datos Mundiales.
| No. | Código CINGEN | Estadio | Nivel Taxonómico | Asignación de Taxón | Probabilidad de Correspondencia (%) |
| 1 | CG-M2-ENT | ADULTO | Filo | NDB | NDB |
| Clase | NDB | NDB | |||
| Orden | NDB | NDB | |||
| Familia | NDB | NDB | |||
| Género | NDB | NDB | |||
| Especie | NDB | NDB | |||
| 2 | CG-M3-ENT | ADULTO | Filo | Arthropoda | 100 |
| Clase | Insecta | 100 | |||
| Orden | Diptera | 100 | |||
| Familia | Calliphoridae | 100 | |||
| Género | Sarconesia | 100 | |||
| Especie | Sarconesia chlorogaster | 100 | |||
| Filo | NDB | NDB | |||
| Clase | NDB | NDB | |||
| 3 | CG-M4-ENT | ADULTO | Orden | NDB | NDB |
| Familia | NDB | NDB | |||
| Género | NDB | NDB | |||
| Especie | NDB | NDB | |||
| Filo | Arthropoda | 100 | |||
| Clase | Insecta | 100 | |||
| 4 | CG-M5-ENT | ADULTO | Orden | Diptera | 100 |
| Familia | Calliphoridae | 100 | |||
| Género | Sarconesia | 100 | |||
| Especie | Sarconesia versicolor | 99,8 | |||
| Filo | Arthropoda | 100 | |||
| Clase | Insecta | 100 | |||
| 5 | CG-M6-ENT | ADULTO | Orden | Diptera | 100 |
| Familia | Fanniidae | 100 | |||
| Género | Fannia | 100 | |||
| Especie | Fannia sp | 94 |
Fuente: (CINGEN, IITCUP).
La muestra identificada como CG-M2-ENT (resaltado en amarillo) no fue encontrada en ninguna de las bases de datos del GenBank y del Boldsystems. Sin embargo se encuentra emparentado con secuencias de Sarconesia chlorogaster (Fig. 2). La muestra identificada como CG-M4-ENT (resaltado en amarillo) no fue encontrada en ninguna de las bases de datos del GenBank y del Boldsystems. Sin embargo se encuentra relacionado con secuencias de Sarconesia versicolor (Fig. 3).
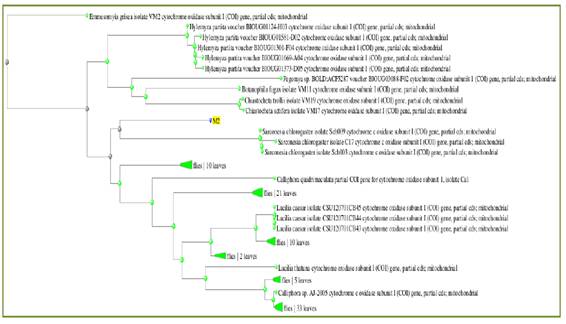
Figura 2 Árbol filogenético de la especie identificada, CG-M2-ENT (resaltado en amarillo), construido con programa Blast Tree View, método de Neighbor Joining (Saitou and Nei 1987) considerando 0.75 de Diferencia máxima de Secuencia y una distancia de Jukes-Cantor (1969). Fuente: (GENBANK)
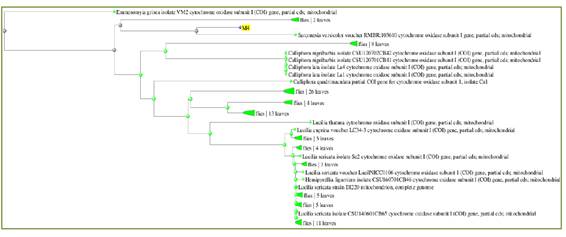
Figura 3 Árbol filogenético de la especie identificada, CG-M4-ENT (resaltado en amarillo), construido con programa Blast Tree View, método de Neighbor Joining (Saitou and Nei 1987) considerando 0.75 de Diferencia máxima de Secuencia y una distancia de Jukes-Cantor (1969).Fuente: (GENBANK).
Se presenta la clasificación taxonómica molecular de las 4 muestras de diferentes estadios inmaduros: huevos, larvas de primer, segundo y tercer estadio. Las muestras de CG-M17-ENT y CG-M19-ENT se observa la P.C. de la especie en un 100% y las muestras CG-M18-ENT y CG-M20-ENT presentan un P.C. de la especie del 99.8%. (Tabla 2)
Tabla 2 Resultados de los algoritmos de las bases de datos mundiales BoldSystems y GenBank, de las diferentes fases del ciclo biológico del insecto
| Código CINGEN | Estadio | Nivel Taxonómico | Asignación de Taxón | Probabilidad de Correspondencia (%) | |
| Filo | Arthropoda | 100 | |||
| INSTAR | Clase | Insecta | 100 | ||
| 1 | CG-M17-ENT | PRIMER | Orden | Diptera | 100 |
| Familia | Calliphoridae | 100 | |||
| Género | Sarconesia | 100 | |||
| Especie | Sarconesia chlorogaster | 100 | |||
| Filo | Arthropoda | 100 | |||
| Clase | Insecta | 100 | |||
| 2 | CG-M18-ENT | SEGUNDO | Orden | Diptera | 100 |
| INSTAR | Familia | Calliphoridae | 100 | ||
| Género | Sarconesia | 100 | |||
| Especie | Sarconesia chlorogaster | 99,8 | |||
| Filo | Arthropoda | 100 | |||
| Clase | Insecta | 100 | |||
| 3 | CG-M19-ENT | TERCER | Orden | Diptera | 100 |
| INSTAR | Familia | Calliphoridae | 100 | ||
| Género | Sarconesia | 100 | |||
| Especie | Sarconesia chlorogaster | 100 | |||
| Filo | Arthropoda | 100 | |||
| Clase | Insecta | 100 | |||
| 4 | CG-M20-ENT | HUEVO | Orden | Diptera | 100 |
| Familia | Calliphoridae | 100 | |||
| Género | Sarconesia | 100 | |||
| Especie | Sarconesia chlorogaster | 99,8 |
Fuente: CINGEN, IITCUP
Discusión
En vista de que es crucial la identificación de la fauna entomológica cadavérica para ayudar a establecer el IPM en el curso de la investigación de los hechos, la determinación precisa de las especies de las larvas halladas en los cadáveres, así como de las pupas que los rodean es de suma importancia y, pese a la escases de estudios que emplean secuencias de COI para identificar factores de importancia forense16, los resultados obtenidos del análisis genético de la presente investigación muestra la caracterización taxonómica de cinco especímenes adultos obteniendo una probabilidad de correspondencia igual al 100% en dos muestras y de 99.8% en una muestra (Tabla 1), dos muestras no fueron encontradas en las bases de datos del GenBank y del Boldsystems. En la figura 1, la muestra M2 (resaltado en amarillo) se encuentra en la rama de especímenes Sarconesia chlorogaster. No obstante esta secuencia es diferente a los especímenes comparados llevándonos a sugerir una subespecie. En la figura 2, la muestra M4 (resaltado en amarillo), secuencia no hallada en las bases de datos internacionales, tiene semejanza a la secuencia de referencia Sarconesia versicolor.
Así mismo, se ha logrado realizar el análisis genético de diferentes fases del ciclo biológico del insecto (huevo, instar 1, instar 2 e instar 3), en vista de, que la información específica contenida en la molécula de ADN y en particular en el gen COI, se caracteriza por su inmutabilidad durante todos los cambios del insecto (tabla 2). Las secuencias de COI de cada especie fueron únicas y distinguibles entre sí, aunque mostraron alta homología. Este trabajo puso de manifiesto que la identificación de especies de dípteros inmaduros por análisis de secuencias del ADN resulta sencilla, economiza tiempo al eliminar la necesidad de esperar a la emergencia del adulto y elimina el requerimiento de un conocimiento altamente especializado de claves morfológicas. Además, proporciona información, no sólo del Intervalo Post-Mortem, sino también de las condiciones ambientales que rodean a los cuerpos17.
Conclusiones
Teniendo en cuenta los resultados obtenidos, podría concluirse que el uso del Citocromo Oxidasa Subunidad I (COI) como herramienta en la identificación taxonómica se mostró una región de fácil amplificación y secuenciación, que pueden ser utilizadas como complemento en la identificación de dípteros de interés forense, lo que daría un mayor soporte y consistencia a las investigaciones criminales. La metodología utilizada y propuesta por el sistema de identificación universal "ADN Barcode "fue eficiente en la identificación de la mayoría de las especies de la Familia Calliphoridae y Muscidae, objetos de estudio del presente trabajo, sobre todo en la identificación de los especímenes de diferentes fases del ciclo biológicos del insecto.
La presente investigación ha identificado secuencias genéticas del gen mitocondrial Citocromo Oxidasa I, COI (COI Barcode) no incluidos en las bases de datos mundiales de GenBank y Boldsystems, por lo tanto se considera un aporte científico al incorporar nuevos datos al sistema.
Sin embargo su aplicación no estaría completa si no se cuentan con los especímenes identificados en otras épocas del año, y en las distintas regiones geográficas del país para tener una base de datos completa, esta situación obliga a que se realicen más investigaciones tomando en cuenta todas las características mencionadas, para así tener una mejor aplicabilidad por el personal involucrado en investigación judicial: policía, médicos forenses, criminalistas, etc. Y lograr aplicar como pericia de manera eficiente en un marco estrictamente académico y científico.