Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Médica del Hospital Nacional de Niños Dr. Carlos Sáenz Herrera
versión impresa ISSN 1017-8546
Rev. méd. Hosp. Nac. Niños (Costa Rica) vol.40 no.2 San José ene. 2005
Revisión de la bibliografía sobre AmpC: Una importante β-lactamasa
Cendry Alfaro Rojas 1
1 Laboratorio Clínico, Hospital Nacional de Niños "Dr. Carlos Sáenz Herrera", Caja Costarricense de Seguro Social. Correo electrónico: calfaror@hnn.sa.cr
Introducción
Las β-lactamasas son enzimas sintetizadas por las bacterias, ya sea Gram negativo o Gram positivo y que actϊan como un mecanismo de defensa contra los antibióticos β-lactámicos. Su mecanismo de acción se fundamenta en la unión irreversible al antibiótico, inhabilitando la capacidad de romper la pared bacteriana y permitiendo a la bacteria continuar con su crecimiento, aún en presencia del antibiótico (1,6). Actualmente, debido a la gran cantidad de diferentes β-lactamasas que se han descrito, se implementaron dos clasificaciones: la de Ambler (1), que incorpora características moleculares, y la de Bush et al.(6), la cual incluye parámetros fenotípicos.
Las β-lactamasas clasificadas como AmpC son enzimas con serinas en el sitio activo y funcionan principalmente como cefalosporinasas. En muchos organismos Gram negativo, la expresión de los genes ampC cromosomales es baja, pero inducible en respuesta a algunos estímulos (14,18). Otros organismos, como Escherichia coli, presentan un gen ampC cromosomal que se expresa de manera constitutiva. Sin embargo, recientemente se ha descrito la transferencia horizontal de los genes ampC desde Enterobacter cloacae, Citrobacter freundii, Morganella morganii y otros, hacia especies como E. coli, Klebsiella pneumoniae y Salmonella ssp, a través de plásmidos que acarrean los componentes para esta inducción (4,11,47,54,55). Desde el punto de vista clínico, esto trae implicaciones muy grandes, con la falla en el tratamiento con antimicrobianos, que antiguamente eran utilizados en las bacterias que acarreaban estos genes de resistencia (7).
La inducción de AmpC requiere de la exposición de la bacteria a drogas β-lactámicas o a otros estímulos, lo cual está ligado a la vía de reciclaje de la pared celular (10,17); esta inducción abarca de AmpC incluye diferentes productos génicos asociados a esta vía, los cuales incluyen AmpR, AmpD y AmpG, además de precursores muropéptidos, que al parecer actúan como cofactores (9,10,17,40,48,53,56).
Actualmente, algunas publicaciones mencionan la relevancia de cuatro tipos de enzimas que están llegando a ser importantes en nuevos aislamientos clínicos y en la resistencia en bacterias Gram negativo. Estos son: las β-lactamasas de espectro extendido (ESBL), las β-lactamasas con sensibilidad reducida a los inhibidores de β-lactamasas, las β-lactamasas AmpC mediadas por plαsmidos y las β-lactamasas que hidrolizan carbapenems (perteneciendo a los grupos C, A y D, A y B de la clasificación de Ambler (1) o a los grupos 1, 2 2be y 3a, 3b, y 3c de la clasificación de Bush) (6,51).
En este resumen se hará énfasis en las β-lactamasas del tipo AmpC, las cuales pasaron de ser elementos cromosomales de cierto grupo de bacterias, a formar parte de elementos móviles, como los plásmidos, y a llegar a formar parte importante de la multiresistencia a cefalosporinas de bacterias muy prevalentes en aislamientos clínicos, como E. coli y K. pneumoniae. A nivel de laboratorio clínico, este conocimiento es fundamental para la implementación de nuevas técnicas de diagnóstico y para el manejo clínico de pacientes portadores de cepas de este tipo (36,39).
Inducción de la β-lactamasa AmpC
Las β-lactamasas AmpC han sido objeto de estudio desde la dιcada de los años 70. La mayoría de estas enzimas son cefalosporinasas, pero son capaces de hidrolizar todos los antibióticos β-lactámicos. Muchos estudios han analizado las características de ambas AmpC, inducibles y no inducibles, como las propiedades físicas, actividad hidrolítica, los mecanismos involucrados en la expresión cromosomal, además de estudios comparativos entre géneros en el potencial inductor de la enzima (14). En las tablas 1 y 2 se muestran los géneros más estudiados y la capacidad de inducción.
Tabla Nº 1
Enterobacteriaceae productoras de β-lactamasas AmpC induciles*
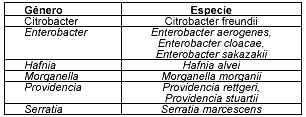
Tabla Nº 2
Potencial inductor consenso para antibióticos β-lactámicos*

En los años 80, estos genes cromosomales inducibles fueron detectados en plásmidos (la mayoría aún sin las capacidades de inducción) y transferidos a organismos que típicamente no expresan este tipo de enzimas, como E.coli, K. pneumoniae o Salmonella ssp (4,11,35,36,37,41). Esto ha venido a complicar el trabajo en la microbiología clínica, sobretodo en el ámbito hospitalario, ya que actualmente no se puede "predecir" a partir de su identificación, si una bacteria es acarreadora de un gen ampC, lo que le conferiría resistencia a antibióticos β-lactámicos. Otro gran problema que se introduce con esta diseminación horizontal, es que la detección fenotípica de estos plásmidos es muy difícil y se confunde con las presentaciones de las β-lactamasas de espectro extendido (ESBL).
Los mecanismos de expresión del gen ampC han sido analizados usando dos organismos modelos, E. cloacae y C. freundii (2, 3, 10, 12, 15, 16, 23, 25, 26, 27, 28, 29, 30, 31, 32, 38, 44, 48).
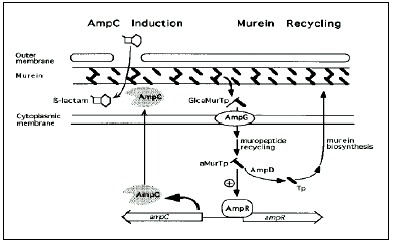
Dicha expresión es un proceso complejo, que primariamente es regulado por el producto del gen ampR, AmpR, además de otros dos genes ampD y ampG, estos últimos expresados en todas las enterobacterias, incluyendo las que no producen AmpC, participando primariamente en el reciclaje de los componentes de la pared bacteriana.
AmpR fue descrito en un inicio en E. cloacae y C. freundii, genéticamente ubicado junto al gen ampC, formando la región control ampRC, la cual se encuentra sólo en las bacterias Gram negativo con producción inducible de AmpC, opuesto a las que tienen una expresión constitutiva, como E. coli, donde el gen ampR fue eliminado (16, 31). AmpR es una proteína que se une al ADN y funciona como proteína reguladora, uniéndose al promotor de ampR y corriente arriba del promotor de ampC, trabajando entonces como estimulador de la expresión de AmpC y como autoregulador negativo. Mutaciones en ampR se han descrito en aislamientos clínicos, donde la expresión de AmpC aumenta a niveles muy altos, aún sin inducción (28). Estas bacterias presentan un fenotipo similar al mostrado por cepas productoras de ESBL.
AmpD es una N-acetilmuramil-L-alanina amidasa, que participa en el reciclaje de los fragmentos de peptidoglicán. AmpD funciona como un regulador negativo de la expresión de ampC y su participación en este proceso se descubrió originalmente en cepas sobreproductoras de AmpC, donde se encontró que la mutación causante de dicho fenotipo se localizaba en ampD, la cual estaba inactiva y por lo tanto, no ejercía su efecto regulador (15,17).
La verdadera inducción de la producción de AmpC, está relacionada con las vías de reciclaje del peptidoglicán. Los antibióticos β-lactámicos producen perturbaciones en la pared, al ser su sitio blanco, obligando a las bacterias a activar sus mecanismos de defensa, en este caso, las β-lactamasas (ver figura 1). Las propuestas mαs estudiadas han postulado como el verdadero inductor de AmpC, a varias moléculas que se acumulan durante el proceso de reciclaje y rompimiento de la pared producido por los antibióticos β-lactámicos, entre ellos el anhidroMurNAc-tripéptido (17) o el GlcNAc-anhMurNAc-peptapéptido (10). Ambas moléculas se han encontrado acumuladas en grandes cantidades en el espacio periplásmico, durante el curso de terapias con antibióticos β-lactámicos, como el imipenem y la cefoxitina y se les ha relacionado con la activación de AmpR y la consecuente expresión de AmpC (49).
Figura 1
Interconexión entre las vías de reciclaje de peptidoglicán y la inducción de AmpC*
β -lactamasas de tipo AmpC acarreadas por plásmidos, un nuevo problema en multirresistencia
Las AmpC que han demostrado ser codificadas cromosomalmente, han sido descritas en bacterias como Acinetobacter spp., Aeromonas spp., Chromobacterium violaceum, C. freundii, Enterobacter spp., E. coli, Morganella morganii, Proteus rettgeri, Providencia stuartii, Pseudomonas aeruginosa, Serratia marcescens, Yersinia enterocolitica, entre otras (5,13,18,29). La resistencia a antibióticos, aparece inicialmente en organismos como Enterobacter cloacae, Citrobacter freundii, Serratia marcescens, y Pseudomonas aeruginosa (19,20,34,42,43), que podrían sobreproducir su β-lactamasa AmpC por mutación, generándose fenotipos de resistencia a ambos oxiimino- y 7-β-metoxicefalosporinas y monobactamas (46). En otros organismos, como Klebsiella pneumoniae, Escherichia coli, Salmonella spp., y Proteus mirabilis, fue apareciendo un fenotipo similar, el cual fue atribuido a ESBL en plásmidos, pero a diferencia de AmpC, no eran resistentes a los 7-β-metoxicefalosporinas (cefoxitin y cefotetan) y sí eran bloqueadas por el ácido clavulánico, sulbactam o tazobactam. Con el uso de cefoxitin y cefotetan, así como de los inhibidores de β-lactámicos para contrarrestar estas nuevas cepas, los plásmidos acarreadores de β-lactamasas AmpC empezaron a ser descritos.
La primera prueba de que una β-lactamasa AmpC habνa sido capturada en un plásmido, fue demostrado por Papanicolaou et al. en 1988, quienes describieron una enzima (MIR-1) con propiedades bioquímicas idénticas y con 90% de identidad con el gen ampC de E. cloacae, expresada en K. pneumoniae. Dichos reportes se han realizado a lo largo del mundo. Estas cepas fueron consistentemente resistentes a aminopenicilinas (ampicilina y amoxicilina), carboxipenicilinas (carbenicilina o ticarcilina) y ureidopenicilinas (piperacilina), a cefalosporinas de los grupos oxiimino (ceftazidime, cefotaxime, ceftriaxone, ceftizoxime, cefurotime) y al grupo metoxi (cefoxitin, cefotetan, cefmetazole, moxalactam). Las enzimas también fueron activas contra aztreonam, aunque la sensibilidad contra cefepime y cefpirome fue poco afectada y no cambió para los carbapenems (imipenem y meropenem) (4,11,45,47,54,55).
Altos niveles de expresión de AmpC
La resistencia de los miembros de la familia Enterobacteriacea a las cefalosporinas de tercera generación constituye un serio problema clínico. Esta resistencia se da por la sobreexpresión constitutiva del gen ampC cromosomal, que originalmente, en estas cepas, es de bajo nivel y no es inducible, y no solo por la presencia de un plásmido que acarrea un gen ampC procedente de otros géneros, como se trató en la sección anterior. En E. coli se han encontrado mutaciones en las cajas -35 y -10 y en los segmentos Inter. cajas de las regiones promotoras (8,35,50,52), lo cual lleva a un promotor tan fuerte, que desencadena una sobre expresión de AmpC. En estos casos, las mutaciones en estos segmentos del ADN que forman parte de la región promotora del gen (cajas), provoca que la ARN polimerasa se una con más afinidad al ADN, provocando una mayor síntesis de ARNm y por lo tanto, llevando a una mayor síntesis de β-lactamasa. Los patrones fenotípicos encontrados van desde resistencia solamente a cefoxitin, hasta multi resistencia a cefalosporinas de tercera generación, dependiendo del tipo de mutación que se haya producido.
Cepas sobreproductoras se han encontrado además en E. Cloacae (20,23,28,46) y P. Aeruginosa (21,22,24,33,57), donde también se corre el peligro de que estas nuevas porciones mutadas se pasen a elementos móviles, como plásmidos, y se diseminen a otros géneros bacterianos.
Conclusiones
El conocimiento de los mecanismos genéticos responsables de los patrones de resistencia, que actualmente encontramos en los aislamientos clínicos, es fundamental, como parte integral del gran problema y la epidemia de multi resistencia que se está presentando en los centros hospitalarios a nivel mundial, donde Costa Rica no es la excepción. A partir de dicho conocimiento, se pueden planear estrategias de vigilancia epidemiológica y control de infecciones, para monitorear la aparición de nuevas cepas multi resistentes, con patrones de susceptibilidad presuntivos de la presencia de una β-lactamasa AmpC. Otro aspecto muy importante, es el planeamiento, a partir del personal mιdico, de las estrategias terapéuticas ideales para utilizar en estas situaciones, donde la ayuda brindada por el personal de laboratorio es indispensable para analizar y atender estos casos.
A modo de recomendación para el personal médico, el reporte del laboratorio respecto a la presencia de una cepa productora de AmpC o de una ESBL, conlleva el no utilizar ningún antibiótico β-lactαmico. Por otro lado, si no se hace una búsqueda específica de estos elementos moleculares, la cepa puede ser reportada como sensible a los β-lactámicos y una vez que se induzca en el paciente la producción de los elementos moleculares antes mencionados y se tenga el desarrollo de resistencia en el paciente, se documentaría eso como un fallo terapéutico. Recordemos que muchos de los procesos moleculares descritos, ocurren a niveles muy bajos, ya que son inducidos y es en el paciente donde se manifiestan y pueden pasar desapercibidos, si no existe una cultura en el laboratorio, de hacer una búsqueda expresa de ellos.
Bibliografía
1. Ambler, R. The Structure of β-lactamases. Philos Trans R Soc Lond Biol Sci. 289: 321, 1980. [ Links ]
2. Barlow, M. & Hall, B. Origin and Evolution of the AmpC β-Lactamases of Citrobacter freundii. Antimicrob Agents Chemother. 46: 1190, 2002. [ Links ]
3. Barnaud, G., et al. Cloning and Sequencing of the Gene Encoding the AmpC β-lactamase of Morganella morganii. FEMS Microbiol Letter. 148:15,1997. [ Links ]
4. Barnaud, G., et al. Salmonella enteritidis: AmpC Plasmid-Mediated Inducible β-lactamase (DHA-1) with an ampR Gene from Morganella morganii. Antimicrob Agents Chemother. 42: 2352, 1998. [ Links ]
5. Bonnet, R., et al. Inducible AmpC β–Lactamase of a New Member Enterobacteriaceae. Antimicrob Agents Chemother. 46: 3316, 2002. [ Links ]
6. Bush, K., et al. A Functional Classification Scheme for β-Lactamases and Its Correlation with Molecular Structure. Antimicrob Agents Chemother. 39:1211, 1995. [ Links ]
7. Bush, K. New β-Lactamases in Gram-Negative Bacteria: Diversity and Impact on the Selection of Antimicrobial Therapy. Clin. Infec. Dis. 32:1085, 2001. [ Links ]
8. Corvec, S., et al. Mutation in the ampC Promoter Increasing Resistance to β-lactamase in a Clinical Escherichia coli Strain. Antimicrob Agents Chemother. 46: 3265, 2002. [ Links ]
9. Dietz, H., et al. Location of N-Acetylmurayl-L-Alanyl-D-Glutamylmesodiaminopimelic Acid, presumed Signal Molecule for β-Lactamase induction, in the Bacterial Cell. Antimicrob Agents Chemother. 40: 2173, 1996. [ Links ]
10. Dietz, H., et al. The Signal Molecule for β-lactamase Induction in Enterobacter cloacae Is the Anhydromuramyl- Pentapeptide. Antimicrob Agents Chemother. 41: 2113, 1997. [ Links ]
11. Fortineau, N., et al. Plasmid-mediated and Inducible Cephalosporinase DHA-2 from Klepsiella pneumoniae. J. Antimicrobial Chemother. 47: 207, 2001. [ Links ]
12. Genereux, C., et al. Mutational analysis of the catalytic centre of the Citrobacter freundii AmpD N-acetylmuramyl-L-alanine amidase. Biochem J. 377: 111, 2004. [ Links ]
13. Handson, N. AmpC β-lactamase: what do we need to know for the future? J. Antimicrobiol Chemother. 52: 2, 2003. [ Links ]
14. Handson, N. & Sanders, C. Regulation of Inducible AmpC β-lactamase Expression Among Enterobacteriaceae. Curr. Pharmac Design. 5, 11: 881, 1999. [ Links ]
15. Holtje, J., et al. The Negative Regulator of β-lactamase induction AmpD is a N- acetyl-anhydromuramyl-L-alanine amidase. FEMS Microbiol Letter. 122: 159,1994. [ Links ]
16. Honoré, N., et al. Inducible cephalosporinase production in clinical isolates of Enterobacter cloacae is controlled by a regulatory gene that has been deleted from Escherichia coli. EMBO J. 5: 3709, 1986. [ Links ]
17. Jacobs, C., et al. Cytosolic Intermediates for Cell Wall Biosynthesis and Degradation Control Inducible β-lactam Resistance in Gram-Negative Bacteria. Cell. 88: 823,1997. [ Links ]
18. Jacoby, J. & Muñoz, L. The New β-Lactamase. N. Engl J. Med. 352: 380, 2005. [ Links ]
19. Jones, R. Important and Emerging β-lactamase-mediated Resistances in Hospital- based Pathogens: The Amp C Enzymes. Diagn Microbiol Infect Dis. 31: 461,1998. [ Links ]
20. Juan, C., et al. Molecular Mechanisms of β-lactam resistance Mediated by AmpC Hyper production in Pseudomonas aeruginosa Clinical Strains. Antimicrob Agents Chemother. 49: 4733, 2005. [ Links ]
21. Juan, C., et al. Stepwise up regulation of the Pseudomonas aeruginosa Chromosomal Cephlosporinase Conferring High-Level β-Lactam Resistance Involves Three AmpD Homologues. Antimicrob Agents Chemother. 50: 1780,2006. [ Links ]
22. Kadima, T. & Weiner, J. Mechanism of Suppression of Piperacillin Resistance in Enterobacteria by Tazobactam. Antimicrob Agents Chemother. 41: 2177,1997. [ Links ]
23. Kaneko, K., et al. Gene mutations Responsible for Over expression of AmpC β-lactamase in Some clinical Isolates of Enterobacter cloacae. J. Clin Microbiol. 43, 6: 2955, 2005. [ Links ]
24. Kong, K., et al. Pseudomonas aeruginosa AmpR Is a Global Transcripcional Factor That Regulates Expresión of AmpC and poxB β-lactamases, Porteases, Quorum Sensing, and Other Virulence Factors. Antimicrob Agents Chemother. 49: 4567, 2005. [ Links ]
25. Kopp, U., et al. Sequence of Wild-Type and Mutant ampD Genes of Citrobacter freundii and Enterobacter cloacae. Antimicrob Agents Chemother. 37, 2: 224-228,1993. [ Links ]
26. Korfman, G., et al. Altered Phenotypes Associated with ampD Mutations in Enterobacter cloacae. Antimicrob Agents Chemother. 35, 2: 358,1991. [ Links ]
27. Korfman, G. & Sanders, C. ampG Is Essential for High-Level Expression of AmpC β-lactamase in Enterobacter cloacae. Antimicrob Agents Chemother. 33: 1946, 1989. [ Links ]
28. Kuga, A., et al. ampR Gene Mutations That Greatly Increase Class C β-lactamase Activity in Enterobacter cloacae. Antimicrob Agents Chemother. 44: 561, 2000. [ Links ]
29. Lindberg, F. & Normark, S. Common Mechanism of ampC β-lactamase Induction in Enterobacteria: Regualtion of the cloned Enterobacter cloacae P99 β-Lactamase Gene. J. Bacteriol. 169, 2: 758, 1987. [ Links ]
30. Lindberg, F., et al. Inactivation of the ampD Gene Causes Semi constitutive Overproduction of the Inducible Citrobacter freundii β-lactamase. J. Bacteriol. 169: 1923, 1987. [ Links ]
31. Lindberg, F., et al. Regulatory Components in Citrobacter freundii ampC β-lactamase induction. Proc Natl Acad Sci. 82: 4620, 1985. [ Links ]
32. Lindquist, S., et al. Binding of the Citrobacter freundii AmpR Regulator to a Single DNA Site Provides Both Auto regulation and Activation of the Inducible ampC β-lactamase Gene. J. Bacteriol. 171, 7: 3746, 1989. [ Links ]
33. Lodge, J., et al. Investigation of the Pseudomonas aeruginosa ampR gene and its role at the chromosomal ampC β-lactamase promoter. FEMS Microbiol Letter. 111: 315,1993. [ Links ]
34. Mahlen, S., et al. Analyses of ampC gene expression in Serratia marcescens reveal new regulatory properties. J. Antimicrobiol Chemother. 51: 791, 2003. [ Links ]
35. Mammeri, H., et al. AmpC β-lactamase in an Escherichia coli Clinical isolate Confers Resistance to Expended-Spectrum Cephalosporins. Antimicrob Agents Chemother. 48:450, 2004. [ Links ]
36. Nasim, K., et al. New Method for Laboratory Detection of AmpC β-lactamase in Escherichia coli and Klebsiella pneumoniae. J. Clin Microbiol. 42, 10: 4799, 2004. [ Links ]
37. Nelson, E. & Elisha, B. Molecular basis of AmpC Hyper production in Clinical Isolates of Escherichia coli. Antimicrob Agents Chemother. 43: 957, 1999. [ Links ]
38. Oliva, B. et al. Penicillin-Binding Protein 2 Is required for Induction of the Citrobacter freundii Class I Chromosomal β-lactamase in Escherichia coli. Antimicrob Agents Chemother. 33: 1116, 1989. [ Links ]
39. Pérez, F. & Handson, N. Detection of Plasmid-Mediated AmpC β-lactamase Genes in Clinical isolates by Using Multiplex PCR. J. Clin Microbiol 40: 2153, 2002 [ Links ]
40. Pfeifle, D., et al. Role of Penicilin-Binding Proteins in the initiation of the AmpC β- lactamase Expression in Enterobacter cloacae. Antimicrob Agents Chemother. 44: 169, 2002. [ Links ]
41. Philippon, A., et al. Plasmid-Determinated AmpC-type β-Lactamases. Antimicrob Agents Chemother. 46: 1, 2002. [ Links ]
42. Poirel, L., et al. Cloning, Sequence Analyses, Expression, and Distribution of ampC-ampR from Morganella morganii Clinical Isolates. Antimicrob Agents Chemother. 43: 769, 1999. [ Links ]
43. Power, P., et al. Biochemical and Molecular Characterization of Three New Variants of AmpC β-lactamase from Morganella morganii. Antimicrob Agents Chemother. 50: 962, 2006. [ Links ]
44. Preston, K., et al. Nucleotide Sequence of the chromosomal ampC Gene of Enterobacter aerogenes. Antimicrob Agents Chemother. 44, 11: 3158, 2000. [ Links ]
45. Reisbig, M., et al. Factors Influencing Gene Expression and resistance for gram- negative organisms expressing plasmid-encoded ampC Genes of Enterobacter origin. J. Antimicrobiol Chemother. 51: 1141, 2003. [ Links ]
46. Reisbig, M. & Handson, N. Promoter Sequences Necessary for High-Level Expression of the plamid-Associated ampC β-Lactamase Gene blaMIR-1. Antimicrob Agents Chemother. 48: 4177, 2004. [ Links ]
47. Reisbig, M. & Handson N. The ACT-1 plasmid-encoded AmpC β-lactamase is inducible: detection in a complex β-lactamase background. J. Antimicrobiol Chemother. 49: 557, 2002. [ Links ]
48. Sanders, C., et al. Penicillin-Binding Proteins and induction of AmpC β-lactamase. Antimicrob Agents Chemother. 41: 2013, 1997. [ Links ]
49. Schmidtke, A., et al. Model System To Evaluate the Effect of ampD Mutations on AmpC-Mediated β-Lactam Resistance. Antimicrob Agents Chemother. 50: 2030, 2006. [ Links ]
50. Siu, L., et al. High-level Expression of AmpC β-Lactamase Due to Insertion of nucleotides between –10 and –35 Promoter Sequences in Escherichia coli Clinical isolates: cases Not Responsive to Extended-Spectrum-Cephaloporin Treatment. Antimicrob Agents Chemother. 47: 2138, 2003. [ Links ]
51. Thomson, K. & Moland, E. Version 2000: The New β-lactamases of Gram-negative bacteria at the dawn of the new millennium. Microb Infec. 2: 1225, 2000. [ Links ]
52. Tracz, D., et al. Increase in ampC promoter stregth due to Mutations and deletion of the attenuator in a Clinical Isolate of cefoxitin-resistant Escherichia coli as determinated by RT-PCR. J Antimicrobiol Chemother. 55: 768, 2005. [ Links ]
53. Uehara, T. & Park, J. Role of the murein Precursor UDP-N-Acetylmuramyl-L-Ala-γ-D-Glu-meso-Diaminopimelic Acid-D-Ala-D-Ala in repression of β-Lactamase Induction in Cell Division Mutants. J. Bacteriol. 184, 15: 4233, 2002. [ Links ]
54. Verdet, C., et al. A novel Integron in Salmonella enterica Serovar Enteritidis, carrying the blaDHA-1 Gene and Its Regulator Gene ampR Originated from Morganella morganii. Antimicrob Agents Chemother. 44: 222, 2000. [ Links ]
55. Verdet, C., et al. Emergence of DHA-1-Producing Klebsiella spp. in the Parisian Region: genetic Organization of the ampC and ampR Genes Originating from Morganella morganii. Antimicrob Agents Chemother. 50, 2: 607, 2006. [ Links ]
56. Weidemann, B., et al. Induction of β-lactamase in Enterobacter cloacae. Clin Inf Dis. 27 (suppl 1): S42-47, 1998. [ Links ]
57. Yousef, T., et al. An ampD Gene in Pseudomonas aeruginosa Encodes a Negative Regulator of AmpC β-lactamese Expression. Antimicrob Agents Chemother. 42: 3296, 1998. [ Links ]