Introducción
El ñame (Dioscorea sp.) es un género de la familia Dioscoreaceae, con más de 600 especies (Méndez et al. 2013). Las dioscoreáceas constituyen una importante fuente alimentaria y están distribuidas en regiones tropicales, subtropicales y templadas de todo el mundo (Vega 2012). El género Dioscorea principalmente es utilizado para la alimentación humana en lugares como África, el sur de Asia, Islas del Pacífico y la región Caribe Colombiana, en donde se considera un producto básico en la dieta alimenticia de la población por presentar un alto contenido nutricional, con una fuente rica de carbohidratos, almidón, sales minerales como: el calcio, hierro, fósforo, vitamina A y C, las cuales le otorgan un valor como anti-escorbuto, también contiene riboflavina, niacina, piridoxina y carotenos. Los tubérculos poseen la mayor parte de los aminoácidos esenciales tales como: arginina, leucina, isoleucina y valina, por lo que se encuentra en menor proporción la histidina, triptófano y metionina (Vega 2012), y se convierte en un valioso alimento para el consumo humano y los animales. Entre otros usos, también es utilizado como materia prima para fines industriales, que genera empleo rural y fuente de ingresos (Vega 2012, Pinzón et al. 2013, Marroquín et al. 2016). El incremento de la demanda de este producto ha generado la necesidad de mejorar las condiciones de manejo agronómico para incrementar los rendimientos.
La agricultura actual enfrenta graves problemas de contaminación en el medio ambiente, así como disminución en la calidad de los recursos de la tierra, debido al uso excesivo de fertilizantes de síntesis químicas (Ibrahim et al. 2011, Abbasdokht y Gholami 2010), que aunque proporcionan nutrientes a las plantas e incrementan el rendimiento de los cultivos, el uso indiscriminado de estos genera contaminación de cuerpos de agua, además del incremento de los costos de producción y el impacto negativo en la salud humana y animal (Clavijo et al. 2012). Las nuevas investigaciones se han orientado hacia el desarrollo de nuevas biotecnologías enmarcadas en el manejo sostenible con el fin de reducir el impacto ambiental negativo, y en este sentido, el uso de Rizobacterias Promotoras De Crecimiento Vegetal pueden contribuir a este propósito.
Las PGPRs interactúan en el suelo al establecer relaciones variables y complejas que se traducen en la participación en los ciclos geoquímicos de nutrientes, los cuales determinan su disponibilidad para las plantas, comunidades microbianas del suelo y estimulan el desarrollo de las plantas de manera directa e indirecta, mediante relaciones benéficas (Camelo et al. 2011). Estos microorganismos se encuentran en el suelo y pueden colonizar los espacios intercelulares de los tejidos radicales e incluso algunos alcanzan el tejido vascular (Achari y Ramesh 2014); entre los diversos mecanismos usados por las PGPRs, que estimulan el desarrollo vegetal, se destacan la fijación del nitrógeno atmosférico, producción de fitohormonas (auxinas, giberelinas, etileno, ácido abscísico y citoquinas), solubilización de fosfatos, síntesis de antibióticos y/o compuestos fúngicos y producción de sideróforos (Marquina et al. 2018, Walpola y Arunakumara 2015), mecanismos que generan un efecto benéfico y desempeñan un papel clave en la toma de nutrientes y mantenimiento de la salud radicular, que favorecen el rendimiento de los cultivos (Rives et al. 2007, Tahir y Sarwar 2013).
De los mecanismos usados por las PGPRs, la producción de fitohormonas como la auxina: Ácido Indol Acético (AIA) y la solubilización de nutrientes como el fósforo han sido estudiadas ampliamente (Kumar y Audipudi 2015, Ecundayo et al. 2016, Khalifa et al. 2016, Roy et al. 2017). El AIA promueve el desarrollo de raíces al estimular el crecimiento vegetal y son responsables de la división, expansión, diferenciación de las células y regulación de muchos parámetros fisiológicos de la planta (Martínez-Viveros et al. 2010, Soler et al. 2012). Existen reportes en donde el AIA es sintetizado por algunos microorganismos como Azospirillum, Azotobacter, Pseudomonas, Rhyzobium, entre otros, que generan incrementos en parámetros de crecimiento vegetal (Patten y Glick 1996, Egamberdiyeva 2005, Mehnaz et al. 2010, Barua et al. 2012, Khalifa et al. 2016). El fósforo, después del nitrógeno, es un elemento crítico para la producción agropecuaria; su disponibilidad es cada vez más limitada, debido a la progresiva insuficiencia de sus fuentes naturales y su baja movilidad si se compara con otros nutrientes (Beltrán 2014). El fósforo disponible para las plantas se encuentra en forma de ortofosfatos, que son formas inorgánicas solubles, donde se encuentran iones monobásicos (H2PO4 -1) y dibásicos (HPO4 -2) (Banerjee et al. 2010). La acción de los microorganismos solubilizadores de fosfato es esencial, ya que constituyen un grupo importante, pues están involucrados en un amplio rango de procesos que afectan la transformación del fósforo, al ser componentes integrales del ciclo edáfico de este nutriente (Beltrán 2014) y así facilitar la adquisición del nutriente por parte de las plantas e incrementar el crecimiento y su productividad; géneros como Pseudomonas y Bacillus se reportan como solubilizadores de este nutriente, al favorecer el desarrollo vegetal (Gulati et al. 2008, Swain et al. 2012, Diep y Hieu 2013, Roy et al. 2017). En ese contexto, el uso de microorganismos productores de AIA y con capacidad de solubilización de fosfatos representa una alternativa viable para incorporar en las tecnologías de manejo agronómico, al representar una opción amigable desde el punto de vista ambiental, la cual implementa prácticas agrícolas sostenibles.
Para la selección de PGPRs, además de la evaluación de sus capacidades como promotores de crecimiento in vitro e in vivo, conocer su identidad es fundamental para evitar seleccionar microorganismos con algún riesgo en la salud humana, por tanto, con el avance actual de la tecnología, la caracterización molecular de los microorganismos se ha convertido en una herramienta de gran utilidad. La secuenciación parcial del gen ARNr 16S se usa generalmente para la identificación bacteriana, este gen contiene regiones conservadas que proporcionan polimorfismos de secuencia que sirven como "firmas" únicas para un género o especie (Mendes et al. 2014, Persing et al. 2016), sin embargo, se ha encontrado que el grado de divergencia observado dentro de la molécula de ARNr 16S, puede no ser suficiente para distinguir algunas especies estrechamente relacionadas (Nemergut et al. 2013, Wang et al. 2018), por tanto, actualmente se utilizan otros marcadores moleculares que tienen regiones conservadas que flanquean zonas variables, las cuales permiten separar mejor a especies estrechamente relacionadas, entre estos se incluye el gen rpoB (subunidad β de ARN polimerasa) (Persing et al. 2016), que recientemente se utiliza como un marcador robusto para el análisis de diversidad bacteriana a nivel intraespecífico (Gopinath et al. 2018, Wang et al. 2018).
Al tener en cuenta los anteriores argumentos, la presente investigación tiene como objetivo el aislamiento de bacterias asociadas a la rizosfera de Dioscorea rotundata, caracterización molecular mediante secuenciación del gen rpoB, caracterización de capacidades de promoción de crecimiento vegetal por producción de AIA y solubilización de fósforo y evaluación del efecto a nivel de invernadero de su inoculación sobre el crecimiento de plántulas in vitro Dioscorea rotundata.
Materiales y métodos
Toma de muestra. Este estudio se realizó en el Centro de Investigación Turipaná (Corpoica) km 13 vía Montería-Cereté (Colombia), las coordenadas geográficas corresponden a los 8°31'16'' de latitud norte y 75°58'11' de longitud oeste. Las muestras fueron tomadas a partir de suelo rizosféricos del cultivo de ñame (Dioscorea rotundata), en la región Caribe Colombiana y las zonas Montes de María, Sabanas Colinadas y Valle del Sinú, para un total de 15 fincas productoras representativas de este cultivo (Cuadro 1).
Cuadro 1 Predios seleccionados para el aislamiento de bacterias asociadas a cultivo de ñame de la región del Caribe Colombiano.
| Zonas | Código | Nombre Predio | Vereda / Corregimiento | Municipio / Departamento | Coordenadas | ||
| N | W | ||||||
| Montes de María | Ñ05 | Acapulco | Arroyo Arena | Carmen de Bolívar/Bolívar | 09°43′35” | 75°09′23” | |
| ÑO6 | La Lucha | Caracoli Grande | Carmen de Bolívar/Bolívar | 09°44′24” | 75°13′28” | ||
| Sabanas Colinadas | Ñ01 | Quita Sueño | Los Algarrobos | Chinú/Sucre | 09°09′39″ | 75°23′53″ | |
| Ñ02 | Nueva Esperanza | Los Algarrobos | Chinú/Sucre | 09°11′37″ | 75°23′54″ | ||
| Ñ03 | Parcela 3 | Mateo Pérez | Sampués/Sucre | 09°21′69″ | 75°38′14″ | ||
| Ñ04 | Florida | Segovia | Sampués/Sucre | 09°10′59″ | 75°22′44″ | ||
| Ñ07 | El Rosario | Las Tinas | Corozal /Sucre | 09°17´9.0” | 75°19´8.0” | ||
| Ñ08 | Puerta Roja | Las Palmas | Corozal/Sucre | 09°15´47” | 75°20´29” | ||
| Ñ09 | San Rafael | Las Nubes | San Antero/Córdoba | 09° 20´48” | 75° 47´48” | ||
| Ñ10 | Marbella | Las Nubes | San Antero/Córdoba | 09° 20´51” | 75° 47´40” | ||
| Ñ11 | Guasimal | Guasimal | San Antero/Córdoba | 09°20´47” | 75° 45´17” | ||
| Ñ12 | Sol y Sombra | Mosquito | San Antero/Córdoba | 09°20´10” | 75° 44´52” | ||
| Ñ13 | Villa Marela | Bonguita | San Antero/Córdoba | 09° 21´ 24” | 75° 45´41” | ||
| Ñ14 | Padilla | Bajo Grande | San Antero/Córdoba | 09° 20´24” | 75° 45´49” | ||
| Valle Del Sinú | Ñ15 | C.I Turipaná | Retiro de los indios | Cereté/Córdoba | 08°50´48” | 75°48´57” | |
Las muestras de suelo fueron tomadas aplicando un patrón sistemático de muestreo en forma de X, se tomaron 5 puntos en cada lote de la rizosfera a una profundidad de 20 cm, determinada por las raíces del cultivo entre 150-200 gr de suelo. Las muestras se almacenaron en bolsas plásticas, refrigeradas y transportadas al Laboratorio de Microbiología Agrícola del C.I. Turipaná para su procesamiento.
Aislamientos. Se tomó una muestra de 10 g de suelo y se diluyó en 90 ml de NaCl estéril al 0,85%, con una agitación de 100 rpm durante 15 min. Se realizaron diluciones seriadas con base 10 desde 10-1 hasta 10-7, se inoculó 0,1 ml en medios de cultivo Agar Triptona Soya (TSA)® para aislamientos de mesófilos arábicos y medio King B (King et al. 1954) para aislamientos de pseudomonas; para aislamientos de Azotobacter se utilizó medio Ashby, en el que se realizó la siembra de gránulos de suelo en cajas Petri (Fenglerowa 1965). Las colonias puras obtenidas de cada aislamiento fueron conservadas en viales con medio aceite mineral estéril (Carrillo et al. 1998).
Determinación de la actividad PGPR S in vitro de los aislados. La cuantificación química de la auxina: ácido indol acético (AIA) se realizó en un un medio de cultivo líquido con K-lactato y suplementado de triptófano a 100 mg.l-1 (Sánchez et al. 2012). El pH fue ajustado a un valor de 7,0 y esterilizado a 121°C durante 15 minutos; los caldos inoculados por triplicado con las cepas fueron incubados durante 72 h a 150 rpm en la oscuridad. La determinación de la producción del AIA se llevó a cabo por método colorimétrico al utilizar el reactivo de Salkowski preparado a partir de cloruro férrico en ácido sulfúrico (FeCl37H2O12 gl-1en H2SO4 7,9M) en una relación 1:1 con la suspensión bacteriana, se dejó reaccionar durante 30 min en oscuridad (Glickmann y Deessaux 1995) y se realizó la lectura a una absorbancia de 450 nm.
La determinación cuantitativa de solubilización de fósforo se hizo en medio líquido SRSM-PR modificado con roca fosfórica al 0,5% como fuente de fósforo (g.l-1: glucosa 10; extracto de levadura 0,5; NH4(SO4)2 0,50; KCl 0,2; MgSO47H2O 0,3; MnSO4 7H2O 0,004; FeSO47H2O 0,2; pH: 7,2). Los aislamientos se cultivaron en caldo SRSM-PR e incubaron durante 5 días a 150 rpm, posteriormente 24 ml de los cultivos se centrifugaron a 3,000 rpm, al emplear 0,5 ml del sobrenadante. La medición y lectura se realizó mediante la técnica de azul de fosfomolibdeno a una absorbancia de 712 nm (Fiske y Subbarow 1925).
Para la prueba de compatibilidad todas las cepas fueron cultivadas en erlenmeyer durante 24 h y ajustadas a una OD540= 0,500, posteriormente, la primera cepa se colocó en placas de agar LB y se cultivaron durante 5 días a 30ºC. La segunda cepa se diluyó el caldo en agar LB [1,5% (peso / vol)] y enfrió a una temperatura de 30°C y luego se vertió en placas hasta solidificación, luego, se cortaron trozos de la segunda cepa y se voltearon sobre la primera cepa. Después del crecimiento durante 24 horas a 30°C, se observaron las placas, la presencia de halos indicó incompatibilidad de las cepas (Shank et al. 2011).
Caracterización molecular de los aislamientos. Se realizó mediante la amplificación y secuenciación del gen rpoB. La extracción de ADN de las bacterias seleccionadas se realizó con el kit QIAamp® DNA Mini and Blood Mini Handbook de QUIAGEN (Alemania) a partir de una suspensión bacteriana cultivada en medio Luria Bertani (LB) toda la noche. La amplificación del gen rpoB se realizó al seguir las instrucciones del fabricante, mediante el Kit PCR Master Mix (ThermoScientific). Los primers utilizados en la PCR fueron rpoB1698f (59-AACATCGGTTTGATCAAC-3) y rpoB2041r (59-CGTTGCATGTTGGTACCCAT-3) (Dahllof et al. 2000). La PCR se realizó en un termociclador Multigene Optimax de Labnet. El programa para la PCR fue como sigue: denaturación a 94°C por 3 minutos, seguido por 10 ciclos de denaturación a 94°C por 1 minuto, alineación a 50°C por 1,5 minutos, extensión por 2 minutos a 72ºC seguido, por 25 ciclos de denaturación por 1 minuto a 94ºC, alineación a 50ºC por 1,5 minutos y extensión por 2 minutos a 72ºC y una extensión final a 72°C por 10 minutos. Los productos de PCR se enviaron para su secuenciación a Macrogen-Korea. Las secuencias obtenidas se limpiaron y ensamblaron en una secuencia consenso que fue alineada con secuencias de la base de datos GenBank del NCBI, mediante la herramienta BLASTn.
Con las secuencias obtenidas se realizó el análisis filogenético en el software MEGA7 (Kumar et al. 2016), las secuencias se alinearon mediante el programa MUSCLE (Edgar 2004), las distancias evolutivas se calcularon al utilizar el método de máxima verosimilitud compuesta (Tamura et al. 2004) al tener en cuenta transiciones y transversiones. La construcción del árbol filogenético se infirió al usar el método Neighbor-Joining (Saitou y Nei 1987) y la evaluación del árbol se realizó mediante el método Bootstrap con 1000 permutaciones para asignar niveles de confianza a los nodos en el árbol (Felsenstein 1985).
Evaluación en invernadero de aislamientos asociados al cultivo de ñame espino (Dioscorea rotundata) por su promoción de crecimiento. El experimento se mantuvo bajo condiciones de invernadero con plántulas de ñame espino de Dioscorea rotundata obtenidas in vitro, según protocolo descritos por Quintero et al. (2003), estas plántulas in vitro fueron ubicadas en invernadero durante 8 días con riego constante para su aclimatación y sembradas en bandejas de 28 alveolos (Figura 1).

Figura 1 A. Plántulas ñame in vitro en proceso de aclimatación en invernadero. B. Plantas de ñame a los 60 días de establecidas en el invernadero. C. Raíces de las plantas de ñame con minitubérculos.
Se utilizó un suelo sin esterilizar, procedente de las zonas donde fueron aisladas las bacterias: Montes de María, el suelo exhibía una textura franco arcillosos, pH=5,89; 3,23% MO; 3,48 P (mg.kg-1); 0,24 K; 7,54 Mg; 20,95 Ca (Cmol.kg-1). Sabanas Colinadas, el suelo mostraba una textura franco limoso, pH=6,46; 2,02% MO; 2,69 P (mg.kg-1); 0,36 K; <1,00 Mg; 21,74 Ca (Cmol.kg-1) y Valle Del Sinú, el suelo presentaba una textura franco arcillosos, el suelo pH=6,58; 1,42% MO; 5,87 P (mg.kg-1); 6,49 K; 0,43 Mg; 16,26 Ca (Cmol.kg-1).
Se empleó un diseño completamente al azar con 5 repeticiones por tratamiento con (3 plantas/unidad experimental). Se determinó un control sin inocular para cada zona. Las plántulas fueron inoculadas 2 veces a los 8 días de establecido el experimento y a los 30 días directamente al suelo con 5 ml de suspensión bacteriana medida con una absorbancia a 540 nm, correspondiente a una población bacteriana de 108 UFC.ml-1 en medio nutritivo (g.l-1: glucosa 0,5; extracto de levadura 0,5; peptona 0,5; caseína 0,5; almidón 0,5; K2HP04 0,30; MgS04 0,05; 17,0 pH: 7). Se realizó el muestreo destructivo a los 60 días y se evaluó las variables de respuesta: altura de la parte aérea y radical (cm), masa seca de la parte aérea y radical (g).
Análisis estadístico. Los datos fueron sometidos a una evaluación estadística al emplear un análisis de varianza (ANOVA) y la prueba de Tukey con 95% de confianza.
Resultados y discusión
Aislamiento, selección y caracterización molecular de bacterias asociadas a rizosfera de Dioscorea sp. Se aislaron un total 106 morfotipos bacterianos, se seleccionaron 19 morfotipos con características asociadas a los géneros Bacillus, Brevibacillus, Azotobacter, Pseudomonas, Stenotrophomonas, Enterobacter y Rhizobium, los cuales fueron caracterizados molecularmente mediante la amplificación y secuenciación del gen rpoB (Cuadro 2), el árbol filogenético generado a partir del análisis de las secuencias se presenta en la Figura 2.
Cuadro 2 Caracterización molecular de cepas bacterianas aisladas de la rizosfera de cultivos de Dioscorea rotundata.
| Zona | Cepa Bacteriana | Identificación Molecular | % De Identidad | # De Accesión |
| Montes de María | DCB9 | Azotobacter vinelandii | 98% | CP005095.1 |
| DCB12 | Azotobacter chroococcum | 88% | CP010415.1 | |
| DCB13 | Enterobacter cloacae | 99% | CP009756.1 | |
| Sabanas Colinadas | DCR11 | Azotobacter vinelandii | 99% | CP005095.1 |
| DCR13 | Azotobacter vinelandii | 98% | CP005095.1 | |
| DCR21 | Stenotrophomonas maltophilia | 96% | HE798556.1 | |
| DSC1 | Bacillus licheniformis | 98% | CP014781.1 | |
| DSC6 | Enterobacter cloacae | 99% | CP009756.1 | |
| DSC10 | Enterobacter asburiae | 99% | CP011591.1 | |
| DSC17 | Enterobacter cloacae | 99% | CP009756.1 | |
| DSC18 | Enterobacter cloacae | 99% | CP009756.1 | |
| DSC19 | Brevibacillus laterosporus | 99% | CP007806.1 | |
| DSC21 | Brevibacillus laterosporus | 99% | CP007806.1 | |
| DSC62 | Pseudomonas denitrificans | 94% | CP004143.1 | |
| DSC72 | Azotobacter vinelandii | 99% | CP005095.1 | |
| DSC82 | Bacillus subtilis | 100% | CP011534.1 | |
| DSC85 | Rhizobium sp. | 98% | HG518322.1 | |
| Valle Del Sinú | DVS9 | Azotobacter vinelandii | 99% | CP005095.1 |
| DVS10 | Pseudomonas denitrificans | 96% | CP004143.1 |
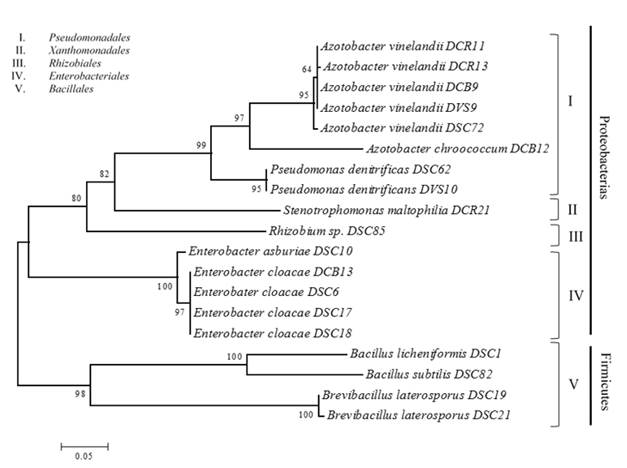
Figura 2 Árbol filogenético generado por el método Neighbor-Joining basado en el gen rpoβ para 19 cepas bacterianas aisladas de la rizosfera de Dioscorea rotundata de zonas productoras del Caribe Colombiano con características de promoción de crecimiento vegetal evaluadas in vitro y en invernadero.
El árbol filogenético (Figura 2) está dibujado a escala, con longitudes de rama en las mismas unidades que las distancias evolutivas utilizadas para inferir el árbol, las cuales se encuentran en las unidades del número de sustituciones de bases por sitio. El análisis involucró 19 secuencias de nucleótidos. Se eliminaron todas las posiciones que contenían lagunas y datos faltantes. El porcentaje de árboles replicados en el que los taxones asociados se agruparon en la prueba Bootstrap (1000 réplicas) se muestran junto a los nodos en el árbol.
En términos generales, la secuenciación del gen rpoB permite diferenciar las bacterias obtenidas por grupos correspondientes a sus características biológicas, más que por su procedencia. El 79% de los microorganismos aislados se agrupan por pertenecer al Phylum Proteobacterias, que es un grupo diverso de bacterias Gram negativas, dentro de este amplio grupo, los microorganismos se distribuyen en los órdenes Xanthomonadales, Rhizobiales, Enterobacteriales y Pseudomonadales (Figura 2), este último grupo es al que pertenecen el mayor número de aislados obtenidos en este estudio. El 21% de microorganismos restantes se agrupan por pertenecer al Phylum Firmicutes, que son las bacterias Gram positivas con bajo contenido G+C (<50%), los microrganismos pertenecen a clase Bacilli, Orden Bacillales y se diferencian en 2 grupos correspondientes a la división a nivel de género que se puede observar en el árbol filogenético (Figura 2). Todos los géneros encontrados en esta investigación han sido reportados como PGPRs con capacidad para favorecer el crecimiento, rendimiento y calidad de los cultivos (Selvakumar et al. 2012, Ahemad y Kibret 2014, Kumar y Audipudi 2015, Ecundayo et al. 2016, Khalifa et al. 2016, Pérez y Sánchez 2017, Roy et al. 2017).
Determinación de la actividad PGPRs in vitro de las cepas bacterianas
Se realizaron pruebas de habilidades de promoción de crecimiento in vitro, como la producción de AIA y solubilización de fósforo a las 19 bacterias seleccionadas y caracterizadas (Cuadro 3).
Cuadro 3 Determinación de la actividad promoción de crecimiento in vitro a cepas bacterianas de la rizosfera de cultivos de Dioscorea rotundata.
| Zona | Cepa Bacteriana | Identificación Molecular | (AIA) (µg.ml-1) | Fosfatos Disponible (ppm) |
| Montes De María | DCB9 | Azotobacter vinelandii | 24,29±1,30 | 18,94±0,42 |
| DCB12 | Azotobacter chroococcum | 27,70±4,77 | 15,90±0,56 | |
| DCB13 | Enterobacter cloacae | 101,42±1,57 | 15,1±0,33 | |
| Sabanas Colinadas | DCR11 | Azotobacter vinelandii | 67,62±7,45 | 18,68±0,31 |
| DCR13 | Azotobacter vinelandii | 37,39±3,15 | 14,36±0,40 | |
| DCR21 | Stenotrophomonas maltophilia | 46,59±4,84 | 19,03±1,32 | |
| DSC1 | Bacillus licheniformis | 35,15±1,36 | 18,18±0,19 | |
| DSC6 | Enterobacter cloacae | 63,25±4,54 | 16,78±0,41 | |
| DSC10 | Enterobacter asburiae | 28,05±0,9 | 10,96±0,09 | |
| DSC17 | Enterobacter cloacae | 25,52±2,40 | 18,32±1,42 | |
| DSC18 | Enterobacter cloacae | 49,01±1,31 | 18,88±0,88 | |
| DSC19 | Brevibacillus laterosporus | 7,06±0,98 | 18,58±0,68 | |
| DSC21 | Brevibacillus laterosporus | 25,09±0,46 | 19,03±1,32 | |
| DSC62 | Pseudomonas denitrificans | 18,72±4,64 | 18,37±0,32 | |
| DSC72 | Azotobacter vinelandii | 19,92±0,70 | 7,19±0,12 | |
| DSC82 | Bacillus subtilis | 7,59±0,36 | 20,78±1,14 | |
| DSC85 | Rhizobium sp. | 25,98±1,18 | 18,40±0,84 | |
| Valle Del Sinú | DVS9 | Azotobacter vinelandii | 25,71±2,11 | 18,33±0,32 |
| DVS10 | Pseudomonas denitrificans | 67,93±0,89 | 21,92±0,19 |
Los resultados obtenidos muestran que las cepas presentan algunos mecanismos directos de promoción de crecimiento in vitro como la producción de AIA y solubilización de fósforo, los cuales contribuyen de manera positiva al crecimiento de las plantas inoculadas y las convierte en una alternativa para mejorar los cultivos, que evitan el abuso de fertilizantes de síntesis químicas.
Las cepas bacterianas que presentaron los mayores valores de AIA fueron Enterobacter cloacae (DCB13) aisladas de la zona de Montes de María con 101,42 µg.ml-1, valor muy cercano al reportado por Khalifa et al. (2016), que indica que la cepa promotora de crecimiento vegetal Enterobacter cloacae MSR1 aislada de alfalfa alcanzó un valor de AIA de 112,00 µg.ml-1. En el Valle del Sinú se destacó la bacteria Pseudomonas denitrificans (DVS10) con una producción de AIA de 67,93 µg.ml-1, Egamberdiyeva (2005), reporta cepas de Pseudomonas denitrificans positivas para la producción de AIA, entre otros estudios para este género bacteriano. Malik y Sindhu (2011) reportan que la cantidad de AIA producida por aislados de Pseudomonas varía en un rango de 10,20 a 31,20 µg.ml-1, valores inferiores a los obtenidos en esta investigación con la cepa DVS10.
La cepa Azotobacter vinelandii (DCR11), aislada de la zona de Sabanas Colinadas, alcanzó una producción de AIA in vitro de 67,62 µg.ml-1, valor superior al reportado por Barua et al. (2012), por una cepa de referencia de Azotobacter vinelandii (MTCC 2460), que alcanzó una producción de AIA de 23,16 µg.ml-1, de igual forma, el valor de AIA obtenido por la cepa DCR11 fue superior al reportado por Lara et al. (2014) en sus estudios realizados en la zona del Valle del Sinú del Caribe Colombiano, en donde las bacterias más eficientes en la producción de AIA fueron del género Azotobacter, donde se destacó la cepa A14 de la zona de San Carlos con el mayor valor de 44,276 y las cepas SN1 y S32 de la zona de San Pelayo con 39,342 µg.ml-1 y 37,209 µg.ml-1 respectivamente.
Por último, se destaca que la cepa aislada de la zona de Sabanas Colinadas era de Stenotrophomonas maltophilia (DCR21), con una producción AIA de 46,59 µg.ml-1, lo cual es un valor superior a lo reportado por Mehnaz et al. (2010), quienes encontraron que las cepas de Stenotrophomonas maltophilia aisladas de caña de azúcar mostraron las más bajas producciones de AIA en un rango de 0,30 a 6,00 µg.ml-1, en contraste a estos resultados Kumar y Audipudi (2015) reportaron que la cepa Stenotrophomonas maltophilia AVP27 presentó una producción de AIA de 93,0 µg.ml-1, valor superior al obtenido para la cepa DCR21 en esta investigación, sin embargo, este resultado puede atribuirse a la cantidad de triptófano usado en el medio de cultivo. Para el caso de Kumar y Audipudi (2015) utilizaron 1000 µg.ml-1 de triptófano, mientras que en esta investigación se utilizó 100 µg.ml-1. Se ha informado que la producción de AIA por bacterias puede variar entre los diferentes géneros y especies, y también está influenciada por condiciones de cultivo, la etapa de crecimiento y la disponibilidad de sustrato (Sánchez et al. 2012).
Todas las bacterias presentaron la capacidad de sintetizar AIA, la cual es una auxina natural que se encuentra presente en la mayoría de las plantas; estas auxinas son hormonas vegetales que regulan diversos procesos del desarrollo vegetal. La síntesis de estas sustancias resulta de gran importancia, ya que la aplicación de estas bacterias puede constituir una alternativa viable en el contexto de una agricultura ecológica (Castillo et al. 2005), se perfilan a las bacterias aisladas de la rizosfera de ñame que son potenciales promotoras de crecimiento vegetal.
La solubilización de fósforo, medida como la disponibilidad de ortofosfatos a partir de roca fosfórica, se encuentra en un rango de 7- 21 ppm. Para la zona del Valle del Sinú se destaca la bacteria Pseudomonas denitrificans (DVS10) con el mayor valor de solubilización de fósforo de 21,92 ppm, resultados que sustentan lo afirmado por Roy et al. (2017), quienes plantean la solubilización de fósforo como una característica de esta especie bacteriana. Otros estudios realizados por Gulati et al. (2008) mencionan que este género bacteriano Pseudomonas presenta la capacidad de solubilizar diferentes fuentes de fosfatos (tricálcico y roca fosfórica), en el que se destaca la cepa Pseudomonas BIHB que presentó solubilización en un rango de 8,05 ppm a 0,06 ppm, valores inferiores a los obtenidos con la cepa DVS10 en esta investigación.
En la zona de Sabanas Colinadas se destacan diferentes cepas aisladas. La cepa Bacillus subtilis (DSC82) logró solubilizar hasta 20,78 ppm, valor que se encuentra dentro del rango reportado por Swain et al. (2012), quienes indican la capacidad de solubilización de fosfatos en cepas de Bacillus subtilis en el rango de 20 a 30 ppm, de igual forma, Diep y Hieu (2013) reportan que cepas de Bacillus subtilis alcanzan valores de 19,01 ppm.
La cepa Brevibacillus laterosporus (DSC21) presentó capacidad de solubilizar in vitro hasta un valor de 19,03 ppm, resultados que apoyan lo obtenido por Ecundayo et al. (2016), que reportan cepas de Brevibacillus laterosporus con capacidad para solubilizar fósforo de tipo inorgánico por la disminución del pH en el medio de cultivo NBRIP. Los resultados obtenidos para la cepa Stenotrophomonas maltophilia (DCR21) indican que fue capaz de solubilizar fósforo a partir de roca fosfórica hasta un valor de 19,03 ppm. Según Gomes et al. (2014) la Stenotrophomonas maltophilia es capaz de solubilizar fósforo en un rango de 46-79 ppm a partir de roca fosfórica, resultados superiores a los obtenidos en esta investigación, lo que sugiere que esta especie bacteriana tiene una alta capacidad de solubilización de fósforo y está ampliamente reportada como solubilizadora de fósforo (Liba et al. 2006, Xiao et al. 2009).
Las bacterias relacionadas en el experimento fueron capaces de solubilizar la roca fosfórica presente en medio de cultivo in vitro, estas bacterias pueden ser una alternativa para los suelos agrícolas que, aunque contienen grandes cantidades de P, se encuentran en formas no asimilables que pueden ser solubilizados por estos grupos bacterianos (Restrepo-Franco et al. 2015).
Los resultados de compatibilidad indican que existe competencia entre cepas evaluadas, por tanto, no se deben mezclar en Montes de María las cepas DCB12-DCB13, en Sabanas Colinadas DCR11-DCR14, DSC1-DSC6, DSC17-DSC19, DSC18-DSC19 y DSC62-DSC72.
Como se puede observar en la Cuadro 4, la inoculación de las bacterias genera un efecto en los parámetros de crecimiento de plántulas de ñame después de 60 días de establecidas en invernadero. Los resultados evidenciaron que la inoculación con algunas bacterias de este estudió influyó positivamente sobre la longitud de la parte aérea de las plantas, para la zona Montes de María en la localidad del Carmen de Bolívar, la cepa Azotobacter vinelandii (DCB9) presentó un incremento 28% respecto al control sin inocular. En la zona de Sabanas Colinadas en localidad de corozal, la Stenotrophomonas maltophilia (DCR21) presentó diferencias significativas (p≤0,05) de 106% respecto al control sin inocular. En la localidad de Chinú, la cepa Enterobacter cloacae (DSC17) presentó un incremento del 36%.
Cuadro 4 Respuesta vegetal a la inoculación de bacterias aisladas de la rizosfera de Dioscorea rotundata en invernadero.
| Zona | Localidad | Cepa Bacteriana | Longitud Aérea (cm) | Longitud Radical (cm) | Peso Seco Aéreo (g) | Peso Seco Radical (g) |
| Montes De María | Carmen De Bolívar | Control | 26,77a | 15,66a | 0,24a | 0,050a |
| DCB9 | 34,40a | 16,58a | 0,25a | 0,048a | ||
| DCB12 | 26,75a | 15,31a | 0,21a | 0,053a | ||
| DCB13 | 26,16a | 18,40a | 0,27a | 0,062a | ||
| Sabanas Colinadas | Corozal | Control | 26,68b | 19,00a | 0,35b | 0,36b |
| DCR11 | 19,81b | 16,98ab | 0,34b | 0,75a | ||
| DCR13 | 16,14b | 15,29b | 0,48ab | 0,60ab | ||
| DCR21 | 55,19a | 18,06ab | 0,54a | 0,55ab | ||
| Chinú | Control | 17,19ab | 18,59ab | 0,20b | 0,21b | |
| DSC1 | 31,21a | 21,47a | 0,31ab | 0,40ab | ||
| DSC6 | 9,10b | 20,01ab | 0,29ab | 0,52a | ||
| DSC10 | 12,83b | 17,73b | 0,33a | 0,40ab | ||
| Sampués | Control | 55,94ab | 20,09a | 0,40a | 0,29a | |
| DSC17 | 76,47a | 19,92a | 0,57a | 0,39a | ||
| DSC18 | 48,47ab | 18,73a | 0,53a | 0,44a | ||
| DSC19 | 43,43b | 19,07a | 0,52a | 0,57a | ||
| DSC21 | 44,26b | 17,52a | 0,52a | 0,41a | ||
| San Antero | Control | 46,43a | 22,39a | 0,45ab | 0,42b | |
| DSC62 | 42,73a | 20,81a | 0,47ab | 0,62ab | ||
| DSC72 | 29,95ab | 21,73a | 0,50ab | 0,65ab | ||
| DSC82 | 15,00b | 19,96a | 0,33b | 0,79a | ||
| DSC85 | 38,44ab | 22,15a | 0,52a | 0,59ab | ||
| Valle del Sinú | Cereté | Control | 33,69a | 21,31a | 0,45a | 0,23b |
| DVS9 | 32,88a | 18,83ab | 0,39ab | 0,52a | ||
| DVS10 | 26,76a | 18,32b | 0,28b | 0,34a |
Medias con letras no comunes dentro de cada variable difieren estadísticamente según prueba de Tukey para p≤0,05 (n=15).
Con respecto a la longitud radicular en los Montes de María en la localidad de la zona del Carmen de Bolívar Azotobacter vinelandii (DCB9) presentó un incremento del 6%, al igual que la cepa Enterobacter cloacae (DCB13) con incrementos del 17% con respecto al control sin inocular. En la zona de Sabanas Colinadas en la localidad Chinú, la cepa Bacillus licheniformis (DSC1) presentó incrementos en la longitud radicular de15%, respecto al control sin inocular, la cepa Enterobacter cloacae (DSC6) logró incrementos del 7% con respecto al cultivo sin inocular en esta misma localidad (Cuadro 4). Estas PGPRs estimulan y modifican los procesos fisiológicos de las plantas, al incrementar el desarrollo de raíces adventicias y pelos absorbentes, los cuales son importantes para la absorción del fósforo, significativas en el empleo de la agricultura (Vazallo et al. 2013).
Se observó que la variable denominada peso seco aéreo, localizada en Carmen de Bolívar, mostró incrementos en la cepa Azotobacter vinelandii (DCB9) con un 4% respecto al control sin inocular. La cepa Enterobacter cloacae (DCB13) también presentó incrementos de un 12% con respecto al control sin inocular. En la zona de Sabanas Colinadas en localidad de Corozal Azotobacter vinelandii (DCR13) presentó incrementos 140% con respecto al control sin inocular y la cepa Stenotrophomonas maltophilia (DCR21) presentó diferencias significativas (p≤0,05) de 170%. En la localidad de Chinú el Bacillus licheniformis (DSC1) generó un crecimiento del 55% al compararlo con el control sin inocular. En Sampués la cepa Brevibacillus laterosporus (DSC21) presentó incrementos del 30% en referencia con el control sin inocular. En San Antero se destacaron la cepa de Rhizobium sp. (DSC85) con aumentos de 15%, la cepa Azotobacter vinelandii (DSC72) con 11% y Pseudomonas denitrificans (DSC62) con 4% a partir del testigo sin inocular (Cuadro 4).
La mayoría de las cepas por zona presentaron incrementos, principalmente el peso seco radicular de las plántulas de ñame en comparación con el control sin inocular, con la excepción del Azotobacter vinelandii (DCB9) que fue inferior en este parámetro, según el control sin inocular. Para la zona de los Montes de María con la inoculación de Enterobacter cloacae (DCB13) se logró un crecimiento de 24% en el peso seco radicular (Cuadro 4). Para la zona de Sabanas Colinadas en la localidad de Corozal se destaca la bacteria Azotobacter vinelandii (DCR11) con un incremento significativo (p≤0,05) de 108% en el peso seco radical. La bacteria Enterobacter cloacae (DSC6) de Chinú generó un aumento significativo (p≤0,05) de 147% en peso radicular sobre el control sin inocular, para Sampués se logró un crecimiento de 96% con la inoculación de la bacteria Brevibacillus laterosporus (DSC19) y para San Antero la inoculación de Bacillus subtilis (DSC82) permitió un aumento significativo (p≤0,05) de 88% y con la inoculación de la cepa Azotobacter vinelandii (DSC72) se alcanzó el 54% en el peso seco radical de los tubérculos (Cuadro 4). Para la zona del Valle del Sinú con la inoculación de las 2 cepas se obtuvo un incremento estadísticamente significativo (p≤0,05) en el peso seco radicular de ñame al alcanzar un 126% con la inoculación de Azotobacter vinelandii (DVS9) y un 48% con la inoculación de Pseudomonas denitrificans (DVS10) (Cuadro 4).
El incremento del peso seco radicular con la inoculación de bacterias es un reflejo directo de la actividad promotora de crecimiento de estos microorganismos, resultados similares en Dioscorea rotundata fueron reportados por Swain et al. (2007), quienes encontraron un efecto positivo de Bacillus subtilis en el peso seco radicular al alcanzar un incremento máximo de 76% respecto al control sin inocular, valor inferior al encontrado en este estudio con la bacteria DSC82 (Cuadro 4).
Respecto al resto de bacterias evaluadas, Arcos y Zúñiga (2016) reportaron que la inoculación de la bacteria Bacillus Bac17M8, suplementada con materia orgánica, presenta rendimientos superiores en 145,69%, en comparación al control no inoculado en la papa variedad Ccompis; Naqqash et al. (2016) informaron que la inoculación con Enterobacter sp. TN38 produjo efectos positivos significativos en los parámetros de crecimiento al alcanzar aumentos máximos de 76% en la raíz. En esta investigación se obtuvieron incrementos superiores en el peso seco de tubérculos de ñame de hasta 147% con la inoculación de Enterobacter cloacae, en comparación a los obtenidos en papa con cepa del mismo género bacteriano.
Conclusiones
Se aislaron 19 rizobacterias potenciales como biofertilizantes asociadas al cultivo de ñame (Dioscorea sp.), pertenecientes a subregiones Montes de María, Sabanas Colinadas y Valle del Sinú; las cuales fueron identificadas para Montes de María (Azotobacter vinelandii, Azotobacter chroococcum y Enterobacter cloacae), Sabanas Colinadas (Bacillus licheniformis, Enterobacter cloacae, Enterobacter asburiae, Azotobacter vinelandii, Stenotrophomonas maltophilia, Azotobacter chroococcum, Brevibacillus laterosporus, Pseudomonas denitrificans, Bacillus subtilis y Rhizobium sp.) y Valle del Sinú (Azotobacter vinelandii y Pseudomonas denitrificans); las cuales sintetizan auxinas y solubilizan fósforo al facilitar la promoción del crecimiento y desarrollo de la plantas de ñame, lo cual sería una alternativa prometedora como biofertilizantes para este cultivo y una agricultura sostenible al tener en cuenta que disminuiría el impacto sobre el medio ambiente al reducir el uso excesivo de fertilizantes de síntesis química.