Introducción
La satisfacción del consumidor de carne bovina depende de un conjunto de propiedades organolépticas de la carne, como lo son la jugosidad, el sabor y la terneza, esta última es la principal característica asociada con el criterio de calidad de la carne (Soria y Corva 2004). Las características de calidad de la carne se encuentran influenciadas por factores genéticos y ambientales (Soria y Corva 2004) que definen los rasgos en el animal. La predicción de rasgos en el animal, por medio de la genética molecular (Casas et al. 2011), es un tema de investigación de vanguardia para la producción vacuna, donde la detección de las variables involucradas en los rasgos económicamente relevantes, es una necesidad para la optimización del sistema productivo y donde características de calidad de carne como marmoleo, color, capacidad de retención de agua, se encuentran influenciadas por factores genéticos (Li et al. 2013, Paredes-Sánchez et al. 2015).
La investigación de los genes candidatos es una técnica eficiente dentro de la predicción de los rasgos, ya que identifica los genes que, en teoría, se encuentren asociados a la expresión del rasgo y define su variación genética (Karisa et al. 2013, Paredes-Sánchez et al. 2015). El desarrollo de marcadores moleculares permite la detección de genes favorables en los animales y por medio de estos se han determinado características de calidad de carne vacuna (Casas et al. 2006, Moreno-Sánchez et al. 2012). Las asociaciones de los genes del sistema proteolítico del músculo esquelético, calpaína y calpastatina (CAPN1 y CAST), y las características de terneza de la carne vacuna se evalúan por medio de SNP´s Polimorfismo de nucleótido simple (Page et al. 2002, Moreno-Sánchez et al. 2012, Motter et al. 2013), en donde se buscan modificaciones en los genes (Juszczuk-Kubiak et al. 2004, Calvo et al. 2014) y estos genes mantienen una relación con características de sabor, jugosidad, terneza, grasa intramuscular, área del ojo de lomo y fuerza de corte (Casas et al. 2006, Motter et al. 2013, Allais et al. 2014, Calvo et al. 2014, Lee et al. 2014). Adicionalmente, se han estudiado las asociaciones de estos genes con características de crecimiento, tal como el peso vivo (Motter et al. 2013).
Las características de crecimiento en el ganado vacuno de carne y su valor genético se evalúan por medio de la estimación de diferencias esperadas de la progenie (DEP´s) (NBCEC 2010). En Costa Rica, la evaluación genética en animales Bos indicus se realiza a través del programa de mejoramiento genético de bovinos de carne de la Corporación Ganadera (CORFOGA), de donde se obtiene información a partir de los registros de las mediciones realizadas a nivel de finca de peso y circunferencia escrotal de los animales evaluados, así como de todos los parientes incluidos en el pedigrí para el análisis (CORFOGA 2016). Las DEP’s son las predicciones genéticas, que se pueden estimar para cualquier característica del animal, que a su vez, pueda ser medida con precisión. Los DEP´s son desviaciones de un valor base determinado individualmente para cada raza y con ello es posible la predicción del valor genético de cada individuo (mérito genético). Los resultados son empleados por los productores para la toma de decisiones en la selección de los animales (NBCEC 2010, Pires 2010).
Actualmente en Costa Rica el mérito genético de los animales es calculado al considerar solamente la información fenotípica medida en el animal (CORFOGA 2016). No obstante, existe un interés en la generación de marcadores genéticos para la precisión de la predicción del mérito genético de animales para características que son difíciles de medir (tales como características de canal y calidad de la carne), debido a que la metodología adecuada para calcular en forma precisa el mérito genético de un animal es combinar la información fenotípica medida en el animal con la información de los marcadores genéticos (Casas et al. 2006). Por lo anterior, los objetivos de este estudio fueron evaluar en animales Brahman la presencia de polimorfismos de nucleótido simple (SNP´s) en genes de calpaína y calpastatina previamente asociadas a atributos de terneza en animales Bos taurus, y con base en lo anterior, relacionar la presencia de genes de terneza con las diferencias estimadas de la progenie (DEP´s) positivas para características de crecimiento.
Materiales y métodos
Ubicación y animales
Se genotiparon 82 toros Brahman de registro (animales portadores de un registro genealógico, debidamente inscrito ante la Asociación de Criadores de Ganado Cebú de Costa Rica), seleccionados dentro del programa de mejoramiento genético de la Corporación Ganadera. Las muestras fueron obtenidas de animales cuyo sistema de alimentación fue pastoreo con suplementación, en 7 haciendas ganaderas distribuidas en las regiones Huetar Norte, Huetar Atlántica, Chorotega y región Brunca de Costa Rica. La distribución de los animales muestreados por región geográfica se detalla en el Cuadro 1.
Como criterio de inclusión de animales en el presente estudio se estableció como umbral mínimo para el mérito genético total (MGT) individuos con valores positivos (DEPMGT>0).
Recolección de muestras
El muestreo fue realizado durante el periodo 2014-2016. Las muestras de sangre fueron tomadas a nivel de finca, de la vena caudal o la vena yugular de cada animal. Se recolectaron 3.0 mL de sangre por animal, en un vaccutainer® con ácido etilendiaminotetraacético (EDTA) como anticoagulante. Posteriormente, las muestras fueron mantenidas en refrigeración y enviadas al laboratorio de biotecnología molecular, ubicado en la Sede San Carlos del ITCR, donde se mantuvo la cadena de frío hasta el momento de la extracción del ácido desoxirribonucleico (ADN); todas las muestras fueron procesadas en un lapso máximo de 7 días pos colección.
Marcadores utilizados
Se emplearon marcadores específicos para la detección de polimorfismos de nucleótido simple (SNP´s) para los genes de CAPN 1 (calpaína) y CAST (calpastatina). La descripción de las secuencias de imprimadores (“primers”) utilizados se especifica en el Cuadro 2.
El polimorfismo CAPN316 (Genebank AF252504.2 ) consiste en una transversión que implica la sustitución de bases nitrogenadas Citosina/Guanina (C/G) y, a su vez, se traduce en un cambio aminoacídico de glicina por alanina en la cadena de aminoácidos de la calpaína, mientras que el marcador CAPN4751 (Genebank AF248054.2) se encuentra en el intrón #18 de la región regulatoria del mismo gen (Torres-Rodríguez et al. 2015, White et al. 2005), que es el resultado de una inserción/eliminación de Citosina/Timina (C/T) (Casas et al. 2006, Torres-Rodríguez et al. 2015).
Por otra parte, el polimorfismo CAST2959 (Genebank AF159246.1) que se ubica en la región no traducida 3’ del gen de la Calpastatina (CAST), consta de una transición entre Adenina/Guanina (Barendse 2002).
Cuadro 2 Descripción de secuencias de oligonucleótidos “primers” utilizados como marcadores indicadores específicos (Carvalho et al. 2007).
| Marcador | Primers | Referencias |
|---|---|---|
| CAPN 316 | F: GAGTGGCCCTCATAAGATAA R: CCCATCCTCCATCTTGACC | Casas et al. 2005 |
| CAPN 4751 | F: AAGGGACAGATGTGGACAGG R: GAGGGGTGTTCTCTGAGTGC | White et al. 2005 |
| CAST 2959 | F: ACATTCTCCCCACAGTGCC R: GACAGAGTCTGCGTTTTGCTC | Li et al. 2010 |
Purificación de ADN genómico y secuenciación
Se realizó la purificación de ADN genómico, en la que se verificó que las muestras reunían las condiciones requeridas para proceder con la extracción de ADN. Se realizaron las extracciones y purificaciones de ADN mediante los kits de extracción (DNeasy Blood & Tissue) de Qiagen® y el kit de purificación (Wizard SV Genomic DNA Purification System) de Promega®. A los productos de la purificación de ADN obtenidos se les cuantificó la concentración de ADN obtenida mediante el sistema NanoDrop 8000® de Thermo Scientific.
Se estandarizaron los parámetros de reacción en cadena de la polimerasa (PCR) para los 3 pares de imprimadores seleccionados (CAPN316, CAPN4157 y CAST2959). Se realizaron reacciones mediante gradientes térmicos para determinar la temperatura de alineamiento apropiada con el fin de mejorar los productos amplificados. Una vez optimizados los parámetros de PCR se procedió a la amplificación del total de las muestras seleccionadas (con los 3 juegos de imprimadores definidos) para dicho proyecto. Las muestras amplificadas fueron secuenciadas por la empresa Macrogen (Corea del Sur) (Mediante códigos EZ-Seq adquiridos por CORFOGA) sobre las cuales se inició el proceso de análisis de los SNP´s. A partir de los productos de PCR obtenidos de las reacciones con los pares de imprimadores seleccionados, se realizó el análisis de los fragmentos de ADN secuenciados para la obtención de los genes presentes en cada fragmento de ADN. Se realizó la lectura de los resultados de la secuencia de ADN para los 3 SNP´s empleados en este estudio y la información se unificó en conjuntos de datos para cada uno de los animales.
Diferencias estimadas de la progenie
Las características de crecimiento evaluadas como diferencias esperadas en la progenie de un reproductor, que se utilizaron para relacionar con los marcadores moleculares para los genes CAPN y CAST, fueron el peso al nacimiento (PN), peso al destete (PD), leche (LEC) expresado como el efecto de genes de producción de leche sobre el crecimiento de la progenie; materno total (MTO), expresado como el efecto genético sobre el peso al destete, debido a los genes para crecimiento y a los genes para producción de leche conjuntamente; peso al año (PA), circunferencia escrotal al año (CEA), peso a los 18 meses (P18), circunferencia escrotal a los 18 meses (CEB), y el mérito genético total (MGT) que es un índice estandarizado y combinado de las características anteriores ponderadas.
Análisis estadístico
Se realizó la estimación de las frecuencias genotípicas y las frecuencias alélicas para cada SNP´s, por medio de conteo directo. Para la estimación de las asociaciones entre DEP´s y SNP´s, se revisaron las diferencias esperadas de progenie de los animales, cuya sangre fue procesada en el laboratorio. Se establecieron valores numéricos para cada genotipo y marcador, y se correlacionaron por medio de coeficiente de correlación lineal de Pearson. Se realizó análisis multivariante con el propósito de identificar subpoblaciones del gen CAST2959 del conjunto completo de datos. El primer paso fue realizar un análisis de componentes principales (PCA). Para seleccionar el número de componentes principales a utilizar en el próximo paso del análisis, se siguió el criterio de seleccionar solo aquellos con un valor propio (varianza extraída para ese componente principal particular) >1 (criterio de Kaiser). Además, se comprobó la factibilidad del análisis factorial (AF), mediante la prueba de esfericidad de Bartlett para contrastar la hipótesis nula de que la matriz de correlaciones es una matriz identidad y el índice KMO (Kaiser-Meyer-Olkin), que determina el cálculo de las correlaciones entre 2 variables, una vez eliminada la influencia que las restantes variables ejercen sobre ellas y que indica la conveniencia de realizar el AF. Todos los análisis anteriores se realizaron utilizando el programa InfoStat (v. 2017) (Di Rienzo et al. 2017).
Para calcular el efecto sobre las diferencias estimadas de la progenie de los individuos clasificados como positivos para genes de terneza se clasificaron aquellos que resultaron homocigotos para los marcadores CAPN4751 y CAST2959, mediante una clave bivariante (SNP +; SNP-). Se realizó un análisis bayesiano y se asumió que el efecto de zona y los residuos de los modelos se distribuían normalmente de forma independiente. Se asumieron a priori planos acotados para todos los efectos y varianzas (Blasco 2017). Se consideró el efecto aleatorio del animal y el efecto de finca como un efecto de ruido al no provenir todos los individuos de una misma finca. Las distribuciones posteriores marginales se estimaron utilizando el muestreo de Gibbs, se probó la convergencia para cada cadena con el criterio Z de Geweke y los errores de muestreo de Monte Carlo se calcularon con el uso de procedimientos de series temporales (Sorensen y Gianola 2002). Se usaron cadenas de 60 000 muestras con un burn-in de 10 000. Para el análisis se utilizó el programa Rabbit desarrollado por el Instituto de Ciencia y Tecnología Animal (Valencia, España). Los parámetros obtenidos de las distribuciones posteriores marginales de las diferencias fueron la media (D), la región de densidad posterior más alta al 95% (HPD95%) y la probabilidad de que los individuos con SNP+ y SNP- sean diferentes, calculada como la probabilidad de que la diferencia entre SNP negativos y positivos sea mayor que cero, dado el valor absoluto de D (P0). Se pueden encontrar más detalles de estos parámetros bayesianos en Blasco (2001, 2005).
Resultados y discusión
Secuencias de ADN
La extracción de ADN de las muestras produjo un mínimo de concentración de ADN de 7, 306 ng.μL-1 y a partir de esa concentración, se obtuvo un total de 241 secuencias. Se determinó la secuencia de bases para 79 muestras del marcador CAPN316; 79 secuencias para el marcador CAPN4751 y 81 de muestras para el marcador CAST2959.
Frecuencias genotípicas
Para el gen CAPN 1, se consideró el genotipo Citosina/Citosina (CC) como favorable en los marcadores CAPN316 y CAPN4751, para características relativas a la calidad de carne (Cuetia et al. 2012). Para el marcador CAPN316 las frecuencias alélicas de C y G fueron 0,006 (0,6%) y 0,994 (94,4%) respectivamente, con frecuencias genotípicas para CC de 0,000 (0%), para CG de 0,013 (1,3%) y para GG 0, 987 (98,7%) (Cuadro 3), lo cual indica equilibrio Hardy-Weinberg (EHW).
Lo anterior apunta una segregación inferior del alelo C en comparación con el alelo G; el alelo C, para el marcador CAPN316, se encuentra asociado con fuerza de corte y terneza (White et al. 2005), y el genotipo CC se encuentra asociado a la característica de terneza y aceptación del cliente (Avilés 2013). Lo encontrado en este estudio es coincidente con lo expuesto por varios autores, donde la segregación del alelo C (5%, 21%, 21,9%) es inferior a la segregación del alelo G (White et al. 2005, Cuetia et al. 2012, Pinilla 2014).
Cuadro 3 Frecuencias alélicas y genotípicas para los genes µ -Calpaína (CAPN) y Calpastatina (CAST) en la población de estudio.
| CAPN 1 | CAST | ||||||
| CAPN316 | CAPN3751 | CAST2959 | |||||
| C | G | C | T | A | G | ||
| 0,006 | 0,0094 | 0,165 | 0,835 | 0,667 | 0,333 | ||
| CC CG GG | CC CT TT | AA AG GG | |||||
| 0,000 0,013 0,987 | 0,025 0,279 0,696 | 0,432 0,469 0,099 | |||||
En el presente estudio no se encontraron individuos con genotipos CC y se evidenció una muy baja cantidad de individuos heterocigotos (CG), esto concuerda con investigaciones anteriores en donde la población de individuos con genotipo CC era prácticamente nula y una sub representación de animales con genotipo CG (12%) (Torres-Rodríguez et al. 2015). En otro estudio, realizado por Casas et al. 2006, se indicó una frecuencia de 0% para el genotipo CC en el gen CAPN 1 específicamente en una población Brahman.
El genotipo CG es presentado en un único individuo de la población estudiada, lo cual sugiere escasez del genotipo en la población, esto concuerda con el estudio desarrollado por Calvo et al. 2014, donde fue mostrado un déficit de individuos heterocigotos en el análisis de SNP´s en el gen CAPN 1. Sin embargo, a pesar de que el gen podría estar en muy baja frecuencia en la población, es necesario continuar con trabajos de muestreo y secuenciación para determinar con mayor precisión la frecuencia genotípica en la población. Por otra parte, el genotipo GG se presentó en la mayoría de la población estudiada para el marcador CAPN 316. Este resultado coincide con lo encontrado por varios autores (Allais et al. 2011, Pinilla 2014, Torres-Rodríguez et al. 2015), quienes encontraron valores sobre 80% del genotipo GG en las poblaciones vacunas.
En investigaciones anteriores se han encontrado efectos significativos (p<0,05) entre el alelo G y la dureza de la carne (Allais et al. 2011, Torres-Rodríguez et al. 2015). Es posible que los genotipos GG, al relacionarse con mayores procesos de contracción muscular, produzcan carne con menor terneza en comparación con animales con el genotipo CC (Casas et al. 2006). El marcador CAPN316 (μ-calpaína) tiene una asociación positiva con características de calidad de la carne bovina (White et al. 2005, Avilés et al. 2009, Gruber et al. 2011); no obstante, en los estudios sobre este marcador han sido encontradas bajas frecuencias genotípicas en poblaciones Bos indicus y Bos taurus (Pinilla 2014). Asimismo, específicamente en la raza Brahman, se ha encontrado que este gen se haya en una baja frecuencia (White et al. 2005), o bien, que los hallazgos encontrados para este marcador no son significativos (Robinson et al. 2012).
El marcador CAPN4751 (μ-calpaína) también se encuentra asociado a la característica de terneza en carne vacuna (White et al. 2005, Gruber et al. 2011, Robinson et al. 2012). Un genotipo favorable en este marcador en poblaciones Bos indicus, Bos taurus y mestizas, se encuentra asociado significativamente a una menor fuerza de corte de la carne (p<0,001) (White et al. 2005). Para el marcador CAPN4751 las frecuencias alélicas fueron de 0,165 (16,5%) para C y 0,835 (83,5%) para T, asimismo, las frecuencias genotípicas para CC, CT y TT fueron 0,025 (2,5%), 0,279 (27,9%) y 0,696 (69,6%) respectivamente, por lo que la población estudiada para este marcador se encuentra en EHW, según resultados de la ecuación empleada. Se ha reportado baja presencia del alelo C y genotipo CC (16% y 49% respectivamente) (Torres-Rodríguez et al. 2015), lo cual coincide con lo reportado en este trabajo, sin embargo, otros autores han encontrado una mayor frecuencia (55%) para el alelo C en el marcador CAPN 4751 (Cuetia et al. 2012). La variación en la frecuencia del alelo C en diferentes poblaciones tiene una explicación en el efecto particular del marcador específico entre cada raza, ya que se encuentran diferencias entre marcadores moleculares para el gen CAPN 1 y la raza (Allais et al. 2011). Asimismo, Torres-Rodríguez et al. (2015) señalan que la variabilidad de la raza de la población estudiada influye en la aparición de genes favorables para terneza de la carne.
Las frecuencias del SNP CAST2959 en el presente estudio fueron de 0,333 (33,3%) para el alelo G y 0,667 (66,7%) para el alelo A, a su vez fueron de 0,099 (9,9%) para GG, 0,469 (46,9%) para AG y de 0,432 (43,2%) para AA (EHW). El alelo A en los genotipos AA y AG en el gen CAST y marcador molecular CAST2959, han demostrado un mejor desempeño en características de calidad de carne (Leal-Gutiérrez et al. 2015). Los alelos de estos genotipos se encuentran asociados a características de calidad de carne como fuerza de corte, retención de agua y terneza (Cuetia et al. 2012, Avilés 2013, Pinilla 2014, Leal-Gutiérrez et al. 2015).
La mayor proporción del alelo A encontrada coincide con estudios anteriores (Cuetia et al. 2012, Pinilla 2014, Torres-Rodríguez et al. 2015), con valores de 66%, 69% y 65% respectivamente, con una sub representación del alelo G (Calvo et al. 2014). En el presente estudio la mayor proporción de animales presentaron el genotipo AG (46,9%), seguido del genotipo AA (43,2%), lo que difiere con estudios previos donde el genotipo AA se ha presentado en mayor proporción en población con valores de 53% (Cuetia et al. 2012) y 80% (Torres-Rodríguez et al. 2015) y diferencias con respecto a los heterocigotos. No obstante, se ha descrito que el genotipo AA se presenta con mayor frecuencia en animales Bos taurus (Cuetia et al. 2012), lo cual es apoyado por lo encontrado por Pinilla (2014), donde poblaciones de animales con madre Brahman presentaron una frecuencia de 35% para el alelo AA; lo anterior justifica la dominancia del genotipo AG en la población estudiada (Bos indicus).
El genotipado de los genes asociados con la proteólisis Miofibrilar (CAPN1 y CAST) es utilizado para predecir la terneza de la carne y la fuerza de corte (Avilés et al. 2013). Sin embargo, existe una variación entre las poblaciones Bos indicus y Bos taurus que afecta la terneza y la detección de marcadores (gen CAPN 1), lo cual sugiere la necesidad de desarrollar el uso de marcadores diferenciados por especie (Casas et al. 2005). Adicionalmente, existe un efecto particular del marcador específico entre cada raza al existir diferencias entre los marcadores moleculares en el gen CAPN 1 y CAST, en relación con la fuerza de corte y terneza (Allais et al. 2011). Asimismo, los marcadores deben ser validados en poblaciones locales, ya que cada grupo existente posee patrones genéticos particulares (Torres-Rodríguez et al. 2015), lo cual puede alterar la interpretación de los resultados.
Subpoblaciones con genes favorables (SNP + ) y desfavorables (SNP - )
Los individuos que resultaron con genes favorables para las características de suavidad de la carne en los genes CAPN4751 y CAST2959, y los individuos que no presentaron genes favorables, se clasificaron en 2 subgrupos con el propósito de analizar el efecto de la presencia de genes de terneza sobre las características de crecimiento a través de las diferencias estimadas de la progenie. En el Cuadro 4, se presentan las distribuciones marginales de las diferencias entre las subpoblaciones con presencia o ausencia del gen CAPN-4751 favorable para terneza. La diferencia entre los grupos que presentaron genes favorables y desfavorables para terneza sólo fue significativa en el caso del peso al nacimiento (P0=0,79). Esto indica que los animales con genes favorables, en promedio, tienden a tener mayores diferencias estimadas de la progenie (0,7 kg) para esa característica y, además, esa diferencia entre grupos es relevante con un 70% de probabilidad. Lo anterior, pone de manifiesto que es posible iniciar procesos de selección genética basados en genes de suavidad de carne y como resultado de una selección indirecta, mejorar o incrementar por término medio parámetros de crecimiento animal, tal y como se ha descrito en la literatura (Van Eenennaam et al. 2007, Curi et al. 2009). No hubo diferencias significativas entre los grupos favorables y desfavorables para el gen CAPN-4751 para las características de crecimiento en Brahman.
Cuadro 4 Características de las distribuciones posteriores marginales estimadas de las diferencias entre subpoblaciones para el gen CAPN4751 con polimorfismos de un solo nucleótido positivos y negativos (SNP- - SNP+) para las diferencias estimadas de la progenie DEP´s.
| DEP | D1 | HPD95% 2 | P0 3 | R4 | PR 5 |
| PN | -0,70 | -2,44 - 1,03 | 0,79 | 0,25 | 0,70 |
| PD | -1,15 | -5,58 - 6,64 | 0,69 | 0,72 | 0,58 |
| LEC | 0,17 | -1,98 - 2,31 | 0,56 | 0,31 | 0,45 |
| MTO | -0,028 | -0,45 - 0,38 | 0,56 | 0,06 | 0,44 |
| PA | 1,97 | -6,64 - 11,54 | 0,67 | 1,15 | 0,57 |
| CEA | -0,02 | -1,84 - 1,82 | 0,52 | 0,23 | 0,41 |
| P18 | 1,76 | -10,35 - 13,90 | 0,61 | 1,68 | 0,49 |
| CEB | -0,25 | -1,71 - 1,18 | 0,64 | 0,21 | 0,52 |
| MGT | -0,03 | -0,45 - 0,38 | 0,56 | 0,06 | 0,44 |
1D: media de la distribución marginal posterior de las diferencias entre SNP´s (SNP- - SNP+); 2HPD95%: región de alta densidad posterior al 95% de probabilidad; 3P0, probabilidad de que la diferencia sea >0 si la D es positiva, o <0 si es negativa; 4R, valor relevante estimado como 1/3 de desviación típica; 5PR, probabilidad de que la diferencia sea >R si D es positiva, o <R si D es negativa. DEP: diferencia estimada de la progenie; PN: peso al nacimiento; PD: peso al destete; LEC: leche; materno total (MTO); peso al año (PA); circunferencia escrotal al año (CEA); peso a los 18 meses (P18); circunferencia escrotal a los 18 meses (CEB); mérito genético total (MGT).
En el Cuadro 5, se presentan las distribuciones marginales de las diferencias entre las subpoblaciones con presencia o ausencia del gen CAST2959 favorable para terneza. Las diferencias entre subpoblaciones con presencia o ausencia del gen favorable fueron significativas para el peso al nacimiento, peso al destete, leche, peso al año, peso a los 18 meses y circunferencia escrotal a los 18 meses, sin embargo, sólo en el caso de peso al nacimiento, peso a los 18 meses y circunferencia escrotal a los 18 meses, esas diferencias fueron relevantes (PR>0,80). El peso al nacimiento y al destete fue mayor en los individuos con presencia de genes favorables para terneza, lo que sugiere que en esa población los pesos en edades tempranas están relacionados con la presencia del marcador CAST2959, aunque fuese en baja frecuencia (Figura 1). En el caso del peso a los 18 meses y la circunferencia escrotal a los 18 meses los individuos con genes desfavorables mostraron mayores valores de DEP´s, lo cual indica que la presencia o ausencia del gen CAST2959, es decir, la regulación de su expresión génica se podría modular en función del crecimiento fisiológico del animal, sin embargo, esto requiere de mayor investigación a futuro.
Cuadro 5 Características de las distribuciones posteriores marginales estimadas de las diferencias entre subpoblaciones para el gen CAST2959 con polimorfismos de un solo nucleótido positivos y negativos (SNP- - SNP+) para las diferencias estimadas de la progenie DEP´s.
| D1 | HPD95% 2 | P0 3 | R4 | PR 5 | |
| PN | -0,67 | -1,36 - 0,11 | 0,96 | 0,25 | 0,87 |
| PD | -0,80 | -2,66 - 1,11 | 0,80 | 0,72 | 0,53 |
| LEC | 0,31 | -0,55 - 1,24 | 0,76 | 0,31 | 0,50 |
| MTO | 0,017 | -0,15 - 0,21 | 0,58 | 0,06 | 0,32 |
| PA | -1,06 | -4,83 - 2,69 | 0,72 | 1,15 | 0,48 |
| CEA | -0,15 | -0,89 - 0,61 | 0,66 | 0,23 | 0,42 |
| P18 | 3,98 | -0,79 - 9,24 | 0,94 | 1,68 | 0,82 |
| CEB | 0,59 | -0,04 - 1,21 | 0,97 | 0,21 | 0,89 |
| MGT | 0,017 | -0,15 - 0,21 | 0,58 | 0,06 | 0,32 |
1D: media de la distribución marginal posterior de las diferencias entre SNP´s (SNP- - SNP+); 2HPD95%: región de alta densidad posterior al 95% de probabilidad; 3P0, probabilidad de que la diferencia sea >0 si la D es positiva, o <0 si es negativa; 4R, valor relevante estimado como 1/3 de desviación típica; 5PR, probabilidad de que la diferencia sea >R si D es positiva, o <R si D es negativa. DEP: diferencia estimada de la progenie; PN: peso al nacimiento; PD: peso al destete; LEC: leche; materno total (MTO); peso al año (PA); circunferencia escrotal al año (CEA); peso a los 18 meses (P18); circunferencia escrotal a los 18 meses (CEB); mérito genético total (MGT).
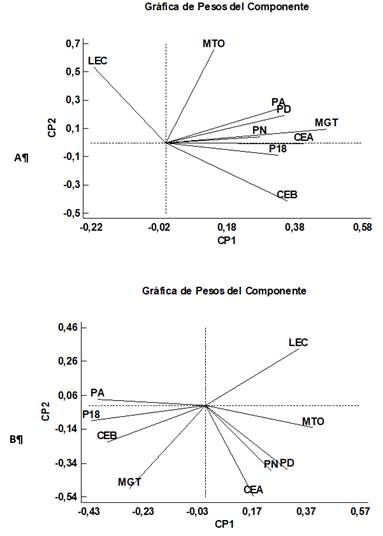
Figura 1 Proyección de las características en el plano definido por 2 componentes principales. A: Subpoblación con genes desfavorables para CAST2959; B: Subpoblación con genes favorables para CAST2959; PN: peso al nacimiento; PD: peso al destete; LEC: leche; materno total (MTO); peso al año (PA); circunferencia escrotal al año (CEA); peso a los 18 meses (P18); circunferencia escrotal a los 18 meses (CEB); mérito genético total (MGT).
Asociación genes favorables
En el Cuadro 6 se presenta la estimación de los coeficientes de correlaciones lineales de Pearson para las diferencias esperadas de progenie y la presencia de los genes favorables para suavidad de la carne. En general, se observan correlaciones bajas entre las diferencias esperadas de progenie y la aparición de genes favorables para terneza. La correlación entre marcadores del gen CAPN 1 (CAPN 316 y CAPN 4751) fue -0,070. Tampoco hubo asociación lineal entre los marcadores del gen CAPN 1 y CAST. Con respecto a la aparición de genes favorables y las diferencias esperadas de progenie, el marcador CAPN316 presentó una correlación con la característica de mérito genético total de 0,23; aunque es la correlación más alta encontrada de todas las características de crecimiento, lo cual era de esperar al ser esta característica un índice ponderado de las demás, se debe interpretar con cautela, ya que en el presente estudio solamente el 1,21% de la población presentó un genotipo favorable para este marcador (heterocigoto). Otros autores han descrito que no existe una asociación (p>0,05) entre los marcadores moleculares de los genes del sistema calpaína con el crecimiento en raza Brahman (Cafe et al. 2010). Esto podría implicar que la presencia de genes favorables para terneza no influya sobre el crecimiento y características de la canal del ganado Brahman. Sin embargo, algunos efectos evidentes para el estado CAPN1 indican la necesidad de nuevos estudios acerca de los efectos de los marcadores sobre la producción.
Cuadro 6 Coeficientes de correlación lineal de Pearson entre características de crecimiento expresadas como diferencia estimadas de la progenie y marcadores moleculares (índice d de Cohenα) para suavidad de la carne.
| CAPN4751 α | CAST2959 | PN α | PD | LEC | MTO | PA | CEA | P18 | CEB | MGT | |
| CAPN4751 | 1 | ||||||||||
| CAST2959 | -0,3079 | 1 | |||||||||
| PN | 0,1222 | 0,1961 | 1 | ||||||||
| PD | 0,1856 | -0,1193 | 0,5127* | 1 | |||||||
| LEC | -0,0233 | -0,0488 | -0,2562 | -0,2672 | 1 | ||||||
| MTO | 0,1468 | -0,1427 | 0,2601 | 0,6835* | 0,5208* | 1 | |||||
| PA | -0,0979 | 0,1330 | 0,1176 | 0,1330 | -0,0740 | 0,0617 | 1 | ||||
| CEA | 0,1315 | -0,1635 | 0,2873 | 0,4590* | -0,2332 | 0,2300 | 0,5416* | 1 | |||
| P18 | 0,0755 | -0,2324 | -0,1122 | 0,0227 | -0,3189 | -0,2214 | 0,4026* | 0,3640* | 1 | ||
| CEB | 0,1752 | -0,3940* | -0,0180 | 0,1723 | -0,5835* | -0,2893 | 0,2773 | 0,5708* | 0,7382* | 1 | |
| MGT | 0,1251 | -0,1306 | 0,3989* | 0,5480* | -0,3458 | 0,2235 | 0,7258* | 0,8046* | 0,6716* | 0,6591* | 1 |
*Significancia estadística, p-valor <0,05. α Valores de correlación para marcadores expresados como el índice de asociación d de Cohen. PN: peso al nacimiento; PD: peso al destete; LEC: leche; materno total (MTO); peso al año (PA); circunferencia escrotal al año (CEA); peso a los 18 meses (P18); circunferencia escrotal a los 18 meses (CEB); mérito genético total (MGT).
El marcador CAST2959 presentó una correlación baja y negativa con la característica circunferencia escrotal a los 18 meses (-0,28). Lo anterior podría deberse al proceso de selección realizado por los ganaderos orientado a características de crecimiento y desfavoreciendo otras características relevantes, como las reproductivas, donde se seleccionan animales en edades tardías; no obstante, no se indican correlaciones moderadas o altas para ninguna de las variables de crecimiento y aparición de genes favorables para terneza. Al analizar las asociaciones existentes entre mediciones fenotípicas y los genes para rasgos de suavidad de la carne, se concluye que la selección de animales basada en características cuantitativas no causa efecto adverso sobre los genes del animal (Wolcott et al. 2009); es decir, las características no se encuentran correlacionadas. Por otra parte, al evaluar marcadores moleculares del gen CAST, ninguno de los marcadores se encontró asociado con el peso vivo, terneza y otras características de calidad de carne, tal y como se ha descrito por Motter et al. (2013). Esto sugiere que es necesario continuar con la búsqueda de marcadores moleculares que reflejen la variabilidad presentada por cada raza.
Conclusiones
Las frecuencias alélicas y genotípicas para el alelo favorable C en el gen CAPN316 fueron muy bajas, incluso nulas en algunos casos en la población Brahman estudiada.
Para los marcadores genéticos CAPN4751 y CAST2959, se encontraron animales distribuidos en los 3 genotipos.
En el análisis de asociación entre SNP´s favorables y DEP´s, se encontraron asociaciones muy débiles o incluso ausencia de correlación.
No se encontraron asociaciones relevantes entre los polimorfismos de nucleótido simple entre sí, ni entre los polimorfismos de nucleótido simple (CAPN 4751 y CAST 2959) y las diferencias estimadas de la progenie para las características de crecimiento en Brahman