El coronavirus del síndrome respiratorio agudo grave tipo 2 (SARS-CoV-2) es un miembro de la familia Coronaviridae, género Betacoronavirus, subgénero Sarbecovirus, y es el agente etiológico de la enfermedad causada por el SARS-CoV-2 (COVID-19).1 Esta patología suele cursar con tos, fiebre, disnea y fatiga, lo cual puede progresar a cuadros más graves de neumonía y muerte.2 Dicho patógeno se ha diseminado globalmente y ha generado un problema alarmante de salud pública que engloba a más de 768 millones de infectados y más de 6.9 millones de muertes al 26 de julio del 2023 (World Health Organization. WHO COVID-19 dashboard. [Internet]. Geneva: WHO; 2020 [accesado 30-07-2023]. Disponible en: https://covid19.who.int/). Debido a estas cifras, tanto la vigilancia epidemiológica como el uso de métodos diagnósticos eficientes y prácticos resultan fundamentales para el entendimiento de la evolución de la pandemia y la propagación del virus.
Las pruebas de ácidos nucleicos que detectan el genoma de ácido ribonucleico (ARN) del SARS-CoV-2 han sido una herramienta clave en el manejo de la pandemia, siendo la reacción en cadena de la polimerasa con transcriptasa reversa (RT-PCR) la prueba considerada como el estándar de oro en el diagnóstico del cuadro.
Sin embargo, pese a su rendimiento satisfactorio, 3 esta prueba no está exenta de limitaciones; se ha visto que el RT-PCR puede generar resultados falsos negativos hasta en un 30% de los casos, ya sea por una toma de muestra deficiente o por muestreos en lapsos avanzados o insuficientes tras la infección o progresión del cuadro hacia vías respiratorias inferiores.4,5 Además, los resultados de la RT-PCR detectan la presencia del virus en una muestra, mas no reflejan la respuesta inmune o la exposición pasada del paciente al virus. Otras limitaciones son los extensos periodos de respuesta, los elevados costos de operación (sea por reactivos o equipo especializado) y la necesidad de personal calificado para ejecutar la prueba.
Por otra parte, la medición de la respuesta humoral o anticuerpos se ha empleado como complemento de las pruebas moleculares, principalmente en el diagnóstico decasos sospechosos negativos por RT-PCR o en infecciones pasadas de pacientes con síntomas no específicos o asintomáticos.6 Las pruebas serológicas específicamente presentan una serie de ventajas sobre la detección de ARN, como son la facilidad de ejecución e interpretación de los resultados, la mayor accesibilidad económica y la rapidez en la generación de datos. La utilidad de estos ensayos en la detección de infecciones agudas se ve limitada, pues, en su mayoría, los anticuerpos son detectables aproximadamente tras dos semanas de infección; 6,7 sin embargo, presentan una serie de aplicaciones fundamentales para el buen manejo de la pandemia. Por ejemplo, estas pruebas permiten estudiar la respuesta inmunológica poblacional mediante seroprevalencia, estimar tasas de infección, indicar el estado inmunitario y/o de infección en el paciente, identificar a individuos que desarrollan una respuesta de anticuerpos robusta (como potenciales donadores para terapias con suero convaleciente), evaluar posibles respuestas protectoras contra la infección (mediante correlación con anticuerpos neutralizantes) y, de gran importancia actual, valorar la eficacia del proceso de vacunación.8-10 Para comprender y aplicar los conocimientos derivados de estos procesos, se vuelve necesario contar con ensayos serológicos prácticos de bajo costo, pero confiables y de alto rendimiento analítico.
Con base en dicha necesidad, este estudio expone la estandarización y la evaluación del desempeño de un ensayo por inmunoadsorción ligado a enzima (ELISA) para la detección semicuantitativa de inmunoglobulinas M (IgM) e inmunoglobulinas G (IgG), empleando como sustrato el dominio de unión a receptor (RBD) de la proteína de espícula del SARS-CoV-2. La espícula (S) del SARS-CoV-2 es una glicoproteína en conformación de homotrímero, constituida por las subunidades S1, S2 y el RBD, altamente inmunogénica y conservada entre los Sarbecovirus.10 Como parte de esta investigación, se detalla el protocolo de producción de los antígenos recombinantes empleados como sustrato en el ELISA, el análisis de una serie de características demográficas y clínicas propias de la población ensayada que pueden relacionarse al desarrollo de la respuesta inmunológica contra la infección, así como la posible interferencia por reacción cruzada con otras virosis de alta prevalencia en Costa Rica, como el dengue (Pan American Health Organziation. PAHO/WHO Data -Costa Rica- Casos de dengue. [Internet]. Washington D. C.: PAHO/WHO; 2014 [accesado 10-06-2023]. Disponible en: https://www3.paho.org/data/index.php/es/temas/indicadoresdengue/dengue-subnacional/537-cri-dengue-casos-es.html). Lo anterior tiene el fin de determinar la posible aplicación de dichos ensayos como herramientas de apoyo diagnóstico y evaluar la respuesta inmune ante la infección por SARS-CoV-2 o su respectivo proceso de vacunación en costarricenses.
Materiales y métodos
Expresión y purificación de la proteína de SARS-CoV-2
El método de producción de la proteína recombinante se realizó con base en un protocolo previamente publicado y modificado por nuestro laboratorio.10, 11 De forma breve, la secuencia del nucleótido optimizado por codones de células de mamíferos que codifica por la proteína de espícula del SARS-CoV-2 (GenBank:MN908947.3) fue sintetizada comercialmente. El RBD (aminoácidos 319-541; RVQP…CVNF), junto a un péptido señal (aminoácidos 1-14; MFVF…VSSQ) y una etiqueta de hexahistidina, se clonaron en el vector de expresión en células de mamífero pCAGGS. La versión soluble de la proteína de espícula (aminoácidos 1-1,213; MFVF…IKWP), que incluye un sitio de escisión de trombina C-terminal, el dominio de trimerización de la fibritina T4 y la etiqueta de hexahistidina también se clonaron en el vector pCAGGS. La secuencia de la proteína se modificó para eliminar la escisión polibásica (RRAR a A) y se introdujeron dos mutaciones estabilizadoras (K986P y V987P; tipo salvaje). Las proteínas recombinantes se produjeron en células Expi293F (Thermo Fisher Scientific; Waltham Massachusetts, Estados Unidos) por medio de transfecciones de ADN purificado con el reactivo Polietilenimina (Polyethylenimine 25 kDa linear PEI, cat. No. 23966, Polysciences Inc.; Warrington Pennslvania, Estados Unidos) según protocolo.11, 12 Tres días después de la transfección, se recogieron los sobrenadantes de las células por centrifugación a 4000 g durante 20 minutos y luego se incubaron con 6 mL de agarosa Ni-NTA (Qiagen; Venlo, Países Bajos) durante 1-2 horas a temperatura ambiente. El Ni-NTA se recogió con columnas de flujo por gravedad y luego se eluyó la proteína. Cada proteína se concentró en unidades de centrifugación Amicon (EMD Millipore; Burlington Massachusetts, Estados Unidos) y se resuspendió en solución salina tamponada con fosfatos (PBS).
Las proteínas recombinantes se analizaron a través de un peso molecular correcto y de una electroforesis en gel de poliacrilamida con dodecilsulfato de sodio (SDS-PAGE) para comprobar su integridad. Se mezcló 1 μg de proteína con tampón Laemmli 2x, que contiene β-mercaptoetanol al 5% en proporción de 1:1. Las muestras se calentaron a 100°C por 15 minutos y se cargaron en un gel de poliacrilamida (5-20%; Bio-Rad; Hercules California, Estados Unidos). Los geles se incubaron con un anticuerpo anti-histidina murino (Sigma-Aldrich MA1-21315; St. Louis Missouri, Estados Unidos) y un anticuerpo anti-ratón acoplado con peroxidasa (Life Technologies; Carlsbad California, Estados Unidos) y se revelaron con Lumi-Light Plus Western Blotting Substrate (Roche; Basel, Suiza) con el equipo Chemidoc (Bio-Rad; Hercules California, Estados Unidos).
Desarrollo, estandarización y ejecución de los ensayos tipo ELISA
El proceso de generación y la ejecución del ELISA se realizó modificando un protocolo previamente reportado.10 De forma breve, se recubrieron placas de 96 pozos (Nunc MaxiSorp M9410, Thermo Fisher Scientific; Waltham
Massachusetts, Estados Unidos) con 50 μL por pocillo de una solución de 2 μg/mL de cada proteína suspendida en PBS y se incubaron a 4 °C durante una noche. A la mañana siguiente, se eliminó la solución de recubrimiento, se lavó tres veces la placa con solución de lavado PBS con Tween 20 al 0.1% (PBST) y se agregaron 100 μL por pocillo de leche descremada al 3%, preparada en PBST como solución de bloqueo. Se incubó a temperatura ambiente durante 1 h y se lavó nuevamente tres veces con solución de lavado antes de la adición de las muestras.
Las muestras de suero se inactivaron previamente a 56 °C durante 1 h para reducir el riesgo de posibles virus residuales y, además, para desactivar el complemento. Se realizaron diluciones de las muestras (sueros o controles) en leche descremada al 1% preparada en PBST. Se agregaron por triplicado (n=3) 50 μL de cada dilución a las placas e incubaron a 4 °C durante una noche. Posteriormente, las placas se lavaron tres veces con 200 μL por pocillo de PBST al 0,1%. Se agregaron 100 μL a cada pozo de una dilución 1:50 000 de IgG anti-humano preparado en cabra, conjugado con enzima peroxidasa de rábano picante (HRP) (Sigma; St. Louis Missouri, Estados Unidos) preparado en PBST al 0,1%, dejando incubar por 1 hora con protección contra la luz. Las placas se lavaron nuevamente tres veces con PBST al 0,1%. A continuación, se agregaron 50 μL de sustrato TMB (3,3’,5,5’-tetrametilbenzidina; Sigma; St.
Louis Missouri, Estados Unidos) a cada pocillo. Esta solución se dejó en las placas durante 10 min y se detuvo la reacción mediante la adición de 50 μL por pocillo de ácido sulfúrico 1 M. La densidad óptica (OD) se midió a 450 nm y a 630 nm para referencia usando un lector de placas Epoch (BioTek; Winooski Vermont, Estados Unidos).
Como control positivo en el proceso de estandarización del inmunoensayo, se empleó un anticuerpo humano monoclonal recombinante anti-SARS coronavirus, clon CR3022, producido en células HEK293, y obtenido a través de BEI Resources, NIAID, NIH (NR-52481). Como control negativo, se utilizó un pool de 10 sueros prepandémicos, tomados entre 2018 y 2019 por nuestro laboratorio para determinaciones serológicas contra otros virus. La esquematización gráfica del inmunoensayo tipo ELISA desarrollado en el presente estudio se muestra en la Figura 1.
Obtención de los sueros
Un total de 129 muestras de suero provenientes de donantes voluntarios con infección confirmada por SARS-CoV-2 vía RT-PCR mediante hisopado nasofaríngeo, obtenidas y procesadas entre agosto del 2020 y noviembre del 2021, se utilizaron como muestras positivas para evaluar el rendimiento de cada ensayo (Cuadro 1), en concordancia con los permisos brindados por el Comité Ético Científico de la Universidad de Costa Rica (CEC-333-2020, CEC-164-2021) y por el consentimiento informado dado por el Banco Nacional de Sangre al donar sangre para suero convaleciente.
Adicionalmente, se recibieron muestras seriadas de 99 donantes voluntarios para examinar la respuesta de la prueba ante la cinética serológica en distintos contextos de exposición al antígeno viral. De forma paralela, un panel de 196 muestras de suero prepandémicas, recolectadas en Costa Rica antes de diciembre 2019, provenientes de donantes voluntarios y almacenadas en la seroteca del laboratorio de virología, fueron empleadas como muestras negativas por SARS-CoV-2 en la evaluación del desempeño de las pruebas.
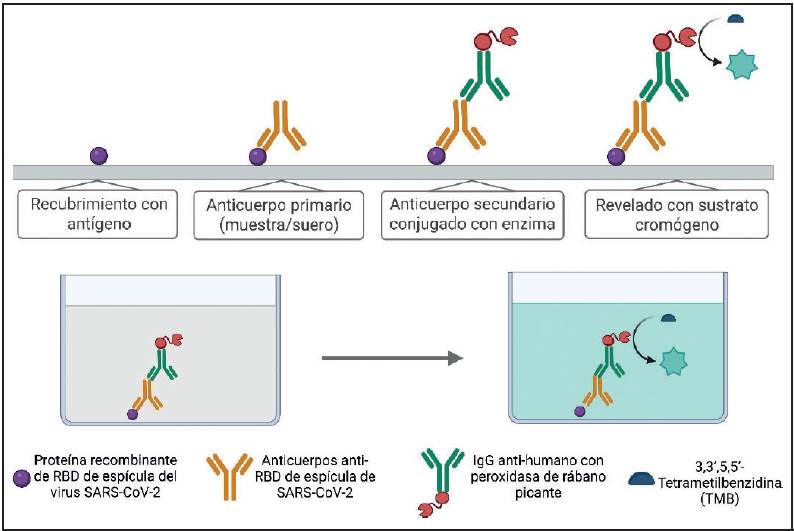
Figura 1 Diagrama de las fases de un ensayo por inmunoadsorción ligado a enzima (ELISA) indirecto para la detección de anticuerpos anti-RBD de proteína de espícula del virus SARS-CoV-2. RBD= Dominio de unión a receptor. Figura de elaboración propia utilizando BioRender.com.
Cuadro 1 Descripción de los especímenes de suero empleados como muestra s positivas, negativas o seriadas para evaluar la respuesta de cinética serológica en el ensayo de detección de anticuerpos anti-RBD de proteína de espícula del virus SARS-CoV-2
| Variable | Característica | Frecuencia | Porcentaje |
| Procedencia de muestras positivas por la infección viral (N=129) | Donantes voluntarios no anónimosa Sueros convalecientes anónimosb | 46 83 | 35.7 64.3 |
| Procedencia de muestras prepandémicas (N=196 | Ensayo de seroprevalencia de virus dengue13 Donantes voluntarios anónimos | 36 160 | 18.4 81.4 |
| Voluntarios con muestras seriadas según número de muestras (N=99) | Dos muestras Tres muestras Cuatro muestras Cinco muestras | 63 22 12 2 | 63.6 22.2 12.1 2.0 |
| Estado inmunitario de voluntarios con muestras seriadas (N=99) | Infección viral pasada Vacunación previa Sin exposición antigénica | 9 47 43 | 9.1 47.5 43.4 |
a. Donantes voluntarios incluidos sin ningún criterio de inclusión clínico o demográfico específico.
b. Muestras donadas por el Instituto Clodomiro Picado (ICP) y el Banco Nacional de Sangre.
El análisis de los datos se llevó a cabo utilizando la versión 4.1.2 de R (R Core Team. R: A language and environment for statistical computing. [Internet]. Vienna: R Foundation for Statistical Computing; 2021 [accesado 08-11-2021]. Disponible en: https://www.Rproject.org/), junto al empleo del paquete tidyverse y ggplot2.14 Todos los valores de OD obtenidos a partir de los ensayos tipo ELISA se normalizaron dividiendo el promedio de las lecturas de cada muestra entre el punto de corte de la técnica, correspondiente al límite de cuantificación o cutoff (promedio de OD del control negativo sumado a diez veces su desviación estándar) de la corrida correspondiente, con el fin de poder hacer comparaciones entre datos de distintas corridas. Se calculó la sensibilidad, especificidad, valor predictivo positivo (VPP) y valor predictivo negativo (VPN) para los dos isotipos de anticuerpo, como marcadores del rendimiento de los ensayos en desarrollo. Para ello se emplearon las siguientes fórmulas, entendiendo en ellas los parámetros de “verdadero” como la concordancia de los resultados del estándar de oro (RT-PCR) con la prueba en evaluación (ELISA RBD) (positivo para infección presente y negativo para infección ausente), y los criterios de “falso” como la discordancia entre ambas pruebas (empleado como referencia el estándar de oro diagnóstico RT-PCR).
Sensibilidad = [verdaderos positivos / (verdaderos positivos + falsos negativos)] x 100
Especificidad = [verdaderos negativos / (verdaderos negativos + falsos positivos)] x 100
VPP = [verdaderos positivos / (verdaderos positivos + falsos positivos)] x 100
VPN = [verdaderos negativos / (verdaderos negativos + falsos negativos)] x 100
Para evaluar la posible asociación entre la seropositividad por dengue y la seropositividad por SARS-CoV-2, se ejecutó una prueba de chi cuadrado. Las diferencias de los valores de OD según sexo y autorreporte de presencia o ausencia de síntomas se analizaron mediante pruebas de los rangos con signo de Wilcoxon. La correlación entre la edad en años y los valores de OD se estimó mediante una prueba de Spearman, donde los valores p <0.05 se consideraron estadísticamente significativos.
Resultados
Estandarización de la técnica de ELISA
Para obtener los mejores resultados, se probaron sistemáticamente distintos tipos y marcas de placas de ELISA (Immunolon, MaxiSorp, etc.), distintas soluciones de bloqueo (leche descremada al 1% y al 3%, caseína, suero fetal bovino al 1%), y se variaron temperaturas y tiempos de incubación utilizando el anticuerpo CR3022 como control positivo (datos no mostrados). Una vez estandarizadas las variables técnicas como están descritas en el apartado de materiales y métodos, se dispuso la estandarización de una dilución del suero muestra para poder disminuir la señal inespecífica y obtener resultados semicuantitativos. Esta dilución de trabajo se estableció como 1:50 para la determinación de IgG y 1:100 para la determinación de IgM.
Cada ELISA se realizó con las mismas variables técnicas establecidas, con la presencia de 6 determinaciones del control positivo y 6 determinaciones del pool de sueros negativos (Figura 2). Todos los sueros fueron probados por triplicado para obtener un promedio de OD con su respectiva desviación estándar. Este valor fue utilizado semicuantitativamente para determinar positividad y, para poder comparar entre ensayos de ELISA, se normalizó el valor de OD de cada voluntario dividiendo el promedio de las lecturas de cada muestra entre el punto de corte de la técnica, correspondiente al límite de cuantificación o cutoff (promedio de OD del control negativo sumado a diez veces su desviación estándar).
Marcadores de desempeño de la prueba para la detección de anticuerpos contra el RBD de la proteína de espícula del SARS-CoV-2
El total de las 129 muestras de suero positivas por SARS-CoV-2 se procesaron mediante el ensayo de detección de anticuerpos IgG anti-RBD de la espícula. Únicamente 43 muestras de suero positivas se incluyeron en el ensayo de detección de anticuerpos de isotipo IgM (por las cantidades de muestra disponibles y por la limitación de no conocer la temporalidad en aparición de síntomas). Por su parte, el panel completo de las 196 muestras de suero prepandémicas fue empleado en la evaluación de ambos ensayos. Los parámetros de desempeño analítico obtenidos para las pruebas de detección de anticuerpos se observan en el Cuadro 2.
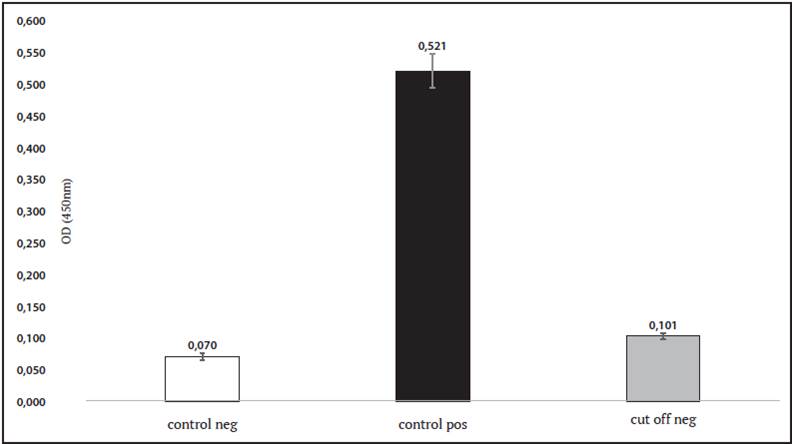
Figura 2 Resultados del ELISA tras la optimización de variables técnicas. El conjunto de sueros negativos se utilizó como control negativo (control negativo) y el anticuerpo CR3022 contra la RBD de SARS-COV-2 se utilizó como control positivo (control positivo). Al promedio de OD obtenido del control negativo, se le sumaron 10 desviaciones estándares para calcular el punto de corte para demostrar positividad. En este ejemplo, todo valor de OD mayor a 0,101 se considera como positivo (N=6). RBD= dominio de unión a receptor. Figura de elaboración propia utilizando la versión 4.1.2 de R (https://www.R-project.org/).
Cuadro 2 Indicadores de desempeño analítico de la prueba para la detección de anticuerpos anti-RBD de proteína de espícula del virus SARS-CoV-2 respecto a las pruebas de detección de ácidos nucleicos como estándar de oro diagnóstico, según isotipo de anticuerpo ensayado
| Detección de IgM | Detección de IgG | |
|---|---|---|
| Sensibilidad (%) | 72.1 | 81.4 |
| Especificidad (%) | 54.1 | 86.2 |
| Valor predictivo positivo (%) | 25.6 | 79.5 |
| Valor predictivo negativo (%) | 89.8 | 87.6 |
Debido a los bajos valores de rendimiento obtenidos para el ensayo de detección de anticuerpos IgM anti-RBD de la espícula en comparación con los anticuerpos de isotipo IgG ensayados, los análisis estadísticos subsiguientes (exceptuando la evaluación de correlación entre seropositividad por virus del dengue y seropositividad por SARS-CoV-2) se realizaron únicamente con los resultados del ensayo de detección de IgG.
Características demográficas y clínicas de las poblaciones de voluntarios incluidas en el estudio
El estudio de características demográficas y clínicas se realizó solamente con una fracción de las 129 muestras de suero positivas de donantes voluntarios (n=46) debido a su carácter no anonimizado (ver Cuadro 1). En dicha subpoblación, un 58.7% correspondía a hombres y un 41.3% a mujeres. Al respecto, se encontró que no existen diferencias significativas en los valores de OD para la serología por IgG entre ambos sexos (W = 242, p = 0.7572). En lo referente a la edad, la subpoblación contemplada presenta una edad promedio de 40 años, con un rango que va de los 5 a los 76 años. El análisis de correlación entre los valores de OD para la serología por IgG y la edad en años obtuvo un rho = 0.2485, indicando que no existe una correlación lineal entre ambas variables.
Al examinar la presencia de sintomatología autorreportada en los voluntarios, se encontró que un 73.9% de los participantes experimentó su infección por SARS-CoV-2 con sintomatología asociada a COVID-19 (principalmente manifestaciones respiratorias y fiebre), mientras que un 26.1% presentó ausencia de síntomas relacionados con el cuadro. Sumado a ello, se detectaron diferencias significativas en los valores de OD para la serología por IgG cuando el donante voluntario reportó tener o no sintomatología (W = 17672, p < 0.0000).
La mayoría de los donantes voluntarios con ausencia de síntomas mostró valores de OD con tendencia cercana a cero para la serología por IgG, mientras que aquellos que experimentaron sintomatología asociada al cuadro clínico tuvieron valores de OD distribuidos con un valor más amplio y mayor, en comparación con los donantes asintomáticos (Figura 3). Adicionalmente, una porción menor, pero notoria, de los voluntarios que reportaron ausencia de sintomatología presentó valores de OD equivalentes hasta cuatro veces en magnitud a los resultados obtenidos por la población asintomática mayoritaria restante.
Para determinar asociaciones entre la respuesta serológica por SARS-CoV-2 y otros patógenos, se procesaron las 36 muestras de otro ensayo de seroprevalencia de dengue tanto para detección de IgM como IgG contra SARS-CoV-2, con el fin de evaluar la posible existencia de correlación o la interferencia inespecífica entre las seropositividades por ambos virus. De la totalidad de las muestras, ninguna resultó positiva en el ensayo de detección de anticuerpos IgG, mientras que un 44.4% de estas (10 de 18 seropositivas por dengue y 6 de 18 seronegativas por dengue) resultaron positivas por anticuerpos IgM anti-RBD de la espícula de SARSCoV-2. El análisis de asociación entre ambas variables indicó que no existe correlación entre la seropositividad por SARS-CoV-2 y la seropositividad por virus del dengue (X2 = 1.8, p = 0.1797), pero que sí hay al menos una reacción cruzada en la detección del isotipo IgM.
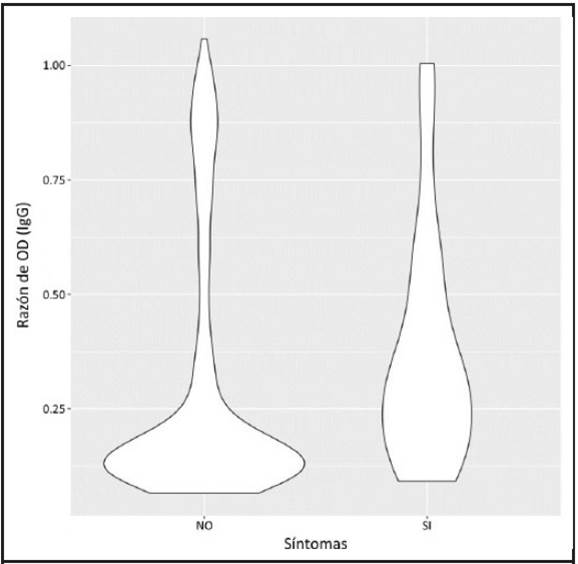
Figura 3 Distribución de los valores normalizados de densidad óptica (OD) en diagrama de violín de muestras de suero positivas evaluadas mediante el ensayo de detección de anticuerpos IgG anti-RBD de proteína de espícula de SARS-CoV-2 según el auto reporte de presencia o ausencia de síntomas asociados al cuadro clínico de COVID-19 manifestado por los donantes voluntarios (n=46). Figura de elaboración propia utilizando la versión 4.1.2 de R (https://www.R-project.org/).
Cinética serológica de voluntarios de la prueba con muestras seriadas durante la duración del estudio
Con la finalidad de evaluar la respuesta del ensayo de detección de anticuerpos IgG ante distintos escenarios de exposición al antígeno viral, se procesaron 250 muestras provenientes de 99 donantes voluntarios que tuvieran múltiples muestras seriadas en el estudio.Los detalles de la cinética serológica mostrada por estos usuarios se observan en la Figura 4.
De los tres estadios inmunológicos presentes en los donantes voluntarios con muestras seriadas, tanto la vacunación contra el SARS-CoV-2 como la infección natural previa por este agente resultaron en incrementos significativos en los valores de OD para la serología por IgG, en comparación con aquellos sin exposición previa al antígeno viral, como era de esperarse. En el caso de los voluntarios no expuestos al antígeno vía vacunación ni por infección, sus valores de OD resultaron bajos a repetición respecto a las condiciones de exposición al antígeno viral (Figura 4). Además, se pudo observar que hay una respuesta heterogénea de anticuerpos IgG dependiendo del donante y del tipo de exposición al antígeno (infección natural y vacunación).
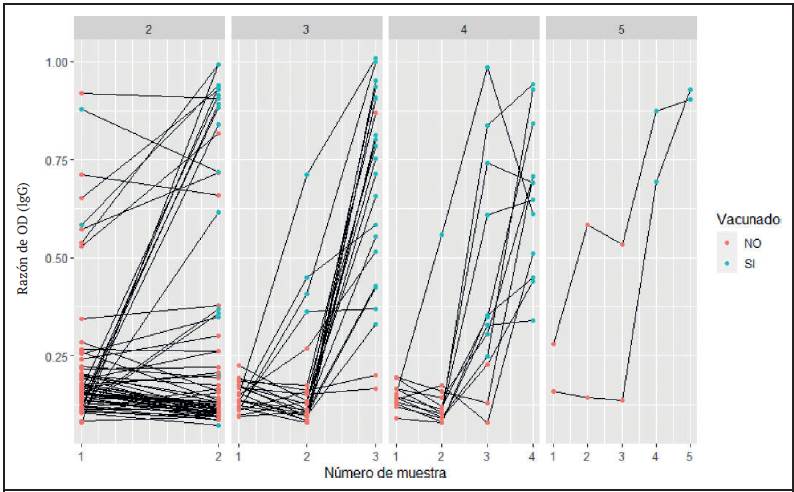
Figura 4 Curvas de cinética serológica de acuerdo con los valores normalizados de densidad óptica (OD) de muestras de suero evaluadas mediante el ensayo de detección de anticuerpos IgG anti-RBD de proteína de espícula de SARS-CoV-2 según número de muestra seriada (2-5 muestras, número en panel superior de la figura) de 99 donantes voluntarios en distintos contextos de seroconversión (infección pasada, vacunación o no exposición al antígeno; n=250). Figura de elaboración propia utilizando la versión 4.1.2 de R (https://www.R-project.org/).
Discusión
El presente estudio expone el protocolo de estandarización de un ensayo serológico tipo ELISA para la detección de anticuerpos IgM e IgG, empleando como sustrato una proteína recombinante del RBD de la espícula del SARS-CoV-2, así como la subsecuente evaluación de su rendimiento y respuesta ante variables demográficas y clínicas de los donantes voluntarios. Los valores obtenidos para los parámetros analíticos del ensayo de detección de IgG denotan un buen valor diagnóstico de la prueba, mientras que el ensayo de detección de IgM demuestra un desempeño pobre, con valores de sensibilidad, especificidad y VPP bajos en comparación a otras pruebas serológicas disponibles en el mercado.9,15,16 Estos valores bajos pueden indicar poca fiabilidad para interpretaciones referentes a la infección por SARS-CoV-2, razón por la cual no se realizaron mayores análisis derivados de los datos obtenidos de la población en estudio mediante este ensayo ni se continuó utilizando. La justificación a este rendimiento puede aludir a la posible presencia de factores que conlleven a la generación de resultados falsos negativos y, sobre todo, falsos positivos, los cuales fueron los más observados en nuestro estudio con los sueros prepandémicos.
Tanto la especificidad como el VPP reflejan el impacto de los falsos positivos en el ensayo, mientras que la sensibilidad y el VPN se ven influenciados por los resultados falsos negativos. Dentro de las causas de falsos negativos, se encuentran el proceso y los períodos normales de seroconversión.
Los anticuerpos IgM son el primer isotipo o clase de inmunoglobulinas generado tras el primer contacto con el antígeno y surgen cerca de las dos semanas después de dicho contacto, permaneciendo aproximadamente de 21 a 28 días en circulación antes de decaer.17 Sucesivamente, se da un cambio de isotipo hacia IgG, los cuales se detectan en el suero hasta 7 meses o más después de la infección.18
Debido a la persistencia tan limitada en el tiempo de los anticuerpos IgM y al hecho de que durante el estudio no se registraron estrictamente las fechas de infección e inicio de los síntomas (además de la naturaleza compleja de la definición de estas), se puede atribuir este proceso como motivo de disminución en la sensibilidad del ensayo. Otro factor reportado como causante de falsos negativos es la ausencia de respuesta serológica, principalmente dada por edades cortas o ante respuestas inmunológicas robustas mediadas por células T.19-21
En el caso de los resultados falsos positivos, múltiples estudios han evaluado sus causas y, entre los hallazgos más relevantes, se tiene el papel del factor reumatoide. Niveles séricos elevados de esta inmunoglobulina (desde 70 hasta más de 1550 UI/mL) han demostrado generar reacción cruzada en los ensayos serológicos de SARSCoV-2.22,23 Adicionalmente, varios autores han encontrado que puede existir cierto grado de reacción cruzada entre anticuerpos contra el dengue y antígenos del SARSCoV-2.24-27 Pese a que nuestros resultados no indican una correlación como tal entre la serología por ambos virus, no se puede omitir que 10 de 18 muestras positivas por dengue mostraron seropositividad por SARS-CoV-2, lo cual puede ser indicativo de reacciones cruzadas, fenómeno que sería recomendable evaluar a profundidad en estudios futuros. Otros agentes reportados en menor frecuencia como responsables de falsos positivos han sido Plasmodium sp. Y otros virus, incluidos coronavirus.9,23,24,27,28 Pese a que los recursos del presente estudio no alcanzan a evaluar la veracidad de estas reacciones cruzadas, tampoco se pueden descartar, dado que no se contemplaron como criterios de exclusión en los participantes. Finalmente, otro factor con potencial contribución a la generación de falsos positivos es el carácter “pegajoso” de ciertas inmunoglobulinas. Se ha reportado que el isotipo IgM de anticuerpos puede generar uniones inespecíficas con distintos sustratos, por lo que la presencia de anticuerpos en el suero de personas no expuestas al patógeno en estudio puede generar una señal de fondo medible en el ensayo sin que necesariamente corresponda a inmunoglobulinas específicas contra el antígeno de interés.29
Entre los hallazgos demográficos relevantes, la ausencia de diferencias entre los valores de OD (proporcionales a los títulos de anticuerpos) según el sexo concuerda con lo reportado en estudios poblacionales.8,30 En cuanto a la edad, la ausencia de correlación lineal entre esta variable y sus valores de OD responde a la variabilidad cualitativa y cuantitativa de la respuesta humoral en los diferentes grupos etarios. Cada estrato de edad tiende a mostrar una respuesta serológica distinta, con diferencias notorias en la respuesta inmune desarrollada dependiendo de si la persona es mayor o menor de 45 años.8,30 Asimismo, se ha observado cierta propensión a una menor seroprevalencia, pero títulos de anticuerpos más elevados en personas de mayor edad que hayan experimentado la infección.8,30,31 Estos hallazgos sugieren que cada grupo etario debería evaluarse de formaaislada para entender la respuesta serológica particular Idealmente, se debería disponer de una cantidad igual de muestras por cada estrato etario, condición con la cual no se contó en este estudio, lo cual limita los resultados.
En lo que respecta a la manifestación clínica, las diferencias existentes entre los valores de OD si la persona presentó o no sintomatología se fundamentan en la proporcionalidad de los títulos de anticuerpos a la gravedad de los síntomas. Está bien descrito que los pacientes asintomáticos o con una clínica más leve muestran títulos de anticuerpos menores que pacientes con manifestaciones más graves.8,19,32 En el estudio, los valores de OD con una tendencia cercana a cero en las personas que experimentaron la infección en ausencia de síntomas pueden responder a títulos de anticuerpos bajos, mientras que los valores de OD distribuidos con un máximo más amplio y elevado (en comparación al escenario anterior) mostrados por la mayoría de las personas que tuvieron síntomas pueden aludirse a una respuesta de anticuerpos más robusta y variable. Para entender esta distribución, es necesario conocer la gravedad clínica de cada individuo, información con la que no se contó en este estudio. Por su parte, la porción de donantes voluntarios sin sintomatología que presentó valores de OD de hasta cuatro veces los valores de la población asintomática restante destaca sobre el comportamiento descrito esperable. Esto es explicado por ciertos autores como un fenómeno inusual pero posible, en donde individuos infectados, pero asintomáticos, levantan títulos de anticuerpos considerables.33 Tales interpretaciones en este estudio deben manejarse con cuidado debido al limitado número de muestras asintomáticas (n=12), por lo que los resultados son susceptibles a verse afectados por valores atípicos.
Basado en la cinética serológica, se puede afirmar que el ensayo responde bien al comportamiento inmunológico esperado ante procesos de seroconversión por exposición antigénica. El fenómeno que la totalidad de las muestras provenientes de contextos de infección previa o vacunación generen valores de OD elevados en comparación con las muestras de donantes sin contacto con el antígeno sucede por la respuesta humoral generada ante el contacto del sistema inmunológico con un componente ajeno (la proteína de espícula). Este proceso desencadena la activación de los linfocitos B y la subsecuente generación de los anticuerpos,34 cuyo título en circulación es proporcional a los valores de OD medidos en los ensayos. Las diferencias de magnitud entre muestras de un mismo estado inmunitario pueden deberse a los principios clínicos anteriormente discutidos; pero, puntualmente, en la población vacunada se ha demostrado que existe cierta variabilidad cuantitativa en la respuesta serológica generada tras la inmunización. Dentro de los factores que determinan dicha respuesta, están las infecciones previas, el número de dosis y el tipo de vacuna.35,36 Por su parte, la subpoblación de voluntarios que reportaron no haber estado en contacto con el virus obtiene valores de OD bajos a repetición respecto a las condiciones donde sí hubo exposición a la proteína de espícula, lo cual es esperable por la ausencia de exposición al antígeno. Este comportamiento representa una virtud del desempeño del ensayo, ya que indica una respuesta constante para muestras distintas en igualdad de condiciones de exposición antigénica.
Dentro de las limitaciones del estudio, se tiene que la inclusión de los participantes no se realizó mediante un muestreo aleatorio, por lo que los análisis demográficos y clínicos son dependientes de un panel no necesariamente representativo de la población. Este mismo aspecto limitó la diversidad de los grupos etarios analizados, motivo por el cual la realización de descripciones más específicas de este parámetro se vio restringida. Asimismo, el muestreo aplicado no fue prospectivo, por lo que las inferencias sobre variables como el tiempo de seroconversión según la fecha de infección o de inicio de los síntomas y la gravedad de los síntomas se limitaron a comparaciones adimensionales.
Este mismo fenómeno lo sufrió el análisis de cinética serológica, ya que no se disponía de lapsos homogéneos entre la recolección de las muestras seriadas, sumado a que la reducida cantidad de muestras por persona limitó el estudio a descripciones observacionales no cuantitativas.
Pese a ello, nuestro estudio representa un avance en la investigación a nivel local sobre la dinámica inmunológica generada ante la exposición al SARS-CoV-2. Se debe destacar que este estudio expone y pone a disposición un diseño de ensayo serológico tipo ELISA capaz de medir la respuesta inmune por IgG contra la RBD de la espícula del virus SARS-CoV-2 de forma apropiada. Además, los resultados aquí expuestos expresan la capacidad de la prueba para evidenciar fenómenos inmunológicos que ameritan mayor profundización, como los son personas asintomáticas que generan una respuesta serológica robusta. Por lo tanto, se aporta información útil para el desarrollo de otras técnicas serológicas a futuro para el estudio de seroprevalencia, respuestas protectoras y evolución respecto a la vacunación en el contexto de la patología de COVID-19.