Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Acta Médica Costarricense
versión On-line ISSN 0001-6002versión impresa ISSN 0001-6012
Acta méd. costarric vol.49 no.2 San José abr. 2007
Infecciones nosocomiales por bacterias productoras de ß lactamasa de espectro ampliado: prevalencia, factores de riesgo y análisis molecular
Carlos Araya-Fonseca1, Ricardo Boza-Cordero2, Laura Arguedas-Soto3, Gloria Badilla-Baltodano4, Fernando García-Santamaría5
Resumen
Justificación y objetivos: La producción de ß-lactamasas es uno de los principales mecanismos de resistencia a antibióticos utilizados por las bacterias Gram negativas. A partir de los años 80 se describe en Escherichia coli y Klebsiella pneumoniae un nuevo tipo de estas enzimas, las ß- lactamasas de spectro ampliado (BLEA) que inactivan todos los antibióticos ß-lactámicos, con excepción de los carbapenémicos. Su diseminación global ha sido muy rápida, lo que ha creado problemas terapéuticos importantes. Este trabajo tiene como objetivos el análisis de la frecuencia de las infecciones producidas por estas bacterias en pacientes internados en el Hospital San Juan de Dios (HSJD), los factores de riesgo para su adquisición y el análisis molecular de las enzimas identificadas.
Metodología: Se revisaron los archivos del laboratorio de bacteriología del HSJD de 2004 y 2005, con el fin de identificar cepas de K. pneumoniae y E. coli productoras de BLEA. La muestra control fue seleccionada al azar entre los cultivos negativos de E. coli y K. pneumoniae por BLEA obtenidos durante mismo período en las bacterias de interés. Solo se incluyeron los reportes positivos por muestra intrahospitalarias. Se analizaron los factores de riesgo para adquirir estas infecciones en pacientes internados entre marzo y julio de 2004. Para la identificación de las bacterias, la sensibilidad a diversos antibióticos, así como la producción de BLEA, se utilizó el equipo automatizado VITEKR BioMérieux. La comprobación de la producción de estas enzimas se efectuó por medio de la prueba de disco empleando cefotaxima, cefotaxima + clavulanato, ceftazidima y ceftazidima + clavulanato.La identificación de las secuencias de los tipos de ß-lactamasas SHV-5, CTX-M y TEM se efectuó por medio de la prueba de reacción en cadena de la polimerasa.
Resultados: En 2004, en el 12% de los aislamientos de E. coli y el 16% de K. pneumoniae se demostró la producción de BLEA. En 2005, el 18% y el 40% de esas bacterias, respectivamente, fueron BLEA+. Se estudiaron 44 pacientes y 47 controles y los factores de riesgo para adquirir infecciones por estos gérmenes fueron la estancia intrahospitalaria (p<0.05); ingresos previos al hospital (p<0.05); presencia de Diabetes Mellitus (p<0.02) o insuficiencia renal crónica (p<0.02); empleo de sondas, catéteres (p<0.001), tubo endotraqueal (p<0.05), el uso de ciprofloxacina (p<0.01), el uso de mezclas de antibióticos (p<0.01). Se demostró alta resistencia a gentamicina (78%), ciprofloxacina (85%), sulfametoxazol-trimetoprim (91%), piperacilina-tazobactam (78%) y cefepime (100%) en las bacterias productoras de BLEA. Los tipos de ß-lactamasa identificados en aislamientos de E. coli fueron TEM un 94%, SHV-55 en un 76%, y CTX-M en un 3% y en K. pneumoniae TEM en el 100%, CTX-M en el 30% y SHV-5 0%. En algunas bacterias se logró demostrar más de un tipo de enzima.
Conclusiones: En el HSJD, la presencia de bacterias productoras de BLEA es un fenómeno que ha ido aumentando en los últimos años. Estas manifiestan baja sensibilidad a gran cantidad de antibióticos, lo que dificulta su manejo. En este estudio, solo el imipenem fue activo en el 100% de los aislamientos. Es llamativo el hecho de que SHV-5, el tipo de enzima más frecuentemente identificada en otras regiones en K. neumoniae, no se detectó en este studio.
Descriptores: ß-lactamasa de espectro ampliado, Klebsiella pneumoniae, Escherichia coli, resistencia a antibióticos.
Key Words: extended spectrum ß-lactamases, Klebsiella pneumoniae, Escherichia coli, antibiotic resistance.
Recibido: 19 de julio de 2006 Aceptado: 8 de febrero de 2007
El amplio uso de antibióticos en humanos y animales, el incremento de la expectativa de vida, el aumento de la sobrevida en los pacientes con enfermedades crónicas gracias al desarrollo de novedosas modalidades terapéuticas como los trasplantes, y la aparición de las unidades de cuidado intensivo, son algunos de los factores que han incidido directamente en la resistencia bacteriana a los diversos antimicrobianos, así como en el aumento de las infecciones nosocomiales.1,2
El incremento de infecciones nosocomiales afecta en especial a los pacientes más severamente enfermos. Se calcula que en los Estados Unidos se producen más de 2 millones de infecciones intrahospitalarias por año, del 50 al 60%, por bacterias resistentes a antibióticos, y 77,000 muertes al año asociadas a este problema, con un costo de aproximadamente 15 mil dólares por paciente infectado y entre los 5 y 10 mil millones de dólares por año.1
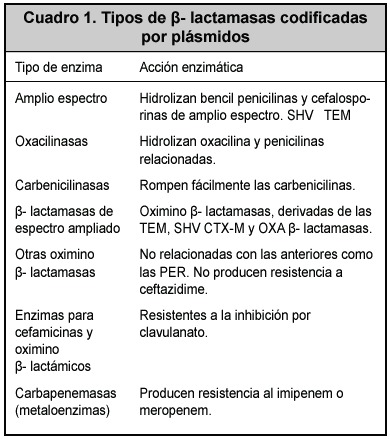
Son varios los mecanismos de resistencia bacteriana a antibióticos, entre los que se encuentra la producción de enzimas inactivadoras de antimicrobianos, y de estas las más ampliamente estudiadas han sido las ß-lactamasas, las que inactivan el anillo ß-lactámico de penicilinas y cefalosporinas a través de la ruptura de la unión amida por medio de un sitio serina activo.3-6 Tanto las bacterias Gram negativas como las positivas pueden expresarlas y su clasificación, de acuerdo con el sustrato sobre el que actúan y su inhibición por el ácido clavulánico, se puede establecer en siete grupos (Cuadro 1).3, 4, 7
Los genes que codifican para la síntesis de estas enzimas se han encontrado en elementos genéticos extracromosómicos móviles, como los plásmidos y transposones, que transportan frecuentemente también genes de resistencia a otros antibióticos como las sulfas y los aminoglucósidos3,6 Ambos elementos requieren unidades de integración dentro del ADN para incorporarse al genoma bacteriano llamados integrones, que son sitios de eventos de recombinación para secuencias no homólogas de ADN y que tienen gran importancia en los mecanismos de diseminación de genes de resistencia a antibióticos.8,9
Algunas enzimas son codificadas por genes que se encuentran en los cromosomas bacterianos, las denominadas Amp-C, que pueden ser inducidas por diversos antibióticos ß-lactámicos y son frecuentes en Enterobacter sp. Estas enzimas son activas contra cefamicinas y oximinocefalosporinas y son resistentes a la acción de clavulanato. Los genes que las codifican no se diseminan usualmente entre especies bacterianas como los encontrados en los elementos móviles descritos en el Cuadro1.3
Con el desarrollo de nuevas penicilinas y cefalosporinas, se observó la aparición casi concomitante de enzimas inactivadoras de estos antibióticos.4 En los años 80 aparecen las cefalosporinas de tercera generación, como un gran avance en el tratamiento de diversas infecciones, principalmente las producidas por bacterias Gram negativas, y en 1983 se describen las primeras ß-lactamasas activas contra las oximinocefalosporinas (ceftazidima, cefotaxima, ceftriaxona) en aislamientos de K. pneumoniae y E. coli.6,7 Se logró demostrar que estas enzimas se derivan de ß- lactamasas ya conocidas y llamadas de espectro amplio, TEM (de Temoniera, nombre del paciente en quien se describió inicialmente) y SHV o sulfihidril variable, ambas con actividad sobre la ampicilina y las cefalosporinas de primera y segunda generación.3,4 La sustitución de uno o varios aminoácidos en estas enzimas dieron origen a las BLEA.3,5 Esta modificación les permitió ampliar su actividad sobre todos los antibióticos ß-lactámicos, con excepción de los carbapenémicos. En la actualidad se han descrito más de 350 tipos de estas enzimas y las más conocidas son las TEM, las SHV, las CTX-M, entre otras, identificadas en la mayoría de las enterobacterias.3, 4, 7
Aunque inicialmente el problema estuvo localizado en las unidades de cuidado intensivo, posteriormente se ha encontrado en diversos sitios.3,10 Su distribución geográfica es mundial, pero no homogénea. En Europa son más frecuentes en la región sur que en la norte, e incluso entre los mismos países los centros de salud identifican diferentes tipos de BLEA.10-12 El problema se ha circunscrito principalmente al medio hospitalario, sin embargo, ya se ha comprobado su presencia en pacientes ambulatorios en diferentes naciones sin que se precisen aún los factores epidemiológicos relacionados.13-15
Schwaber MJ et al16 recientemente publicaron un trabajo en el que demostraron el alto costo clínico de la septicemia asociada a estas infecciones, medido con base en la pobre evolución, incluyendo una gran mortalidad, la alta estancia hospitalaria y el uso inadecuado de antibióticos, así como el costo económico calculado en aproximadamente 10 mil dólares en exceso por paciente. Estos datos, unidos a otras investigaciones,1, 5, 7 evidencian el fuerte impacto que sobre la atención intrahospitalaria tienen las infecciones por estas bacterias.
Han sido identificados diversos factores de riesgo para adquirir estas infecciones en el medio intrahospitalario, tales como el uso de diversos antibióticos, estancia en unidades de cuidado intensivo, estancias hospitalarias prolongadas, uso de catéteres y sondas, y presencia de enfermedades debilitantes.3-5, 17-20
Por otro lado, la actividad de estas enzimas contra antibióticos específicos varía entre países. En Latinoamérica se ha descrito mayor actividad hidrolítica para cefotaxime que para ceftazidime, contrario a lo descrito en Norteamérica,10 lo que probablemente se relaciona con el tipo de enzima prevalente en la región. 11, 12
Además, se ha observado que las cepas productoras de BLEA presentan también con frecuencia resistencia aumentada a otros antibióticos como los aminoglucósidos, las fluoroquinolonas, las tetraciclinas y sulfametoxazol- trimetoprim, ya que genes que transmiten estas características están contenidos en elementos extracromosómicos junto a los genes que codifican para BLEA.3-7
El trabajo tiene como objetivos el análisis de las características epidemiológicas de las infecciones relacionadas con estas bacterias aisladas en pacientes internados en el Hospital San Juan de Dios, en el período comprendido entre marzo y julio de 2004, así como los factores de riesgo y la estructura molecular de las enzimas identificadas.
Materiales métodos
Estudio de casos y controles
Se revisaron los archivos del laboratorio de Bacteriología del HSJD en los años 2004 y 2005, con el fin de determinar el número de aislamientos de K. pneumoniae y E. coli productoras de LEA.
Se estudiaron pacientes internados en el HSJD (hospital clase A, 720 camas), entre el 1 de marzo y el 31 de julio de 2004, con infecciones intrahospitalarias, en quienes se aislaron E. coli y K. pneumoniae de diversos sitios, productoras de BLEA. Se revisaron los expedientes médicos para obtener los datos clínicos respectivos.
La muestra control correspondió a aislamientos de E. coli y K. pneumoniae no productoras de BLEA obtenidos de pacientes con infecciones en sitios, servicios y fechas similares a aquellas asociadas a bacterias productoras de BLEA.
Se compararon, entre pacientes y controles, las siguientes variables: edad, sexo, diagnóstico de ingreso, tiempo de estancia intrahospitalaria, ingreso previo en este internamiento en la Unidad de Cuidados Intensivos, ingresos previos al hospital, clases de antibióticos utilizados y tiempo de uso de estos, sensibilidad a los antibióticos de las bacterias aisladas, uso de catéteres y sondas y enfermedades concomitantes.
Identificación bacteriana:
La identificación bacteriana fue realizada por medio del equipo para análisis bacteriológico respectivo (VITEK® BioMérieux), a través de la tarjeta GNI (código de software utilizado por el equipo, también GN-113 y GNS-120). Una vez realizada la identificación de las bacterias, se efectuó la prueba de sensibilidad a los antibióticos usando la tarjeta GN-113. Por medio del sistema experto computarizado, se notificó la sospecha de la presencia de la ß-lactamasa; la confirmación de la producción de enzimas se realizó con la tarjeta GNS-120.
Identificación de BLEA
Posteriormente, se trasladaron los aislamientos a platos de agar sangre para la confirmación de la presencia de BLEA, al laboratorio de Bacteriología de la Facultad de Microbiología de la Universidad de Costa Rica. Ahí se realizó un cultivo líquido en agar cerebro corazón más un 20% de glicerol para congelación.
La prueba de sensibilidad a antibióticos se ejecutó mediante difusión de disco (OXOID) (21). Se realizó una dilución de las cepas a McFarland 0.5 y se rayó en placas de Petri con 24 ml de agar Mueller-Hinton, y se colocaron 4 discos a 5cm con cefotaxime 30ug, cefotaxime 30ug más ácido clavulánico 10ug, ceftazidime 30ug y ceftazidime 30ug más ácido clavulánico 10ug. Se incubaron a 37 º C por 24 horas y se interpretó el resultado de la prueba como positivo por BLEA si al valuarse el halo de inhibición para el disco de antibiótico más inhibidor resultaba igual o mayor de 5 mm que aquel sin inhibidor. En el caso de que el halo no aumentara, se sospechó de la presencia de Amp C.5,6
Identificación de las secuencias de BLEAs (tipos) en las cepas positivas
Para la identificación de las secuencias SHV-5,TEM, Amp-C y CTX-M, se empleó la técnica descrita.21 Brevemente, se utilizaron 12.5ul de Master Mix R (Fermentas); 0.1 (25UM) de primer Mix y 1ul de ADN 1/10 (21). Las condiciones de PCR fueron las siguientes: un ciclo prePCR o de pre desnaturalización a 94 º C por 5 minutos, 35 ciclos de 30 segundos a 94 º C, 30 segundos a 55 º C, 2 minutos a 72 ºC y un ciclo final de extensión por 15 minutos a 72 ºC. Los primers utilizados fueron para el grupo TEM: OT-1 (5-TTGGGTGCACGAGTGGGTTA-3) y OT-2 (3-TAATTGTTGCCGGGAAGCTA-5'). Para los SHV-5: SHV-A (5-ACTGAATGAGGCGCTTCC-3) y SHV-B (3-CGCACCCCGCTTGCT-5). Para la detección de AmpC (otro tipo de ß-lactamasa que no se afecta por el inhibidor) se utilizaron los siguientes primers: AMPC-F (5 ´-TGGGTTCAGGCCAACATGGATGC-3 ´), AMPC-R (3 ´-TGCCCAACATTACGATGCCAAGG-5 ´).
Para las secuencias CTX-M se utilizaron las mismas cantidades anotadas en el párrafo anterior y con las siguientes condiciones del termociclador: un ciclo inicial de predesnaturalización a 95 º C por tres minutos, 30 ciclos de 30 segundos a 95 º C, 30 segundos a 55 º C, 30 segundos a 72 º C y un ciclo final de extensión por 3 minutos a 72 ºC. Los primers utilizados fueron: CT-F (5-TGGGTTCAGGCCAACATGGATGC-3) y CT-R (3- TTACAAACCGTCGGTGA-5).
Identificación de integrones
Para la identificación de integrones de clase 1 se utilizaron los siguientes primers: 5CS (5-GGCATCCAAGCAAGCAGAAG-3) y 3CS (5- AAGCAGACTTGACTTGACCTGA-33)
Todos los resultados de PCR se revelaron en un gel de agarosa al 0.8% con buffer X TBE conteniendo bromuro de etidio.
Análisis estadístico
Se utilizó la prueba chi cuadrado para comparar los diferentes factores de riesgo entre pacientes y controles y se consideró el valor p<0.05 como significativo.
Resultados
En el período analizado se identificaron 33 aislamientos de E. coli y 11 de K. pneumoniae productores de BLEA. Los sitios de donde se obtuvieron las bacterias fueron orina: (50%), esputo (25%), herida quirúrgica (20%), y otros (5%). Se estudiaron 47 aislamientos de E. coli y K. pneumoniae no productores de BLEA como controles.
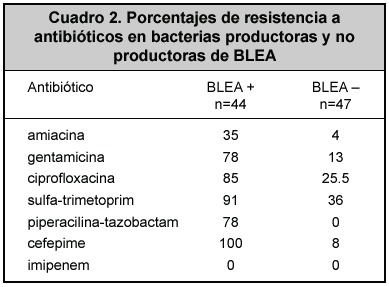
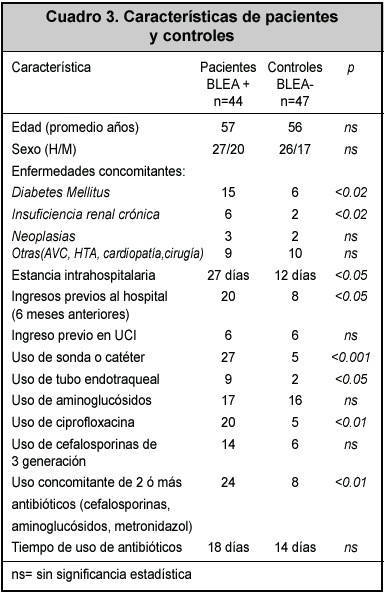
La resistencia a antibióticos de los aislamientos productores y no productores de BLEA se muestra en el Cuadro 2. En el análisis de los factores de riesgo (Cuadro 3) para adquirir bacterias productoras de BLEA, la edad, el sexo, el diagnóstico de ingreso, la estancia previa en la unidad de cuidados intensivos, el tiempo de uso de antibióticos, el empleo de aminoglucósidos, no fueron significativamente diferentes entre pacientes y controles.
Se demostraron como factores de riesgo para adquirir infecciones por estas bacterias: la estancia intrahospitalaria, ingresos previos al hospital, presencia de patologías concomitantes como la diabetes mellitus y la insuficiencia renal crónica, uso de tubos endotraqueales, empleo de ciprofloxacina y de mezclas de antibióticos, el uso de sondas o catéteres (Cuadro 3).
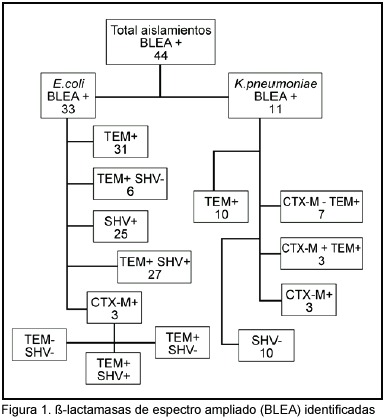
Con respecto al análisis de las ß-lactamasas, el tipo de BLEA identificado según cada bacteria se muestra en la Figura 1. Se demostró en los 44 casos, la presencia de SHV- 5 en 25 aislamientos de E. coli (76%), pero en ninguno de K. pneumoniae; de TEM en 31 aislamientos de E. coli (94%) y en los 11 de K. pneumoniae; para CTX-M en 3 de E. coli (99%) y 3 (30%) K. pneumoniae. En 28 de los casos por E. coli y 10 de los casos por K. pneumoniae se identificó más de un tipo de BLEA.
Durante el análisis de integrones de clase 1, se identificaron dos distintos, uno de 600 a 700 pares de bases y el otro de 1500 a 2000 pares de bases en 6 cepas de K. pneumoniae. En 8 aislamientos E.coli se demostraron tres diferentes tipos de integrones de 600 a 700 pares de bases, 900 a 1000 y de 1500 a 2500 pares de bases cada uno. En total, en 14 cepas (32%) fue posible identificar integrones de clase 1.
Discusión
Entre 1997 y 1999, un 45.4% de K. pneumoniae aisladas en Latinoamérica, un 24.6% en el Pacífico Occidental, un 22.6% en Europa, un 7.6% en Estados Unidos y 4.9% en Canadá fueron productoras de BLEA; asimismo, entre un 4.2% y un 8.4% de las E. coli aisladas en ese mismo tiempo y regiones presentaron esta característica (11). En el San Juan de Dios en 2004, el 12% de los aislamientos de E. coli y el 16% de K. pneumoniae fueron BLEA+, mientras que en 2005, de 1530 aislamientos de E. coli, un 18% fueron BLEA+ y un 40% de 524 aislamientos de K. pneumoniae se les demostró dicha característica, lo que indica la importancia mundial de este problema.
Se han descrito múltiples factores de riesgo para adquirir las bacterias productoras de BLEAs en el medio hospitalario. En el presente estudio se encontró que la estancia intrahospitalaria prolongada, los ingresos previos al hospital, así como el uso de ciprofloxacina y de mezclas de antibióticos diversos, fueron los factores más importantes, ya demostrados en otros estudios.2, 3, 5, 7 Sin embargo, la presencia de enfermedades como la diabetes mellitus y la insuficiencia renal crónica, el uso de catéteres y sondas, se constituyeron también en factores de riesgo. Todos estos factores han sido demostrados en otras investigaciones previas.18-20 El uso previo de cefalosporinas de tercera generación no fue un factor de riesgo, lo que sí se ha observado en otros trabajos. Es posible que el tamaño de la muestra en este estudio influyese para este resultado.
De gran importancia es la resistencia a múltiples antibióticos que se ha encontrado en estas bacterias, lo que se explica por la adquisición de elementos extracromosómicos con genes de resistencia a varios antibióticos, facilitada por la presencia de integrones, demostrados en una tercera parte de los aislamientos en el trabajo. En esta casuística, además de la resistencia a los antibióticos ß-lactámicos, se comprobó una gran resistencia a los aminoglucósidos, sulfametoxasol- trimetoprim, ciprofloxacina y cefepime. Llama la atención el alto porcentaje de resistencia encontrado a este último antibiótico, ya que en diversas publicaciones se ha recomendado su empleo con base en la alta sensibilidad demostrada en otras latitudes a este antibiótico.22-25
La baja sensibilidad observada a piperacilina-tazobactam (22%) es preocupante, dado que en muchas ocasiones es uno de los pocos antibióticos disponibles para emplear en estos casos.
Tal hallazgo podría explicarse no por la presencia de BLEA, sino de otro tipo de ß-lactamasas que inactivan también a los inhibidores, con genes codificadores localizados en cromosomas, pero también en plásmidos, frecuentemente identificados en algunas enterobacterias.6,7 Este fenómeno de identificación de varios tipos de ß-lactamasas en un solo aislamiento bacteriano ha sido notificado antes.7
La multirresistencia plantea retos importantes desde el punto de vista terapéutico, ya que la única opción es el uso de carbapenémicos, con el riesgo de ejercer una presión selectiva y la posibilidad de aparición de resistencia a ellos.22,23
Se ha discutido mucho la relevancia clínica de la presencia de BLEA.24,25 Se ha señalado que para el tratamiento de las infecciones por estos gérmenes debe utilizarse carbapenémicos, basados en la adecuada respuesta clínica y bacteriológica observada en diversos estudios, principalmente en el caso de infecciones severas.3-6,25 La experiencia refiere que, en pacientes con infecciones severas (septicemia, neumonía nosocomial) debe utilizarse carbapenémicos, pero en infecciones leves, como sepsis urinaria, pueden utilizarse cefalosporinas de tercera generación, aminoglucósidos o ciprofloxacina, con un seguimiento clínico-bacteriológico estricto. De cualquier forma, siempre el criterio médico será el que guíe las decisiones.
El análisis de las BLEA identificadas permite concluir que la diseminación de los genes de resistencia es policlonal, no es un brote epidémico de un solo origen, más bien podría tratarse de un fenómeno endémico con múltiples orígenes, lo que se comprueba posteriormente por el aumento en la incidencia entre 2004 y 2005.
Las medidas de control con el fin de evitar la diseminación de estas bacterias en el medio hospitalario, han sido ampliamente descritas2,5 las que incluyen educación al personal médico, pacientes y sus familiares, aislamiento de contacto con lavado adecuado de manos antes y después de examinar al paciente, uso de guantes, cubrebocas y batas al examinar al paciente, colocación de desechos en bolsas separadas, ubicación del paciente en un cuarto aislado o con pacientes con infecciones por bacterias similares, y restricción al uso de cefalosporinas de tercera generación. Es de resaltar que estas mismas medidas han sido implementadas para combatir la resistencia bacteriana a otros antibióticos como la oxacilina y la vancomicina en Staphylococcus aureus y Enterococcus spp.26
Se ha estimado que al menos una tercera parte de las infecciones nosocomiales podrían evitarse a través de estas sencillas acciones.
Debe destacarse lo demostrado en el presente estudio y que ha sido observado desde hace muchos años: las largas estancias hospitalarias (mayor a tres semanas) se constituyen en uno de los principales factores de riesgo para adquirir estas y otras infecciones bacterianas.
Abstract
Aim: Production of ß-lactamases is a very frequent mechanism of resistance to antibiotics in Gram-negative bacteria. Since the 80s, new types of these enzymes, extended spectrum ß-lactamases (ESBL) were described in isolates of Escherichia coli and Klebsiella pneumoniae in Europe and in North America. These bacteria are resistant to all ß-lactam antibiotics, except the carbapenems. Its global dissemination has been fast, creating an important therapeutic challenge. The objectives of this study were to analyze the prevalence of infections with these bacteria in patients admitted to the Hospital San Juan de Dios, a 700 beds tertiary care hospital, the risk factors associated and to perform a molecular analysis of the identified enzymes.
Methods: Records from the Bacteriological Laboratory were reviewed in order to identify E.coli and K.pneumoniae ESBL-producers between March and July 2004
In a case - control study, risks factors to acquire these bacteria in hospitalized patients were analyzed.
Automated equipment (Vitek-bioMerieux ®) was used to identify bacteria, susceptibilities to antibiotics as well as ESBL production. Verification of enzyme production was done by the disc diffusion susceptibility testing using cefotaxime, cefotaxime + clavulanate, ceftazidime and ceftazidime + clavulanate. The sequence analysis of ß- lactamases types SHV-5, CTX-M and TEM was done using the PCR amplification technique.
Results: ESBL were demonstrated in 12% isolates of E.coli and 16% isolates of K.pneumoniae in 2004 and 18% of E.coli and 40% K.pneumoniae in 2005.
Forty four patients and 47 controls were studied during the study period. The risks factors for infections due to ESBL- producing organisms were: long hospitalization stay (p<0.05), prior use of ciprofloxacin (p<0.001), use of various antibiotic mixtures (p<0.01), the presence of a central venous catheter or a urinary catheter (p<0.01), the presence of diabetes mellitus or chronic renal insufficiency (p<0.02). High resistance to gentamicin (78%), ciprofloxacin (85%), trimetoprim-sulfametoxazol (91%), piperacilin-tazobactam (78%) and cefepime (100%) were found in ESBL-producing E. coli and K. pneumoniae. ESBL types demonstrated in E.coli were TEM (94%), SHV-5 (76%) and CTX-M (3%) and in K. pneumoniae TEM(100%), CTX-M(30%). In some isolates, more than 1 ESBL type was shown.
Conclusion: Infections with ESBL-producing E. coli and K. pneumoniae are an increasing problem in our hospital. Risks factors associated with these infections were long hospital stay, antimicrobial use, the presence of chronic diseases and the use of in-dwelling catheters. High resistance patterns to some antimicrobials were found in these organisms also previously reported in other series. ESBL types identifies were similar to observed in other countries, but the lack of SHV-5 in K. pneumoniae is a remarkable finding. We analyze these findings and discuss their clinical and epidemiologic implications.
Referencias
1. Roberts RR, Scott RD, Cordell , Solomon SL, Steele L Kampe LM, et al The use of economic modeling to determine the hospital costs associated with nosocomial infections Clin Infect Dis 2003; 336: 1424- 1432 [ Links ]
2. Owens RC, Rice L. Hospital-based strategies for combating resistance Clin Infect Dis 2006; 42: S173-181 [ Links ]
3. Jacoby GA, Muñoz-Price LS. The new ß-Lactamases N Engl J Med 2005; 352: 380-391
4. Paterson DL, Bonomo RA. Extended-Spectrum ß-Lactamases: a Clinical Update Clin Microbiol Rev 2005; 118: 657-686
5. Stürenburg E, Mack D. Extended-spectrum ß-Lactamases: implications for the clinical microbiology laboratory, therapy, and infection control. J Infect 2003; 47: 273-295
6. Bradford PA. Extended-Spectrum ß-Lactamases in the 21st Century: Characterization, Epidemiology, and Detection of This Important Resistance Threat. Clin Microbiol Rev 2001; 114: 933-951
7. Bush K. New ß-Lactamases in gram-negative bacteria: diversity and impact on the selection of antimicrobial therapy Clin Infect Dis 2001; 332: 1085-1089 [ Links ]
8. Fluit AC, Schmitz FJ. Resistance integrons and super-integrons Clin Microbiol Infect Dis 2004; 10: 272-288 [ Links ]
9. Leverstein van Hall MA, Blok HEM, Donders RT, Paauw A, Fluit AC, Verhoef J. Multidrug resistance among enterobacteriaceae Is strongly associated with the presence of integrons and Is independent of species or isolated origen. J Infect Dis 2003; 187: 251-259 [ Links ]
10. Paterson DL Ko WC, von Gottberg A, Mohapatra S, Casellas JM, Goossens H, et al. International prospective study of Klebsiella pneumoniae bacteremia: Implications of extended-spectrum ß- Lactamase Production in nosocomial infections. Ann Intern Med 2004; 140: 26-332
11. Winokur PL, Canton R, Casellas JM, Legakis N. Variations in the prevalence of strains expressing an extended-spectrum ß-Lactamase phenotype and characterization of isolates from Europe, the Americas, and the western Pacific Region. Clin Infect Dis 2001; 32(Suppl 2): S94-103
12. Silva J, Gatica R, Aguilar C, Becerra Z, Garza-Ramos U, Velásquez M, et al. Outbreak of infection with Extended-Spectrum ß-Lactamase-Producing Klebsiella pneumoniae in a Mexican Hospital. J Clin Microbiol 2001; 39: 3193-3196 [ Links ]
13.Rodríguez-Baño J, Paterson D. A change in the epidemiology of infections due to Extended-Spectrum ß-Lactamases-Producing organisms. Clin Infect Dis 2006; 42: 935-937
14. Ben-Ami R, Schwaber MJ, Navon-Venezia S, Schwartz D, Giladi M, Chmelnitski I, et al. Influx of Extended-Spectrum ß-Lactamase- producing enterobacteriaceae into the hospitals. Clin Infect Dis 2006; 42: 925-934
15. Arpin C, Dubois V, Maugein J, Jullin J, Dutilh B, Brochet JP, et al. Clinical and molecular analysis of extended-spectrum ß- Lactamase-producing enterobacteria in the community setting. J Clin Microbiol 2005; 43: 5048-5054
16. Schwaber MJ, Navon-Venezia S, Kaye KS, Ben-Ami R, Schwartz D, Carmeli Y. Clinical and economic impact of bacteremia with extended-spectrum-ß-Lactamase-producing Enterobacteriacea. Antimicrob Agents Chemother 2006; 50: 1257-1262
17.Schiappa DA, Hayden MK, Matushek MG, Hashemi FN, Sullivan J, Smith KY, et al. Ceftazidime-resistant Klebsiella pneumoniae and Escherichia coli bloodstream infections: a case-control and molecular epidemiologic investigation. J Infect Dis 1996; 174: 529-536 [ Links ]
18. Graffunder EM, Preston KE, Evans AM, Venezia R. Risk factors associated with extended-spectrum ß-lactamase-producing organisms at a tertiary care hospital J Antimicrob Chemother 2005; 56: 139- 145
19. Lautenbach E, Patel JB, Bilker WB, Edelstein PH, Fishman. NO Extended-Spectrum ß-Lactamase-Producing Escherichia coli and Klebsiella pneumoniae: Risk factors for infection and impact of resistance on outcomes Clin Infect Dis 2001; 32: 1162-1171
20. Bermejo J, Lesnaberes P, Arnesi N, Gianello M, Notario R, Borda N, et al. Factores de riesgo asociados a infecciones por Klebsiella pneumoniae resistentes a ceftacidima. Enferm Infecc Microbiol Clin 2003; 21: 72-76 [ Links ]
21. Steward CD, Rasheed JK, Hubert SK, Biddle JW, Raney PM, Anderson GJ, et al. Characterization of Clinical Isolates of Klebsiella pneumoniae from 19 laboratories using the national committee for clinical laboratory standards extended-spectrum ß-Lactamase Detection Methods lin Microbiol 2001; 39: 2864-2872
22. Paterson DL, Ku WC, von Guttberg A, Mohapatra S, Casellas JM, Goossens H, et al. Antibiotic therapy for Klebsiella pneumoniae bacteremia: implications of production of extended-spectrum ß- lactamases Clin Infect Dis 2003; 39: 31-37
23. Wong-Beringer A, Hindler J, Loeloff M, Queenan AM, Lee N Pegues DA, et al. Molecular correlation for the treatment outcomes in bloodstream Infections caused by Escherichia coli and Klebsiella pneumoniae with reduced susceptibility to ceftazidime. Clin Infect Dis 2002; 34: 135-146 [ Links ]
24. Branger C, Zamfir O, Geoffrey S, Laurans G, Arlet G, Vu Thien H, et al. Genetic background of Escherichia coli and extended-spectrum ß-lactamase type. Emerg Infect Dis 2005; 11: 54-61
25. Ramphal R. Ambrose PG. Extended-spectrum ß-Lactamases and clinical utcomes: current data. Clin Infect Dis 2006; 42: S164-172
26. Salas-Vargas A, Boza-Cordero R, Bustamante-García W, García-Santamaría F, Barrantes-Valverde E. Prevalencia e identificación genotípica de enterococos vancomicina-resistentes en pacientes en un medio hospitalario. Acta Méd Costar 2003; 46: 19-26 [ Links ]
Sección de Medicina1 y Servicio de Infectología, HSJD2, Centro de Investigación en enfermedades tropicales, Universidad de Costa Rica3,5, Laboratorio de Bacteriología, SJD4
Abreviaturas: BLEA, ß -lactamasa de espectro ampliado; ESBL, extended spectrum ß -lactamase; HSJD, Hospital San Juan de Dios; PCR, polymerase chaín reaction o reacción en cadena de la polimerasa.
Correspondencia:
Carlos Araya-Fonseca carayaf20cr@yahoo.com.mx