Introducción
Los cangrejos Mitrácidos pertenecen a la superfamilia Majoidea Samouelle, 1819, en donde se incluyen las especies comúnmente conocidas como cangrejos araña (Assugeni et al., 2017), presentan gran riqueza de especies, diversidad morfológica y ecológica (Ng, Guinot, y Davie, 2008). La familia Mithracidae se encuentra representada por 86 especies y 14 géneros; de los cuales aproximadamente 35 especies se encuentran en el Golfo de México y en la región occidental del Atlántico (De Grave et al., 2009; Klompmaker, Portell, Klier, Prueter y Tucker, 2015; Ng, Ho, Lin y Yang, 2018; Windsor y Felder, 2014), 27 especies en el Pacífico oriental y 30 especies en el Pacífico Indo-occidental (Windsor y Felder, 2014). Su distribución abarca zonas tropicales y subtropicales, desde zonas intermareales hasta los 450 m de profundidad, formando parte de las comunidades de arrecifes coralinos y también de escombros rocosos (Klompmaker et al., 2015). En Costa Rica, se ha reportado la presencia de cangrejos mitrácidos en zonas rocosas y arrecifes poco profundos (Alvarado y Vargas-Castillo, 2012; Vargas-Castillo y Vargas-Zamora, 2020).
El tamaño del cuerpo, los estilos de vida, hábitat y coloración varían de forma considerable entre las especies de esta familia, incluso dentro de una misma especie es posible observar variaciones morfológicas importante, por lo cual la identificación taxonómica de los representantes de este grupo es compleja (Cruz y Campos, 2003). Lo anterior ha llevado al uso de herramientas moleculares basadas en el código de barras de ADN, para resolver las relaciones filogenéticas que se mantenían inconclusas a partir de estudios morfológicos y ecológicos (Assugeni et al., 2017; Bucklin, Lindeque, Rodríguez-Ezpeleta, Albaina y Lehtiniemi, 2016; Bucklin, Steinke y Blanco-Bercial, 2011; Geiger et al., 2016; Toon, Finley, Staples, y Crandall, 2009; Wagner, 1990; Windsor y Felder, 2009).
Recientemente, la familia Mithracidae ha sido ampliamente analizada a partir de caracteres morfológicos e identificación molecular, con el propósito de la reubicación de géneros y establecimiento de nuevas relaciones filogenéticas (Windsor y Felder, 2009, 2014, 2017). Windsor y Felder (2014) reorganizaron algunos géneros de la familia Mithracidae tales como Mithrax Latreille (1816); Mithraculus (White, 1847);. ( Nemausa A Milne-Edwards, 1875); Nonala (Windsor & Felder, 2014); Omalacantha (Streets, 1871) y Petramithrax (Windsor & Felder, 2014). Posteriormente, Klompmaker et al. (2015) añadieron el género Maguimithrax, anteriormente renombrado por Windsor y Felder (2014) como Damithrax. Finalmente, Windsor y Felder (2017) homologaron el género Mithrax con el nombre de Amphithrax, reasignando algunas especies de cangrejos araña de la familia Mithracidae (WORMS, 2020).
La asignación de la identidad taxonómica y el conocimiento de las áreas geográficas en que habitan las especies de cangrejos mitrácidos, mediante predicciones filogenéticas y mapas de distribución espacio-temporal, facilitan los estudios de biodiversidad, la generación de reportes sobre alteración del hábitat por actividades antropogénicas, planes de conservación y medidas de protección de los ecosistemas (Moscoso, 2012; Vargas, 2012; Wehrtmann y Cortés, 2009). Por lo que este estudio busca revelar la riqueza de especies y la distribución espacial-temporal de la familia Mithracidae en las costas del Caribe y Pacífico costarricenses, así como identificar molecularmente, por primera vez en Costa Rica, especies de cangrejos araña de esta familia.
Metodología
Área de estudio
Se obtuvieron muestras de tejido a partir de colecciones del Museo de Zoología de la Universidad de Costa Rica (MZUCR) (cuadro suplementario 1). Además, se seleccionaron puntos de muestreo basados en reportes previos de colectas y observaciones de campo en zonas rocosas intermareales y zonas arrecifales del Caribe costarricense, específicamente en Puerto Viejo, Punta Uva, Manzanillo, Cahuita e Isla Uvita y en la costa pacífica: el Golfo de Nicoya, Isla del Caño, Dominical, Esterillos, Caldera, Isla Negritos, Playa Buenavista, Playa Tambor, Cuajiniquil, Coyote y La Cruz (Moran y Dittel, 1993; Vargas, 2012; Wagner, 1990). Basados en estas localidades, se colectaron individuos de cada especie en ambas costas. Los especímenes se fotografiaron y los tejidos se conservaron en etanol al 70 % y 4˚C, para sus posteriores análisis moleculares.
Mapas de distribución espacial y temporal de cangrejos Mitrácidos
Se realizaron mapas de distribución, a partir de los muestreos exploratorios realizados en la costa Pacífica y en el Caribe de Costa Rica, así como de información obtenida de registros de campo suministrados por el Museo de Zoología de la UCR. Además, se realizaron mapas de registros temporales de colectas en un periodo de 53 años (1963-2016). Para la creación de los mapas se emplearon un total de 11 géneros, 20 especies, y se utilizó el programa estadístico R (R versión 3.4.0, 2017) con el paquete de ggplot2 v. 2.2.1 (Wickham, 2016).
Extracción de ADN y amplificación de genes específicos por PCR
La extracción de ADN total se realizó mediante el kit comercial PureLink® Genomic DNA Mini Kit (Invitrogen, USA) a partir de 30 mg de tejido muscular abdominal y de la coxa del cuarto pereiópodo de cada cangrejo analizado. La amplificación de secuencias mediante reacción en cadena de la polimerasa (PCR) fue dirigida a regiones específicas del gen nuclear: Histona 3 (H3), y tres genes mitocondriales: ADN ribosomal 16S (ADNr 16S), ADN ribosomal 12S, y citocromo oxidasa I (COI). Las reacciones de PCR se optimizaron a un volumen final de 25 μl: 2,5 μl de 10X Taq buffer, 2,5 μl de MgCl2 (25 mM), 0,5 μl de dNTPs (10 mM), 1,875 μl de cada cebador (10 mM), 0,25μl Taq (5U), 2 μl del ADN (100 ng) y 13,5 μl agua ultrapura doble desionizada. Las condiciones de termociclado para los genes fueron: desnaturalización inicial 94°C por 5 min, seguido por 36 ciclos de 94°C por 1 min, 50°C (COI y 12S) o 52°C (16S y H3) por 1 min y 72°C por 1 min, seguido por una extensión final a 72°C por 5 min. Los cebadores utilizados fueron: Histona 3 (H3Af: 5’-ATG GCT CGT ACC AAG CAG ACV GC-3’y H3Ar: 5’-ATA TCC TTR GGC ATR ATR GTG AC-3’) (Colgan et al., 1998), ADNr 16S (16L2: 5’-TGC CTG TTT ATC AAA AAC AT3’ y 1472: 5’AGA TAG AAA CCA ACC TGG3’) (Baeza et al., 2010), ADN 12S (12Sf: 5’-GAA ACC AGG ATT AGA TAC CC3’ y 12S1r: 5’-AGC GAC GGG CGA TAT GTA C-3’) (Mokady, Rozenblatt, Graur y Loya, 1994) y COI (HCO2198: 5’-TAA ACT TCA GGG TGA CCA AAR AAY CA-3’y LCO1490: 5’-GGT CAA CAA ATC ATA AAG AYA TYG G-3’) (Folmer, Black, Hoeh, Lutz y Vrijenhoek, 1994). La confirmación de los productos PCR se realizó mediante movilidad electroforética a 90 V, 400 mA por 45 min, en geles de agarosa al 2 % en buffer TAE 1X. Los amplicones fueron purificados, para posterior secuenciación, mediante el kit de aislamiento y purificación de productos retenidos en geles de agarosa Wizard® SV Gel and PCR Clean-Up System (PROMEGA, USA), según las indicaciones de manufactura.
Secuenciación genética de los productos de PCR y análisis bioinformáticos
La secuenciación de los productos de PCR se realizó en ambas direcciones para cada fragmento con una química Big Dye Terminator Mix v3.1, utilizando el analizador genético ABI 3130 (Applied Biosystems), del Laboratorio de Análisis Genómico (LAGEN) de la Escuela de Ciencias Biológicas, Universidad Nacional. Las secuencias obtenidas fueron editadas y alineadas utilizando el software Geneious® versión R9 (Biomatters Ltda) y sometidas a un análisis de alineamiento local (BLASTn) del NCBI (http://www.ncbi.nlm.nih.gov/blast) para confirmar su identidad. Posteriormente, las secuencias nucleotídicas fueron accesadas en el GenBank con los siguientes números de accesión: KX033404-KX033407 para COI y KU951436-KU951440 para el gen 16S ribosomal, y previamente para los genes Histona 3 (H3) y ADN ribosomal 12S. Para determinar el posicionamiento taxonómico de los especímenes (DNA barcoding) se realizó un análisis filogenético multilocus, concatenando las secuencias de este estudio y posicionándolas con las obtenidas desde Genbank (https://www.ncbi.nlm. nih.gov/genbank) (Cuadro suplementario 2). Inicialmente, se elaboraron matrices de secuencias para cada gen (COI, 12S, 16S y H3) y se alinearon con el programa MAFFT v7.308 (Katoh y Standley, 2013) con el método de refinamiento iterativo (G-INS-i) y el parámetro 1 PAM/k=2. Seguidamente, se realizó una selección de bloques conservados mediante parámetros menos estrictos empleando el programa Gblocks (Castresana, 2000). El mejor modelo de sustitución nucleotídica se determinó mediante el programa ModelFinder (Kalyaanamoorthy, Minh, Wong, von Haeseler, y Jermiin, 2017) lo que arrojó que el mejor modelo fue el TVM+F+G4 con asignación de frecuencia de bases empírica, modelo GAMMA discreto de cuatro categorías.
La elaboración de inferencias de posicionamiento taxonómico se realizó mediante la plataforma en línea IQ-TREE (http:// iqtree.cibiv.univie.ac.at) (Nguyen, Schmidt, von Haeseler y Minh, 2015; Trifinopoulos, Nguyen, von Haeseler y Minh, 2016) con el método de máxima verosimilitud (ML) empleando un algoritmo estocástico. Los parámetros utilizados para la inferencia filogenética fueron el uso de heterogeneidad de tasa libre, cálculo de soporte de los nodos derivado del método bootstrap UFboot de 5000 permutaciones (Minh, Nguyen y von Haeseler, 2013), con un máximo de 5 000 iteraciones y una prueba de rama única (SH-aLRT) con 5 000 réplicas. Se utilizó a Micippa thalia como grupo externo. Los árboles obtenidos se visualizaron y editaron con el programa FigTree v1.4 (Rambaut, 2010). Finalmente, se garantizó que no existieran topologías individuales en conflicto entre los árboles obtenidos y se procedió a concatenar las matrices alineadas de los genes COI, 12S, 16S y H3 con 658, 351, 419 y 328 posiciones informativas, respectivamente, mediante el programa SequenceMatrix v1.7.8 (Vaidya, Lohman y Meier, 2011) con el fin de obtener una topología multigénica consenso mediante el método ML y el modelo TVM+F+G4.
Análisis y resultados
Los mapas de ubicación geográfica generados a partir de los registros de muestreo de los especímenes de la familia Mithracidae permitieron determinar la distribución y riqueza espacio-temporal de las especies de cangrejos araña en la costa Caribe y Pacífico de Costa Rica, revelando la presencia de las siguientes 24 especies: Ala cornutaStimpson, 1860; Maguimithrax spinosissimusLamarck, 1818(syn. Mithrax spinosissimus); Hemus finneganae Garth, 1958; Microphrys branchialis Rathbun, 1898; Microphrys platysomaStimpson, 1860; Mithraculus cinctimanusStimpson, 1860; Mithraculus corypheHerbst, 1801; Mithraculus denticulatusBell, 1836; Mithraculus fórceps A.Milne-Edwards, 1875; Mithraculus sculptus Lamarck, 1818; Amphithrax aculeatusHerbst, 1790(syn. Mithrax aculeatus); Amphithrax tuberculatusStimpson, 1860(syn. Mithrax tuberculatus); Nemausa sinensisRathbun, 1892(syn. Mithrax sinensis); Omalacantha bicornuta Latreille, 1825 (syn. Microphrys bicornutus); Omalacantha interruptaRathbun, 1920(syn. Microphrys interruptus); Petramithrax pygmaeus Bell, 1836 (syn. Mithrax pygmaeus); Teleophrys cristulipesStimpson, 1860; Teleophrys tumidus Cano, 1889; Thoe puellaStimpson, 1860; Thoe erosa Bell, 1836; Pitho lherminieriDesbonne in Desbonne & Schramm, 1867; Pitho pictetiDe Saussure, 1853 ; Pitho quinquedentata Bell, 1836 y Pitho sexdentata Bell, 1836 (Cuadro suplementario 1).
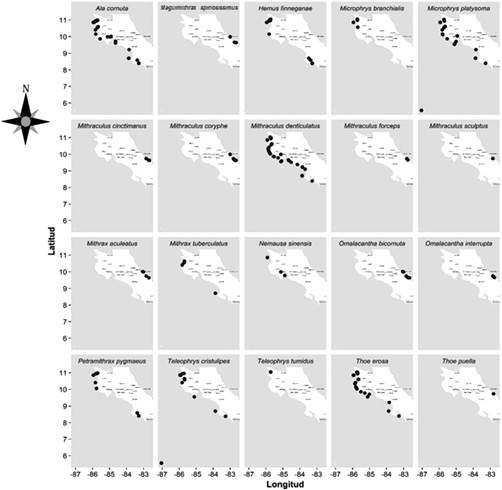
Nota: Fuente propia de la investigación (2016).
Figura 1 Mapas de distribución por especie de cangrejos mitrácidos de la región noreste del Caribe y el suroeste del Pacífico costarricense periodo (1963-2016).
Se muestra una distribución individual de los reportes de cada una de las especies de la familia Mithracidae, en la región Caribe o Pacífico, incluidas islas continentales (Isla Uvita, Isla del Caño, Islas Murciélago, Isla Juanilla, Isla David, Isla Bolaños, Islas Huevos) y oceánicas (Isla del Coco) (Figura 1). Las localidades que presentaron la mayor cantidad de especies reportadas en este estudio, según la región son: Caribe, provincia de Limón (Puerto Viejo de Talamanca, Isla Uvita, Manzanillo, Punta Uva, Cahuita, Puerto Vargas, Portete), Pacífico Norte, provincia de Guanacaste (islas Murciélago, Cuajiniquil, Golfo Santa Elena, Camaronal, Santa Cruz, Samara, Nandayure, La Cruz, Bahía Culebra, Bahía Junquillal, Bahía Huevos, Carrillo) y Pacífico Central, provincia de Puntarenas (Golfo de Nicoya, Esterillos, Herradura, Jacó, Manuel Antonio, Tambor, Agujas e Isla del Coco), Pacífico Sur, Provincia de Puntarenas (Isla del Caño, Golfo Dulce, Punta Dominical, Bahía Ballena) (Cuadro suplementario 1).
En la distribución temporal, se observa una mayor incidencia de registros u observaciones de este grupo de cangrejos a partir de 1990 y hasta la actualidad, lo cual puede deberse a un aumento en el esfuerzo de muestreo, producto de las investigaciones referentes a la diversidad y riqueza de crustáceos marinos en diferentes zonas del país, y a través de muestreos exhaustivos en las costas del Pacífico y el Caribe costarricenses (Moran y Dittel, 1993; Vargas, 2012; Wehrtmann y Cortés, 2009) (Figura 2). Se observaron especies cuya aparición en el tiempo es baja, tales como T. puella, T. tumidus, O. interrupta, A. aculeatus (syn. Mithrax aculeatus) y M. sculptus, lo cual puede deberse a una baja prevalencia en el país a lo largo de los años, además de las dificultades asociadas a su localización en el campo como consecuencia de su camuflaje con los corales y las algas de los sustrahábitats de pastos marinos y arrecifes de coral en localidades evaluadas en la península Yucatán, México y la bahía de Nenguange, Caribe y Colombia (González-Gómez, Briones-Fourzán, Álvarez-Filip y Lozano-Álvarez, 2018; Gutiérrez-Salcedo, Aguilar-Pérez, Bermúdez, Campos y Navas, 2007), y en el Atlántico Occidental, abarcando la región de Florida (EE. UU), las Antillas y Brasil; en São Paulo (Mantelatto et al., 2020) y Ilha Vitória (Alves et al., 2012). Por su parte, T. puella se reporta como abundante en las costas poco profundas del litoral de Santiago de Cuba asociado a rocas coralinas y arrecife de coral (García y Capote, 2015). En contraposición, existen múltiples registros locales de especies como M. denticulatus, M. coryphe, M. platysoma, A. cornuta y T. erosa, lo cual podría ser un indicador de la abundancia de estas especies o de la cantidad de territoritos coralinos que dificulta su observación. Sin embargo, está descrito que la abundancia de M. sculptus y M. coryphe es alta en y hábitats en los que se pueden localizar. Mientras que la aparición contemporánea de algunas especies como M. cinctimanus, M. sculptus, M. branchialis y P. pygmaeus puede deberse a la direccionalidad del muestreo hacia la búsqueda de estas especies en específico (Figura 2).
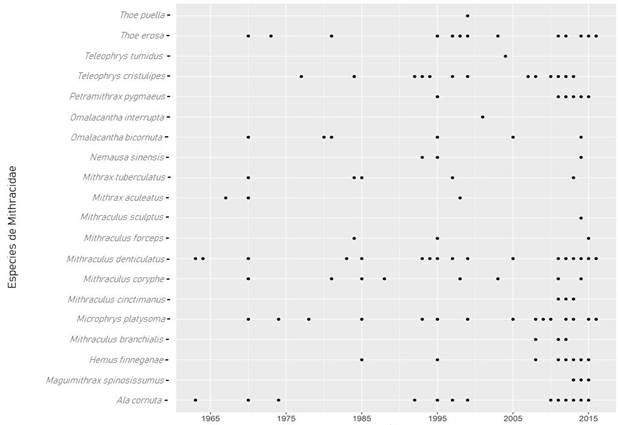
Nota: Fuente propia de la investigación.
Figura 2 Registro de distribución temporal de mitrácidos en Costa Rica, según los registros de campo obtenidos por medio de colectas de especies de cangrejos araña en un periodo de 53 años (1963-2016).
Los mapas de distribución espacio-temporal constituyen una importante herramienta para realizar estudios de monitoreo local sobre la prevalencia y estado de las poblaciones, la elaboración de metodologías de muestreo y la planificación de expediciones en campo dirigidas en la búsqueda de especímenes. Así, por ejemplo, T. cristulipes y M. platysoma se encuentran, además de en la zona continental, en la Isla del Coco a 500 km del continente. Mientras que M. spinosissimus, especie de consumo local y de explotación comercial a pequeña escala, abarca los principales puntos de pesca y turismo de la región Caribe como Cahuita, Puerto Viejo, Punta Uva, Cocles y Manzanillo, lo cual la hace vulnerable a la sobreexplotación. Igualmente, especies como M. cinctimanus y M. coryphe se localizan en el Caribe y son utilizados en el mercado de la acuariofilia (Rhyne, Fujita y Calado, 2004), por lo que son potencialmente susceptibles a su reducción poblacional.
La monofilia de Mithracidae ha sido ampliamente discutida, algunos reportes plantean esta condición dentro del grupo (Hultgren y Stachowicz, 2008; Hultgren et al., 2009), mientras que otros se inclinan hacia la ausencia de un ancestro común (Martin y Davis, 2006). Asimismo, las clasificaciones internas son aún más discutidas y menos estables, requiriendo de investigaciones enfocadas a esclarecer las relaciones taxonómicas de este grupo (Hultgren y Stachowicz, 2008). Nuestros hallazgos permitieron agrupar 9 géneros y 16 especies de la familia Mithracidae, mediante topología concatenada multigénica de secuencias nucleotídicas obtenidas en este estudio; regiones mitocondriales 12S (27 secuencias), 16S (21) y COI (33) y la región nuclear H3 (17), así como secuencias reportadas en el GenBank (Cuadro suplementario 1), lo que revela géneros monofiléticos como Thoe (Bell, 1836), Hemus (A. Milne-Edwards, 1875) y Mithraculus (Windsor y Felder, 2014). El género Maguimithrax se posiciona taxonómicamente cercano al género Ala y se agrupa dentro de un mismo nodo, formando un clado con alto soporte según el análisis de máxima verosimilitud. El género Omalacantha se agrupa en un mismo clado con el género Microphrys y Omalacantha
+ Microphrys es un clado hermano al género Teleophrys. Microphrys y Omalacantha son muy similares desde el punto de vista morfológico (Colavite et al., 2020; Hollier, 2020; Carmona-Suárez y Poupin, 2016; Windsor y Felder, 2017). Por otra parte, el género Mithraculus, representado por cuatro especies en nuestro árbol filogenético (M. cinctimanus, M. forceps, M. coryphe y M. denticulatus) representa un clado monofilético bien soportado. Este género no está cercanamente relacionado a los géneros Maguimithrax y Petramithrax (Figura 3). Por otra parte, los géneros Petramithrax, Hemus y Thoe se agrupan en clados independientes, con valores altos de bootstrap.
Topología inferida mediante máxima verosimilitud (ML), modelo TVM+F+G4 con 5000 permutaciones. Las secuencias obtenidas en este estudio se identifican como: (Costa Rica). Los valores de soporte de cada rama se muestran como SHaLRT (Test de rama única) / UFBoot (boostrap ultra rápido), en el cual la confianza en un clado que se interpreta como >80 y >90, respectivamente. Valores de soporte de las ramas menores al
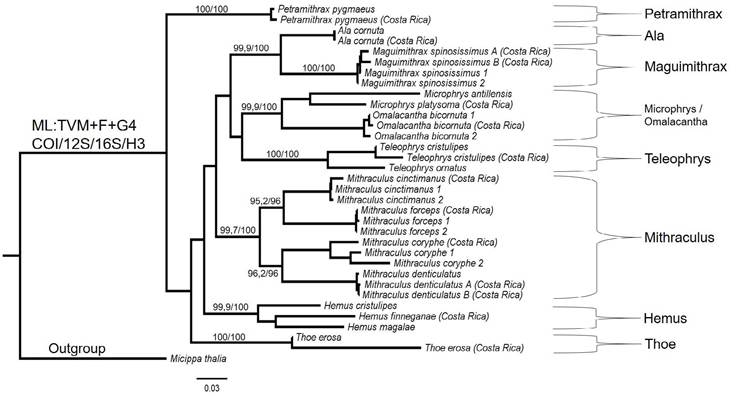
Figura 3 Árbol de posicionamiento taxonómico de cangrejos araña (Familia Mithracidae) de Costa Rica, basado en secuencias parciales de una matriz génica concatenada (COI, 12S, 16S y H3).
80 % no se muestran en la topología. Micippa thalia (Herbst, 1803) corresponde al outgroup. La barra de escala corresponde a 1 sustitución nucleotídica por cada 30 sitios. Los números de accesión de las secuencias obtenidas del GenBank se muestran en el Cuadro suplementario 2, generado con datos de esta propia investigación (2020).
Baeza et al. (2010) analizaron las relaciones filogenéticas entre especies de los géneros Mithrax y Mithraculus mediante el marcador molecular gen 16S (ADNmt), reportando que el género Mithraculus posee 4 especies representativas monofiléticas (M. cinctimanus, M. coryphe, M. sculptus y M. forceps). Posteriormente, Windsor y Felder (2014) reportaron una filogenia con un mayor número de secuencias genéticas, así como el análisis de la estructura del espermatozoa, e incorporan los géneros Hemus y Pitho dentro de la familia Mithracidae, siendo Hemus un clado hermano de los géneros Thoe y Petramithrax, con lo cual se sugiere la validez de los géneros Omalacantha, Nonala y Petramithrax. Además, la especie M. spinosissimus (Lamarck, 1818) fue trasladada al género Maguimithrax (Klompmaker et al. 2015). El género Mithrax quedaría compuesto por tres especies: Mithrax hispidus (Herbst, 1790), Mithrax pleuracanthus (Stimpson, 1871), Mithrax tortugae (Rathbun, 1920). Además, incluyeron 10 especies a un nuevo género llamado Amphithrax; el cual contiene: Amphithrax aculeatus (Herbst, 1790), Amphithrax armatus (De Saussure, 1853), Amphithrax bellii (Gerstaecker, 1857), Amphithrax besnardi (Schimdt de Melo, 1990), Amphithrax braziliensis (Rathbun, 1892), Amphithrax caboverdianus (Türkay, 1986), Amphithrax clarionensis (Garth, 1940), Amphithrax hemphilli (Rathbun, 1892), Amphithrax leucomelas (Desbonne, 1867), Amphithrax tuberculatus (Stimpson, 1860).
Windsor y Felder (2014) concluyen que Petramithrax es genéticamente distinto de Mithrax, y lo agrupan en su topología filogenética con Hemus y Thoe, aun siendo morfológicamente distinto de ambos; pues lo consideran, entonces, como un taxón único dentro de Mithracidae y con su posición aún no resuelta dentro de la familia. Los resultados arrojados en nuestro estudio ubican a Petramithrax separado de los demás cangrejos araña analizados, lo cual se justifica por su morfología única. Sin embargo, a pesar de que estos géneros no parecen compartir caracteres morfológicos que evidencien parentesco, pueden existir similitudes ecológicas. Windsor y Felder (2017) mencionan que a nivel morfológico existe una relación entre Hemus y Thoe, dada por la presencia de una amplia antena basal que forma el piso orbital y el desarrollo de una cerradura dactilar en los apéndices ambulatorios presentes en cangrejos Mithracidae. En nuestro caso, contrario a lo establecido por Windsor y Felder (2017) y Assugeni et al. (2017) mediante herramientas moleculares, el género Hemus se encuentra localizado en un clado independiente separado filogenéticamente de Petramithrax y Thoe. Por otra parte, nuestros resultados muestran que algunos géneros se resolvieron con alto soporte filogenético, como es el caso de Petramithrax, Ala, Maguimithrax, Microphyrs, Omalacantha, Teleophrys y Thoe.
Por otro lado, el género Ala se ubica en cercanía filogenética con Maguimithrax, como potenciales grupos hermanos. Estos resultados concuerdan con los obtenidos por Windsor y Felder (2017) y Assugeni et al. (2017) en los cuales se describe a Ala y Nonala en posiciones cercanas entre sí, y a su vez agrupados con Maguimithrax y Nemausa como grupos hermanos. La posición de M. spinosissimus separado de Mithraculus es igualmente concordante con los resultados obtenidos por ambos autores, en los cuales se posiciona a estas especies separadas de Mithraculus, contrario a lo reportado por Baeza et al. (2010), quienes se basan en un único locus genético. El género Microphrys, por su parte, se posiciona cercanamente de Omalacantha, lo cual puede deberse a la homología que han presentado estos géneros (Hollier, 2020; Windsor y Felder, 2017), y sus similitudes morfológicas (Carmona-Suárez y Poupin, 2016; Colavite et al., 2020). Además, nuestros resultados revelan diferencias en las distancias genéticas entre especies del Caribe y del Pacífico; siendo mayor entre O. antillensis (Caribe, Costa Rica) y M. platysoma (Pacífico, Costa Rica). De la misma forma, Windsor y Felder (2017) encontraron que la especie del Pacífico Microphrys branchialis se encuentra lejana filogenéticamente de los taxones del Atlántico. Basados en caracteres morfológicos, los mismos autores revelan diferencias menores, pero consistentes entre el clado Atlántico; comprendido en su estudio por O. antillensis, O. bicornuta y especies distribuidas en el Pacífico Oriental, lo que los lleva a postular una restricción provisional de especies del Pacífico al género Microphrys s.s. y remoción de especies de distribución atlántica al género Omalacantha (Streets, 1871).
La posición taxonómica de Teleophrys dentro de Mithracidae ha quedado en muchas ocasiones sin resolver. Estudios moleculares con bajo soporte de los nodos ubican este género como un grupo hermano de Hemus y Thoe, fundamentado por caracteres morfológicos y similitudes ecológicas (Windsor y Felder, 2014). Por otra parte, aspectos únicos en la morfología de Teleophrys distinguen al género de otros cangrejos mitrácidos (Windsor y Felder, 2017), lo que es consistente con nuestros datos moleculares que ubican a Teleophrys distante de Hemus y Thoe, pero relacionado genéticamente a Omalacantha (Assugeni et al., 2017). De manera concluyente, estudios de taxonomía molecular posicionan a Thoe como grupo hermano de Petramithrax, formando ambos un clado cercanamente relacionado con Hemus (Assugeni et al., 2017; Windsor y Felder, 2014), lo cual difiere con nuestros hallazgos. Las similitudes morfológicas y ecológicas entre Thoe y Hemus sugieren una mayor relación y cercanía entre ambos géneros (Števcic, 1994; Windsor y Felder, 2014). La información reportada en este estudio podría ser utilizada como línea de base para futuras identificaciones taxonómicas de crustáceos decápodos mediante herramientas moleculares (i.e., código de barras molecular). Además, es la antesala de potenciales estudios de filogenética, estructura genética de poblaciones, filogeografía, genética de la conservación y ampliación de rangos de distribución de especies de cangrejos araña (Baeza et al., 2019; Mantelatto et al., 2020).
Conclusiones
Este es el primer reporte de distribución y riqueza de cangrejos mitrácidos en las costas Caribe y Pacífico de Costa Rica. Estos hallazgos representan una importante contribución en cuanto a la actualización de reportes de especímenes de cangrejos de la familia Mithracidae y su localidad de observación o captura en Costa Rica. Este estudio respalda la metodología de una herramienta molecular de inferencia taxonómica (DNA barcoding) para cangrejos araña de la familia Mithracidae en Costa Rica, basado en aproximaciones filogenéticas multigénicas bien soportadas, empleando las regiones mitocondriales (12S, 16S y COI) y nucleares (H3). Además, nuestros hallazgos confirman las relaciones filogenéticas previamente reportadas tales como las cercanías entre los clados Ala-Maguimithrax, Omalacantha-Microphyrs, Omalacantha-Teleophrys y el clado bien soportado de las especies representativas del género Mithraculus. En contraste, la proximidad genética entre Thoe-Petramithrax-Hemus no se obtuvo en nuestros resultados. La información molecular obtenida, por primera vez, de poblaciones de cangrejos Mithracidae en Costa Rica, visualiza perspectivas a futuro en cuanto a estudios de genética de poblaciones, filogeografía, flujo génico regional y divergencia entre especies Caribe y Pacífico (reloj molecular). Mediante este reporte de distribución espacio-temporal y riqueza de poblaciones de cangrejos araña, es posible identificar localidades susceptibles a la disminución de la diversidad de especies y desequilibrio ecológico, producto de procesos como la destrucción del hábitat y la sobreexplotación de los recursos; información que resulta relevante para los entes tomadores de decisiones a nivel país en cuanto al establecimiento de zonas protegidas potencialmente aptas en la dinámica poblacional de individuos de la familia Mithracidae.
Cuadros suplementarios
Los cuadros suplementarios están disponibles en sitio del artículo: https://dx.doi.org/10.15359/ru.36-1.18
Agradecimiento
Agradecemos a Óscar Ramírez-Alan (QEPD) por su aporte técnico en la elaboración de gráficos de este estudio. A Karolina Ramos y Daniela González por la colaboración en los ensayos moleculares. A Sergio Cambronero, Allan Artavia y Antonio Cambronero por el apoyo en los muestreos exploratorios de cangrejos araña en Costa Rica. Al Museo de Zoología de la Universidad de Costa Rica (UCR), por facilitar el acceso a las colecciones de crustáceos Majidos. J. Antonio Baeza agradece el soporte económico de la Universidad Nacional de Costa Rica (UNA) que permitió pasantías al Laboratorio de Análisis Genómico (LAGEN) así como el desarrollo de este manuscrito.
Declaración de la contribución de los autores
Todos los autores afirmamos que se leyó y aprobó la versión final de este artículo. El porcentaje total de contribución para la conceptualización, preparación y corrección de este artículo fue el siguiente: C.S.B. 20 %, L.E.S.A. 15%, J.A.M. 15%, R.V.C. 15%, J.A.B. 15 %, R.U.C. 20%.