Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Agronomía Mesoamericana
On-line version ISSN 2215-3608Print version ISSN 1659-1321
Agron. Mesoam vol.25 n.2 San Pedro Jul./Dec. 2014
Análisis y Comentarios
Burkholderia glumae en el cultivo de arroz en Costa Rica
Burkholderia glumae in the rice crop in Costa Rica
Burkholderia glumae in the rice crop in Costa Rica
*Dirección para correspondencia:
Resumen
El objetivo de este trabajo fue determinar la presencia de Burkholderia glumae en arroz en Costa Rica. La bacteria Burkholderia glumae está asociada al cultivo del arroz en el que provoca la enfermedad llamada añublo bacterial. Bajo condiciones ambientales favorables, la densidad bacteriana aumenta, lo que provoca que, bajo un sistema de regulación denominado quorum sensing, se expresen sus mecanismos de virulencia mediante la activación de genes responsables para la síntesis de la toxoflavina, que bloquea el flujo de nutrientes, para la biogénesis de flagelos y la respuesta quimiotáctica, y la producción de la enzima catalasa. Las plantas desarrollan la sintomatología que finalmente conlleva a un vaneamiento del grano provocando pérdidas económicas importantes. Se investigó la situación referente a la contaminación del grano de arroz causado por esta bacteria en Costa Rica durante los años 2009 y 2010, mediante un convenio entre la Corporación Nacional Arrocera y el Laboratorio de Fitopatología del Centro de Investigación en Protección de Cultivos de la Universidad de Costa Rica. Se usó la metodología de PCR de punto final recomendada por investigadores del Centro Internacional de Agricultura Tropical en Colombia y se reforzó la identificación, por medio de técnicas de microbiología convencional. Se obtuvieron resultados que indican la presencia de la bacteria en Costa Rica, la primera información sobre la prevalencia de un fitopatógeno bacteriano de gran importancia para el sector arrocero.
Palabras clave: añublo bacterial, vaneamiento en arroz, panícula de arroz.
Abstract
The objective of this study was to determine the presence of B. glumae in the rice crop in Costa Rica. The bacterium Burkholderia glumae is associated with the cultivation of rice, causing the disease known as bacterial blight. Under favorable weather conditions, the bacterial density increases, thus causing the expression, under a system called quorum sensing regulation, of its virulence mechanisms, by activating genes responsible for toxoflavine synthesis, which blocks the flow of nutrients for flagellum biogenesis and the chemostatic response, and the production of the enzyme catalase. Plants develop symptoms that eventually lead to empty hulls, causing significant economic losses. The current level of contamination of rice seeds with this bacteria in Costa Rica was studied during 2009 and 2010, through an agreement between the National Rice Corporation, the Plant Pathology Laboratory of the Crop Protection Research Center at the University of Costa Rica. The Endpoint PCR methodology recommended by researchers at the International Center for Tropical Agriculture in Colombia, the identification was reinforced with conventional microbiology. Results indicating the presence of the bacteria in Costa Rica were obtained, providing the first information on the prevalence of a bacterial plant pathogen of great importance for the rice sector.
Keywords: bacterial blight, sterility, rice panicle.
Introducción
El arroz es, después del trigo, el segundo cultivo en importancia económica y nutricional en la alimentación de los seres humanos. Según datos de Coorporación Nacional Arrocera (CONARROZ, 2012) en Costa Rica, en el periodo 2011-2012 se obtuvo una producción mayor a 260 000 toneladas métricas del grano, además en el mismo periodo se estimó un consumo per-cápita de 53,71 kg y un promedio mensual de 20 658 toneladas, bastante superior al registrado en los períodos anteriores. Este cultivo genera numerosos empleos desde el campo hasta la agroindustria en diferentes regiones del territorio nacional, siendo la Región Chorotega la principal con el 35% del área total sembrada, en segundo lugar se ubica la Región Huetar Norte con 25%, que para el periodo 2011-2012 desplazó a la Región Brunca que tradicionalmente había ocupado ese lugar. Para este período su participación pasó a 23%, ocupando el tercer lugar en importancia, seguida por el Pacífico Central con 11% y la región Huetar Atlántica con 3% (CONARROZ, 2012).
Su cultivo intensivo es afectado por numerosos factores bióticos, destacándose particularmente los agentes infecciosos, incluyendo virus, bacterias y hongos, así como plagas por diversas especies de artrópodos. Burkholderia glumae es una bacteria Gram-negativa responsable de la enfermedad conocida como añublo bacterial que afecta la panícula del arroz, provocando grandes pérdidas económicas anualmente en los países productores del grano (Prado, comunicación personal, 2011), por lo que este problema infeccioso amerita un abordaje integral para evitar un impacto en la productividad nacional. En Costa Rica no existen datos de las pérdidas económicas atribuidas específica y directamente a las infecciones por B. glumae. El objetivo de este trabajo fue determinar la presencia de Burkholderia glumae en arroz en Costa Rica y generalidades de esta bacteria.
Metodología
Se realizó una revisión bibliográfica sobre la bacteria B. glumae, incluyendo generalidades, fisiología, genética, modos de transmisión, patología, así como de su impacto en la producción de arroz a nivel nacional y mundial. Por otra parte, se recopiló la información generada por el Laboratorio de Fitopatología (CIPROC-UCR) en el año 2009 y el año 2010, en el que se analizaron 242 muestras usando la metodología recomendada por el CIAT, usando los imprimadores diseñados por Takeuchi et al. (1997) específicos de las regiones espaciadoras 16S-23S de ADN ribosomal, con las secuencias GL-13f 5’-ACA CGG AAC ACC TGG GAT-3’ y GL-14r 5’-TCG CTC TCC CGA AGA GAT-3’. Durante el año 2009 se analizaron un total de 113 muestras de diferentes variedades, entre ellas, Puitá, Palmar 18 y SEN 250 entre otras, todas provenientes de las región Chorotega y en el año 2010 se analizaron 129 muestras provenientes de las regiones Chorotega, Brunca y Pacífico Central, incluyendo, entre otras, las variedades Palmar 18, CFX 18, CR 5272, CR 4477.
Generalidades de B. glumae
El género Burkholderia fue establecido en 1992 (Yabuuchi et al., 1992) y está conformado por especies previamente incluidas en el género Pseudomonas. Para el año 2009 habían sido reportadas 59 especies (Coenye, 2009), las cuales se caracterizan por ser bacterias Gram-negativas, pertenecientes al phylum β-Proteobacteria, tener forma de bacilo, un metabolismo oxidativo y ser móviles (Schaad et al., 2001). Comprende patógenos de plantas, especies asociadas a humanos y otros mamíferos; por ejemplo, B. mallei es de importancia a nivel veterinario, causante de una infección típica en equinos, es rara en humanos pero puede ser transmitida provocando infección pulmonar aguda. B. pseudomallei es el agente causal de meloidosis en equinos y la enfermedad fue descrita como una septicemia que compromete pulmones, hígado y riñones (Mahon et al., 2007).
A nivel agronómico son importantes las especies B. cepacia asociada a cebolla, B. andropogonis que infecta entre otros el sorgo y el maíz, B. caryophylli que se encuentra en clavel y B. gladioli que se encuentra asociado a gladiola y arroz, causando pudrición suave y necrosis severa en tejidos (Viallard et al., 1998).
Burkholderia glumae (Pseudomonas glumae Kurita & Tabei 1967, Burkholderia glumae Urakami et al., 1994. comb. nov) se asocia al suelo, a la rizosfera y a la superficie de diversas plantas donde se considera epífita, sin provocar daño al hospedero, pero constituyendo un importante reservorio que puede dar origen a patologías bajo ciertas condiciones en numerosos cultivos (Goto y Ohata, 1956; Kurita y Tabei, 1967; Urakami et al., 1994; Compant et al., 2008). En el arroz esta bacteria causa la enfermedad conocida como añublo bacterial de la panícula del arroz, que es de gran impacto económico en diferentes partes del mundo, provocando pérdidas de hasta un 40% de la producción (Iwai et al., 2002; Rush et al., 2003). Además, esta especie es causante del marchitamiento en muchos cultivos de campo como tomate, chile, berenjena, papa, girasol (Uematsy et al., 1976).
Uno de los mayores avances en la comprensión de las características biológicas de B. glumae se ha logrado mediante la secuenciación completa de su genoma a partir de la cepa B. glumae BGR1, aislada de una panícula de arroz enferma. Esta consiste en dos cromosomas y cuatro plásmidos. El primer cromosoma contiene 3 906 529 pares de bases (G+C=68,11%) con 3290 secuencias codificantes, 144 pseudogenes, 3 operones de ARNr y 56 de ARNt (Lim et al., 2009).
El cromosoma 2 contiene 2 827 355 pares de bases (G+C=68,76%) con 2079 secuencias codificantes, 192 pseudogenes, dos operones de ARNr y ocho de ARNt. Este es de particular interés, debido a que su origen de replicación posee una estructura muy similar a la observada en plásmidos, lo que sugiere que se originó a través de la transferencia horizontal de un plásmido, que eventualmente adquirió genes codificantes para ARNr y ARNt para convertirse funcionalmente en un cromosoma. Además, en el cromosoma 2 se ubican los genes asociados a su capacidad para causar daño en plantas, lo que a su vez sugiere que la fitopatogenicidad de esta bacteria no es una característica inherente, sino adquirida (Lim et al., 2009).
Factores de virulencia
Se reportan varios factores de virulencia en B. glumae, entre ellos, la catalasa, la biogénesis flagelar, un sistema de secreción tipo III y una fitotoxina llamada toxoflavina (Ham et al., 2011); sin embargo, el principal mediador de daño es la toxoflavina, una toxina con propiedades físicas y químicas similares a la riboflavina y que se evidencia en las cepas productoras de la toxina por la pigmentación amarilla que produce en los medios de cultivo (Nandakumar et al., 2009).
La toxoflavina actúa como un transportador de electrones entre NADH y el oxígeno sin la intermediación de los citocromos, generando peróxido de hidrógeno (Latuasan y Berends, 1961). Este es altamente tóxico para diversos tipos de células, provocando daño tisular y teniendo además, un efecto antimicrobiano. Este mecanismo de acción explica la ausencia de toxicidad de la toxoflavina bajo condiciones anaeróbicas (Suzuki et al., 2004).
La síntesis de toxoflavina depende de la presencia de dos operones (conjuntos de genes cuya transcripción ocurre coordinadamente) ligados genéticamente y ubicados en el cromosoma 2 de la bacteria. La ausencia de ambos operones es responsable de la ocurrencia de cepas avirulentas de B. glumae (Cottyn et al., 2001). El primer operón está constituido por cinco genes (toxABCDE) y es necesario para la biosíntesis de la toxoflavina, mientras que el segundo operón contiene cuatro genes (toxFGHI) que son requeridos para el transporte de la toxoflavina hacia el espacio extracelular. Adicionalmente se requieren de dos genes, denominados toxJ y toxR, los cuales codifican para proteínas que regulan la expresión genética de los dos operones antes mencionados (Shingu y Yoneyama, 2004).
La producción de la toxina depende de dos factores. Por un lado, la biosíntesis de la toxoflavina en B. glumae depende de la temperatura y ocurre entre 30 °C y 37 °C (Matsuda y Sato, 1988). A temperaturas inferiores a ese rango puede suceder la multiplicación bacteriana pero sin darse la síntesis de la toxina. Interesantemente, la temperatura óptima para la producción de la toxina por parte de B. glumae coincide con las temperaturas que el Instituto Meteorológico Nacional ha reportado para la provincia de Guanacaste, donde la temperatura máxima promedio es de 32,4 °C (Guzmán, 2006). Por otro lado, la síntesis de la toxoflavina depende de la densidad de la población bacteriana y es regulada mediante un mecanismo conocido como quorum sensing (Kim et al., 2004).
Este fenómeno implica la síntesis de moléculas de señalización intercelular que, en el caso de bacterias Gram-negativas como B. glumae, son N-acil-homoserina-lactonas. A mayor densidad bacteriana, mayor es la concentración de las moléculas de señalización, las cuales controlan, mediante mecanismos de transducción celular, la expresión de diversos genes. En consecuencia, las bacterias coordinan la expresión genética de manera que se desarrolle un comportamiento social. En el caso de B. glumae, la síntesis y el transporte de toxoflavina, así como la síntesis de catalasa, la biogénesis de flagelos y la formación de biopelículas, dependen de la densidad bacteriana involucrando un mecanismo de quorum sensing. De esta forma, cuando B. glumae se encuentra sobre las plantas en densidades bajas no causa daño al tejido vegetal. Cuando la población bacteriana tiene una alta densidad, superior a 1 x 108 bacterias por mililitro, se inducen los factores de patogenicidad y el grado de daño en la planta es alto (Fuqua y Greenberg, 2002).
Las bacterias aerobias tienen la capacidad de sobrevivir en presencia de oxígeno molecular gracias a un conjunto de enzimas que las protegen de especies reactivas de oxígeno, como peróxido y superóxido, incluyendo superóxido dismutasa, alquil-hidroperóxido reductasa y catalasa (Imlay, 2003). Las catalasas son enzimas antioxidantes que catalizan la degradación de peróxido de hidrógeno en agua y oxígeno. Además, las catalasas juegan un papel sumamente importante en la virulencia de muchas bacterias patógenas, por cuanto protegen a las bacterias en los tejidos del hospedero de las especies reactivas de oxígeno que se generan durante la respuesta inmune. B. glumae posee la enzima catalasa KatG que resulta esencial para su sobrevivencia en ambientes aerobios y expuestos a la luz solar. Además, la expresión de KatG es regulada por el fenómeno de quorum sensing y se requiere para la protección de la bacteria del daño oxidativo generado por la toxoflavina. Mutantes de B. glumae deficientes de KatG tienen una menor capacidad de causar daño en ensayos de patogenicidad en arroz (Chun et al., 2009).
El desplazamiento de bacterias en fluidos o superficies y la capacidad de coordinar la motilidad como respuesta a estímulos quimiotácticos resultan esenciales para su sobrevivencia en diferentes ambientes. B. glumae posee dos flagelos polares, lo que le permite realizar motilidad (swimming) en fluidos y un desplazamiento colectivo (swarming) sobre superficies. Los flagelos son estructuras muy complejas y su biogénesis representa un gasto energético importante para el metabolismo bacteriano, por lo que la expresión de los flagelos y los patrones de motilidad están fuertemente controlados. Al igual que la síntesis de la toxoflavina y la enzima catalasa, la biogénesis de flagelos depende del fenómeno de quorum sensing anteriormente descrito. Interesantemente, mutantes de B. glumae incapaces de sintetizar flagelos o de responder a estímulos quimiotácticos no producen patología en arroz. Presumiblemente, la motilidad en B. glumae podría ser necesaria para iniciar el proceso infeccioso en el arroz (Kim et al., 2007).
Las bacterias poseen diversos sistemas de secreción que les permite exportar proteínas desde su citoplasma hacia el exterior de la célula bacteriana. Los sistemas de secreción tipo III son utilizados por bacterias Gram-negativas patógenas para traslocar proteínas directamente al interior de células eucariotas (Galan y Wolf-Watz, 2006). En el caso de bacterias fitopatógenas, los sistemas de secreción tipo III son necesarios para causar daño en plantas susceptibles y provocar respuestas de hipersensibilidad en las hojas de plantas no hospederas o en plantas resistentes (Alfano y Collmer, 2004). Se ha reportado que B. glumae posee un sistema de secreción tipo III que puede mediar tanto daño en el arroz así como también respuesta de hipersensibilidad. Sin embargo, aún no se determinado claramente cuáles son los factores proteicos producidos y luego traslocados por el sistema de secreción tipo III en B. glumae (Kang et al., 2008).
Transmisión
Burkholderia glumae es una bacteria que se transmite principalmente mediante semilla infectada, y es por medio de esta que se disemina a diferentes regiones, debido a la exportación e importación de semilla (Sayler et al., 2006).
B. glumae invade las semillas germinadas, inhabilita las raíces y las vainas inferiores y de esta manera empieza a crecer sobre la planta como un organismo epífito. La sintomatología se presenta durante la floración, cuando la bacteria se multiplica activamente al invadir las espigas de la planta a través de los estomas o de pequeñas heridas en la epidermis de las glumas (Sayler et al., 2006; Zhou et al., 2011).
Posteriormente, coloniza las espigas y se multiplica rápidamente utilizando los azúcares que son necesarios para el desarrollo del grano (Hikichi et al., 1994). De hecho, la bacteria es más virulenta en panícula que en plántulas (Nandakumar et al., 2009), debido probablemente a la mayor densidad bacteriana.
Hay evidencia además que sugiere que esta enfermedad es transmitida por semilla, desde plantas infectadas en el año previo a la siembra (Weinberg et al., 2007), la bacteria se localiza en la parte basal del lodículo y al interior de la lema del grano de arroz (Tsushima, 2011), pudiendo sobrevivir dentro de la semilla hasta por tres años (Tsushima et al., 1989).
La dispersión de la bacteria se puede dar por salpicadura, también es arrastrada por el viento durante la lluvia y además por contacto entre panículas, resultando de todo lo anterior un foco de infección, posteriormente, las altas temperaturas en combinación con alta humedad y lluvias frecuentes favorecen el desarrollo de un brote (Zhou et al., 2011).
Aunque se sugirió que B. glumae era un componente más del complejo ácaro Steneotarsonemus spinki - hongo Sarocladium oryzae - bacteria, en Luisiana EE.UU en el año 2005 después de un monitoreo en todas las zonas productoras de arroz no se encontró el ácaro, pero el cultivo sí presentaba la enfermedad añublo bacterial de la panícula (Correa-Victoria, 2006). En otros casos se ha aislado la bacteria de campos que presentan síntomas, pero la presencia del ácaro tampoco se reporta.
Posteriormente, se realizó un monitoreo poblacional del ácaro tampoco en diferentes variedades en Colombia y su relación con la bacteria y los rendimientos del 2008 al 2010 y los resultados obtenidos indicaron que no se encontró correlación entre la presencia del artrópodo y la incidencia del añublo bacterial (Pérez y Saavedra, 2011).
Patología
El daño causado por B. glumae en las plantas depende de su capacidad para multiplicarse abundantemente, lo cual, a su vez, depende de las condiciones ambientales y la disponibilidad de nutrientes. Así, la enfermedad se desarrolla solamente en condiciones de alta temperatura, especialmente por la noche, aunado a alta humedad relativa y con precipitaciones frecuentes. La bacteria se puede multiplicar sobre la superficie de las panículas emergentes y sobre las flores justamente después de abrirse. Además, la bacteria puede ingresar a la planta a través de los estomas y multiplicarse en los espacios intercelulares del parénquima. Como se indicó anteriormente, la multiplicación bacteriana en altas densidades genera la producción de la toxoflavina, la cual, mediante generación de peróxido de hidrógeno, provoca daño tisular y una obstrucción de los haces vasculares en las plantas de arroz, impidiendo la llegada de monosacáridos y disacáridos esenciales para la formación del almidón en la panícula (Mahenthiralingam et al., 2005).
La obstrucción de las haces vasculares reduce el crecimiento del follaje y tejido radical en las plántulas e induce síntomas de clorosis en las panículas en la fase de pudrición del grano, siendo responsable de causar marchitez bacteriana (Sato et al., 1989; Suzuki et al., 2004; Shingu y Yoneyama, 2004).
Sintomatología
Esta enfermedad se caracteriza en el arroz por la presencia de panículas café o decoloradas debido a un proceso de clorosis. Las ramas y panículas se mantienen verdes al inicio, sin lesiones ni presencia de decoloración, y las flores afectadas detienen el crecimiento o son abortadas por la planta. Las panículas afectadas pueden tener pocas o todas las flores enfermas (Yang, 2004). Los granos pueden mostrar diferentes grados de decoloración dependiendo de la intensidad de la infección (Zhou et al., 2011) (Figura 1).
Cuando la enfermedad es severa, las plantas permanecen erguidas y la panícula se mantiene vertical, debido a la ausencia de ganancia de peso en los grados (Yang, 2004; Sayler y Yang, 2006). Así, el estado más crítico de la enfermedad se presenta en el momento que emerge la panícula y el efecto más claro es la esterilidad de los granos y su decoloración (Correa y Victoria, 2006).
B. gladioli es una bacteria que comparte muchas características fenotípicas con B. glumae, tales como la temperatura de crecimiento (Nandakumar et al., 2009), el número de flagelos, producción de toxoflavina y la producción de síntomas similares en las plantas de arroz, debido a lo anterior, B. gladioli se considera un agente causal del añublo bacterial que provoca que los granos de arroz tomen tonalidades verdes y se encojan tornándose luego amarillo oscuro y finalmente café antes de secarse (Ura et al., 2006).
Técnicas de diagnóstico
El diagnóstico microbiológico para encontrar bacterias viables en los tejidos, se realiza usualmente mediante técnicas de cultivo; posteriormente, se realizan los aislamientos bacterianos, los cuales deben ser inicialmente caracterizados por tinción de Gram y por pruebas bioquímicas convencionales como oxidasa y catalasa (B. glumae bacilo Gram-negativo oxidasa-variable, catalasa-positivo) (Shaad et al., 2001).
B. glumae crece en medios de cultivos convencionales como King B (20 g de peptona, 1,5 g KH2PO4, 1,5 g MgSO4 7 H2O, 15 ml glicerol) y el medio selectivo S-PG (1,3 g KH2PO4, 1,2 g Na2HPO4, 5,0 g (NH4)2SO4, 0,25 g MgSO4. 7 H2O, 24,0 mg Na2MoO4.2H2O, 10,0 mg EDTA-Fe, 10,0 g Sorbitol, 1,0 mg metil violeta, 20,0 mg rojo de fenol), formando colonias circulares y convexas de color amarillo en el primero y con centro púrpura-rojizo en el segundo, variando un poco la morfología colonial según la cepa.
Se han utilizado perfiles bioquímicos (capacidad de crecer en presencia de diferentes tipos de sustratos) para realizar la identificación fenotípica de la bacteria. La técnica de Biolog es considerada como una herramienta muy útil y confiable en la identificación de aislamientos de B. glumae (Jeong et al., 2003; Yang, 2004; Nandakumar et al., 2009). Alternativamente se puede utilizar perfiles de ácidos grasos para realizar la identificación de la bacteria, obteniéndose muy buenos resultados con el sistema Sherlock Microbial (Jeong et al., 2003; Weinberg et al., 2007).
Además se han diseñado técnicas moleculares para la identificación de B. glumae basándose en secuencias específicas de ADN en la región espaciadora entre los genes que codifican para los ARNr 16S y ARNr 23S (Takeuchi et al., 1997). Así, mediante el diseño de oligonucleótidos apropiados esa región específica puede ser amplificada mediante técnicas de reacción en cadena de la polimerasa (PCR), permitiendo la identificación de la bacteria de una manera rápida y precisa. Adicionalmente se han utilizado otros genes, como gyrB y rpoD, con secuencias específicas que permiten identificar y discriminar B. glumae y otras especies de Burkholderia que pueden estar presentes en los cultivos, incluyendo B. gladioli y B. plantarii (Kato et al., 1992; Maeda et al., 2006).
La aplicación de métodos moleculares permiten un análisis de una manera segura, rápida y con una alta especificidad y sensibilidad, detectando hasta diez células bacterianas en una muestra (Sayler et al., 2006). La amplificación por PCR es muy útil en la identificación de aislamientos bacterianos obtenidos de tejidos vegetales que presentan lesiones. Sin embargo, se deben aplicar con reserva cuando se analizan directamente muestras de plantas sin hacer cultivo bacteriano, dado que se puede detectar ADN proveniente de bacterias no viables presentes en las muestras (Nandakumar et al., 2009).
Además, es importante considerar si la técnica de PCR a utilizar es de punto final o de tiempo real, ya que la primera detecta solamente presencia o ausencia, mientras que la segunda permite realizar una cuantificación del número de copias de las moléculas detectadas (Millar y Moore, 2004).
Adicionalmente, estas técnicas moleculares pueden ser utilizadas no solo para detectar B. glumae, o bien otros fitopatógenos de interés, sino también su ADN directamente en semillas y determinar si la semilla está libre de fitopatógenos (Sayler et al., 2006).
Las técnicas de diagnóstico molecular tienen un papel importante en bacteriología clínica, sin embargo, nunca reemplazarán las metodologías de microbiología convencional que siguen siendo la piedra angular de la bacteriología moderna (Millar y Moore, 2004).
Los aislamientos de B. glumae deben ser analizados por su capacidad para causar daños en tejidos vegetales, ya que puede estar presente en plantas de arroz y no causar ningún daño debido a que su virulencia depende principalmente de la producción de la toxoflavina. De hecho se reporta que es común la presencia de cepas no virulentas en plantas (Cottyn et al., 2001) y en algunas investigaciones se ha recuperado hasta un 5% de cepas de B. glumae no patógenas (Nandakumar et al., 2009).
Para verificar esto, se inoculan suspensiones de la bacteria utilizando una jeringa o por aspersión sobre plantas de arroz sanas y luego de un período de incubación se observa la aparición de síntomas de la enfermedad. En caso de aparecer, el agente infeccioso debe ser aislado e identificado nuevamente, para corroborar su asociación causal con la sintomatología observada (Shaad et al., 2001; Yang, 2004; Arauz, 2011).
Detección de B. glumae en Costa Rica
El Laboratorio de Fitopatología del Centro de Investigación en Protección de Cultivos de la Universidad de Costa Rica, estandarizó la metodología para la detección de B. glumae en muestras de arroz empleando la técnica de PCR de punto final y utilizando imprimadores de regiones altamente conservadas, específicamente la región 16S, siendo este el más adecuado debido a que delimitan una región de 1000 pb, lo cual hace más robusto el análisis filogenético. La metodología empleada es la recomendada por el Centro Internacional de Agricultura Tropical (CIAT) en Colombia para la identificación de la bacteria a partir de semilla, usando imprimadores específicos de las regiones espaciadoras 16S-23S de ADN ribosomal, denominados GL-13f (5 ACA CGG AAC ACC TGG GAT 3) y GL 14r (5 TCG CTC TCC CGA AGA GAT 3) (Takeuchi et al., 1997; Fory y Prado, 2008).
Durante el periodo 2009-2010 se analizaron muestras de plantas recolectadas en el campo por técnicos de CONARROZ, las cuales presentaban la sintomatología descrita previamente. Las muestras fueron maceradas y posteriormente, se realizó la extracción de ADN empleando el kit de GIAGEN®, mini protocolo para tejido vegetal y se siguieron las indicaciones del fabricante. El PCR se realizó con la técnica estandarizada en el CIAT con las condiciones de amplificación: volumen final de 25 µl conteniendo el buffer Taq polimerasa Promega® (1X) (10 mM Tris HCl, pH 8,3; 50 mM KCl; 0,1% Triton® X-100); 1,5 mM MgCl2; 200 µM de cada dinucleótido (dATP, dCTP, DGTP, dTTp), 0,2 µM de cada uno de los cebadores mencionados, una (1) unidad de la enzima y 20 ng de la muestra de ADN que se extrajo previamente. El programa empleado en la reacción fue amplificado en un termociclador y el programa utilizado fue 94 ºC por dos minutos, seguido de veintinueve ciclos a 94 ºC por un minuto, 56 ºC por 45 segundos, 72 ºC por un minuto y una extensión final de 72 ºC por siete minutos, finalizando el programa a 10 ºC por diez minutos. Los productos obtenidos se analizaron mediante electroforesis en geles de agarosa al 1,5% teñidos con bromuro de etidio, en donde la presencia de una banda de amplificación de 400 pb se consideró positivo para B. glumae.
Algunas de las muestras de plantas se procesaron además por métodos convencionales realizando un aislamiento en medios de cultivo. Se realizó una desinfección previa al tejido vegetal que presentaba sintomatología, se depositó en beakers pequeños con alcohol 70%, se lavó y se agregó hipoclorito de sodio al 1% con tres lavados de agua destilada. Se colocó tejido en medio de cultivo King B y se incubó 48 horas a 32 ºC. Posterior a la incubación se realizó prueba de catalasa, oxidasa y se inoculó el medio TSI (Triple Sugar Iron por sus siglas en inglés) para confirmar si eran cepas no fermentadoras de azúcares. Las cepas que resultaron no fermentadoras (K/NC) se analizaron en platos BIOLOG GENIII® según las instrucciones del fabricante.
Resultados
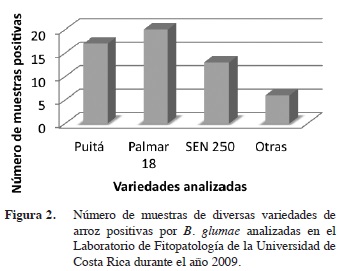
Durante el año 2009 se analizaron un total de 113 muestras de diferentes variedades, Puitá, Palmar 18 y SEN 250 entre otras, todas provenientes de las región Chorotega y de ellas el 49% estaban infectadas con B. glumae (Figura 2).
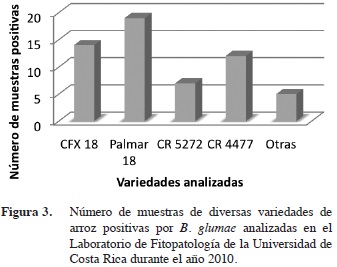
En el año 2010 se analizaron 129 muestras provenientes de las regiones Chorotega, Brunca y Pacífico Central, incluyendo las variedades Palmar 18, CFX 18, CR 5272, CR 4477. El 44% de las muestras estaban infectadas con B. glumae (Figura 3) mediante PCR de punto final y algunas se confirmaron por las técnicas de cultivo e identificación bioquímica. Estos resultados aportan la primera información sobre la prevalencia de un fitopatógeno bacteriano de gran importancia para el sector arrocero nacional.
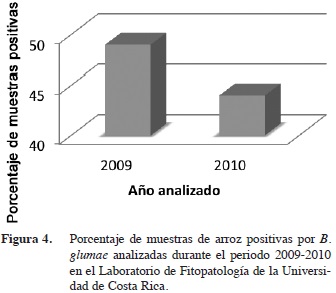
La Figura 4 representa los porcentajes de positividad de las muestras analizadas durante el periodo 2009-2010, en ambos casos, las muestras no superaron el 50% y en el segundo periodo se obtuvo un 5% menos de positividad con respecto al primer año estudiado.
Todas las muestras procesadas presentaban los síntomas descritos para B. glumae, tales como, decoloración del grano y vaneamiento; sin embargo, no todas fueron positivas. Más del 50% de las muestras procesadas arrojaron resultados negativos en el cultivo y en análisis molecular por B. glumae. Los síntomas descritos para las infecciones causadas por B. glumae no son patognomónicos, de hecho, hay microorganismos que pueden causar sintomatología similar como B. gladioli quien también produce la enfermedad pero con menor severidad (Nandakumar et al., 2009).
Durante el periodo analizado se encontraron muestras positivas por B. glumae, en la mayoría de estas se empleó la técnica de PCR de punto final y algunas se confirmaron por métodos convencionales y pruebas bioquímicas. Al diagnosticar la mayoría de positivos por PCR de punto final, se debe aclarar que sólo se detectó el ADN de la bacteria, sin poder determinar su viabilidad, su concentración ni patogenicidad, por lo tanto, es posible que la bacteria no estuviera viable, sólo se detectó su ADN, esto explica porqué a pesar de los positivos obtenidos, no se ha reportado un brote importante en el territorio nacional, pues CONARROZ reportó como principales causas de pérdidas para el periodo 2009-2010 inundación y sequía en las zonas Pacífico Central, Huetar Norte y Huetar Atlántica, sin reportar datos importantes de pérdidas sufridas en las regiones de las que provenían las muestras analizadas en este estudio.
A pesar de que no se ha logrado relacionar la bacteria con algún brote de importancia en territorio nacional, la sola presencia de esta representa una amenaza para el cultivo del arroz en Costa Rica que justifica y amerita un sistema de vigilancia para la detección temprana de brotes que a su vez permite intervenciones pertinentes y oportunas para evitar una mayor diseminación de la enfermedad.
Aunque es imposible controlar las variables meteorológicas como las precipitaciones y la temperatura, el combate de las infecciones por B. glumae se debe realizar con un buen manejo integrado. Se deben realizar fertilizaciones apropiadas, aplicando en cantidades adecuadas de nitrógeno, fósforo y potasio; este último elemento contribuye a detener enfermedades en el cultivo. También es primordial manejar baja densidad de siembra con el objetivo de controlar la humedad ambiental. Un factor de relevancia es el uso de semilla certificada, aquella que haya sido tratada y esté libre de potenciales patógenos microbianos.
Se debe incentivar a los productores costarricenses para el uso de semilla certificada y comprar sólo las variedades inscritas en la Oficina Nacional de Semillas a diferentes empresas que aseguren su calidad.
Con respecto a las técnicas empleadas, la de PCR de punto final permite detectar pocas moléculas de ADN blanco en la muestra y el tiempo de análisis puede ser muy corto en comparación con las técnicas de cultivo, su principal desventaja es que solamente permite detectar presencia o ausencia del material genético y no permite determinar si el agente infeccioso está viable o no, ni se puede conocer la concentración bacteriana en el tejido analizado, lo cual impide realizar un diagnóstico veraz de la situación del cultivo en campo o del análisis de semilla, así como no es posible realizar estudios posteriores de virulencia o de susceptibilidad a intervenciones de control con sustancias químicas, incluyendo antibióticos. En consecuencia, la técnica de PCR de punto final empleada es muy útil, pero solo puede aportar un diagnóstico presuntivo, no brinda uno definitivo.
Existen otras versiones de los métodos de amplificación, como la técnica de loop-mediated isothermal amplification (LAMP) (Caldera-Dominguez et al., 2013) o el PCR en tiempo real (Sayler et al., 2006), que permiten igualmente la detección molecular de B. glumae. La técnica de LAMP es relativamente más barata dado que no requiere termocicladores por llevarse a cabo a una sola temperatura. Sin embargo, no ofrece la posibilidad dada por el PCR en tiempo real de llevar a cabo una cuantificación de la carga bacteriana en las muestras analizadas. Al igual que el PCR de punto final, estas nuevas técnicas moleculares sólo permiten la detección del ADN bacteriano y no aportan mayor información sobre las características del agente infeccioso. Por el contrario, las pruebas bioquímicas se deben realizar posterior a la recuperación del microorganismo en estudio, ya que, para un diagnóstico definitivo se requiere realizar un aislamiento y asegurarse que el patógeno está presente y viable, básico en la identificación bacteriana, pero un tiempo más prolongado para obtener los resultados.
1. Este trabajo se realizó como requisito parcial para optar por el título en Microbiología y Química Clínica en la Facultad de Microbiología de la Universidad de Costa Rica, de la primer autora.
Literatura citada
Alfano, J.R., y A. Collmer. 2004. Type III secretion system effector proteins: double agents in bacterial disease and plant defense. Annu. Rev. Phytopathol. 42:385-414. [ Links ]
Arauz, F. 2011. Fitopatología, un enfoque agroecológico. Editorial de la Universidad de Costa Rica, Costa Rica. [ Links ]
Caldera-Dominguez, M.A., J. Ham, y R. Singh. 2013. Development of a loop-mediated isothermal amplification for detection of Burkholderia glumae. Poster. APS MSA Joint Meeting, August 10-14, 2013.Austin, Texas, USA. [ Links ]
Chun, H., O. Choi, E. Goo, N. Kim, H. Kim, Y. Kang, J. Kim, J. Moon, y I. Hwang. 2009. The quorum sensing-dependent gene katG of Burkholderia glumae is important for protection from visible light. J. Bacteriol. 191:4152-4157. [ Links ]
Coenye, T. 2009. Modern bacterial systematics in practice: Polyphasic taxonomy of the Burkholderia cepacia complex. Laboratory for Pharmaceutical Microbiology Ghent University, Belgium. [ Links ]
Compant, S., J. Nowak, T. Coyene, C. Clement, y E.A. Barka. 2008. Diversity and occurrence of Burkholderia spp. in the natural enviroment. FEMS Microbiol. Rev. 32:607-626. [ Links ]
CONARROZ (Coorporación Arrocera Nacional). 2012. Informe Anual Estadístico. Costa Rica. http://www.conarroz.com/UserFiles/File/INFORME_ANUAL_ESTADISTICO_2011-2012.pdf (Consultado 12 Nov. 2013). [ Links ]
Correa-Victoria, F. 2006. Asociación de la bacteria Burkholderia glumae al complejo acaro-hongo-bacteria en Panamá. Observaciones sobre muestras afectadas por el complejo en campos de arroz de Panamá. Aislamientos y pruebas de patogenicidad. http://ciat-library.ciat.cgiar.org:8080/jspui/bitstream/123456789/6691/1/complejo_acaro_costa_rica.pdf (Consultado 16 set. 2013). [ Links ]
Cottyn, B., E. Regalado, B. Lanoot, M. De Cleene, T. Mew, y J. Swings. 2001. Bacterial populations associated with rice in the tropical environment. Phytopathology 91:282-292. [ Links ]
Fuqua, C., y E. Greenberg. 2002. Listening in on bacteria: acyl homo serine lactona signalling. Nav. Rev. Mol. Cell. Biol. 3:685-695. [ Links ]
Galan, J.E., y H. Wolf-Watz. 2006. Protein delivery into eukaryotic cells by type III secretion machines. Nature 444:567-573. [ Links ]
Goto, K., y K. Ohata. 1956. New bacterial diseases of rice (brown stripe and grain rot). Ann. Phytopathol. Soc. Jpn. 21:46-47. [ Links ]
Guzmán, D. 2006. Manejo agronómico del cultivo del arroz sembrado bajo riego en finca Ranchos Horizonte en Guanacaste, Costa Rica. Sede regional de San Carlos, Instituto Tecnológico de Costa Rica, Alajuela, Costa Rica. [ Links ]
Ham, J.H., R.A. Melanson, y M.C. Rush. 2011. B. glumae: next major pathogen of rice? Mol. Plant. Pathol. 12:329-339. [ Links ]
Hikichi, Y., T. Okuno, y I. Furusawa. 1994. Susceptibility of rice spikelets to infection with P. glumae and its population dinamics. J. Pesticide Sci. 19:1-17. [ Links ]
Imlay, J.A. 2003. Pathways of oxidative damage. Annu. Rev. Microbiol. 57:395-418. [ Links ]
Iwai, T., K. Kaku, R. Honkura, S. Nakamura, H. Ochiai, T. Sasaki, e Y. Ohashi. 2002. Enhanced resistance to seed transmitted bacterial diseases in transgenic rice plants overproducing an oat cell wall bound thionin. Mol. Plant Microbe Interact 15:515-521. [ Links ]
Jeong, Y., J. Kim, S. Kim, Y. Kang, T. Nagamatsu, e I. Hwang. 2003. Toxoflavin produced by Burkholderia glumae causing rice grain rot is responsible for inducing bacterial wilt in many field crops. Plant Dis. 87:890-895. [ Links ]
Kang, Y., J. Kim, S. Kim, H. Kim, J.Y. Lim, M. Kim, J. Kwak, J.S. Moon, e I. Hwang. 2008. Proteomic analysis of the proteins regulated by HrpB from the plant pathogenic bacterium Burkholderia glumae. Proteomics 8:106-121. [ Links ]
Kato, T., T. Tanaka, e Y. Fujita. 1992. Studies on bacterial seedling blight of rice. Classification of bacteria, obtained from disease seedling of rice in Yamagata perfecture. Bull Yamagata Agric Exp Stn 26:103-109. [ Links ]
Kim, J., J. Kang, G. Jang, J. Jog, S. Lim, T Suga, e I. Hwang. 2004. Quorum sensing and the Lys R type transcriptional activator ToxR regulate toxoflavin biosynthesis and transport in B. glumae. Mol. Microbiol. 54:921-934. [ Links ]
Kim, J., Y. Kang, O. Choi, Y. Jeong, J. Jeong, J. Lim, M. Kim, J. Moon, H. Suga, e I. Hwang. 2007. Regulation of polar flagellum genes is mediated by quorum sensing and FlhDC in Burkholderia glumae. Mol. Microbiol. 64:165-179. [ Links ]
Kurita, T., y H. Tabei. 1967. On the casual agent of bacterial grain rot of rice. Ann. Phytopathol. Soc. Jpn. 33:111. [ Links ]
Latuasan HE., y W. Berends. 1961. On the origin of toxicity of toxoflavin. Biochim Biophys Acta 52:502-508. [ Links ]
Lim, J., T. Lee, B. Nahm, Y. Do Choi, M. Kim, e I. Hwang. 2009. Complete genome sequence of Burkholderia glumae BGR1. J. Bacteriol. 191(11):3758. DOI: 10.1128/JB.00349-09. [ Links ]
Maeda, Y., H. Shinojara, A. Kiba, K. Ohnishi, Y. Furuya, Y. Kawamura, T. Ezaki, P. Vandamme, S. Tsushima, y Y. Hikichi. 2006. Phylogenetic study and multiplex PCR based detection of Burkholderia plantarii, B. glumae, B. gladioli using gyrB and rpoD sequences. International Journal of Systematic and Evolutionary Microbiology 56:1031-1038. [ Links ]
Mahenthiralingam, E., T. Urban, y J. Goldberg. 2005. The multifarious, multireplicon Burkholderia cepacia complex”. Nat. Rev. Microbiol. 3:144-156. [ Links ]
Mahon, C., D. Lehman, y G. Manuselis. 2007. Diagnostic microbiology. 3 ed. Elsevier, St Louis Missouri. [ Links ]
Matsuda, I., y Z. Sato. 1988. Relation between pathogenicity and pigment productivity in the causal agent of bacterial grain rot of rice. Ann. Pytopathol. Soc. Jpn. 54:478. [ Links ]
Millar, B.C., y J.E. Moore. 2004. Molecular diagnostics: current options. Methods Mol. Biol. 266:139-166. [ Links ]
Nandakumar, R., A. Shahjahan, X. Yuan, E. Dickstein, D. Groth, C. Clarck, R. Cartwright, y M. Rush. 2009. B. glumae and B. gladioli cause bacterial panicle blight in rice in Sothern United States. Plant Dis. 93:896-905. [ Links ]
Pérez, C., y E. Saavedra. 2011. Avances en el manejo integrado de la bacteria B. glumae en el cultivo del arroz en el caribe colombiano Rev. Colombiana Cienc. Anim. 3(1):111-124. [ Links ]
Rush, M., Q. Shao, S. Zhang, K. Shahjahan, K. O´reilly, D. Shih, D. Groth, y D. Linscombe. 2003. Biotechnology and control of rice diseases. La. Agric. 46:20-23. [ Links ]
Sato, Z., Y. Koiso, S. Iwasaki, I. Matsuda, y A. Shirata. 1989. Toxins produced by P. glumae. Ann. Phytopathol. Soc. Jpn. 55:353-356. [ Links ]
Sayler, R., R. Cartwright, y Y. Yang. 2006 Genetic characterization and Real time PCR detection de B.glumae a newly emerging bacterial pathogen of rice in USA. Plant Dis. 90:603-610. [ Links ]
Schaad, N., J. Jones, y W. Chun. 2001. Laboratory guide for identification of plant pathogenic bacteria. 3rd ed. APS Press, Minnesota, USA. [ Links ]
Shingu, Y., y K. Yoneyama. 2004. Essential regulator gene toxR for toxoflavin biosynthesis of Burkholderia glumae. J. Gen Plant Pathol. 70:108-114. [ Links ]
Suzuki, F., H. Sawada, K. Azegami, y K. Tsuchiya. 2004. Molecular characterization of the tox operon involved in toxoflavin biosynthesis of B. glumae. J. Gen. Plant Pathol. 70:97-107. [ Links ]
Takeuchi, T., H. Sawada, F. Suzuki, e I. Matsuda. 1997. Specific detection for Burkholderia plantarii and Burkholderia glumae by PCR using primers selected from the 16S-23S ADNr spacer regions. Ann. Phytopathol. Soc. Jpn. 63: 455-462. [ Links ]
Tsushima, S. 2011. Study on control and epidemiology of bacterial grain rot of rice. J. Gen. Plant Pathol. 77:358-360. [ Links ]
Tsushima, S., S. Mogi, y H. Saito. 1989. Existence of Pseudomonas glumae on the rice seeds and development of the simple method for detecting P. glumae from the rice seeds. Bull. Kyushu Natl. Agric. Exp. Stn. 25:261-270. [ Links ]
Uematsy, T., D. Yoshimura, K. Nishiyama, T. Ibaragi, y H. Fujii. 1976. Pathogenic bacterium causing seedling rot of rice. Ann. Phytopathol. Soc. Jpn. 42:464-471. [ Links ]
Ura, H., N. Furuya, K. Liyama, M. Hidaka, K. Tsuchiya, y N. Matsuyama. 2006. Burkholderia gladioli associated with symptoms of bacterial grain rot and leaf-sheath browning of rice plants. J. Gen Plant Pathol. 72:98-103. [ Links ]
Urakami, T., C. Ito-Yoshida, H. Araki, T. Kijima, K.-I. Suzuki, y K. Komagata. 1994. Transfer of Pseudomonas plantarii and Pseudomonas glumae to Burkholderia as Burkholderia spp. and description of Burkholderia vandii sp. nov. Int. J. Syst. Bacteriol. 44:235-245. [ Links ]
Viallard, V., I. Poirier, B. Cournoyer, J. Haurat, S. Wiebkin, K. Phhel-Keller, y J. Balandreau. 1998. Burkholderia graminis a novel species of rhizospheric Burkholderia and reassessment of Pseudomonas phenazinium pyrrocinia and Pseudomonas glathei into Burkholderia. Int. J. Syst. Bacteriol. 48:549-563. [ Links ]
Weinberg, J., B. Alexander, J. Majure, L. Williams, J. Kim, P. Van Damme, y J. Lipuma. 2007. Burkholderia glumae infection in an Infant with chronic granulomatous disease. J. Clin. Microbiol. 45:662-665. [ Links ]
Yabuuchi, E., Y. Kosako, H. Oyaizu, I. Yano, H. Hotta, Y. Hashimoto, T. Ezaki, y M. Arakawa. 1992. Proposal of Burkholderia gen. nov and transfer of seven species of the genus Pseudomonas homology group II to the new genus, with the type species Burkholderia cepacia (Palleroni y Holmes 1981) comb. nov. Microbiol. Inmunol 36:1251-1275. [ Links ]
Yang, X. 2004. Identification of bacterial pathogens causing panicle blight of rice in Louisiana. Thesis M.Sc. The Department of Plant Pathology and Crop Physiology, Louisiana State University and Agricultural and Mechanical College, USA. [ Links ]
Zhou, X.G., A.M. Mcclung, M.O. Way, Y. Jo, R.E. Tabien, y L.T. Wilson. 2011. Severe outbreak of bacterial panicle blight across Texas Rice Belt in 2010 [abstract]. Phytopathology 101:S205. [ Links ]
Arauz, F. 2011. Fitopatología, un enfoque agroecológico. Editorial de la Universidad de Costa Rica, Costa Rica. [ Links ]
Caldera-Dominguez, M.A., J. Ham, y R. Singh. 2013. Development of a loop-mediated isothermal amplification for detection of Burkholderia glumae. Poster. APS MSA Joint Meeting, August 10-14, 2013.Austin, Texas, USA. [ Links ]
Chun, H., O. Choi, E. Goo, N. Kim, H. Kim, Y. Kang, J. Kim, J. Moon, y I. Hwang. 2009. The quorum sensing-dependent gene katG of Burkholderia glumae is important for protection from visible light. J. Bacteriol. 191:4152-4157. [ Links ]
Coenye, T. 2009. Modern bacterial systematics in practice: Polyphasic taxonomy of the Burkholderia cepacia complex. Laboratory for Pharmaceutical Microbiology Ghent University, Belgium. [ Links ]
Compant, S., J. Nowak, T. Coyene, C. Clement, y E.A. Barka. 2008. Diversity and occurrence of Burkholderia spp. in the natural enviroment. FEMS Microbiol. Rev. 32:607-626. [ Links ]
CONARROZ (Coorporación Arrocera Nacional). 2012. Informe Anual Estadístico. Costa Rica. http://www.conarroz.com/UserFiles/File/INFORME_ANUAL_ESTADISTICO_2011-2012.pdf (Consultado 12 Nov. 2013). [ Links ]
Correa-Victoria, F. 2006. Asociación de la bacteria Burkholderia glumae al complejo acaro-hongo-bacteria en Panamá. Observaciones sobre muestras afectadas por el complejo en campos de arroz de Panamá. Aislamientos y pruebas de patogenicidad. http://ciat-library.ciat.cgiar.org:8080/jspui/bitstream/123456789/6691/1/complejo_acaro_costa_rica.pdf (Consultado 16 set. 2013). [ Links ]
Cottyn, B., E. Regalado, B. Lanoot, M. De Cleene, T. Mew, y J. Swings. 2001. Bacterial populations associated with rice in the tropical environment. Phytopathology 91:282-292. [ Links ]
Fuqua, C., y E. Greenberg. 2002. Listening in on bacteria: acyl homo serine lactona signalling. Nav. Rev. Mol. Cell. Biol. 3:685-695. [ Links ]
Galan, J.E., y H. Wolf-Watz. 2006. Protein delivery into eukaryotic cells by type III secretion machines. Nature 444:567-573. [ Links ]
Goto, K., y K. Ohata. 1956. New bacterial diseases of rice (brown stripe and grain rot). Ann. Phytopathol. Soc. Jpn. 21:46-47. [ Links ]
Guzmán, D. 2006. Manejo agronómico del cultivo del arroz sembrado bajo riego en finca Ranchos Horizonte en Guanacaste, Costa Rica. Sede regional de San Carlos, Instituto Tecnológico de Costa Rica, Alajuela, Costa Rica. [ Links ]
Ham, J.H., R.A. Melanson, y M.C. Rush. 2011. B. glumae: next major pathogen of rice? Mol. Plant. Pathol. 12:329-339. [ Links ]
Hikichi, Y., T. Okuno, y I. Furusawa. 1994. Susceptibility of rice spikelets to infection with P. glumae and its population dinamics. J. Pesticide Sci. 19:1-17. [ Links ]
Imlay, J.A. 2003. Pathways of oxidative damage. Annu. Rev. Microbiol. 57:395-418. [ Links ]
Iwai, T., K. Kaku, R. Honkura, S. Nakamura, H. Ochiai, T. Sasaki, e Y. Ohashi. 2002. Enhanced resistance to seed transmitted bacterial diseases in transgenic rice plants overproducing an oat cell wall bound thionin. Mol. Plant Microbe Interact 15:515-521. [ Links ]
Jeong, Y., J. Kim, S. Kim, Y. Kang, T. Nagamatsu, e I. Hwang. 2003. Toxoflavin produced by Burkholderia glumae causing rice grain rot is responsible for inducing bacterial wilt in many field crops. Plant Dis. 87:890-895. [ Links ]
Kang, Y., J. Kim, S. Kim, H. Kim, J.Y. Lim, M. Kim, J. Kwak, J.S. Moon, e I. Hwang. 2008. Proteomic analysis of the proteins regulated by HrpB from the plant pathogenic bacterium Burkholderia glumae. Proteomics 8:106-121. [ Links ]
Kato, T., T. Tanaka, e Y. Fujita. 1992. Studies on bacterial seedling blight of rice. Classification of bacteria, obtained from disease seedling of rice in Yamagata perfecture. Bull Yamagata Agric Exp Stn 26:103-109. [ Links ]
Kim, J., J. Kang, G. Jang, J. Jog, S. Lim, T Suga, e I. Hwang. 2004. Quorum sensing and the Lys R type transcriptional activator ToxR regulate toxoflavin biosynthesis and transport in B. glumae. Mol. Microbiol. 54:921-934. [ Links ]
Kim, J., Y. Kang, O. Choi, Y. Jeong, J. Jeong, J. Lim, M. Kim, J. Moon, H. Suga, e I. Hwang. 2007. Regulation of polar flagellum genes is mediated by quorum sensing and FlhDC in Burkholderia glumae. Mol. Microbiol. 64:165-179. [ Links ]
Kurita, T., y H. Tabei. 1967. On the casual agent of bacterial grain rot of rice. Ann. Phytopathol. Soc. Jpn. 33:111. [ Links ]
Latuasan HE., y W. Berends. 1961. On the origin of toxicity of toxoflavin. Biochim Biophys Acta 52:502-508. [ Links ]
Lim, J., T. Lee, B. Nahm, Y. Do Choi, M. Kim, e I. Hwang. 2009. Complete genome sequence of Burkholderia glumae BGR1. J. Bacteriol. 191(11):3758. DOI: 10.1128/JB.00349-09. [ Links ]
Maeda, Y., H. Shinojara, A. Kiba, K. Ohnishi, Y. Furuya, Y. Kawamura, T. Ezaki, P. Vandamme, S. Tsushima, y Y. Hikichi. 2006. Phylogenetic study and multiplex PCR based detection of Burkholderia plantarii, B. glumae, B. gladioli using gyrB and rpoD sequences. International Journal of Systematic and Evolutionary Microbiology 56:1031-1038. [ Links ]
Mahenthiralingam, E., T. Urban, y J. Goldberg. 2005. The multifarious, multireplicon Burkholderia cepacia complex”. Nat. Rev. Microbiol. 3:144-156. [ Links ]
Mahon, C., D. Lehman, y G. Manuselis. 2007. Diagnostic microbiology. 3 ed. Elsevier, St Louis Missouri. [ Links ]
Matsuda, I., y Z. Sato. 1988. Relation between pathogenicity and pigment productivity in the causal agent of bacterial grain rot of rice. Ann. Pytopathol. Soc. Jpn. 54:478. [ Links ]
Millar, B.C., y J.E. Moore. 2004. Molecular diagnostics: current options. Methods Mol. Biol. 266:139-166. [ Links ]
Nandakumar, R., A. Shahjahan, X. Yuan, E. Dickstein, D. Groth, C. Clarck, R. Cartwright, y M. Rush. 2009. B. glumae and B. gladioli cause bacterial panicle blight in rice in Sothern United States. Plant Dis. 93:896-905. [ Links ]
Pérez, C., y E. Saavedra. 2011. Avances en el manejo integrado de la bacteria B. glumae en el cultivo del arroz en el caribe colombiano Rev. Colombiana Cienc. Anim. 3(1):111-124. [ Links ]
Rush, M., Q. Shao, S. Zhang, K. Shahjahan, K. O´reilly, D. Shih, D. Groth, y D. Linscombe. 2003. Biotechnology and control of rice diseases. La. Agric. 46:20-23. [ Links ]
Sato, Z., Y. Koiso, S. Iwasaki, I. Matsuda, y A. Shirata. 1989. Toxins produced by P. glumae. Ann. Phytopathol. Soc. Jpn. 55:353-356. [ Links ]
Sayler, R., R. Cartwright, y Y. Yang. 2006 Genetic characterization and Real time PCR detection de B.glumae a newly emerging bacterial pathogen of rice in USA. Plant Dis. 90:603-610. [ Links ]
Schaad, N., J. Jones, y W. Chun. 2001. Laboratory guide for identification of plant pathogenic bacteria. 3rd ed. APS Press, Minnesota, USA. [ Links ]
Shingu, Y., y K. Yoneyama. 2004. Essential regulator gene toxR for toxoflavin biosynthesis of Burkholderia glumae. J. Gen Plant Pathol. 70:108-114. [ Links ]
Suzuki, F., H. Sawada, K. Azegami, y K. Tsuchiya. 2004. Molecular characterization of the tox operon involved in toxoflavin biosynthesis of B. glumae. J. Gen. Plant Pathol. 70:97-107. [ Links ]
Takeuchi, T., H. Sawada, F. Suzuki, e I. Matsuda. 1997. Specific detection for Burkholderia plantarii and Burkholderia glumae by PCR using primers selected from the 16S-23S ADNr spacer regions. Ann. Phytopathol. Soc. Jpn. 63: 455-462. [ Links ]
Tsushima, S. 2011. Study on control and epidemiology of bacterial grain rot of rice. J. Gen. Plant Pathol. 77:358-360. [ Links ]
Tsushima, S., S. Mogi, y H. Saito. 1989. Existence of Pseudomonas glumae on the rice seeds and development of the simple method for detecting P. glumae from the rice seeds. Bull. Kyushu Natl. Agric. Exp. Stn. 25:261-270. [ Links ]
Uematsy, T., D. Yoshimura, K. Nishiyama, T. Ibaragi, y H. Fujii. 1976. Pathogenic bacterium causing seedling rot of rice. Ann. Phytopathol. Soc. Jpn. 42:464-471. [ Links ]
Ura, H., N. Furuya, K. Liyama, M. Hidaka, K. Tsuchiya, y N. Matsuyama. 2006. Burkholderia gladioli associated with symptoms of bacterial grain rot and leaf-sheath browning of rice plants. J. Gen Plant Pathol. 72:98-103. [ Links ]
Urakami, T., C. Ito-Yoshida, H. Araki, T. Kijima, K.-I. Suzuki, y K. Komagata. 1994. Transfer of Pseudomonas plantarii and Pseudomonas glumae to Burkholderia as Burkholderia spp. and description of Burkholderia vandii sp. nov. Int. J. Syst. Bacteriol. 44:235-245. [ Links ]
Viallard, V., I. Poirier, B. Cournoyer, J. Haurat, S. Wiebkin, K. Phhel-Keller, y J. Balandreau. 1998. Burkholderia graminis a novel species of rhizospheric Burkholderia and reassessment of Pseudomonas phenazinium pyrrocinia and Pseudomonas glathei into Burkholderia. Int. J. Syst. Bacteriol. 48:549-563. [ Links ]
Weinberg, J., B. Alexander, J. Majure, L. Williams, J. Kim, P. Van Damme, y J. Lipuma. 2007. Burkholderia glumae infection in an Infant with chronic granulomatous disease. J. Clin. Microbiol. 45:662-665. [ Links ]
Yabuuchi, E., Y. Kosako, H. Oyaizu, I. Yano, H. Hotta, Y. Hashimoto, T. Ezaki, y M. Arakawa. 1992. Proposal of Burkholderia gen. nov and transfer of seven species of the genus Pseudomonas homology group II to the new genus, with the type species Burkholderia cepacia (Palleroni y Holmes 1981) comb. nov. Microbiol. Inmunol 36:1251-1275. [ Links ]
Yang, X. 2004. Identification of bacterial pathogens causing panicle blight of rice in Louisiana. Thesis M.Sc. The Department of Plant Pathology and Crop Physiology, Louisiana State University and Agricultural and Mechanical College, USA. [ Links ]
Zhou, X.G., A.M. Mcclung, M.O. Way, Y. Jo, R.E. Tabien, y L.T. Wilson. 2011. Severe outbreak of bacterial panicle blight across Texas Rice Belt in 2010 [abstract]. Phytopathology 101:S205. [ Links ]
*Correspondencia a:
2. Centro de Investigación en Protección de Cultivos, Universidad de Costa Rica, San José, Costa Rica. Tel. 2511-8781. andrea.quesada@ucr.ac.cr (Autor para correspondencia).
3. Centro de Investigación en Enfermedades Tropicales, Facultad de Microbiología, Universidad de Costa Rica. San José, Costa Rica. Tel. 2511-8646. fernando.garcia@ucr.ac.cr
Recibido: 12 de diciembre, 2013. Aceptado: 30 de junio, 2014.














