Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agronomía Mesoamericana
versión On-line ISSN 2215-3608versión impresa ISSN 1659-1321
Agron. Mesoam vol.23 no.1 San Pedro jun. 2012
Diversidad de maíces criollos de Nuevo León, México, mediante aflp y caracteres morfológicos
Gilberto Rodríguez-Pérez2*, Francisco Zavala-García2, Carmen Ojeda-Zacarías2, Adriana Gutiérrez-Diez2, José Elías Treviño-Ramírez2, Froylan Rincón-Sánchez3*
*Dirección para correspondencia
Resumen
Diversidad de maíces criollos de Nuevo León, México, mediante AFLP y caracteres morfológicos. El presente trabajo tuvo como objetivo identificar la variabilidad fenotípica y molecular entre poblaciones de maíz Pinto Amarillo y Liebre en Nuevo León, México. En la caracterización fenotípica se estableció un experimento en primavera 2009. Las variables registradas fueron altura de planta, días a floración masculina y femenina, número de hileras por mazorca, granos por hilera de mazorca, longitud y diámetro de mazorca. Para la caracterización molecular, las treinta poblaciones se sembraron en febrero del 2010 en macetas con peat-moss, después de treinta días se colectaron todas las hojas de la plántula, para la técnica AFLP; se realizó un análisis conglomerados utilizando el método de UPGMA. Se identificaron tres grupos; doce poblaciones de Pinto Amarillo y tres de Liebre agrupando genotipos comunes. Los grupos se asociaron más a la raza de maíz que al lugar de colectas.
Palabras clave: Maíz, AFLP, caracterización molecular, caracterización fenotípica.
Abstract
Diversity of maize landraces of Nuevo León, México, using AFLP and morphological characters. The objective of this work was to identify the molecular and phenotypic variability among maize populations Pinto Amarillo and Liebre in Nuevo Leon, Mexico. The phenotypic characterization experiment was established in spring 2009. The variables registered were plant height, days to male and female flowering, number of rows per ear, kernels per row of ears, length and diameter of the corn ear. For the molecular characterization of the 30 populations, the seeds were planted in February 2010 in pots with peat-moss. At 30 days after planting all the leaves were collected and used in AFLP. A cluster analysis was conducted using UPGMA method. Three groups were identified; twelve populations of Pinto Amarillo and three of Liebre. The landraces within each group were associated more to races of maize more than to location.
Keys words: Maize, AFLP, molecular characterization, phenotype characterization.
Introducción
La caracterización de la diversidad genética en maíz es compleja, ya que implica evaluar un concepto multidimensional que involucra elementos etnográficos y la relación con el medio ambiente aplicados a una población, grupo de poblaciones o especies de plantas, y puede ser estudiada a nivel fenotípico, así como de ADN usando marcadores moleculares como AFLP (Amplified Fragment Length Polymorphisms). La caracterización molecular de maíces criollos tiene aplicación práctica en el mejoramiento vegetal tanto para la identificación de genotipos nativos, como para la estimación de relaciones genéticas (Vos et al. 1995).
En algunos estudios de caracterización fenotípica o molecular se han utilizado diversos tipos de información, técnicas de agrupamiento y teorías del origen del maíz (Sánchez y Goodman 1992, Rincón et al. 1996, Benz 1997); así, se han identificado grupos raciales bien definidos (Sánchez et al. 2000b). En muchas re- giones de México, los agricultores que cultivan variedades locales o criollas de maíz en forma tradicional, contribuyen a la conservación y a la generación de la diversidad genética in situ del cultivo (Bommer 1991). Los productores tradicionales mantienen las variedades locales al reproducirlas de una generación a otra (Louette y Smale 1996) y llegan a formarse nuevos tipos, variedades o razas (Hernández 1972, Dobzhans- ky 1982).
Han transcurrido 59 años de la publicación del trabajo clásico de Wellhausen et al. (1952) en el cual se propuso la clasificación de la diversidad genética del maíz en México en veinticinco razas; este y otros estudios permitieron establecer relaciones con otras razas de México (Sánchez y Goodman 1992).
Se estima que existen aproximadamente 400 razas de maíz en todo el mundo, de las cuales entre 250 y 300 razas se encuentran en América y de éstas, aproximadamente el 30% (75 razas) se encuentran en México. La mayoría de los estudios de la diversidad genética de maíz consideran regiones como México (Sánchez et al. 2000a) o el Continente Americano (Sánchez et al. 2000b) y describen las razas y sus agrupamientos de acuerdo con la similitud de sus características morfológicas.
La heterogeneidad ambiental, el aislamiento geográfico, la recombinación entre razas o poblaciones aledañas y la selección que los agricultores han realizado a través de miles de años en el maíz, con tribuyeron a la conservación in situ, al incremento de su diversidad y a su mejoramiento genético (Eagles y Lothrop 1994).
El maíz es el tercer cultivo comercial por área sembrada a nivel mundial, después del trigo y del arroz, con una superficie de 125 millones de hectáreas; en México es el más importante ya que se producen alrededor de 24,3 millones de toneladas en una superficie de 7,3 millones de hectáreas, de las cuales, el 94% corresponde al ciclo primavera-verano y el 6% al ciclo otoño-invierno.
El 88% de la superficie se siembra de temporal o secano y es el que representa a un mayor número de productores (FAOSTAT 2010), los cuales se pueden agrupar en: aquellos que poseen predios de 1 a 5 ha (92%) y aportan el 56,4% de la producción total; la producción se destina al autoconsumo y sus rendimientos fluctúan entre 1,3 y 1,8 t/ha y el resto (8%) corresponden a productores con predios mayores de 5 ha por productor y aportan el 43,6% de la producción nacional; sus rendimientos oscilan de 1,8 a 12 t/ha y únicamente destinan el 13,5% de su producción al autoconsumo.
Nuevo León cuenta con una superficie agrícola de 392, 415 ha abiertas al cultivo, lo que representa casi el 2% de la superficie agrícola total del país, de las cuales el 75% son de temporal y el 25% de riego. Estas zonas se encuentran en los valles y llanos del estado, siendo los municipios del centro y sur de la entidad, los que concentran el 82% de la superficie agrícola, donde el maíz tiene una participación de casi 66 mil ha, seguido de trigo y sorgo con 34 y 26 mil ha, respectivamente (SAGARPA 2009).
Las siembras de las poblaciones criollas de maíz en el estado de Nuevo León se realizan en menor su perficie con tecnología intermedia con variedades de polinización abierta como Pinto Amarillo y Liebre, que cuentan con amplia distribución, precocidad y adaptación a sequía entre otras características.
Según la información obtenida como parte de los resultados de un proyecto financiado por la CONA BIO, Zavala et al. (2010) establecieron que dentro de 75 poblaciones, algunas tienen influencia a la raza ratón o tuxpeño, ratón x tuxpeño, ratón x tuxpeño norteño, tuxpeño x ratón norteño, de estas colectas seis fueron de Güerito, tres de Breve Padilla, cinco de Hualahuises, cuatro de Ligero, cuatro de Blanco Liviano, cuatro de Temprano, cuatro de La Purísima, cuatro de Blanco Grueso, cuatro de Olote Colorado, tres de Pilinque-Saire, ocho de Liebre, 22 de Pinto Amarillo, dos de NL-Amarillo y dos de NL-127.
De las poblaciones de maíces colectadas en las partes Norte del Estado de Nuevo León, existe un alto porcentaje (54,5%) que corresponden a la variedad Pinto Amarillo y Liebre, las cuales pudieran estar emparentadas a pesar de haber sido colectadas en diferentes municipios del estado.
El maíz cultivado en la parte norte del estado ha sido la principal fuente de alimento para la población desde la época de las haciendas coloniales. En esta región, las áreas dedicadas a la producción de maíz de temporal son principalmente valles, cañadas y mesetas que exhiben condiciones fisiográficas distintivas, las cuales han favorecido el desarrollo de un gran número de variedades locales o criollas con adaptación restringida a cada uno de esos ambientes naturales.
En los municipios donde se colectaron las poblaciones la producción obtenida es dedicada mayormente a satisfacer las necesidades de las familias de los agricultores, las cuales lo utilizan como grano tierno (elote y esquites), grano tostado y molido (pinole y atoles), grano fermentado (pozol) y como materia prima para la elaboración de masa nixtamalizada que utilizan para la preparación de tamales y tortillas. Además su uso se extiende a la alimentación del ganado.
Debido a la importancia que representan estas poblaciones entre las colectas del Norte de Nuevo León y la necesidad de identificar las mejores para realizar un mejoramiento genético, se realizó el presente trabajo con el objetivo de identificar la variabilidad fenotípica y molecular entre poblaciones de maíz Pinto Amarillo y Liebre en Nuevo León, México.
Materiales y Métodos
La caracterización molecular se realizó en el Laboratorio de Biotecnología de la Facultad de Agronomía de la Universidad Autónoma de Nuevo León, en el Campus de Ciencias Agropecuarias, ubicado en el Municipio de Escobedo en Nuevo León, mientras que la caracterización fenotípica se llevó a cabo en el Campo Experimental de la Facultad de Agronomía, Campus Marín, ubicado en el Municipio de Marín, Nuevo León, México.
Se utilizaron treinta poblaciones de maíces criollos (Cuadro 1), veintidós de Pinto Amarillo y ocho de Liebre, colectadas en catorce municipios de la parte centro y norte del Estado de Nuevo León.
Caracterización molecular
Las treinta poblaciones se sembraron en febrero del 2010 en macetas con peat-moss, se depositaron seis semillas en cada una. Cuando las plantas alcanzaron una altura de 20 cm, se colectaron todas las hojas de las seis plantas y se colocaron en bolsas de polietileno de cierre hermético previamente identificadas con el número de la muestra, las cuales se almacenaron en un ultracongelador (-70°C) hasta su procesamiento. La extracción de ADN se realizó de acuerdo con el protocolo modificado de Saghai- Maroof y colaboradores (Hoisington et al. 2001).
La cuantificación del ADN se realizó por medio del espectrofotómetro NanoDrop 2000c (Thermo Scientific) y la calidad se verificó en gel de agarosa al 1% teñido con SYBR Green. Para la generación de AFLP se utilizó el estuche comercial IRDye Fluorescent AFLP Kit for Large Plant Genome Analysis (LICOR Biosciences, Nebraska, modelo 4200).
La amplificación selectiva se realizó con un proto- colo de PCR conocido como “con doce combinaciones dúplex” (con dos iniciadores EcoRI marcados con fluorescencia uno a 700 y el otro a 800 nm) en un volumen total de reacción de 11 μl, con el propósito de producir los fragmentos de amplificación de los ácidos nucleótidos de AFLP como adenina, citocina, guanina y tiamina.
La separación de los productos de amplificación se llevó a cabo en un gel de poliacrilamida al 6,5% en un secuenciador de LICOR IR2 4200 (LICOR Biosciencies, Lincoln, NE). El programa para el análisis fue el SAGAmx y se utilizó como criterio de selección la definición e intensidad de las bandas amplificadas.
Se generó una matriz de datos binarios para determinar el nivel de agrupamiento o similitud entre las poblaciones generando dendogramas utilizando el método de UPGMA.
b) Caracterización fenotípica
La caracterización fenotípica se realizó en primavera del año 2009. Se estableció un experimento de bloques completos al azar con parcelas de cinco surcos de cinco metros de longitud a una distancia entre surcos de 0,80 m y cinco plantas por metro lineal con cuatro repeticiones.
Las variables utilizadas para la caracterización fenotípica fueron:
Altura de planta (AP, cm). Medida desde la base de la planta a la punta de la espiga, se utilizó una muestra aleatoria de diez plantas.
Días a floración masculina (DFM). Se consideró cuando más del 50% de las plantas de la parcela estuvieran soltando polen en más del 50% de la espiga.
Días a floración femenina (DFF). Se registró cuando más del 50% de las plantas de la parcela pre sentaron los estigmas receptivos.
Número de hileras (NH). Se estimó contando las hileras de una muestra representativa de cinco mazor- cas de cada parcela; el dato se promedió y se registró como el dato promedio por mazorca.
Número de granos por hilera (# G), se contaron los granos de una hilera representativa de cada muestra representativa de cinco mazorcas de cada parcela. Se obtuvo un promedio y éste dato se utilizó para registrar el dato por mazorca.
Longitud de la mazorca (LM, cm). Medida desde la base a la punta de la mazorca. El tamaño de muestra fue representativa de cinco mazorcas por parcela. Se calculó el promedio en cm y se registró el dato por parcela.
Diámetro de la mazorca (DM, cm). Esta variable se midió con un vernier graduado en cm, se colocó en la parte media de la mazorca y se obtuvo el dato de cinco mazorcas representativas. Se calculó el prome- dio en cm y se registró el dato por parcela.
Análisis de datos
Para el análisis molecular con las bandas de AFLP, se generó la matriz de datos binarios. Se utilizó como criterio de selección cuando en las bandas amplificadas se observara una intensidad oscura fuerte, se seleccionaron las bandas y registraron las bandas como variables binarias utilizando el número “1” para indicar la presencia de patrones amplificados y “0” cuando la intensidad fuera tenue para indicar su ausencia.
En la caracterización fenotípica se generó una matriz promediando los datos agronómicos de cuatro repeticiones como altura de planta, días a floración masculina y femenina, longitud y diámetro de mazorca y número de hileras por mazorca.
Se utilizó el método de UPGMA (media métrica no ponderada) para calcular las distancias y generar los grupos más compactos y homogéneos y así diferenciar los grupos molecular y fenotípicamente dentro de los grupos de las poblaciones. Este método es eficiente, ya que genera conglomerados equilibrados y de tamaño pequeño, además de que tiene interpretación sencilla (Peña 1987).
El método de UPGMA asume que las especies son grupos por sí mismas, luego relaciona los grupos más cercanos basado en la matriz de distancias, recalcula la matriz de distancia y repite el proceso hasta que todas las especies estén conectadas a un único grupo. El método UPGMA realiza todos sus cálculos con la matriz calculada hallando la distancia genética entre las especies. La ventaja de este método es muy sencillo ya que realiza los cálculos basados en la matriz de distancias y no directamente sobre las secuencias es mucho más rápido computacionalmente que los métodos basados en carácter.
Se utilizó el programa Statistic (versión 6.0) para la generación de dendogramas. El criterio tomado para la distancia de corte y definición de los grupos en los dendogramas fue establecido sobre el número óptimo de grupos cuando se produjeron saltos bruscos en las distancias.
Resultados y Discusión
En el análisis molecular se seleccionaron 434 bandas amplificadas de doce combinaciones dúplex de dos iniciadores utilizados: (EcoRl-700) y (EcoRl-800) los cuales produjeron fragmentos de amplificación de los ácidos nucleótidos de AFLP como: (A) adenina, (C) citocina, (G) guanina y (T) tiamina (Cuadro 2).
El análisis de bandas se realizó en el programa SAGAmx que incluyó también un análisis de conglomerados, el cual permitió identificar varios grupos a partir de la generación de una matriz acumulativa.
Desde una perspectiva multivariada se hizo un análisis de componentes principales considerando las 30 poblaciones, 22 de Pinto Amarillo y ocho de Liebre, previa estandarización de los datos y una rotación de tipo Varimax en los componentes resultantes para determinar la correlación con cada uno de los factores.
En la Figura 1 se muestra como ejemplo, el patrón de bandas amplificadas con la combinación de iniciadores M-CTT–E-AAG, seleccionando las bandas y registrándolas como variables binarias utilizando el nú- mero “1” para indicar la presencia de patrones amplificados con la intensidad de color oscuro y “0” cuando la intensidad fuera tenue para indicar su ausencia.
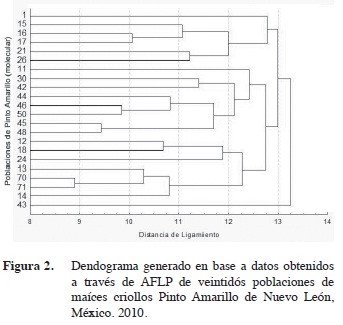
En la Figura 2 se observa la formación de tres grupos principales de las veintidós poblaciones de Pinto Amarillo, las cuales se agruparon específicamente de acuerdo a la presencia de las bandas seleccionadas.
El primer grupo se constituyó por ocho poblaciones, al asociarlos con las razas, el 50% de estas tuvieron asociación a la raza Ratón, el 37,5% a Tuxpeño y 12,5% a Tuxpeño x Ratón. Este grupo se caracterizó por la distribución geográfica alejada entre cada población donde se colectaron, sin embargo; las condiciones climáticas de este grupo indican que el 88% de las colectas se siembran bajo condiciones de temporal lo que agrupó aquellas colectas con un porte de planta con 198 cm, 79 días a la madurez, longitud e hileras de mazorca con 13,5 y 2,3 cm el diámetro de la mazorca. Esta similitud pudo ser influenciada por el medio ambiente en que se han adaptado las variedades a pesar de pertenecer a diferentes municipios debido a la diversidad genética y la posible existencia de intercambio de semilla realizada por los agricultores a través de los años, coincidiendo con lo descrito por Hartings et al. (2008) quienes concluyeron que la diversidad genética de las poblaciones es altamente influenciada por las condiciones ambientales específicas y locales asociadas a su origen geográfico, mas no se establecieron las relaciones en base a las razas de maíz per se.
El segundo grupo lo integraron ocho poblaciones de las cuales se sembraron bajo condiciones de riego distinguiéndose con un porte de altura de planta con 200 cm, a los 78 días de madurez, una longitud de mazorca de 14,1 cm y 13,5 cm de hileras por mazorca con un promedio de 14,2 en número de granos por hilera y 2,4 de diámetro de mazorca.
Los datos mostrados indican que existe variación entre las poblaciones adaptadas a condiciones de tem- poral en climas secos. La definición de patrón etnofitogenético de (Muñoz 2003) es aplicable a la diversidad observada en las ocho poblaciones de maíz ya que es posible dentro de una microrregión diferenciar genotipos en base a sus características.
Las poblaciones se colectaron en siete municipios representando el 50% de estos, indicando la existencia de amplia distribución de la diversidad genética encontrada del lugar de origen, es decir, las poblaciones que se agruparon en este grupo son molecularmente iguales y estos resultados tienen coincidencia con lo reportado por Liu et al. (2009); ellos encontraron alto nivel de la diversidad genética; de 247 colectas de maíces nativos en China formaron grupos diferentes, principalmente enfocados a factores geográficos de siete provincias del sur de China.
El tercer grupo se constituyó por seis poblaciones provenientes de cuatro municipios, que están muy cercanos geográficamente presentando estrecha relación, prevaleciendo de que no hay variación genética entre estas poblaciones (Muñoz 2003). Las siembras de estas poblaciones se realizan bajo condiciones de tem- poral lo que se distinguieron por presentar promedios de altura de planta con 1,98 cm, fueron más precoces a los 76 días, presentaron longitud de mazorca de 13,1 cm y un diámetro de 2,3 cm.
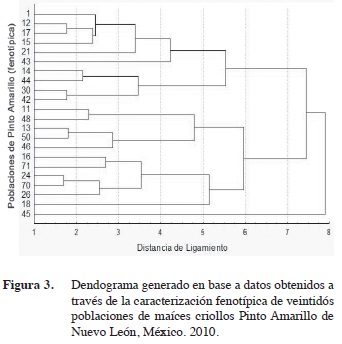
Para el caso de la caracterización fenotípica, la dispersión de los datos mostró variación entre las veintidós poblaciones (Figura 3), se aprecia la formación de tres grupos principales, al igual que en los datos de AFLP, esto indica que las poblaciones que se formaron en cada grupo son fenotípicamente iguales a pesar de pertenecer a distintas áreas geográficas en que se colectaron.
El primer grupo lo formaron siete poblaciones las cuales tuvieron el 57,1% asociación a la raza Tuxpeño x Ratón, el 14,3% a Ratón y otro 28,6% a Tuxpeño. La información proporcionada por los agricultores que aportaron las colectas de estas variedades, comentan que 86% de estas poblaciones se cultivan bajo con- diciones de temporal, contando con un rango entre 350-640 mm anuales de precipitación; de esta manera presentaron altura de plantas de 197 cm, fueron más precoces a los 76 días, obtuvieron longitud de mazorca de 13,0 cm y un duiametro de mazorca de 2,1 cm, el otro 14% de las poblaciones se cultivan bajo riego.
Estas poblaciones presentaron una variabilidad genética estrecha dentro de las regiones en que se colectaron (Ramírez et al. 1988).
El segundo grupo se constituyó por cinco poblaciones con una proporción del 80% a la raza Ratón, el 40% a Tuxpeño x Ratón; las siembras en este grupo de poblaciones se realizan bajo condiciones de temporal presentando un rango entre 380-650 mm anuales de precipitación; en función a la disposición de agua, en este grupo sobresalen los promedios, principalmente por su porte en altura de planta de 189 cm, fueron de ciclo a los 78 días de madurez, mientras que el diámetro de mazorca fue 13,2 cm, el número de hileras por mazorca con 12,8 y granos por hileras fue de 34 y un diámetro de mazorca de 2,2 cm. Por esta razón, parecen ser una excelente fuente de resistencia a sequía. Bajo estas condiciones ambientales en las que se cultivan estas variedades difícilmente prosperarían los híbridos comerciales, razón por la cual son conservadas por los agricultores, conscientes de la eventualidad de las lluvias para la agricultura temporal (Martín et al. 2008).
La distribución de estas colectas presentaron amplia diversidad de las poblaciones como lo reportado por Ron et al. (2008) quienes encontraron amplia diversidad genética, de 127 colectas de maíces nativos de los estados de Jalisco, Michoacán y Nayarit el cual les permitió identificar 53 colectas con 42% influencia de la raza Tuxpeño, 26% a Tabloncillo y 41 colectas con 32% influencia de la raza Mushito.
El análisis de los conglomerados permitió identificar cuatro, tres y cuatro poblaciones de Pinto Amarillo de las Figuras 1 y 2, mostrando similitud molecular y fenotípica de acuerdo al área geográfica en que colectaron estas variedades se encontraron los siguientes grupos: en el primero fueron las poblaciones 18 de Villaldama, 70 de Dr. González, 24 (Los Ramones) y 71 (Paras). El segundo grupo lo integraron las poblaciones 46 (García), 50 (China) y 11 (Pesquería); el tercer grupo se identificaron las poblaciones 21 (Pesquería), 1 y 15 de Villaldama, 17 (Bustamante) y 12 de Hidalgo).
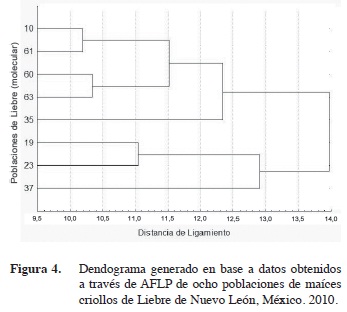
El análisis de agrupamiento utilizando datos moleculares de las ocho poblaciones de Liebre (Figura 4) resultó en la formación de dos grupos, el primero se integró con tres poblaciones 37, 23 y 19 pertenecientes a Los Ramones y Pesqueria, este grupo se caracterizó por presentar asociación a la raza Ratón (Olotillo), sin embargo, las condiciones que presentan las poblaciones son en siembras bajo condiciones de riego distinguiéndose por presentar promedios en altura de planta de 210 cm, 78 días a la madurez, 14,4 cm en longitud de mazorca, 3,7 cm en diámetro de mazorca con un número de granos por hilera por mazorca de 12,1.
El segundo grupo se constituyó por cinco poblaciones, tres de ellas 63, 60 y 61 pertenecen a Cadereyta las cuales tuvieron el 60% asociación a Ratón x Tuxpeño, las dos restantes 10 y 35 pertenecen a Pesqueria con 40% de asociación a la raza Ratón. Este grupo de poblaciones se cultivan con poca disponibilidad de agua entre los 560-800 mm anuales de precipitación; se distinguen por presentar un porte de 200 cm de altura de planta, 74 y 77 días de floración masculina y femenina respectivamente, 13,6 cm de longitud de mazorca, 3,5 en diámetro de mazorca y 11,5 hileras por mazorca.
Las características de las poblaciones tienen relación con las precipitaciones, esto es, que las poblaciones más precoces y menor altura de planta se cultiven en áreas de temporal con menores promedios de precipitaciones, lo anterior refleja una relación entre la diversidad genética del maíz y la variación ecológica ambiental, lo cual refiere (Romero y Muñoz 1996), (Gil et al. 1995).
En este estudio las poblaciones presentaron una estrecha similitud molecular debido a la cercanía de los municipios donde se colectaron y coincide con la investigación por Reif et al. (2006) quienes caracterizaron molecularmente veinticinco razas pertenecientes a once estados de México, entre ellas la raza Tuxpeño; ellos concluyeron que una distribución de la diversidad genética presenta un patrón de aislamiento por distancia; esto es, poblaciones más cercanas genéticamente y geográficamente se parecen más entre sí.
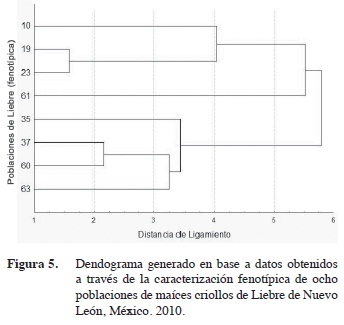
Respecto a la caracterización fenotípica de las ocho colectas de Liebre (Figura 5), se formaron cuatro grupos, caracterizados fenotípicamente iguales; en el primer grupo con dos poblaciones 60, 63, 37 y 35 per- tenecientes a Cadereyta se asociaron el 50% a la raza Tuxpeño x Ratón, la población 35 de Pesqueria, la 37 a Los Ramones se asociaron a la raza Ratón (Olotillo).
De la información proporcionada por los agricultores al momento de las colectas, se pudo notar que tres poblaciones 60, 37 y 63 de este grupo se cultivan bajo condiciones de temporal teniendo un rango de precipitación entre 550-900 mm anuales; se distinguen por tener promedios de planta de 200 cm de altura, fueron más precoces con 74 y 77 días de floración masculina y femenina respectivamente; tuvieron menor tamaño y grosor de mazorca con 14 y 3,6 cm respectivamente, el número de hileras por mazorca fue de 11,5 y con 34 granos por hileras.
Las poblaciones presentaron una marcada diferencia en las características agronómicas al sembrarse en condiciones de escasa precipitación en ambientes de temporal, lo que coincide lo mencionado por (Pecina et al. 2009).
El segundo grupo formado por cuatro poblaciones, 10 y 23 provenientes de Pesqueria, 61 de Cadereyta, y la 19 de Los Ramones, el 75% se asoció a la raza Ratón (Olotillo), el 25% con influencia a la raza Ratón. En este grupo se identificaron dos poblaciones la 23 y 10 del grupo tardío las cuales se han estado sembrando bajo condiciones de riego, se distinguieron por porte de altura de 210 cm, de ciclo tardío a los 77 y 80 días de floración masculina y femenina respectivamente, el tamaño de mazorca fue de 15,6 cm con un grosor de 4,1 cm, el número de hileras por mazorca fue de trece, tuvieron 38,2 granos por mazorca.
Puede deducirse que de acuerdo a esta información, estos dos municipios que están geográficamente cercanos, los agricultores han estado contribuyendo al intercambio de semilla a través de los años que han conducido a una menor variabilidad genética de las características fenotípicas coincidiendo lo mencionado por (González et al. 2006).
Las poblaciones 61 y 19 del grupo precoz se siembran en condiciones de temporal caracterizada por presentar valores bajos de datos agronómicos. Información similar reportan Santacruz et al. (2010), quienes concluyeron que el 66% de poblaciones de maíz se cultivan bajo condiciones de riego correspondientes al grupo tardío, el resto 34% en temporal al grupo precoz presentaron el 60% influencia a la raza Zapalote Chico, mientras que el 40% restante fueron entre Tuxpeño, Vandeño y Tepecintle.
Al asociar los datos moleculares y fenotípicos el análisis conglomerado permitió identificar tres poblaciones (Figuras 3 y 4): la 37 perteneciente a Los Ramones, la 61 de Cadereyta y la 10 de Pesqueria, las cuales están adaptadas a condiciones de temporal con poca disponibilidad de agua.
Componentes principales
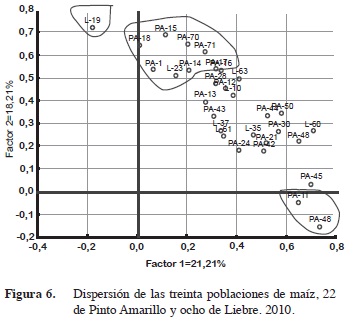
De la variabilidad total observada en las 30 pobla- ciones de maíz, 22 de Pinto Amarillo y ocho de Liebre con base en las cinco variables registradas (Figura 6), los dos factores explicaron el 39,42% (Cuadro 3). El factor 1, 21,1% presentó una mayor asociación positiva con 26 poblaciones, dentro de ellas se agruparon siete poblaciones de Liebre y 19 de Pinto Amarillo, las poblaciones de mayor asociación fueron la población PA-18 (0,67), PA-15 (0,69), PA-70 (0,64), PA-71 (0,63), PA-1 (0,52), PA-14 (0.56), Liebre-23 (0,55), PA-16 (0,58) y PA-17 (0,57); mientras que las poblaciones PA-48 y PA-11 presentaron asociación negativa.
En el factor 2 (18,21%) estuvo determinado principalmente por la población 19 de Liebre.
Estos resultados muestran la diversidad genética de las poblaciones de la zona norte del estado de Nuevo León considerando las características agronómicas de las poblaciones, de tal manera que se pueden considerar como un criterio para realizar un programa de mejoramiento a futuro (Herrera et al. 2000). Al respecto, Gil et al. (2004) señalaron que existe variabilidad genética entre los materiales locales y entre nichos ecológicos, en términos de nivel de precocidad y características agronómicas, la variabilidad atribuible a la selección efectuada durante generaciones por los agricultores (Figura 7).
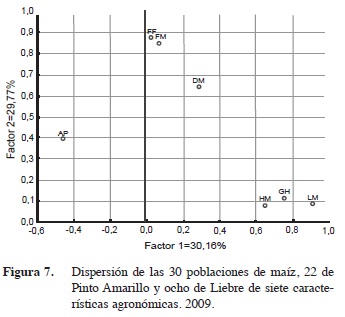
El análisis de componentes principales para las características agronómicas mostró que los dos componentes en conjunto explicaron 59,93% de la variación fenotípica observada de las treinta poblaciones. De acuerdo con los vectores propios, en el primer factor las variables con mayor peso fueron: floración femenina, floración masculina y diámetro de mazorca presentaron asociación positiva con 0,87; 0,84 y 0,64 respectivamente, mientras que hileras por mazorca, granos por hilera y longitud de mazorca presentaron baja asociación.
La diversidad en precocidad es común entre las poblaciones nativas. Al respecto, Ángeles et al. (2010) señalaron que existe variabilidad genética entre los materiales locales y entre nichos ecológicos, en términos de color del grano, nivel de precocidad y características agronómicas, variabilidad que es atribuible a la selección efectuada durante generaciones por los productores, para tipo y color de grano según los usos. Hortelano et al. (2008) también señalaron que en el Valle de Puebla la variación genética existente puede agruparse de acuerdo con influencia para explicar la variación observada en días a floración femenina, número de hojas arriba de la mazorca, área de la hoja de la mazorca, aspecto de planta, longitud de mazorca, número de hileras de grano por mazorca, diámetro de mazorca, longitud de grano, ancho de grano, grosor de grano y diámetro de olote.
Literatura Citada
Ángeles, GE; Ortiz, TE; López, AP; y López, RG. 2010. Caracterización y Rendimiento de Poblaciones de Maíz Nativas de Molcaxac, Puebla. Rev. Fitotec. Mex. Vol. 33 (4): 287–296 pp. [ Links ]
Benz, F. 1997. Diversidad y distribución prehispánica del maíz mexicano. Arqueología Mexicana 25:16-23. [ Links ]
Bommer, R. 1991. The historical development of international collaboration in plant genetic resources. In Van Hintun Th. JL; Frese, L; Perrer, PM. eds. Searching for new concepts for collaborative genetic resources management. Papers of the EUCARPIA/IBPGR Symposium. International Board for Plant Genetic Resources. Roma, Italia. p. 3-12. [ Links ]
Dobzhansky, T. 1982. Genetics and the origin of species. Columbia University Press. Series: The Columbia Classics in Evolution. New York, USA. 364 p. [ Links ]
Eagles, A; Lotrop, E; 1994, Highland maize from central Mexico its origin, characteristics and use inbreeding programs. Crop sci 34:11-19. [ Links ]
FAOSTAT, 2010. FAO Stastitical Databases. Food and Agriculture Organization of the United Nations (en línea). Consultado 7 junio 2010. Disponible http://foastat.fao.org/ [ Links ]
Gil, MA; Muñoz, OA. Carballo, CA; Trinidad, SA. 1995. El patrón varietal en la región sureste de la Sierra Purépecha. I. Variables importantes empleadas en su definición. Rev. Fitotec. Méx. 18: 163-173. [ Links ]
Gil, MA; López, APA; Muñoz, OA; y López, SH. 2004. Variedades criollas de maíz (Zea mays L.) en el Estado de Puebla, México: diversidad y utilización. In Manejo de la diversidad de los cultivos en los agroecosistemas tradicionales. J L Chávez-Servia, J Tuxill y D I Jarvis (eds). Instituto Internacional de Recursos Fitogenéticos. Cali, Colombia. p. 18-25. [ Links ]
González, HA; Sahagún, CJ; Pérez, LDJ; Dominguez, LA; Serrato, CR; Landeros, FV; Dorantes, CE. 2006. Diversidad del maíz Cacahuacintle en el valle de Toluca, México. Rev. Fitotec. Méx. 29(3):255-261. [ Links ]
Hartings, H; Berardo, H; Mazzinelli, F; Valoti, P; Verderio, A; Motto, M: 2008. Assessment of genetic diversity and relationships among maize (Zea mays L.) Italian Landraces by Morphological Traits and AFLP Profiling. Theor Appl Genet 117:831–842. [ Links ]
Hernández, E. 1972. Exploración etnobotánica en maíz. Fitotecnia Latinoamericana 8:46-51. [ Links ]
Herrera, CBE; Castillo, GF; Sánchez, GJJ; Ortega, PGR; Goodman, MM. 2000. Caracteres morfológicos para valorar la diversidad entre poblaciones de maíz en una región: Caso la Raza Chalqueño. Rev. Fitotec. Méx. 23:335-353. [ Links ]
Hoisington, M; Khairallah M; González de León, D. 2001. Laboratory protocols: CIMMYT applied molecular genetics laboratory. CIMMYT. 2 ed. México, D.F. p. 5-9. [ Links ]
Hortelano, SRR; Gil, MA; Santacruz, VA; Miranda, CS y Córdova, TL. 2008 Diversidad morfológica de maíces nativos en el Valle de Puebla. Agric. Téc. Méx. 34:189-200. [ Links ]
Liu, Z; Guo, H; Zhao, R; Cai, L; Wang, G; Cao, J; Wang, H; Shi, S; Song, C; Wang, Y; and Li, Y. 2009. Population structure and genetic diversity of maize landraces from the southwest maize region of China. Maidica 54:63-76. [ Links ]
Louette, D; Smale M; 1996. Genetic diversity and maize seed management in a traditional mexican community: Implications for in situ conservation of maize. NRG papers 96-03. CIMMYT. México. D.F. 21 p. [ Links ]
Martin, LJG; Ron, PJ; Sánchez, GJJ; De la Cruz, LL; Morales, RMM; Carrera, VJA; Ortega, CA; Vidal, MVA y Guerrero, HMJ. 2008. Caracterización agronómica y morfólogica de maíces nativos del noroccidente de México. Rev. Fitotec. Méx. 31(4):331-340. [ Links ]
Muñoz, OA. 2003. Centli-maíz. Prehistoria e historia, diversidad, potencial, origen genético y geográfico. Colegio de Posgraduados, Montecillo, Texcoco, México. 210 p. [ Links ]
Pecina, MJA; Mendoza, CMC; López, SJA; Castillo, GF; Mendoza, RM. 2009. Respuesta morfológica y fenológica de maíces nativos de Tamaulipas a ambientes contrastantes de México. Agrociencia 43 número 7:681-694. [ Links ]
Peña, D. 1987. Estadística. Modelos y métodos. Modelos lineales y series temporales. Vol. 2 Editorial Alianza. Madrid, España. p. 190-191. [ Links ]
Ramírez, FA; Angeles, AH; Molina, GJD. 1988. Selección familial de progenies autofecundadas en una población de maíz (Zea mays L.) de la raza Zapolote Chico. Agrociencia 74:103-114. [ Links ]
Reif, C; Warburton, L; Xia, C; Hoisington, A; Crossa, J. 2006. Grouping of accessions of mexican races of maize revisited with SSR markers. Theoretical and Applied Genetics 113:177-185. [ Links ]
Rincon, F; Johnson, B; Crossa, J; Taba, S. 1996. Cluster analysis, an approach to sampling variability in maize accessions. Maydica 41:307-316. [ Links ]
Romero, PJ; Muñoz, OA. 1996. Patrón varietal y selección de poblaciones de maíz para los sistemas agrícolas en la región de Tierra Caliente. Agrociencia 30:63-73. [ Links ]
Ron, J; Martin, G; Sánchez, J; De la Cruz, L; Morales, M; Carrera, A; Ortega, A; Vidal, A, Guerrero, J. 2008. Caracterización agronómica y morfológica de maíces nativos del noroccidente de México. Rev. Fitotec Mex. 31(4):331-340. [ Links ]
SAGARPA (Secretaría de Agricultura, Ganadería, Desarrollo Rural, Pesca y Alimentación). 2009. Servicio de información y estadística agroalimentaria y pesquera. Producción de maíz en México. p. 28-30. [ Links ]
Sánchez, J; Goodman, M; and Stuber, W. 2000a. Isozymatic and morphological diversity in the races of maize of Mexico. Econ. Bot. 54:43-59. [ Links ]
Sánchez, J; Goodman, M; and Stuber, W. 2000b. Isozymatic diversity of the races of maize of the Americas. Maydica 45: 185-203. [ Links ]
Sánchez, J; and Goodman, M; 1992. Relathionships among the mexican races of maiz. Econ. Bot. 46:72-85. [ Links ]
Santacruz, A; López, G; Muñoz, A; Castillo, T; Córdoba, L; Vaquera, H. 2010. Perfil isoenzimatico de maíces nativos del Istmo de Tehuantepec, Oaxaca, México. Caracterización de grupos. Rev. Fitotecnia Mex. 33(1):1-10. [ Links ]
Vos, P; Hogers, R; Bleeker; M; Reinjans, M; Van de Lee, T; Hornes, M; Frijters, A; Pot, J; Peleman, J; Kuiper, M; Zabeau, M. 1995. AFLP: A new technique for DNA fingerprinting. Nucleic Acids Res. 23:4407-4414. [ Links ]
Wellhausen, J; Roberts, M; Hernández, E. 1952. Races of maiz in Mexico their origin characteristics and distribution. The Bussev Institution of Harvard Univesity. USA. p. 13-44. [ Links ]
Zavala, GF; Salinas, GGE; Piñera, CH FJ. 2010. Conocimiento de la diversidad y distribución actual del maíz nativo en Nuevo León. Informe Final. CONABIO. Universidad Autónoma de Nuevo León, Facultad de Agronomía. 150 p. [ Links ]
*Correspondencia a:
Gilberto Rodríguez-Pérez. Facultad de Agronomía, Universidad Autónoma de Nuevo León, Área de Estudios de Posgrado, Carretera Zuazua-Marín km 17.5, Marín N.L, México. grodriguez263@hotmail.com; francisco.zavala.garcia@gmail.com (Autor para correspondencia); mcgudiez@hotmail.com; eliastrevino_ramirez@hotmail.com
Francisco Zavala-García. Facultad de Agronomía, Universidad Autónoma de Nuevo León, Área de Estudios de Posgrado, Carretera Zuazua-Marín km 17.5, Marín N.L, México. grodriguez263@hotmail.com; francisco.zavala.garcia@gmail.com (Autor para correspondencia); mcgudiez@hotmail.com; eliastrevino_ramirez@hotmail.com
Carmen Ojeda-Zacarías. Facultad de Agronomía, Universidad Autónoma de Nuevo León, Área de Estudios de Posgrado, Carretera Zuazua-Marín km 17.5, Marín N.L, México. grodriguez263@hotmail.com; francisco.zavala.garcia@gmail.com (Autor para correspondencia); mcgudiez@hotmail.com; eliastrevino_ramirez@hotmail.com
Adriana Gutiérrez-Diez. Facultad de Agronomía, Universidad Autónoma de Nuevo León, Área de Estudios de Posgrado, Carretera Zuazua-Marín km 17.5, Marín N.L, México. grodriguez263@hotmail.com; francisco.zavala.garcia@gmail.com (Autor para correspondencia); mcgudiez@hotmail.com; eliastrevino_ramirez@hotmail.com
José Elías Treviño-Ramírez. Facultad de Agronomía, Universidad Autónoma de Nuevo León, Área de Estudios de Posgrado, Carretera Zuazua-Marín km 17.5, Marín N.L, México. grodriguez263@hotmail.com; francisco.zavala.garcia@gmail.com (Autor para correspondencia); mcgudiez@hotmail.com; eliastrevino_ramirez@hotmail.com
Froylan Rincón-Sánchez. Departamento de Fitomejoramiento, Universidad Autónoma Agraria Antonio Narro, Buenavista Saltillo, Coahuila, México. frincon@uaaan. mx
1.Tesis de Doctorado del primer autor. Universidad Autónoma de Nuevo León.
2.Facultad de Agronomía, Universidad Autónoma de Nuevo León, Área de Estudios de Posgrado, Carretera Zuazua-Marín km 17.5, Marín N.L, México. grodriguez263@hotmail.com; francisco.zavala.garcia@gmail.com (Autor para correspondencia); mcgudiez@hotmail.com; eliastrevino_ramirez@hotmail.com
3.Departamento de Fitomejoramiento, Universidad Autónoma Agraria Antonio Narro, Buenavista Saltillo, Coahuila, México. frincon@uaaan. mx
Recibido: 7 de diciembre, 2011. Aceptado: 30 de marzo, 2012.