Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Agronomía Mesoamericana
versão On-line ISSN 2215-3608versão impressa ISSN 1659-1321
Agron. Mesoam vol.21 no.1 San Pedro Jun. 2010
Caracterización molecular de accesiones cultivadas y silvestres de frijol común de Honduras 1
Marcelino Santiago Guachambala-Cando2, Juan Carlos Rosas-Sotomayor2
1 Recibido: 2 de octubre, 2009. Aceptado: 17 mayo, 2010. Trabajo conducido como parte de las actividades de los proyectos de investigación auspiciados por el Programa Dry Grain Pulses CRSP (donación USAID No. EDH-A- 00-07-00005-00). 2 Programa de Investigaciones en Frijol, Escuela Agrícola Panamericana, Zamorano, Honduras. mguachambala@zamorano.edu; jcrosas@zamorano.edu
Resumen
Caracterización molecular de accesiones cultivadas y silvestres de frijol común de Honduras. Para determinar la diversidad genética de las accesiones de Phaseolus que se conservan en el Banco de Germoplasma, un grupo de 69 accesiones de frijol común (Phaseolus vulgaris L.) y de P. coccineus de Honduras, fueron evaluadas con 30 cebadores mediante la técnica de marcadores moleculares RAPD (Polimorfismos de ADN Amplificados al Azar), generándose 341 fragmentos polimórficos de ADN. Las distancias genéticas fueron determinadas según el coeficiente (o índice de similaridad) de Dice y el método UPGMA. Estos análisis moleculares identificaron dos grupos a partir de un coeficiente de Dice (CD) de 0,48, uno perteneciente a P. vulgaris y el otro a P. coccineus. Las accesiones de P. vulgaris, mostraron una división marcada (CD de 0,64) entre las pertenecientes a los reservorios andino y mesoamericano. Por otro lado, las accesiones mesoamericanas se separaron (CD de 0,76) en cuatro grupos, correspondientes a las accesiones silvestres, las variedades de la raza Jalisco, y las variedades mejoradas y criollas de la raza Mesoamérica. Los resultados confirmaron las distancias genéticas esperadas, incluyendo mayor distancia entre las especies de Phaseolus, seguidas de la separación entre las accesiones del reservorio andino y el mesomericano de P. vulgaris. En el reservorio mesoamericano, las dos accesiones de la raza Jalisco presentaron menor similitud que las de la raza Mesoamérica, y dentro de la raza Mesoamérica, los silvestres separados de los cultivados; adicionalmente, las mejoradas relativamente diferentes a las criollas.
Palabras clave: Phaseolus vulgaris, P. coccineus, diversidad genética, marcadores RAPD.
Abstract
Molecular characterization of cultivated and wild accessions of common bean from Honduras. To determinate the genetic diversity of the Phaseolus collection maintained at Zamorano´s Germplasm Bank, 69 accessions of common bean (Phaseolus vulgaris L.) and P. coccineus from Honduras were evaluated with 30 primers using the RAPD (Randomized Amplified Polymorphic DNA) marker technique, generating 341 DNA polymorphic fragments. The study was conducted during 2008-09 in Zamorano; Honduras. The genetic distances were determined by the Dice coefficient (or similarity index) and the UPGMA method. These molecular analyses identified two groups at 0.48 Dice Coefficient (DC), one belonging to the P. vulgaris species and the other to P. coccineus. The group of P. vulgaris accessions shows a clear division (DC of 0.64) between the Andean and the Mesoamerican pools. In the other hand, the Mesoamerican accessions show a separation (DC of 0.76) of four groups corresponding to the wild accessions, the cultivars from the race Jalisco, and the improved and the landrace cultivars from the race Mesoamerica. The results confirmed the expected genetic distances, including a greater distance among the accessions of the two Phaseolus species, followed by a good separation of the accessions from the Andean and Mesoamerican P. vulgaris gene pools. At the Mesoamerican gene pool level, the two accessions from the Jalisco race are less similar from the accessions belonging to the Mesoamerican race; and within the Mesoamerican race, the wild are separated from the cultivated accessions; additionally, in the cultivated the improved are relatively different from the landrace accessions.
Key words: Phaseolus vulgaris, P. coccineus, genetic diversity, RAPD markers.
Introducción
El frijol común (Phaseolus vulgaris L.) es el segundo grano en el consumo alimenticio diario en los países centroamericanos (IICA-Proyecto Red SICTA 2007). En la mayoría de países de América Latina forma parte de alimentos tradicionalmente consumidos principalmente en las zonas rurales y las áreas urbanas pobres (Rosas et al. 2003). La gran diversidad de granos de distintos colores, formas y tamaños, permite la elaboración de una gran diversidad de recetas. El alto contenido de proteínas y minerales como el hierro, hacen del grano de frijol una fuente rica de nutrientes. Un porcentaje importante de las poblaciones rurales en América Latina y África dependen de este cultivo para su sustento diario; por ello, los niveles de autoconsumo de este grano son bastante altos.
El cultivo del frijol común se encuentra ampliamente distribuido geográficamente, y posee una gran adaptación a distintos ecosistemas, debido a su gran diversidad genética representada por tres razas mesoamericanas (Durango, Jalisco y Mesoamérica) y tres razas andinas (Chile, Nueva Granada y Perú) (Singh et al. 1991). Esta diversidad puede ser analizada con marcadores moleculares de ADN. La diversidad genética de los cultivos puede ser entendida de mejor manera usando la variación molecular, química y morfológica. Desde la aparición de las técnicas moleculares como los RFLP (Polimorfismos para Longitudes de Fragmentos de Restricción), RAPD y SCAR (Regiones Amplificadas y Caracterizadas por Secuencia), ha sido posible conocer la diversidad genética de diversos organismos.
Los marcadores RAPD han sido los más usados en análisis de germoplasmas debido a su fortaleza polimórfica (Skroch et al. 1992, Beebe y Pedraza 1998, Beebe et al. 2000). Entre más polimorfismos se identifiquen con la técnica de análisis RAPD, mayor es la capacidad de establecer relaciones genéticas; estos marcadores también permiten identificar QTL (Loci de Carácter Cuantitativo) ligados a la resistencia a enfermedades (Guimaraes et al. 2007). La técnica RAPD ha sido utilizada en estudios de caracterización de la diversidad genética de accesiones de germoplasma de frijol común provenientes de México (Skroch et al. 1998), Chile (Vera et al. 1999), Brasil (Caixeta y Alves 2001, Emygdio et al. 2003), Cuba (Miranda et al. 2006), Centro América (Beebe et al. 1995, Beebe et al. 2000) y El Caribe (Durán et al. 2005), entre otros.
La información genética de las accesiones cultivadas, criollas o mejoradas, o de sus progenitores, permite establecer relaciones filogenéticas; y así obtener información que es de gran utilidad en el mejoramiento de los cultivos. Por ello, se considera importante caracterizar las accesiones cultivadas y silvestres de P. vulgaris y de sus parientes del género Phaseolus que se conservan en el Banco de Germoplasma de Zamorano, con el fin de promover su utilización como cultivares o como progenitores en programas de mejoramiento. La ampliación de la base genética mediante el mejoramiento convencional y la selección asistida con marcadores, es una alternativa para incrementar la estabilidad de las variedades en ambientes diversos como los que se presentan en Centroamérica.
El objetivo de este trabajo fue determinar la diversidad genética del frijol común y sus parientes del género Phaseolus, de la colección hondureña que se conserva en el Banco de Germoplasma de Zamorano, con el fin de planificar su conservación a largo plazo y su uso potencial en actividades de mejoramiento y selección.
Materiales y métodos
Las accesiones de germoplasma utilizadas en este estudio fueron sembradas y analizadas durante el periodo 2008-09, en las instalaciones del Programa de Investigaciones en Frijol (PIF) y el Laboratorio de Biotecnología Aplicada de la Escuela Agrícola Panamericana, Zamorano.
Material genético
Se utilizaron 69 accesiones del género Phaseolus del Banco de Ge rmoplasma, recolectadas durante los períodos 1990-94 y 2001-03 en territorio hondureño, incluyendo varie dades mejoradas desarrolladas en Honduras y de otras regiones. Las accesiones del Banco de Germoplasma fueron elegidas con base en las zonas agroecológicas de procedencia, características agronómicas y comerciales. El germoplasma utilizado en el estudio incluye un total de 64 accesiones de P. vulgaris, constituido por 44 variedades criollas y cinco accesiones silvestres de frijol común del germoplasma hondureño, nueve variedades mejoradas de Honduras, cuatro accesiones andinas, dos accesiones de la raza Jalisco del reservorio mesoamericano, y cinco accesiones de P. coccineus colectadas en Honduras (Cuadro 1). Las 44 accesiones criollas del germoplasma hondureño de frijol común se seleccionaron con base en su procedencia, usando como criterios la altitud de las localidades y que sean provenientes de los depa rtamentos representados en la colección. Las accesiones silvestres y las de P. coccineus, ambas de Honduras, las dos accesiones andinas y las dos de la raza Jalisco, y las variedades mejoradas, se usaron para medir las distancias o diferencias genéticas de las criollas con respecto a éstas.
Siembra
Las accesiones del estudio fueron sembradas en el Invernadero #2, en maceteros individuales conteniendo 4,0 kg de una mezcla de suelo y compost preparado con residuos de plantas y suelo reciclado de invernaderos, en una relación en volumen de 1:1. Las semillas fueron pre-germinadas para garantizar un establecimiento uniforme de las accesiones.
Extracción de ADN
Las muestras de tejido fueron colectadas a los 20 días después de la siembra y la extracción de AND genómico se realizó utilizando el protocolo de Skroch et al. (1998). Se colocaron de cinco a seis primordios de la tercera y cuarta hoja trifolia da en microtubos eppendorf de 1,5 ml, a los cuales se agregó 50 μl de buffer de extracción PEX (etil xantogenato de potasio), y se maceró con pistilos de plástico. A cada muestra macerada se le agregó 450 μl de buffer PEX, seguido de un vortex antes de ser sometidas a 65 °C por una hora en baño maría; luego las muestras fueron centrifugadas a 14 000 revoluciones por minuto (rpm) por 10 minutos para sepa rar el tejido del buffer. El sobrenadante fue transferido a un microtubo limpio de 1,5 ml agregándose 1,0 ml de una solución 6:1 etanol: acetato de amonio 7,5 M, para precipitar los ácidos nucleicos. Se dejó reposar durante 15 minutos y luego fueron centrifugados a 3000 rpm durante 10 minutos para el peletizado de los ácidos nucleicos. El ARN se desnaturalizó media nte una solución de 300 μl de ARNa sa (100 ng/ml) por 60 minutos. Posteriormente, las muestras fueron centrifugadas a 3000 rpm x cinco minutos para colectar los restos de la ARNa sa y del ARN degradado. El sobrenadante fue transferido a un microtubo limpio y después se precipi tó el AND con una solución 3M de etanol: acetato de sodio 10:1 por 15 minutos y se peletizó centrifugando a 3000 rpm por cinco minutos. Una vez peletizada la muestra, se procedió a clarificar el ADN dos veces, usando una solución de etanol al 70%, centrifugando a 14 000 rpm durante 15 segundos, para luego dejar secar las muestras por 13 h. Los pellets secos fueron rehidratados con 100 μl de buffer TE 0,1X en baρo maría a 65 °C por 15 minutos.
Cuantificación y dilución de ADN
Para cuantificar las muestras de ADN se usó un fluorómetro Hoefer DyNA QuantTM 200. El primer paso fue calibrar el fluorómetro usando ADN estándar extraído de timo de bovino a 100 ng/ml. El buffer usado en la cuantificación fue TNE (100 ml TNE 1X y 10 μl de soluciσn de tintura Dye). Una vez obtenido el dato de cuantificación, se procedió a diluir las muestras en 100 μl de buffer TE, hasta obtener una soluciσn de ADN a una concentración de 10 ng/ml usando la fórmula Vi=1000/(Ci-10), donde Vi=volumen inicial y Ci= concentración inicial.
PCR con los cebadores RAPD
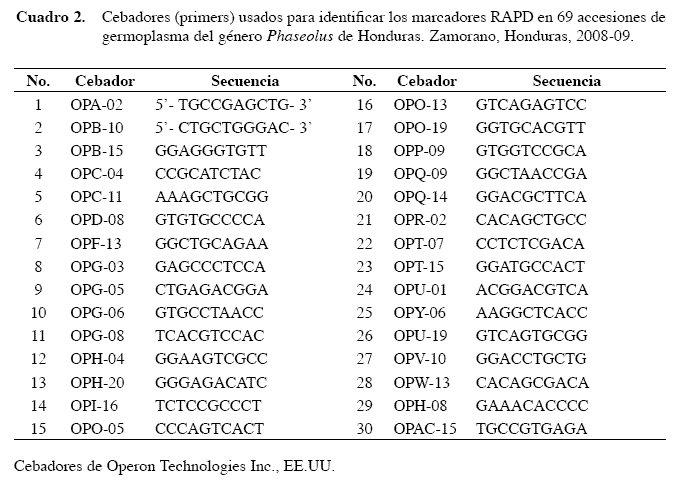
Las muestras de ADN de cada genotipo fueron amplificadas usando 30 cebadores (primers) RAPD de Operon Technologie s, Inc. (Cuadro 2). Las reacciones de amplificación fueron hechas bajo las siguientes condiciones: 3,4 μl de H2O, 1,5 μl buffer green 5X de PCR con MgCl2 (2 mM), 1,5 μl de dNTP (4 mM), 0,6 μl del primer (10 μg/ml), 0,5 μl de Taq Polimerasa GoTaq® Flexi DNA Polymerase, Promega (5 u/μl) y 7,5 μl de ADN (10 ng/ml), para un total de 15 μl por reacciσn. La PCR fue realizada en un termociclador TEC HNE TC- 512, bajo el siguiente perfil térmico: desnaturalización inicia l a 95 °C durante un minuto, 35 ciclos de desnaturalización a 94 °C por 45 segundos, acoplamiento a 35 °C por 30 segundos, extensión a 72º C durante un minuto 15 segundos, una extensión al final de los 35 ciclos a 72 °C por cinco minutos, y mantenimiento y enfriado a 15 ºC por tiempo indefinido.
Electroforesis
Se utilizaron 15 μl de producto de PCR en bandejas con 40 espacios por fila y 10 μl de escalera molecular de 100 pb (100bp DNA ladder ® Promega) diluida al 30% en buffer TE al 0,1X, la cual fue colocada al inicio y al final de las filas para de esa manera poder establecer el tamaño de los polimorfismos amplificados en PCR.
Para la separación de los fragmentos amplificados en PCR se usó un gel de agarosa al 1,2% con buffer TBE 0.5X, en bandejas horizontales Maxicell EC 360M, con un generador de corriente PS 250/2,5 AMP, a 95 V por 90 minutos. Después de la corrida electroforética los geles fueron teñidos durante 10 minutos en una solución de bromuro de etidio (100 mg/ml), y luego lavados en 500 ml de agua destilada, para eliminar el exceso de bromuro, durante 10 minutos. Para capturar las imágenes se usó el sistema EDAS 290 de Kodak, lográndose imágenes claras para la elaboración de las matrices. Las bandas tomadas en cuenta para la generación de las matrices fueron las de mediana a alta intensidad y claridad; las bandas ubicadas en medio de las unidades de medida de la escalera se tomaron como 0,5 del valor de las bandas entre las que se encontraban.
Se utilizaron 30 cebadores previamente empleados en otras caracterizaciones de germoplasma de frijol común mediante la técnica RAPD (Skroch et al. 1998, Beebe et al. 2000). La mayoría de marcadores RAPD generaron un buen número de polimorfismos (Figura 1), los cuales permitieron los análisis para determinar las relaciones genéticas entre los genotipos estudiados. Para la caracterización molecular se usó una matriz tipo 1 con 69 columnas por 341 filas, considerando los valores 1 para la presencia del alelo y 0 para la ausencia del alelo. Para establecer las distancias genéticas de las accesiones se calculó el coeficiente o índice de similaridad de Dice (1945), utilizado para correlaciones de eventos discretos.
La matriz de distancias genéticas se analizó por el método UPGMA (siglas en inglés de Unweighted Pair Group Method with Arithmetic Mean). Se elaboró un dendrograma con los datos analizados utilizando el paquete NTSYSpc 2.1.
Resultados y discusión
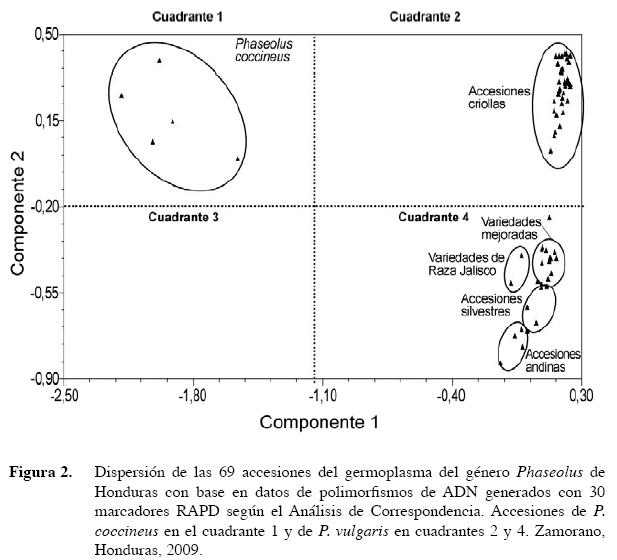
El análisis molecular de las accesiones de Phaseolus mediante la técnica RA PD usando 30 cebadores, permitió generar 341 fragmentos polimórficos de ADN cuya información fue utilizada en los análisis de agrupamiento por el método UPGMA. Según el análisis de correspondencia, considerando los dos primeros componentes, las accesiones se distribuyen en varios grupos de acuerdo al grado de similitud (o distancia) genética, incluyendo al grupo de las accesiones de P. coccineus en el cuadrante 1 superior izquierdo, muy distante de las accesiones de frijol común (P. vulgaris), ubicados en los cuadrantes 2 y 4 (Figura 2). La mayoría de las variedades criollas de Honduras se agruparon en el cuadrante 2, parte superior derecha de la gráfica. Por otro lado, en el cuadrante 4, inferior derecho, se observan relativamente diferenciados los grupos de las accesiones andinas y silvestres. Las variedades mejoradas se ubican hacia la parte superior de este cuarto cuadrante; mientras que, las dos variedades mesoamericanas de la raza Jalisco se separan ligeramente de las mejoradas hacia la izquierda de este cuadrante 4. Estos resultados sugieren las marcadas diferencias genéticas entre las especies P. vulgaris y P. coccineus, seguidas de las diferencias genética entre las accesiones andinas y mesoamericanas de P. vulgaris, como se ha determinado en previos estudios (Skroch et al. 1992, Singh et al. 1991).
En general, los grupos están claramente definidos, de acuerdo con su mayor o menor similaridad en su composición genética (Figura 2). Las variedades mejoradas liberadas en los últimos 15 años en Honduras, se encuentran más cerca de las accesiones andinas y de la raza Jalisco, así como de las silvestres, y todas estas ubicadas en el cuadrante 4; mientras que, la mayoría de las accesiones criollas (38 de un total de 44, es decir el 86,4%) se ubican como un grupo separado en el cuadrante 2. De las seis accesiones criollas restantes (13,6%), cinco se agrupan con las variedades mejoradas en el cuadrante 4, exceptuando a la variedad Arbolito Vaina Blanca. Esta distancia sugiere la importancia en la contribución potencial de estas accesiones criollas al mejoramiento del frijol común en Honduras; ya que los resultados indican marcadas diferencias genéticas con las variedades mejoradas actualmente disponibles, lo cual aparentemente se ha debido al limitado uso de la diversidad genética de las accesiones criollas de Honduras en el mejoramiento por cruzamiento y selección de este importante cultivo.
En el dendograma generado mediante el análisis de agrupamiento del método UPGMA se observan grupos de accesiones bien diferenciados (Figura 3). El primer grupo incluye las cinco accesiones de P. coccineus (F0569, F0571, F0583, F0630 y F0661), que se separan de las accesiones de P. vulgaris a partir de coeficiente de Dice (CD) o índice de similaridad de 0,48, lo cual indica una distancia genética significativa de las accesiones de esta especie con las demás accesiones cultivadas y silvestres de frijol común (P. vulgaris). Sin embargo, la similaridad genética existente entre estas dos especies explica su relación cercana dentro del reservorio genético del género Phaseolus, lo cual explica la factibilidad de la utilización de accesiones de P. coccineus en programas de mejoramiento de P. vulgaris (Beaver et al. 2003). Las accesiones de P. vulgaris presentan varios grupos factibles de identificar por su procedencia u origen. En primera instancia las cuatro accesiones andinas (A) de P. vulgaris forman parte de un grupo separado de las accesiones Mesoamericanas (M) a partir de índice de Dice de 0,64. A su vez, el grupo de las andinas se separa en dos subgrupos relacionados (índice de Dice de 0,78), el primero conformado por AFR 180 y Pompadour J (línea mejorada tipo rojo arriñonada y variedad criolla rojo moteado de la República Dominicana, respectivamente), y el segundo por G05686 y G06727 (ambas accesiones andinas de la raza Nueva Granada son resistentes a la mancha angular).
En las accesiones mesoamericanas se distinguen las agrupaciones siguientes. En las cinco accesiones silvestres de P. vulgaris provenientes del germoplasma de Honduras (M1), se presenta dos bifurcaciones a un CDice de 0,77, es decir con un buen grado de similitud genética pero incluyendo un nivel de diferenciación entre ellas. El siguiente grupo (M2) incluye las accesiones de la raza Jalisco Great Northern 31 y MAM 38, las cuales muestran una similitud genética (CD) de 0,79 entre ellas.
El grupo de las variedades mejoradas de Honduras (M3), está conformando por variedades liberadas en los últimos 20 años, en las que los resultados sugieren una similitud genética entre ellas que las diferencia de las variedades criollas. Este grupo se separa de la variedad criolla Chile de la región de Atlántida (F0419) a un CD de 0,77; y del grupo que incluye a cinco variedades criollas (M4), conformado por Frijol Mantequilla (F0518), Liberal (F0505), Rojo (F0428), Arbolito Vaina Roja (F0155) y Arbolito Vaina Blanca (F0405), procedentes de los departamentos de Atlántida, Olancho y Francisco Morazán de Honduras, a un CD de 0,81.
El grupo que contiene la mayoría de las accesiones criollas de frijol común (M5), incluye variedades criollas de varios departamentos de Honduras, por lo cual requiere una mayor explicación. Este grupo M5 se separa en dos subgrupos; en el primero, se observa la separación de la accesión Gringo (F0245) de Ocotepeque, un frijol de altura tipo habichuela, del resto de las accesiones (Figura 3).
En el grupo que incluye el resto de accesiones criollas, se encuentran un pequeño subgrupo de siete accesiones criollas identificado como C1, compuesto por Seda (F0332), Pronto a la Olla (F0326), Vaina Blanca (F0321), Arbolito (F0264), Negro (F0253), Arbolito Rojo (F0218) y Alilí (F0211). Las accesiones Talete de Lempira (F0201), Talete de Guía (F0190), Cincuenteño (FF0169) y Liberalito (F0162), constituyen el segundo subgrupo de criollas (C2). El tercer subgrupo (C3) está conformado por las accesiones criollas Arbolito Vaina Roja (F0155), Milpero (F0134), Talete de Intibucá (F0130), Chile de Copán (FF0147), Negro Ligero (F00126), Danlí (F0117), Frijol Ñato (F0107), Retinto Vaina Blanca (F0104), Sangre de Toro (F0102), Arbolito ((F0097) y Cuarenteño (F0091).
El cuarto subgrupo (C4) lo constituyen las accesiones criollas Chelito (F0085), Re tinto Rojo (F0080), Frijol Rojo (F0079), Frijol Arbolito (F0077), Desarrural (F0072), Vaina Rosada (F0057), Frijol de Montaña (F0047), Gualiqueme (F0034), Chilito (F0030), Marcialeño (F0026) y Cuarenteño de Olancho (F0022). Por otro lado, La accesión criolla Concha Blanca (F0007) de Yoro, se separa de las accesiones criollas mencionadas anteriormente a un CD de 0,80. Al observar las divisiones de estas accesiones criollas, que en su mayoría se presenta a un CD de 0,85, se puede decir que representan agrupaciones de la gran diversidad genética existente de acuerdo a su distribución en las diferentes regiones de Honduras.
En la Figura 3, se observan dos situaciones contrastantes en cuanto a la similitud genética y sus lugares de recolección. El primero es el caso de las accesiones Frijol Ñato (F0107) y Danlí (F0117) en el subgrupo C3, colectados en comunidades de Comayagua muy cercanas, y que son muy similares (CD de 0,95). Pero por otro lado, las accesiones Arbolito (F0264) y Vaina Blanca (F0321) en el subgrupo C1, también presentan alta similitud genética (CD de 0,96), pero provienen de diferentes departamentos, Ocotepeque y Santa Bárbara, respectivamente. La alta similitud genética en ambos casos, sugiere de que se puede tratar del mismo genotipo o que estos provienen de un ancestro en común. En el segundo caso, se observa efectos de la dispersión y adaptación de estas accesiones a diferentes condiciones.
El germoplasma hondureño evaluado muestra un gran potencial para el mejoramiento genético de frijol común. Algunas de las accesiones criollas estudiadas han sido incluidas en los últimos años en programas de mejoramiento y ampliación de la base genética del frijol común para Centro América. Las accesiones de frijol silvestre y las de P. coccineus representan una fuente de diversidad adicional, siendo las primeras de mayor potencial por su mayor compatibilidad con las formas cultivadas de P. vulgaris.
Teniendo en cuenta los resultados obtenidos en este estudio de caracterización de germoplasma de frijol utilizando la técnica RAPD, y los de estudios anteriores con esta técnica (Skroch et al. 1998, Beebe et al. 2000, Miranda et al. 2006), se recomienda continuar con la caracterización de accesiones de la colección de Honduras, incluyendo un mayor número de accesiones silvestres y de P. coccineus. Adicionalmente, se sugiere ampliar este trabajo incluyendo accesiones de El Salvador y Nicaragua, para determinar si la diversidad genética utilizada por los agricultores en estos países está muy relacionada o es especialmente diversa con relación a la procedente de Honduras; con el fin de promover la recolección, conservación y utilización de la diversidad genética de P. vulgaris y sus parientes existentes en el ámbito regional.
Literatura citada
Beaver, JS; Rosas, JC; Myers, J; Acosta, J; Kelly, JD; Nchimbi- msolla, S; Misangu, R; Bokosi, J; Temple, S; Arnaud-Santana, E; Coyne, DP. 2003. Contributions of the Bean/Cowpea CRSP to cultivar and germplasm development in common bean. Field Crops Research 82:87-102. [ Links ]
Beebe, S; Ochoa, I; Skroch, PW; Nienhuis, J; Tivang, J. 1995. Genetic diversity among common bean breeding lines developed for Central America. Crop Science 35(4):1178-1183. [ Links ]
Beebe, S; Pedraza, F. 1998. Perspectivas para el uso de marcadores moleculares en el mejoramiento del frijol. In: R. Lépiz (Comp.). Taller Internacional de Mejoramiento Genético de Frijol Negro Mesoamericano. PROFRIJOL, Veracruz, México. p. 132-139. [ Links ]
Beebe, S; Skroch, PW; Thome, J; Duque, MC; Pedraza, F; Nienhuis, J. 2000. Structure of genetic diversity among common bean landraces of Middle American origin based on correspondance analysis of RAPD. Crop Science 40(1):264-273. [ Links ]
Caixeta, M; Alves. 2001. Caracterizacao da diversidad e genética em feijao por meio de marcadores RA PD. Pesquisa Agropecuaria Brasileira, Brasilia 36(2):381-385. [ Links ]
Dice, LR. 1945. Measures of the amount of ecological association between species. Ecology 26:297-302. [ Links ]
Duran, LA; Blair, MW; Gi raldo, Z; Ma chia velli, RE ; Prophete, E; Nin, JC; Beaver, JS. 2005. Morphological and molecular characterization of common bean landraces and cultivars from the Caribbean. Crop Science 45(4):1320-1328. [ Links ]
Emygdio, B; Antunes, IF; Choer, E; Nedel, JL. 2003. Eficiencia de coeficientes de similaridade em genotipos de feijao mediante marcadores RAPD. Pesquisa Agropecuaria Brasileira. Brasilia 38(2):243-250. [ Links ]
Guimaraes, E; Ruane, J; Scherf, B; Sonnino, A; Dargie, J. 2007. Marker-assisted selection, current status and future perspectives in crops, livestock, forestry and fish. Food and Agricultural Organization of the United Nations, FAO. Roma, Italia. 471 p. [ Links ]
IICA (Instituto Interamericano de Cooperación para la Agricultura) - Proyecto Red SICTA (Sistema de Integración Centroamericano de Tecnología Agrícola). 2007.
Mapeo de las cadenas agroalimentarias de maíz blanco y frijol en Centroamérica. Proyecto Red SICTA; IICA/ COSUDE. INPASA. Managua. 132 p.
Miranda, S; Rosas, JC; Aranda, LL; Ortíz, R; Ponce, M; Ríos, H. 2006, Análisis molecular de la diversidad genética de frijol común manejada por campesinos de Cuba. Agronomía Mesoamericana 17(3):369-382. [ Links ]
Rosas, JC; Gallardo, O; Jiménez, J. 2003. Mejoramiento del frijol común mediante enfoques participativos en Honduras. Agronomía Mesoamericana 14(1):1-9. [ Links ]
Singh, S; Gepts, P; Debouk, D. 1991. Races of common bean (Phaseolus vulgaris, Fabaceae). Economic Botany 45:379-396. [ Links ]
Skroch, P; Tivang, J; Nienhuis, J. 1992. Analysis of genetics relationships using RAPD marker data. Joint Plant Breeding Symposium Series. Minneapolis, Minnesota, USA. p. 26-30. [ Links ]
Skroch, P; Nienhuis, J; Beebe, S; Tohme, J; Pedraza, F. 1998. Comparison of Mexican common bean (Phaseolus vulgaris L.) core and reserve germplasm collection. Crop Science 38(2):488-496. [ Links ]
Vera, CM ; Paredes, MC ; Becerra, VV. 1999. Estudio comparativo de diversidad morfológica, isoenzimática y RAPDs dentro y entre clases comercia les de fréjol chileno (Phaseolus vulgaris L.). Agricultura Técnica 59(4):247-259. [ Links ]