Introducción
En el desarrollo de la biología molecular y de las ciencias básicas de la salud, en general, ha tenido mucho que ver la aplicación de los conocimientos tecnológicos en el campo del análisis clínico. La posibilidad de detectar, monitorear y cuantificar estructuras subcelulares ha desarrollado el diagnóstico de patologías y propiciado la adecuación de los tratamiento terapéuticos.
Hoy día, se conoce que los estados de salud y enfermedad tienen una base molecular y en su subyacente se encuentra una herramienta interdisciplinaria: la bioquímica. Todo criterio clínico acertado tiene una justificación bioquímica.
Precisamente, sobre este aspecto trata esta revisión, ¿cómo podemos mejorar nuestro enfoque clínico de los problemas de salud utilizando técnicas que nos digan qué está pasando realmente en los tejidos enfermos?
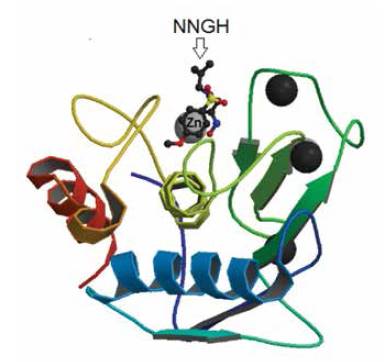
La estructura molecular que aparece en la presentación de este trabajo es una metaloproteinasa de la matriz, la MMP 12. Para conocer sobre este tipo de enzimas, la función que realiza y cómo se relaciona con dos eventos antagónicos: el crecimiento y diferenciación celular que es un dominio de vida, y la metástasis del cáncer, que es dominio de muerte, fue necesario mucho esfuerzo de investigación.
Método
La información sobre el uso de técnicas para el diagnóstico clínico, con independencia del tipo de muestra de que se trate, es realmente abrumadora; en ella se refleja el esfuerzo de toda la comunidad científica; es la historia de cómo curar y preservar la salud; un anhelo que siempre ha impulsado a la mente humana. En esta revisión se emplearon las siguientes herramientas de búsqueda de artículos científicos.
NCBI (National Centerfor Biotechnology Information). Disponible en: http://www.ncbi.nlm.nih.gov/.
• NLM (National Library of Medicine). Disponible en: http://dtd.nlm.nih.gov/.
• EBSCO Health. Disponible en: https://health.ebsco.com/?_ga=1.46795623.1501374684.1472540416.
• Pubmed. Disponible en: www.ncbi.nlm.nih.gov/pubmed.
• IIB-INTECH (Instituto de Investigaciones biotecnológicas-Instituto Tecnológico Chascomús). Disponible en: http://www.iib.unsam.edu.ar/web/papiros.php?a=
• PDB. Banco de Datos de Proteínas:Disponible en: www.rcsb.org/pdb/
• Base de datos BRENDA (Instituto de Bioquímica de la Universidad de Colonia, Alemania). Disponible en: www.brenda.uni-koeln.de
• NIH US National Library of Medicine. Disponible en: https://www.nlm.nih.gov/
Desarrollo
Las células nucleadas constituyen la fuente para obtener el ADN genómico y muchos de los protocolos utilizan la sangre periférica, en particular los leucocitos por ser los eritrocitos y plaquetas células sin núcleo. En estos casos, se aplica la venopunción que resulta dolorosa, invasiva y riesgosa para pacientes y personal médico. En ocasiones este método no es factible, como en personas de avanzada edad (inmunosuprimidos, en estado caquéxico, etc.), pacientes obesos o sometidos a quimioterapia que inutiliza las vías venosas, y en neonatos y prematuros con baja tolerancia a la punción. (Barriga, 2016)
Otro aspecto que no favorece la extracción de sangre como fuente de ADN es que requiere de condiciones especiales de conservación y transporte.
Además puede contaminarse con el hierro hemoglobínico de los eritrocitos; el cual es un inhibidor de la ADN polimerasa, enzima empleada en la reacción en cadena de la polimerasa (PCR), lo cual afecta el análisis posterior del ADN. (Hernández, 2012)
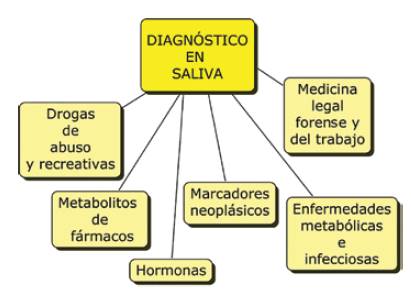
En poblaciones con características geográficas, étnicas y socioculturales semejantes, del mismo sexo y rango de edades se presentan diferencias interindividuales en la respuesta a drogas medicamentosas. Estas diferencias tienen una base genética y son identificadas por la farmacogenética, ciencia de la medicina personalizada. El conocimiento de la composición genética de los pacientes permite establecer qué prescripción de medicamentos es segura y cuáles pueden resultar ineficaces o dar lugar a efectos no deseados. Varias de estas pruebas de fenotipado se realizan en muestras de saliva y permiten minimizar los errores en la prescripción y ajuste de dosis.
También resulta evidente que estas pruebas reducen los costos de salud asociados a las intoxicaciones con fármacos. (Gutiérrez, 2004)
Los estudios genéticos inician con la recolección de ADN y en los laboratorios de Genómica y Proteómica de la Universidad de Pittsburgh han desarrollado la tecnología ORAGENE.ONE, que permite colectar muestras de saliva en tubos plásticos que contienen un reactivo que estabiliza el ADN y lo conserva para su transporte a temperatura ambiente. Se ha comprobado que para ensayos genéticos el ADN de la saliva es equivalente al ADN de muestras de sangre, con la ventaja de que la recolección de muestras es indolora y no invasiva. (Abraham, 2012)
Hoy día, la saliva se considera una matriz con amplias posibilidades para ser utilizada en investigación y diagnóstico en campos como la odontología, genética, farmacología, epidemiología, entre otros.
Esto por la presencia de ADN genómico y biomarcadores de patologías orales y sistémicas. La dad en su colección, conservación y transporte la convierte en un sustrato muy conveniente para estudios poblacionales en gran escala.
En países como Canadá se emplea para realizar prescripciones medicamentosas individualizadas en el tratamiento de enfermedades psiquiátricas.
Esta aplicación se basa en la utilización del ADN genómico de la saliva en estudios de polimorfismo de nucleótido simple (SNP); que permiten establecer las variaciones en genes que expresan enzimas del tipo citocromo P-450 (CYP-450). Estas enzimas intervienen en la Fase I del metabolismo hepático de antidepresivos y antipsicóticos. (Hasan, 2016)
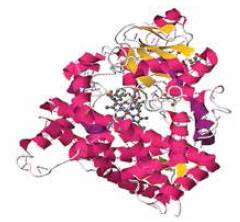
Figura 2 CYP3A4. Participa en el metabolismo de más de 40 fármacos. Estructura cristalográfica en PDB.
Los polimorfismos del tipo SNP pueden dar lugar a tres tipos de afectaciones.
• Aumento en la tasa metabólica, con lo cual una dosis normal del medicamento se torna ineficaz.
• Disminución en la capacidad del metabolismo de la enzima, lo que puede originar un incremento en la fracción libre del fármaco y efectos adversos.
• La no expresión de la enzima, lo que provoca que la persona no pueda metabolizar el fármaco, lo que genera intoxicación aguda.
El polimorfismo genético es una característica monogénica, solo afecta a un gen, y se puede presentar con una frecuencia que afecta entre 1 y 2 % de una población.
Esto significa que este tipo de variación en la secuencia de ADN entre individuos no es rara y tiene una elevada incidencia en poblaciones sometidas a tratamiento médico. (Roma, 2016)
Los estudios genéticos de pacientes psiquiátricos o con trastornos de conducta, que se realizan en Canadá, utilizando muestras de saliva, han permitido establecer el tipo de ansiolítico o anti-psicótico que le funciona a la persona o le puede ocasionar fracaso terapéutico. Literalmente, esta práctica está convirtiendo la medicina personalizada en una realidad. (Bahlo, 2010).
Análisis genético en saliva
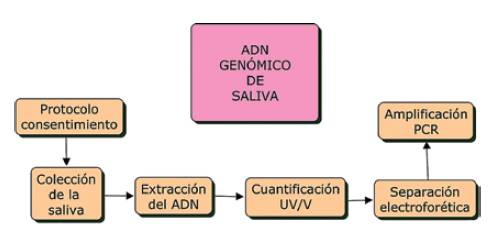
El manejo de muestras biológicas con fines de estudios genéticos se realiza con apego a legislaciones vigentes supervisadas por comités de ética. Implica consentimiento de pacientes y condiciones por las que se rigen los procedimientos.
Aplican protocolos de muestreo, conservación y transporte al lugar de análisis. La no contaminación del ADN extraído puede establecerse, en primera opción, con un espectrofotómetros UV/V. A 280 nm se comprueba contaminación por proteínas y a 230 nm, por compuestos inorgánicos. La señal de ADN se reporta a 260 nm con la presencia de un solo pico espectroscópico.
Un criterio de la calidad del ADN es la razón A260/A280 entre 1,8-2,0.
El empleo de la electroforesis en gel tiene como objeto mejorar la pureza del ADN, verificar que no se han producido procesos de degradación y determinar el tamaño de fracción con el uso de marcadores de peso molecular. Por último, se verifica la funcionalidad del ADN con la reacción en cadena de la polimerasa (PCR), que permite obtener, en pocas horas, una gran cantidad de copias de una secuencia de ADN previamente seleccionada.
La amplificación permite crear bancos de ADN para estudios epidemiológicos, forenses, de paternidad, fármacogenéticos, etc.
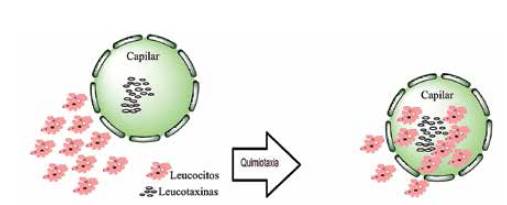
La fuente de ADN en las muestras de saliva no proviene, en buena medida, de las células del epitelio bucal como se podría suponer; ya que se ha confirmado que, aproximadamente, el 74% del ADN de las muestras se obtiene de leucocitos, que se transfieren del plasma a la saliva mediante la red capilar que rodea a los acinos de las glándulas salivales. Un factor que propicia la entrada de los leucocitos es la leucotaxina salival, una sustancia que aumenta la permeabilidad capilar favoreciendo la diapédesis. Esta sustancia también se encuentra en los exudados inflamatorios junto con otros mediadores de la respuesta inmune inespecífica como bradicinina y prostaglandinas. (James, 2011)
La leucotaxina es un péptico que se forma por hidrólisis enzimática de la albúmina, que es la principal fracción de globinas de la sangre, y constituye alrededor del 60% de las proteínas plasmáticas, aunque el porcentaje de la fracción varía de acuerdo con la técnica electroforética empleada. (SEBIA, 2015).
A pesar del elevado tamaño de la albúmina, peso molecular de 65 000 y 69 000 Da, en el plasma solo se encuentra el 40 % de esta proteína, que tiene una amplia distribución extravascular que la expone a la acción de proteasas responsables de la formación de la leucotaxina. El poder quimiotáctico de este péptido se puede comprobar en un sencillo experimento. (Doménech, 2013)
Al colocar una suspensión de leucocitos en plasma cerca de un capilar que contiene al péptido los leucocitos migran al capilar, al cabo de cierto tiempo.
Histolizados
Es un término empleado para designar hidrolizados enzimáticos provenientes de tejidos, órganos y glándulas que contienen péptidos de bajo peso molecular con actividad fisiológica; un ejemplo es la leche materna, que provee a los lactantes de esos péptidos y beneficia el desarrollo del sistema inmunitario. En forma experimental se han obtenido resultados en la regulación de la respuesta inmunitaria en ratones alimentados con hidrolizados de lactoalbúmina. (Baró, 2001)
Hay varios puntos de coincidencia entre la leche materna y la saliva, pues ambas contienen:
• Proteínas de defensa como lisozimas, hidrolasa que rompe enlaces glucosídicos de las paredes bacterianas; lactoferrina, sintetizada por neutrófilos y células acinares, que es un ligando de hierro libre con acción antimicrobiana; transferrina, otra globina fijadora de hierro Fe+3 de la dieta, que al fijarse a receptores de superficies celulares experimenta endocitosis; anticuerpos del tipo IgA, muy abundante en la leche materna y en menor medida en la saliva.
• Células blancas provenientes del plasma sanguíneo, que les confieren propiedades inmunitarias.
• Ambos fluidos tienen un pH promedio menor al plasma sanguíneo. En el caso de la leche materna alrededor de 6,6 y la saliva entre 6,5-7,5; en la no estimulada, aunque el pH de la saliva estimulada puede ser un poco mayor por incremento en la secreción de HCO3.
• Mediante ambos fluidos se excretan fármacos, preferentemente bases débiles, por mecanismo de difusión pasiva.
• Tanto las células de los acinos de glándulas salivales como en las glándulas mamarias se expresan transportadores de iones ATP dependientes mediante los cuales se regulan los niveles de iones como Na+, K+, Cl- y HCO3-. (Mathews, 2013)
En cierta medida, estas coincidencias permiten considerar a la saliva como una especie de histolizado, con péptidos fisiológicamente activos al igual que la leche materna.
Enfermedad periodontal
Un factor importante en las defensas de la saliva en contra de la enfermedad periodontal es la lactoferrina secretada por los gránulos de neutrófilos, Este péptido tiene actividad comprobada in vitro sobre bacterias a concentraciones fisiológicas de la cavidad oral; entre las que se cuentan Aggregatibactes actinomycetemcomitans, Porphyromonas gingivalis, Prevotella intermedia y Prevotella nigrescens. (Castrillón, 2011). La presencia en la saliva y el fluido crevicular de enzimas como elastasa, colagenasa, gelatinasa, proteinasa y la lactato deshidrogenasa (LDH), constituyen biomarcadores de la enfermedad periodontal. Estos péptidos pueden ser detectados en muestras de saliva, mediante la cromatografía líquida de alta resolución (HPLC), la espectrometría de masa y con técnicas de imunoensayo y fluorescencia. (Todorovic, 2006).
Para la detección de biomarcadores en la saliva se requieren técnicas muy sensibles, pues sus niveles son más bajos que en el plasma y la utilidad de esos ensayos consiste en comprobar que tienen buena correlación con los resultados obtenidos en sangre. Una vez comprobado esto, la saliva es la mejor opción; por cuanto es una técnica no invasiva que permite obtener un gran número de muestras en pacientes pediátricos, geriátricos y ambulatorios. A esto se une que el ADN de saliva tiene concentración, fragmentación, tamaño y funcionalidad comparable al de células sanguíneas, que es el referente para fenotipado. (Cajaraville, 2003)
Los niveles de proteínas en saliva son del orden de 200 mg/ml, lo que constituye, aproximadamente, 3% de la concentración plasmáticas de proteínas. Esto se debe a la baja incidencia de los mecanismos de transporte de proteínas plasmáticas hacia la saliva, que se desarrolla preferentemente por difusión pasiva, como es el caso de la albúmina sérica, que se encuentra a muy bajas concentraciones en saliva. Por esta razón, los fármacos, hormonas y metabolitos lipofílicos, que requieren de proteínas para su transporte plasmático, se detectan en su forma libre a muy bajas concentraciones en la saliva. (Hernández, 2012)
Matriz extracelular
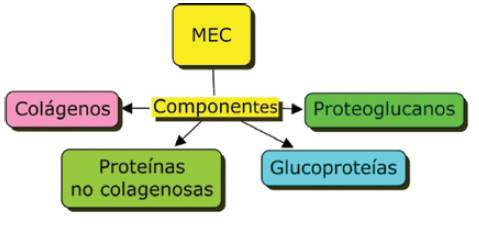
La matriz extracelular (MEC) constituye una compleja red de macromoléculas relacionadas con la proliferación, diferenciación, migración e interacciones celulares, que proporciona una armazón estructural a los tejidos, y forma un lecho gelatinoso donde medran las células. Los cambios en su composición y disposición se asocian con enfermedades como cáncer, artritis, aterosclerosis y fibrosis.
Los colágenos constituyen, aproximadamente, el 30% de la masa proteínica del cuerpo y se reportan más de 20 tipos, y son las principales proteínas de la MEC. Las proteínas no colagenosas contemplan: elastina, confiere elasticidad a los tejidos extensibles y de soporte, como el ligamento periodontal; fibronectina, implicada en la adhesión, migración y morfología celulares y lamininas que participan en el anclaje de las células y componentes de la MEC a la lámina basal.
Las glucoproteínas tienen un polipéptido unido a oligosacáridos (glucanos) de hasta veinte unidades, ramificados, sin repeticiones de secuencias en su estructura; un ejemplo es la mucina salival. Son macromoléculas de la superficie de membranas plasmáticas y sus glucanos estabilizan a la proteína, protegiéndola de proteólisis y aumentando su solubilidad. Actúan como señales de reconocimiento para el transporte y la comunicación celular.
Los proteoglucanos unen glucanos de alto peso molecular, lineales (no ramificados) con unidades de disacáridos que se repiten. Son macro-estructuras de soporte para el tejido conectivo y el cartílago.
Todos los componentes de la MEC son secretados por células del tejido conectivo como fibroblastos, condrocitos y células epiteliales. (Murray, 2013)
La MEC tiene una función de contención que mantiene a las células en su lugar y evita la entrada de moléculas y partículas extrañas a los tejidos. La proliferación de infecciones o la metástasis de células malignas se desarrolla con alteración concomitante de la integridad de esa matriz. Este hecho explica la aparición de sus componentes en la saliva o en el líquido crevicular durante el daño periodontal o desarrollo de cáncer oral.
En el crecimiento y diferenciación celular se producen remodelaciones en la MEC por metaloproteinasas de la matriz (MMPs), que al degradar las proteínas de la MEC permiten la migración celular. Estos cambios en la matriz son necesarios, pero deben ser regulados por procesos de activación-inhibición complejos; cuando estos procesos se alteran la MEC pierde su función de contención lo que sucede en ciertas formas de cáncer y arteriosclerosis. (Stryer, 2015)
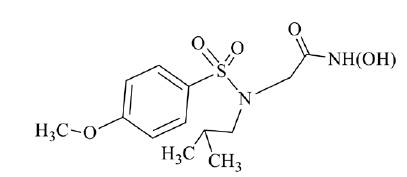
En el control de las MMPs resulta crítica la participación de señales inhibidoras de estas proteasas como es el caso del NNGH, ácido Nisobutil-N-(4-metoxifenilsulfonil)-glicilhidroxámico. Esta molécula es un inhibidor de amplio espectro que puede unirse al sitio activo devarias MMPs inhibiendo a la proteasa.
La utilización de esta sustancia en la inhibición de la MMP 3 suprimió de forma significativa la expresión de la enzima iNOS, óxido nítrico sintetasa inducible, que produce óxido nítrico a partir de L-arginina, una especie reactiva de nitrógeno y oxígeno (ERNOS) de la respuesta inmunitaria inespecífica; así como de las citoquinas pro-inflamatorias; también se redujo la actividad de NFκB, factor activador de la transcripción de genes para síntesis de citoquinasas. La inhibición de la MMP 3 también disminuyó la actividad en la microglia de MAPK, proteincinasa activada por mitógeno.
Estas supresiones confirman una función importante de la MMP 3 en la neuroinflamación. (Christianson, 2012)
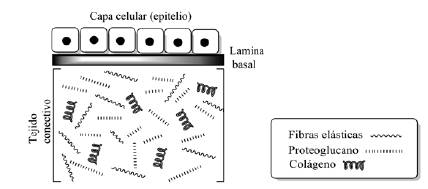
Figura 6 Esquema del tejido conectivo de la matriz extracelular subyacente a la lámina basal que sustenta la capa epitelial
Técnicas para detectar mmps
En relación con la presencia de las MMPs en muestras biológicas, hay más de 20 tipos diferentes, y es evidencia de daño en la MEC y existen técnicas de laboratorio que permiten su detección.
• Zimografía en gelatina. Es una técnica electroforética para separar enzimas en condiciones suaves, sin agentes reductores, a fin de no afectar su actividad. Una vez concentrada la enzima por electroforesis se determina su actividad sobre una matriz de gelatina (colágeno hidrolizado). Después de un tiempo de incubación donde la enzima degrada a la gelatina, el revelado con una tinción para proteína muestra una zona no coloreada donde se fijó la enzima. Está técnica permite determinar la presencia de proteasas que destruyen la MEC. (Novoa-Herrán, 2011)
• Transferencia de energía por resonancia de fluorescencia (FRET). La fluorescence resonante energy transfer es una técnica de fluorescencia que
utiliza grupos cromóforos fluorescentes (fluoróforos).
La técnica emplea un sustrato de la proteína que constituye el biomarcador de daño tisular. Esta molécula sustrato tiene en su estructura un • Transferencia de energía por resonancia de fluorescencia (FRET). La fluorescence resonante energy transfer es una técnica de fluorescencia que utiliza grupos cromóforos fluorescentes (fluoróforos).
La técnica emplea un sustrato de la proteína que constituye el biomarcador de daño tisular. Esta molécula sustrato tiene en su estructura un grupo fluoróforo a una distancia menor de 100 angström de un grupo inhibidor de la emisión.
Cuando se irradia el sustrato con la longitud de onda de excitación el inhibidor impide la fluorescencia del fluoróforo por su proximidad, ya que disipa su energía; pero cuando el sustrato se trata con la enzima biomarcadora, esta lo hidroliza, cortando la molécula de sustrato, con lo cual separa el inhibidor del fluoróforo y este comienza a disipar su energía de excitación mediante fluorescencia. De esta forma, a mayor actividad de la enzima sobre su sustrato mayor será la intensidad de la fluorescencia.
Esta técnica permite determinar la presencia de proteasas que dañan membranas celulares. (Devlin, 2015)
Inmunohistoquímica
Es una técnica muy laboriosa, que utiliza anticuerpos monoclonales obtenidos in vitro que se dirigen contra un solo determinante antigénico en una estructura tisular compleja o molécula secretada por tejidos patológicos, para producir una inmunorreacción con baja incidencia de reacciones cruzadas inespecíficas.
El éxito de la técnica no solo estriba en lograr la especificidad de la reacción anticuerpo-antígeno, sino en conocer el lugar exacto donde se desarrolla (membranas, citoplasma o núcleo) y qué tipo de antígeno en el tejido debe o no interactuar con la sonda del anticuerpo. Por citar un ejemplo, en el daño periodontal aparecen biomarcadores como resultado de la destrucción de la MEC (colágeno, elastina, laminina) y esos antígenos tienen localización extracelular.
Dada las características de la técnica de inmunohistoquímica los servicios de análisis deben realizarse por laboratorios acreditados con personal entrenado, tanto en la parte histopatológica como de procedimientos protocolizados para inmunoensayos. (Buys, 2007)
La inmunohistoquímica tiene un amplio espectro de aplicación en estudios histopatológicos para diagnóstico y clasificación de tumores con presencia o no de metástasis. Con los anticuerpos monoclonales se pueden establecer pronósticos y predicciones sobre tumoraciones; hacer estudios de antígenos virales o tumorales; identificar proteínas tisulares y detectar complejos inmunitarios, lográndose tratamientos más eficaces.
Un ejemplo de la capacidad discriminatoria de esta técnica de inmunoexpresión se informa en la diferenciación lograda al estudiar la respuesta apoptótica de antígenos de caspasa 3 en carcinoma de labio, que resulto más baja, en comparación con la respuesta en cáncer intraoral. (Leite, 2016)
Las caspasas son proteasas de cisteína encargadas de destruir células en exceso o con daño irreparable en su ADN antes de que proliferen. Una deficiencia en la apoptosis de estas células contribuye al desarrollo de cáncer.
El exfoliado activo de caspasa-3 segmenta a una proteína Bid del complejo Bcl-2 de la apoptosis. La activación de Bid aumenta la permeabilidad de la membrana mitocondrial, provocando la salida del citocromo c, el efector intracelular que amplifica la apoptosis. Así, la imunohistoquímica permite establecer diferencias en la respuesta apoptótica en tejido oral enfermo. (Nelson y Cox, 2014)
Análisis de adn microarrays
El ADN en micromatrices, chips de ADN o CMA (chromosomal microarrays analysis) tiene diversas aplicaciones y permite, con alta resolución, diferenciar genes expresados por células sanas y cancerígenas.
El microarrays es del tamaño de un portaobjeto de microscopio con miles de microcavidades. En cada una hay múltiples copias de un gen. Una computadora, acoplada al escáner del microarrays (Scan Array) tiene un software con el nombre del gen que hay en cada microcavidad, una especie de mapa genético.
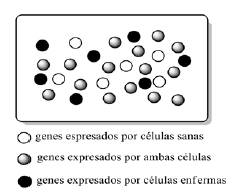
El ADN de células sanas y enfermas se tiñe de diferente color (azul y rojo, respectivamente). La lectura de microarrays, mediante técnica láser, permite ver en qué microcavidad se fija ADN de células sanas, en cuál ADN de células enfermas y en cuál ambos tipos de ADN (amarillo) por la mezcla de los colores.
Los genes “rojos”, que se expresan únicamente en células enfermas, son oncogenes asociados a las alteraciones del ciclo celular de la neoplasia. (Lieberman, 2013)
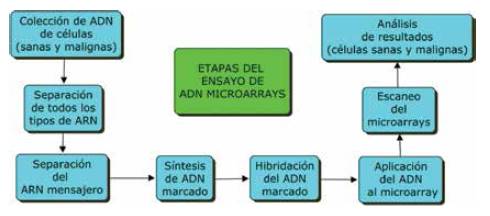
Para el ensayo ADN microarrays se colectan células sanas y enfermas del mismo tipo de tejido. La separación del ARN, de otros componentes celulares, se hace utilizando solventes orgánicos, agitación en vórtex y centrifugación. En el sobrenadante del centrifugado queda la disolución de ARN.
Para separar el ARNm de los otros tipos, el extracto se eluye mediante unas columnas que contienen oli-gonucleótidos que fijan el mensajero, por su extremo 3´-poly-A (secuencia de poliadenilación), única del mensajero, eluyendo los otros ARN. Después la columna se trata con un buffer para obtener por lavado el ARNm.
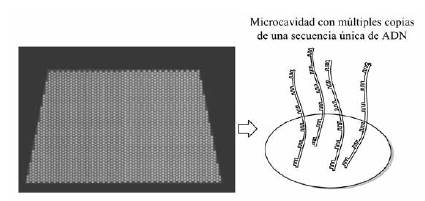
Figura 8 Izquierda: superficie del microarrays con miles de microcavidades. Derecha; una microcavidad del microarrays con copias insertadas de un gen específico.
En las soluciones de ARNm de células sanas y enfermas se obtiene ADN marcado, empleando mezclas reactivas que contienen cebador, transcriptasa inversa y nucleótidos de ADN, además de pigmentos de color diferente (azul y rojo). La reacción de la transcriptasa inversa sintetiza ADN de una hebra y, después de la copia, el ARN molde se degrada. La hibridación consiste en la unión de hebras de ADN complementarias para formas dúplex. Los ADN coloreados se esparcen en la superficie del microarrays y se procede al escaneo.
El código de colores y la identificación en el software de los genes de cada microcavidad, permiten establecer qué genes son expresados de forma exclusiva por las células enfermas. (Baynes, 2015)
La colección de ADN genómico en poblaciones para realizar estudios epidemiológicos y clínicos es una práctica en franco desarrollo, gracias al avance de la biología moleculary de los estudios de genómica, proteómica y fenotipado. Esta práctica requiere de la colección de muestras de alta calidad que sean representativas de las poblaciones objeto de estudio, y con los tamizajes se conoce la influencia genética individual en la respuesta a los tratamientos. (Hansen, 2007)
Un reto que presentan estos estudios es lograr una alta tasa de cumplimiento en las donaciones de los pacientes y un sistema de colección, conservación y transporte de muestras, eficiente y de bajo costo, que permita establecer bancos de ADN genómico para respaldar los estudios a gran escala.
En este sentido, resultan evidentes una serie de ventajas del muestreo de saliva.
• Tiene una adecuada concentración de leucocitos que son las células nucleadas que más se han empleado en la obtención de ADN genómico.
• La calidad, tamaño y funcionalidad del ADN obtenido en muestras de saliva es comparable al de muestras de sangre periférica, que se han venido utilizando en estos estudios.
• No es una técnica invasiva ni dolora, sencillamente el paciente escupe en un tubo plástico que contiene reactivos para conservación del ADN hasta su utilización en los laboratorios.
• Evita la venopunción y los riesgos de infección asociados, tanto para pacientes como para el personal de salud.
• El mismo paciente brinda la muestra por lo que no se requieren de técnicas ni personal especializado en la colección.
• El transporte de las muestras se realiza a temperatura ambiente y se puede utilizar el servicio de correos para realizarlo.
• La colección de muestras no es afectada por condiciones especiales de los pacientes como adultos mayores, neonatos y prematuros o personas con compromiso vascular.
• La colección de muestras se puede realizar tantas veces como sea necesario si se cuenta con el consentimiento de los pacientes.
• Permite realizar un gran número de determinaciones, además del uso del ADN genómico, debido al gran desarrollo tecnológico experimentado en el campo del análisis clínico y toxicológico.
Aunque las muestras de saliva presentan algunas desventajas en relación con las de sangre, estas se han podido resolver en gran medida sin afectar sus ventajas.
• Las menores concentraciones de los biomarcadores en saliva se han ido resolviendo con el diseño de técnicas más sensibles y con menores límites de detección. Siempre que los resultados analíticos en saliva se puedan correlacionar de forma efectiva con los detectados en sangre se prefieren los análisis en saliva.
• La actividad enzimática de algunas bacterias orales pueden interferir en algunas pruebas.
• La presencia de restos de alimentos y de moco en las muestras se ha contrarrestado con el aumento en el volumen de las muestras y procesos de filtración.
Conclusiones
El reto que plantea el uso diagnóstico de la saliva para estudios clínico y genéticos a gran escala es que requiere de laboratorios acreditados, personal entrenado y técnicas que resultan relativamente costosas.
Pero los beneficios son varios: utilizar el potencial humano que egresa de las universidades, desarrollar bases de datos aplicables a estudio epidemiológicos y realizar mejoras en el diagnóstico y los tratamientos. El desarrollo de políticas en este sentido tendría un impacto en la odontología, tanto a nivel de planes de estudios como en la práctica clínica.
José Manuel Rivera Pérez, Profesor de Bioquímica oral y docente del Posgrado de Endodoncia, Universidad Latina de Costa Rica; profesor de Biofarmacia, Análisis de drogas y laboratorio de Toxicología, Universidad de Iberoamérica, Costa Rica. fonticiella3@hotmail.com