Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Costarricense de Salud Pública
Print version ISSN 1409-1429
Rev. costarric. salud pública vol.20 n.2 San José Jul./Dec. 2011
Original Breve
Resistencia antimicrobiana de cepas de Staphylococcus aureus, Costa Rica
Antimicrobial resistance of Staphylococcus aureus, Costa Rica
Resumen
Objetivo: Determinar y comparar los perfiles de resistencia de cepas de S. aureus aisladas de quesos, producidos en
Materiales y Métodos: Se analizaron 35 muestras de queso fresco, adquiridas durante los meses de setiembre y octubre del 2010 en la zona de San Vito de Coto Brus. A cada muestra se le realizaron recuentos de coliformes totales, coliformes fecales y Staphylococcus aureus. Adicionalmente se analizó presencia/ausencia de Listeria monocytogenes en
Resultados: El promedio obtenido para el recuento de coliformes totales fue de 9,7 X 106 UFC/g, para coliformes fecales de 6,7 X 105 y para S. aureus de 2,8 X 105 UFC/g, obteniéndose un 83 % de muestras positivas por esta bacteria. En cuanto a la resistencia antimicrobiana, se obtuvieron porcentajes de resistencia mayores en las cepas de origen clínico. Se encontró también que 23 de las cepas (96%) provenientes de muestras clínicas, presentaban resistencia a más de un antibiótico, mientras que siete de las obtenidas a partir de queso (27%) presentaban esta característica. Con respecto a los betalactamicos (ampicilina, oxacilina y penicilina) se observó la existencia de una diferencia estadísticamente significativa (p<0,01) entre las cepas de ambas fuentes, presentándose mayor resistencia en las de origen clínico.
Conclusión: Ninguna de las cepas analizadas mostró resistencia a vancomicina, trimethoprim/sulfa ni linezolid.
Palabras clave: Productos Lácteos, Staphylococcus aureus, Farmacorresistencia Microbiana. (fuente: DeCS, BIREME)
Abstract
Objective: determined and compared the resistance patters of S. aureus strains isolated from cheese produced in the southern zone of Costa Rica and from clinical samples isolated at the hospital center of the same geographic zone.
Materials and Methods: Total and fecal coliform counts, and S. aureus counts were performed to 35 fresh cheese samples acquired at the zone of San Vito de Coto Brus. Also, the presence of Listeria monocytogenes was analyzed in
Results: The total coliform average count obtained was 9,7 X 106 UFC/g, for fecal coliforms 6,7 X 105 and for S. aureus 2,8 X 105 UFC/g. 83 % of the samples were positive for S. aureus. Antibiotic´s resistance averages were greater for clinical strains. Also, most of the clinical strains 23 (96 %) presented resistance to more than one antibiotic, compared with only seven strains isolated from cheese that presented this characteristic.
Conclusions: None of the strains analyzed showed resistance to vancomycin, trimethoprim/sulfa and linezolid.
Key words: Dairy Products, Staphylococcus aureus, Drug Resistance, Microbial. (source: MeSH, NLM)
El queso es un alimento que ha sido involucrado en numerosos brotes de origen alimentario provocados por microorganismos como S. aureus, Salmonella, Listeria monocytogenes, E. coli productora de verotoxina (1).
La contaminación de los alimentos por S. aureus puede ocurrir directamente desde los animales de consumo, los cuales pueden estar infectados, o puede resultar de la manipulación o manejo inadecuado de alimentos durante su procesado, almacenamiento o comercialización, ya que los seres humanos pueden ser portadores de este microorganismo (2). La intoxicación por este agente se produce al consumir dosis que van desde los 20 ng hasta < 1 μg de toxina. Los síntomas, que incluyen náuseas, calambres abdominales, diarrea y vómito, pueden presentarse de una hasta seis horas luego de haber ingerido el alimento, lo cual varía dependiendo de la susceptibilidad individual y la dosis tóxica ingerida (3). La mayoría de las personas que se ven afectadas por este tipo de intoxicación resuelven el cuadro satisfactoriamente y las muertes por el mismo son sumamente raras, sin embargo la intoxicación estafilocócica, sí produce una considerable carga económica y social (2).
Adicional a la capacidad de producir intoxicación y probablemente debido al uso intensivo de antibióticos en los sistemas de producción animal y en Salud Pública, desde hace muchos años se ha observado una mayor resistencia a los antibióticos en cepas de S. aureus, tanto de origen clínico como de uso industrial (cultivos iniciadores) (4).
En este sentido, se conoce que la resistencia a los antimicrobianos es un problema de Salud Pública en muchos países debido a la persistente circulación de cepas resistentes en el medio ambiente y la posible contaminación de agua y alimentos (2). Ante este panorama los investigadores han postulado que eventualmente los microorganismos ingeridos diariamente con los alimentos podrían estar sirviendo de reservorios para el mantenimiento de la resistencia a los antibióticos (2).
Por esa razón, en el presente trabajo se determinaron y compararon los perfiles de resistencia obtenidos de cepas de S. aureus aisladas a partir de quesos, producidos en
Material y métodos
Se analizaron un total de 35 muestras de queso fresco, de siete diferentes marcas comerciales (identificadas con las letras de
Los quesos fueron transportados al Laboratorio de Microbiología de Alimentos y Aguas de la Universidad de Costa Rica en refrigeración y se analizaron en un máximo de 24 horas post colecta.
A cada muestra se le realizaron los siguientes recuentos: coliformes totales, coliformes fecales y Staphylococcus aureus. Adicionalmente se analizó presencia/ausencia de Listeria monocytogenes en
Se utilizó la metodología descrita en el Compendio de Métodos para el examen Microbiológico de Alimentos (5). Brevemente, se pesaron
De cada muestra también se pesaron
Identificación y prueba de sensibilidad a antibióticos para cepas de S. aureus
A partir de los crecimientos obtenidos en agar Baird Parker se picaron las colonias sospechosas, se les realizó tinción de Gram, catalasa y coagulasa.
Los aislamientos que resultaron Gram, coagulasa y catalasa positivos fueron posteriormente identificados mediante el sistema Vitek I (BioMerieux). A las cepas cuya identificación fue S. aureus se les realizó la prueba de sensibilidad a los antibióticos mediante el mismo sistema Vitek y la interpretación de los datos se realizó siguiendo las pautas del Clinical and Laboratory Standards Institute antimicrobial susceptibility testing 2011 (7).
Adicionalmente, se recolectaron datos acerca de la sensibilidad de las cepas de S. aureus aisladas e identificadas mediante el sistema automatizado Vitek en el Hospital de San Vito de Coto Brus en el período comprendido entre setiembre y octubre del 2010.
Para la comparación de los perfiles de resistencia se utilizó la prueba U de Mann-Whitney.
Resultados
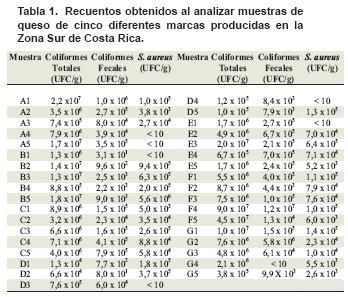
El promedio obtenido para el recuento de coliformes totales fue de 9,7 X 106 UFC/g, para coliformes fecales fue de 6,7 X 105 y para S. aureus el recuento promedio fue de 2,8 X 105 UFC/g.
Con respecto a S. aureus, se encontró que sólo seis muestras, de 35 analizadas, fueron negativas para esta bacteria, lo cual indica que la positividad fue de 83 % (tabla 1). De ninguna de las muestra analizadas fue posible aislar Listeria monocytogenes.
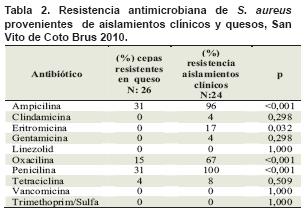
En cuanto a la resistencia antimicrobiana, se obtuvieron porcentajes de resistencia mayores en las cepas de origen clínico, como se muestra en la tabla 2. Es importante destacar también, la marcada susceptibilidad mostrada por las cepas provenientes de quesos en especial la diferencia encontrada para penicilina, oxacilina y ampicilina.
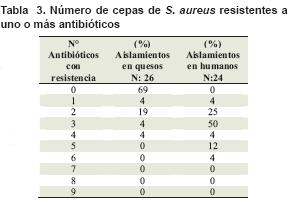
Se encontró también que 23 de las cepas (96 %) provenientes de muestras clínicas presentaban resistencia a uno o más antibióticos, mientras que siete de las obtenidas a partir de queso (27 %) presentaban esta característica (Tabla 3). No se obtuvieron datos para amoxacilina y cefazolin.
Discusión
El reglamento Técnico Centroamericano RTCA 67.04.50:08 “Criterios microbiológicos para la inocuidad de los alimentos”, en el grupo 1 subgrupo 9, quesos frescos no madurados, establece como límite máximo permitido para el recuento de S. aureus 103 UFC/g, por lo cual cuando se observan los resultados, dos muestras de la marca A, dos de
Adicional a lo anterior, el porcentaje de positividad encontrado en este estudio para S. aureus (83 %) resulta bastante alto si se compara con resultados obtenidos por otros investigadores, como es el caso de Rosengren y colaboradores que reportan un 12 % de positividad por S. aureus y Little y colaboradores no encontraron positividad en quesos frescos elaborados a partir de leche pasteurizada en el Reino Unido. Otro estudio realizado en Mérida, Venezuela reporta una positividad de 69 % para este microorganismo (8).
El mismo reglamento indica que estos quesos no deben tener contaminación fecal (E. coli < 10 UFC/g) y aunque en este estudio no se determinó la presencia de dicha bacteria si se observan altos recuentos de coliformes fecales, lo cual llama la atención ya que la literatura reporta que el 90 % de estos pueden llegar a ser E. coli (5). Siendo así recuentos tan altos como los obtenidos resultan inaceptables. Estos datos contrastan con lo reportado por Little y colaboradores quienes encontraron E. coli únicamente en tres (0,7 %) de 412 muestras de queso analizadas, o con lo reportado por Rosengren y colaboradores (9) quienes informan de un 3 % de positividad por E coli en quesos elaborados a partir de leche pasteurizada. Es importante también recordar que el grupo coliforme es sensible a la pasteurización por lo cual su presencia en el queso elaborado a partir de leche pasteurizada, denota mal manejo post proceso o inadecuada pasteurización de la leche (5).
Con respecto a microorganismos patógenos, llama la atención la no detección de L. monocytogenes, ya que reportes anteriores en Costa Rica le asignan a este microorganismo una positividad de 10 % (10) y 45 % (11) en quesos. En Perú se reporta un 4 % de positividad en este producto (12) sin embargo otras investigaciones, llevadas a cabo en Europa (9) y en Chile (13) reportan también la no detección de este patógeno en queso fresco. Con respecto a esto, algunos autores (12,13) indican que las bacterias lácticas son capaces de producir ácidos orgánicos, peróxido de hidrógeno y bacteriocinas que tiene efecto antagónico contra Listeria. En este estudio el menor valor promedio del recuento de bacterias lácticas fue de 8,6 X 106 UFC/g y el mayor fue de 1,2 X 108 UFC/g (resultados no mostrados), hecho, que pudiera haber influido en la no detección de este patógeno en las muestras analizadas.
En lo que respecta a los perfiles de resistencia a antibióticos se observaron diferentes niveles de resistencia en las cepas de S. aureus provenientes de humanos y de muestras de queso, hallazgo también reportado por Yucel y colaboradores (14). Los porcentajes de resistencia más altos encontrados en este estudio fueron para los B-lactámicos penicilina, ampicilina y oxacilina. La resistencia a penicilina está ampliamente reportada en la literatura tanto en cepas clínicas como provenientes de diversos alimentos (2,14-16), sin embargo el porcentaje de resistencia (31 %) obtenido para cepas provenientes de quesos es bastante más bajo que lo reportado por otros autores (9,14,15). Con respecto a la resistencia observada para oxacilina ninguna de las cepas aisladas de quesos presentó concentraciones mínimas inhibitorias (MICs) superiores a cuatro, mientras que 15 de las 16 cepas de origen clínico sí mostraron MICs mayores a cuatro, lo cual amerita una identificación presuntiva como S. aureus meticilina resistente según el CLSI, por lo que se recomienda que estas cepas de origen clínico sean más estudiadas. En este sentido las cepas provenientes de queso no serían sospechosas de ser resistentes a meticilina lo que concuerda con la baja prevalencia reportada para MRSA en alimentos (17).
Los S. aureus provenientes de queso no mostraron resistencia a eritromicina ni gentamicina sin embargo otros autores (14-16) reportan bajos porcentajes de resistencia a estos antibióticos en cepas provenientes de diversos alimentos.
El 69 % de las cepas estudiadas provenientes de quesos fueron sensibles a todos los antibióticos probados pero ninguna cepa de origen clínico mostró esta característica. Sólo siete cepas aisladas de quesos presentaron resistencia a dos o más antibióticos mientras que de los aislamientos clínicos el 96 % (23 cepas) presentó multiresistencia, hallazgo reportado también por otros autores (15-17). Ninguna de las cepas analizadas mostró resistencia a vancomicina, trimethoprim/sulfa ni linezolid dato totalmente concordante con otros reportes a nivel mundial (2,14-16).
Los resultados obtenidos en este trabajo ponen de manifiesto el potencial riesgo que supone el consumo de este producto debido a la contaminación presente.
No obstante, permite ver que la transmisión de determinantes de resistencia a los antibióticos a partir de quesos frescos no es aún significativa. El hallazgo de cepas de S. aureus con resistencia a más de un antibiótico es una llamada de atención a promover el uso racional de estos con el fin de evitar la aparición y propagación de cepas resistentes.
Referencias
1. Little CL, Rhoades JR,
2. Normanno G,
3. Le Loir Y, Baron F, Gautier M. Staphylococcus aureus and food poisoning. Genet. Mol. Res. 2003; 2: 63–67. [ Links ]
4. Resch M, Nagel V, Hertel C. Antibiotic resistance of coagulase-negative staphylococci associated with food and used in starter cultures. International Journal of Food Microbiology 2008; 127: 99-104. [ Links ]
5. Pouch, F. Compendium of methods for the Microbiological Examination of Foods 4th. Ed American Public Health Association 800 I
6. FDA. Bacteriological and Analytical Manual. AOAC. 18th. Ed. New York.1995. [ Links ]
7. Clinical and Laboratory Standards Institute. Antimicrobial susceptibility testing standards M02-A10 and M07-A8.2011. [ Links ]
8. Díaz C, González B. S. aureus en queso blanco fresco y su relación con diferentes microorganismos indicadores de calidad sanitaria. Rev Salud Pública y Nutrición. 2001; 2(3). [ Links ]
9. Rosengren A, Fabricius A, Sylvén S, Lindqvist R. Occurrence of food borne pathogens and characterization of Staphylococcus aureus in cheese produced on farm-dairies. International Journal of Food Microbiology 2010; 144: 263-269. [ Links ]
10. Ellner R, Utzinger D, García V. Aislamiento de Listeria sp. de diversos alimentos en Costa Rica. Rev. Costarric. Ci. Med.1991; 12: 33-39. [ Links ]
11. Arias ML, Antillón F. Contaminación microbiológica de los alimentos en Costa Rica. Una revisión de 10 años. Rev. Biomed. 2001; 1: 113-122. [ Links ]
12. Espinoza A, De
13. Schöbitz R, Marín M, Horzella M, y Carrasco, E. Presencia de Listeria monocytogenes en leche cruda y quesos frescos artesanales. Agro.
14. Yucel N, Citak S, Bayhün S. Antimicrobial resistance profile of Staphylococcus aureus isolate from clinical samples and foods of animal origin. Foodborne pathogens and disease. 2011; (8): 427-431. [ Links ]
15. Pereira V, Lopes L, Castro A, Silva J. Gibbs P, Teixeira P. Characterization for enterotoxin production, virulence factors, and antibiotic susceptibility of Staphylococcus aureus isolates from various foods y Portugal. Food Microbiology. 2009; 26: 278-282. [ Links ]
16. Aydin A, Muratoglu K, Sudagidan M, Bostan K, Okuklu B, Harsa S. Prevalence and antibiotic resistance of foodborne Staphylococcus aureus isolates in
17. Normanno G, Corrente M,
*Correspondencia a: 1 Microbiólogo. Hospital San Juan de Dios. Caja Costarricense de Seguro Social
2 Microbiólogo. Hospital de San Vito. Caja Costarricense de Seguro Social.
Recibido 12 diciembre 2011 Aceptado 23 diciembre 2011