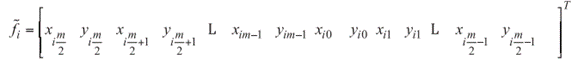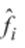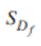Introducción
La economía en Centroamérica depende de la agricultura. En países como Costa Rica, en donde indicadores macroeconómicos muestran un incremento en la diversificación de bienes y servicios, un 10,3% de las actividades económicas se atribuyen al sector agropecuario (1). La supervivencia de este sector depende de la mejora continua que permita alcanzar, mantener e innovar los procesos, superando los estándares de calidad y cantidad demandados por los mercados locales e internacionales (2).
En la búsqueda de biocontroles para enfermedades en plantas se ha perseguido automatizar parcial o totalmente el análisis de muestras con modernos métodos de campo de visión por computador y aprendizaje de máquina, para así potenciar el impacto del tiempo invertido por expertos en el análisis de dichas muestras (3), (4).
Métodos que utilizan explícitamente información de la forma de organismos para determinar indicadores de interés requieren suficiente material de entrenamiento, que es en general costoso de preparar manualmente. Este artículo propone una técnica novedosa de emulación de la variación de siluetas, que se produce con un bajo costo computacional. El método propuesto parte de un conjunto de datos inicial que define el dominio válido de formas, y a partir de allí sigue un recorrido aleatorio por dicho dominio. El proceso genera una sucesión de formas sintéticas nuevas que complementan el conjunto de datos original, con el objetivo de poder utilizarlo en procesos de entrenamiento y validación de sistemas de aprendizaje automático.
Si bien es cierto, el concepto presentado en este artículo se desarrolla en el contexto de análisis de imágenes de nematodos como caso particular, el concepto es aplicable a cualquier otra área en que se utilicen modelos explícitos de forma, como el análisis facial (5) o el análisis de imágenes médicas (6), entre otros.
El artículo se estructura de la siguiente manera: una revisión de trabajos relacionados se presenta en la siguiente sección.; el fundamento teórico y algorítmico utilizado en la generación de las nuevas formas se describe en la tercera sección; en la cuarta, se analizan los resultados del método propuesto. Conclusiones y trabajo futuro presentados en la última sección cierran el artículo.
Trabajos relacionados y modelo propuesto
Varias estrategias se han propuesto para clasificar, detectar, simular y extraer estructuras en imágenes de objetos con formas de gusano. En “Active shape models…” (7) se sientan las bases de los métodos capaces de expresar explícitamente formas válidas por medio de modelos estadísticos de distribución de puntos, con base en el análisis de componentes principales, para la reducción de la dimensión de vectores representativos de la formas. Sin embargo, en ese mismo trabajo se demuestra que una distribución normal de puntos es incapaz de restringir las formas representadas a formas vermes válidas y deja por fuera siluetas anatómicamente plausibles en nematodos y otros organismos vermiformes.
Un método de descripción de las contorsiones de la cabeza del C. elegans se expone en “Automated detection…” (8). Dicho método propone cómo medir los ángulos de contorsión en la cabeza, en la descripción del forrageo (movimientos exploratorios). La información espaciotemporal se aprovecha en “Tracking the swimming motions…” (9) para predecir la posición y ajustar la forma del organismo en procesos de segmentación y rastreo, utilizando para ello la evolución de los ángulos y vértices del esqueleto morfológico. Estos conceptos se toman como base en este trabajo para ser utilizados en la dirección contraria, y sintetizar con ello nuevas formas.
Otros métodos para representar el espacio de formas válidas de nematodos basados en técnicas de aprendizaje de diccionarios y de aprendizaje de variedades se exploran en ”Active dictionary models…” (10) y “Segnema: nematode segmentation…”(11). Un concepto basado en un modelo generativo que aprende la geometría de la variedad a partir de los datos se propone en “Geometry based data generation” (12), en donde se procura ignorar la densidad de estos, utilizando mapas de difusión para establecer la estructura geométrica de la variedad. Dicha estrategia no es apta para el tipo de datos presentes en este artículo, donde la dimensión del espacio es elevada con respecto al número de datos disponible, pues la difusión no cuenta con suficiente soporte para ser estimada de forma robusta.
Como alternativa para dar más peso a la geometría de la variedad que a la densidad de puntos disponibles de entrenamiento, se utiliza en la presente propuesta una técnica similar a los k vecinos más cercanos móviles, expuesta en “Analysis and evaluation of V*-kNN…” (13) para un contexto de consultas en bases de datos espaciales. El método es similar al presentado aquí en el hecho de que usa tanto la información de la posición actual en la caminata como del vecindario para la decisión del siguiente paso; pero en el presente caso la estrategia elegida para saltar a la siguiente posición está orientada a producir formas disímiles al punto de partida, manteniendo su validez, en vez de procurar la descripción geométrica de la variedad.
En otra dirección, la simulación completa del organismo de un nematodo de la especie C. elegans se persigue en el proyecto OpenWorm (14). En ese proyecto ya se ha generado el movimiento locomotivo de un espécimen, por medio de la simulación de 302 neuronas y 95 células musculares. Los datos producidos en dicha simulación se pueden acoplar a renderizadores que generen las siluetas o las imágenes, según se requiera. Si bien es cierto, esta simulación de abajo hacia arriba (bottom-up) permite alcanzar altos niveles de realismo anatómico en los detalles, el consumo de recursos computacionales no la hace viable en contextos donde la generación de formas es parte de otros procesos, como la segmentación de nematodos o su rastreo en imágenes.
En este trabajo se propone una estrategia para la generación de una sucesión de formas a partir de una caminata aleatoria estimada sobre un conjunto de datos de entrenamiento que se asume pertenecen a una variedad topológica embebida en un espacio de alto número de dimensiones, en donde el reto es generar formas suficientemente disímiles de las existentes en el cuerpo de entrenamiento, pero que sigan siendo válidas en el dominio de aplicación, todo ello con un bajo consumo de recursos computacionales, que permita insertar el proceso generativo en otras tareas, como la segmentación o rastreo de nematodos en imágenes.
Simulación de movimientos vermiformes
La generación de nuevas formas aleatorias con el método propuesto persigue restringir los resultados a estructuras vermiformes válidas. Se parte de un modelo donde el dominio completo de Df formas válidas se particiona en subdominios SDf, para los que se define una relación de vecindad. Cada paso realizado en una caminata aleatoria discreta produce saltos a subdominios vecinos. Esto asegura que cada forma generada en cada paso de la caminata es única en su estructura, diferenciándose de los pasos previos, pero además que es una forma válida. En las siguientes secciones se detalla el proceso de generación de formas y la definición del camino aleatorio.
Conjunto de datos para la simulación
Los modelos de forma son prototipos paramétricos representados a través de un conjunto Df de formas cuyo i - ésimo elemento se representa vectorialmente por 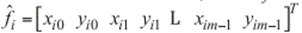 donde cada par
donde cada par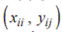 representa las coordenadas cartesianas de un hito Pij en la imagen. Los hitos deben estar ubicados secuencialmente a lo largo del contorno del objeto que representan (ver figura 1).
representa las coordenadas cartesianas de un hito Pij en la imagen. Los hitos deben estar ubicados secuencialmente a lo largo del contorno del objeto que representan (ver figura 1).
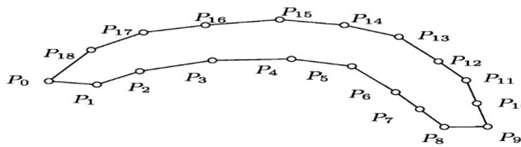
Figura 1 Secuenciación de hitos de una forma verme Cada forma en el conjunto de datos se construye a partir de una forma cruda representada con un número arbitrario de hitos colocados manualmente por un anotador humano sobre el borde de cada organismo en imágenes digitales. Las imágenes provienen de muestras reales de suelo, capturadas con un microscopio óptico en trece secuencias con características visuales variadas.
Las formas crudas son normalizadas en varios pasos. Primero se asegura que los hitos anotados sigan una secuencia en sentido antihorario (15), (16), iniciando siempre en la cabeza o la cola. Un número constante m de hitos equidistantes para cada forma del conjunto Df se genera por medio de interpolación con trazadores segmentarios cúbicos (17), (18) a partir de los hitos anotados. Además, el conjunto de datos se extiende (o aumenta) con formas generadas por reflexiones de las formas interpoladas, e intercambiando las posiciones de cabeza y cola, con
Finalmente, todo el conjunto de formas se alinea y re-escala utilizando el análisis de Procrustos descrito en “Active shape models…” (7). En adelante, la i-ésima forma normalizada del conjunto total de datos se denota con  .
.
Dominio y subdominios de formas vermes
El dominio Df es un conjunto de cardinalidad infinita en el que cada elemento 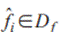 es una forma verme válida. Asimismo, se define un subdominio
es una forma verme válida. Asimismo, se define un subdominio 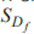 como un subconjunto de individuos
como un subconjunto de individuos 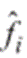 que por medio de algún estadístico de proximidad o distancia, agrupa formas similares. De este modo el dominio se particiona en un conjunto de subdominios disjuntos. En el presente trabajo, cada subdominio se representa por medio de un n-símplex (19), es decir, una envoltura convexa de n dimensiones engendrada por n + 1 puntos en
que por medio de algún estadístico de proximidad o distancia, agrupa formas similares. De este modo el dominio se particiona en un conjunto de subdominios disjuntos. En el presente trabajo, cada subdominio se representa por medio de un n-símplex (19), es decir, una envoltura convexa de n dimensiones engendrada por n + 1 puntos en 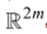 dada por
dada por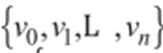 con vectores
con vectores 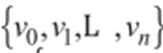 linealmente independientes, de modo que
linealmente independientes, de modo que 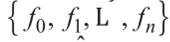 sean formas vermes válidas similares. Se parte de la premisa de que formas válidas
sean formas vermes válidas similares. Se parte de la premisa de que formas válidas 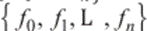 dentro del subdominio
dentro del subdominio 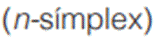 se engendran como combinaciones baricéntricas:
se engendran como combinaciones baricéntricas:
Con 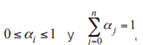 y que dicho subdominio embebido en el espacio
y que dicho subdominio embebido en el espacio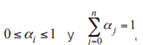 es (aproximadamente) tangente a una variedad topológica n-dimensional correspondiente al dominio
es (aproximadamente) tangente a una variedad topológica n-dimensional correspondiente al dominio 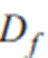 que contiene todas las formas válidas. Dos subdominios que poseen al menos un vértice del n-símplex en común se consideran vecinos.
que contiene todas las formas válidas. Dos subdominios que poseen al menos un vértice del n-símplex en común se consideran vecinos.
Por medio del algoritmo de k - medias (20) se encuentran k conglomerados 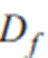 , que agrupan todas las formas disponibles de acuerdo a su similitud. Los k centroides asociados a los conglomerados conforman el conjunto C de posibles vértices de los n-símplex (ver figura 2).
, que agrupan todas las formas disponibles de acuerdo a su similitud. Los k centroides asociados a los conglomerados conforman el conjunto C de posibles vértices de los n-símplex (ver figura 2).
Generación de movimientos aleatorios vermes
Los movimientos vermes se simulan con una caminata aleatoria (21), definida en este caso como una sucesión de saltos aleatorios entre subdominios 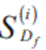 vecinos (como se muestra en la figura 3), que deforman gradualmente la silueta original. Este recorrido entre subdominios vecinos permite que cada elemento
vecinos (como se muestra en la figura 3), que deforman gradualmente la silueta original. Este recorrido entre subdominios vecinos permite que cada elemento 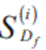 conserve información de la forma de al menos uno de los vértices que los subdominios tienen en común, obligando así en cada cambio de subdominio a una deformación paulatina, sin cambios abruptos.
conserve información de la forma de al menos uno de los vértices que los subdominios tienen en común, obligando así en cada cambio de subdominio a una deformación paulatina, sin cambios abruptos.
Se parte entonces de una forma verme inicial 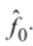 . A partir de ella se calcula el n-símplex
. A partir de ella se calcula el n-símplex 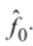 engendrado por el conjunto
engendrado por el conjunto 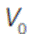 de los n centroides en C más cercanos a
de los n centroides en C más cercanos a 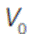 . Esta tarea utiliza el método aproximado de búsqueda de vecinos más cercanos expuesto en “Fast approximate nearest neighbors…” (22).
. Esta tarea utiliza el método aproximado de búsqueda de vecinos más cercanos expuesto en “Fast approximate nearest neighbors…” (22).
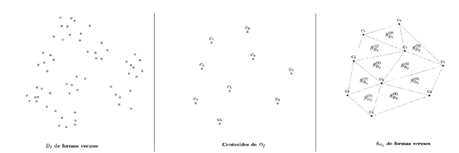
Figura 2 Obtención de 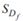 a través de k - medias. El subconjunto de n vértices se elige de modo que el n-símplex engendrado no contenga ningún otro vértice. La elección del número k > n de conglomerados define entonces indirectamente el volumen de los símplex, y qué tan disímiles serán las formas en subdominios vecinos.
a través de k - medias. El subconjunto de n vértices se elige de modo que el n-símplex engendrado no contenga ningún otro vértice. La elección del número k > n de conglomerados define entonces indirectamente el volumen de los símplex, y qué tan disímiles serán las formas en subdominios vecinos.
Ahora, se selecciona el siguiente subdominio 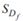 determinado por el conjunto V
1 de los n centroides más próximos a uno de los vértices (seleccionado aleatoriamente) de V
0. Este nuevo subdominio de formas permitidas corresponde a un n-símplex vecino del anterior. Dentro de este subdominio
determinado por el conjunto V
1 de los n centroides más próximos a uno de los vértices (seleccionado aleatoriamente) de V
0. Este nuevo subdominio de formas permitidas corresponde a un n-símplex vecino del anterior. Dentro de este subdominio  se toma aleatoriamente la siguiente forma
se toma aleatoriamente la siguiente forma 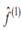 de la sucesión, utilizando coeficientes baricéntricos generados aleatoriamente.
de la sucesión, utilizando coeficientes baricéntricos generados aleatoriamente.
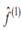
Figura 3 Caminata aleatoria de 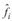 entre
entre 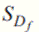 vecinos El proceso se repite iterativamente hasta satisfacer un criterio de parada, como, por ejemplo, un número predeterminado de pasos, o haber superado alguna medida de distancia con el punto de partida. El algoritmo completo se resume en la figura 4.
vecinos El proceso se repite iterativamente hasta satisfacer un criterio de parada, como, por ejemplo, un número predeterminado de pasos, o haber superado alguna medida de distancia con el punto de partida. El algoritmo completo se resume en la figura 4.

Figura 4 Algoritmo de caminata aleatoria de formas válidasEl algoritmo propuesto permite simular las deformaciones a partir de una instancia inicial y que continúen de manera natural conservando información de forma entre las iteraciones, y dando a la secuencia el carácter de movimiento aleatorio de un organismo vermiforme. En la figura 5, se aprecia un ejemplo del movimiento generado por el recorrido aleatorio basado en el salto entre subdominios vecinos. Se parte de una forma inicial arbitraria y se muestran para nueve iteraciones las deformaciones generadas, que el método asegura sean formas vermes válidos.
Transformación de formas y proyección a la variedad
Para poder evaluar el alcance de las formas generadas en la caminata aleatoria, es necesario resolver dos problemas:
¿Cómo generar eficientemente formas arbitrarias que se puedan considerar válidas?
¿Cómo proyectar una forma verme cualquiera al dominio de formas válidas?
El primer problema se ataca para el caso de los nematodos, con la simulación de deformación de la anatomía. El segundo problema se resuelve con el algoritmo de la estimación de las coordenadas baricéntricas correspondientes a una forma dada dentro del n-símplex más cercano. Los detalles se desglosan en las siguientes dos secciones.
Método de deformación de nematodos
Para deformar un nematodo 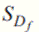 , se utiliza el esqueleto Ei (ver figura 6) definido como un vector compuesto por las posiciones de las vértebras, cada una dada por el promedio de los dos hitos correspondientes opuestos
, se utiliza el esqueleto Ei (ver figura 6) definido como un vector compuesto por las posiciones de las vértebras, cada una dada por el promedio de los dos hitos correspondientes opuestos 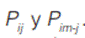 . Se toman los hitos sobre la cabeza y cola como la primera vértebra
. Se toman los hitos sobre la cabeza y cola como la primera vértebra  y última vértebra
y última vértebra 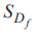 , respectivamente, donde
, respectivamente, donde 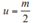 y m es el número de hitos de
y m es el número de hitos de 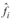 . El conjunto de todos los esqueletos Ei de
. El conjunto de todos los esqueletos Ei de 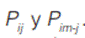 se denota E, y cada uno se expresa vectorialmente por
se denota E, y cada uno se expresa vectorialmente por
La posición de cada vértebra intermedia está dada por
La modificación de forma se concentra en dos fragmentos del esqueleto, definidos por las u´ vértebras 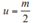 más cercanas a la cabeza y a la cola de
más cercanas a la cabeza y a la cola de 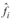 . Para las vértebras Vij del fragmento de la cabeza se cumple
. Para las vértebras Vij del fragmento de la cabeza se cumple 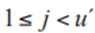 , y para el fragmento de cola
, y para el fragmento de cola 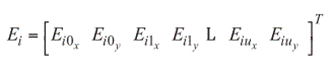 .
.
Para lograr la deformación deseada (e inspirada en el forrageo (8)) se toman los fragmentos u´ con vértebras de cola y cabeza, y se manipulan (junto a los hitos correspondientes a cada vértebra) como una cadena cinemática directa. En dicha cadena, cada vértebra cuenta con un grado de libertad rotacional. Los ángulos de rotación θ están restringidos a ser iguales en todas las vértebras.
Dado el número u´ de vértebras que modificar, el algoritmo toma inicialmente la vértebra del fragmento de cabeza 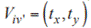 como fija y aplica la transformación al resto de vértebras e hitos en la cadena cinemática.
como fija y aplica la transformación al resto de vértebras e hitos en la cadena cinemática.
Luego, dada la nueva posición de la vértebra 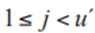 se le aplica M
v
al resto de elementos de la subcadena cinemática hacia la cabeza. Posteriormente, se repite el proceso a partir de la vértebra
se le aplica M
v
al resto de elementos de la subcadena cinemática hacia la cabeza. Posteriormente, se repite el proceso a partir de la vértebra 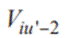 y así sucesivamente hasta llegar al hito de la cabeza. De forma análoga, se sigue el procedimiento de la vértebra colocada en la posición u - u´+ 1 hacia la cola. Las contorsiones vermes de la cola resultante se ilustran en la figura 7.
y así sucesivamente hasta llegar al hito de la cabeza. De forma análoga, se sigue el procedimiento de la vértebra colocada en la posición u - u´+ 1 hacia la cola. Las contorsiones vermes de la cola resultante se ilustran en la figura 7.
Proyección al dominio de formas válidas
La proyección de una forma 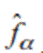 al dominio de formas válidas se divide en dos etapas. La primera es determinar el subdominio al que pertenece
al dominio de formas válidas se divide en dos etapas. La primera es determinar el subdominio al que pertenece 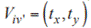 , y la segunda es calcular los pesos baricéntricos que mejor aproximan la forma en dicho subdominio.
, y la segunda es calcular los pesos baricéntricos que mejor aproximan la forma en dicho subdominio.
Para la primera etapa se calcula el conjunto de los n+1 vecinos más cercanos 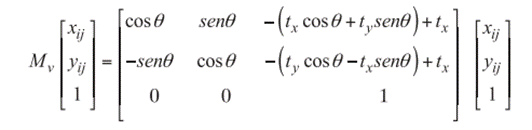 a
a 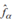
ordenados según su lejanía. Estos vecinos son elementos del conjunto de posibles vértices obtenido con el algoritmo de k-medias (sección 3.2). Luego, se aplica la variación del método matching pursuit (25) propuesta en “Active dictionary models…” (10) para estimar los pesos baricéntricos 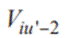 que mejoraproximan la forma
que mejoraproximan la forma 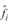 dada por
dada por 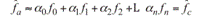
Así 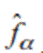 es la aproximación más cercana a
es la aproximación más cercana a 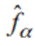 que se encuentra dentro del n-símplex con vértices
que se encuentra dentro del n-símplex con vértices 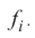
Resultados y discusión
En esta sección, se muestran resultados sobre la versatilidad del método propuesto para la generación de sucesiones aleatorias de formas vermes que simulen el movimiento de serpenteo. Primero, se analiza la similitud entre formas de la secuencia en función del número de centroides utilizados. Luego se revisa el método utilizado para estimar los pesos baricéntricos.
Para la experimentación se utilizó una base de datos de entrenamiento con 10 932 formas vermes distintas, generadas a partir de un conjunto inicial de formas crudas, normalizadas y aumentadas según lo descrito en la sección 3.1, con m=40 hitos por contorno.
Los algoritmos se implementaron en el lenguaje C++, utilizando la biblioteca FLANN (23) para el cálculo eficiente de los centroides y de búsqueda de vecinos cercanos, así como la biblioteca LTI-Lib2 (24) como base para el procesamiento de imágenes.
Distancia entre formas en función de la dimensión del subdominio
En el primer experimento se evaluó la distancia entre las formas producidas con los saltos entre subdominios, en función de la dimensión de dichos subdominios. Dicha distancia entre formas se cuantificó con el promedio de la distancia euclídea entre hitos correspondientes de dos formas sucesivas 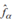 dada por
dada por 
El cuadro 1 resume los resultados obtenidos usando 1000 iteraciones del algoritmo de generación de formas (figura 4) con subdominios n-dimensionales (n-símplex), con n = 5,10,20∙∙∙,100 vértices tomados de k centroides, definidos sobre el conjunto de datos con el método de k medias (sección 3.2) para k > 200 en múltiplos de 100.
El resultado confirma la correlación existente entre la distancia entre las formas sucesivas y la dimensión n de los subdominios. Entre mayor la cantidad de vértices utilizada, mayor la distancia entre los hitos correspondientes a dos deformaciones sucesivas.
Esto puede explicarse así: manteniendo el número k de puntos disponibles como vértices constante, entonces mientras mayor sea el número n, mayor será la distancia entre dichos vértices, de modo que también aumente el tamaño del paso realizado de una forma a otra.
Nota: Se tomaron los k centroides correspondientes a los conglomerados encontrados por el algoritmo de k-medias, y de ellos se seleccionaron subdominios de n dimensiones (n-símplex) embebidos en un espacio de 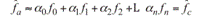 (40 hitos por forma).
(40 hitos por forma).
Si el número de vértices del subdominio es 5, el desplazamiento de los hitos será menor a 8 píxeles en promedio, lo que exhibe baja variabilidad promedio de cambios independientemente del número de centroides.
Si se elige un número de vértices de los subdominios mayor a 40, las deformaciones sucesivas con distancias superiores a 13 pixeles por hito se perciben abruptas y se pierde la noción de continuidad de la simulación, independientemente del número k de centroides utilizado. Simulaciones aceptables con distancias promedio entre hitos de alrededor de 12 pixeles se pueden generar con n-símplex con dimensión n entre 10 y 30, independientemente del número de centroides.
Validez y alcance del dominio de formas
El segundo experimento persigue cuantificar de qué modo cualquier forma verme 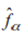 puede ser aproximada en el dominio de formas válidas
puede ser aproximada en el dominio de formas válidas 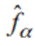 .
.
Se utilizó una muestra de 1000 formas vermes 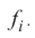 generadas aleatoriamente usando el método de deformación de nematodos ya descrito, normalizadas según el método indicado para la normalización de las formas crudas, utilizando como punto de partida formas no presentes en la base de datos de entrenamiento.
generadas aleatoriamente usando el método de deformación de nematodos ya descrito, normalizadas según el método indicado para la normalización de las formas crudas, utilizando como punto de partida formas no presentes en la base de datos de entrenamiento.
Se utilizó el método para la proyección al dominio de formas válidas, para calcular la aproximación 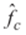 de cada forma
de cada forma  . Se evaluó entonces la distancia promedio entre hitos (dada en la sección 4.1) en función de la dimensión n de los símplex, para varios valores de k centroides de donde se tomaron los n + 1 vértices de los símplex. En los resultados que se muestran en la figura 8 hay tendencia a una disminución de la distancia entre la aproximación
. Se evaluó entonces la distancia promedio entre hitos (dada en la sección 4.1) en función de la dimensión n de los símplex, para varios valores de k centroides de donde se tomaron los n + 1 vértices de los símplex. En los resultados que se muestran en la figura 8 hay tendencia a una disminución de la distancia entre la aproximación 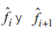 cuando aumenta el número k de centroides que determinan los subdominios. Desde una partición con k = 200 conglomerados del
cuando aumenta el número k de centroides que determinan los subdominios. Desde una partición con k = 200 conglomerados del 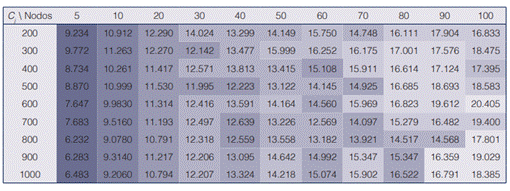 para formar los subdominios, se muestra que aproximadamente la distancia por hito entre las siluetas originales y las calculadas es menor a 1 pixel promedio, por lo que se concluye que son muy cercanas en forma. Por otro lado, al aumentar la dimensión de los n-símplex, la distancia de desvío entre hitos tiende a aumentar también para un mismo número de centroides k, puesto que si se mantiene k, entonces la distancia entre los vértices crece con el aumento de la dimensión n de la variedad. Obsérvese que este efecto se reduce conforme aumenta el número de centroides.
para formar los subdominios, se muestra que aproximadamente la distancia por hito entre las siluetas originales y las calculadas es menor a 1 pixel promedio, por lo que se concluye que son muy cercanas en forma. Por otro lado, al aumentar la dimensión de los n-símplex, la distancia de desvío entre hitos tiende a aumentar también para un mismo número de centroides k, puesto que si se mantiene k, entonces la distancia entre los vértices crece con el aumento de la dimensión n de la variedad. Obsérvese que este efecto se reduce conforme aumenta el número de centroides.
Conclusiones y trabajo futuro
Los experimentos realizados conducen a dos resultados: primero, el modelo basado en n-símplex para aproximar la variedad topológica que cubre el dominio 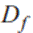 de formas válidas permite alcanzar cualquier forma con un desvío promedio de menos de un pixel por hito entre las formas válidas y sus proyecciones a dicha variedad. Segundo, la estrategia de caminata aleatoria propuesta, que utiliza saltos entre subdominios vecinos, permite generar secuencias de movimientos de serpenteo realistas y (como consecuencia directa del primer punto) capaces de alcanzar en el proceso cualquier forma válida.
de formas válidas permite alcanzar cualquier forma con un desvío promedio de menos de un pixel por hito entre las formas válidas y sus proyecciones a dicha variedad. Segundo, la estrategia de caminata aleatoria propuesta, que utiliza saltos entre subdominios vecinos, permite generar secuencias de movimientos de serpenteo realistas y (como consecuencia directa del primer punto) capaces de alcanzar en el proceso cualquier forma válida.
Las características de las formas generadas en la caminata aleatoria dependerán tanto de la dimensión n de la variedad, que determina el número de vértices de cada símplex, como del número k de centroides utilizados como candidatos a vértices de los símplex.
Estas características le permiten al método propuesto generar formas aleatorias que pueden ser utilizadas en procesos de segmentación y clasificación de siluetas vermes. De igual manera, el método de deformación propuesto es capaz de generar formas vermes distintas a las existentes en el cuerpo de entrenamiento.
La distancia entre formas consecutivas en la secuencia de pasos es ajustable con el método propuesto por medio de los parámetros n (dimensión de la variedad topológica) y k (total de conglomerados cuyos centroides constituyen los posibles vértices de los n-símplex). Dichos parámetros controlan además la complejidad total del proceso, para integrar el método en otras estrategias de segmentación y rastreo.