Introducción
Candida guillermondi, también conocida como Pichia guilliermondii o Meyerozyma guilliermondii [1], es una levadura ascomicete ampliamente distribuida en el medio ambiente natural, es de fácil aislamiento a partir de frutas y bayas, con alto contenido de azúcar y también, es parte de la piel humana y la microflora mucosa[2].
A esta levadura se le atribuyen varias propiedades que le hacen ser de interés biotecnológico, por ejemplo, para control biológico [3], para la bioconversión de xilosa en xilitol [4] a partir de hidrolizados hemicelulósicos, como fuente de energía [5]. Así mismo, C. guilliermondii es una fuente prometedora de enzimas, por ejemplo, inulinasa [6] y glutaminasa [7], así como biocombustibles [8], generación de aromas y su importancia clínica en la sobreproducción de riboflavina (RF, vitamina B2) [4]. Además, tiene potencial para ser utilizada en un proceso de fermentación en estado sólido para obtener biomasa proteica para consumo humano y animal [9].
Sin embargo, esta especie de levadura también tiene efectos negativos, por ejemplo en la industria del vino la presencia masiva de C. guilliermondii reduce o detiene el proceso de fermentación y aumenta la concentración de alcohol y acetoína, por lo tanto es importante usar uvas sanas en la cosecha y filtrar el mosto si fuera necesario para reducir la presencia de C. guilliermondii[10].
Múltiples levaduras pueden experimentar cambios en sus patrones replicativos y morfología, para producir células elongadas enlazadas formando filamentos. La formación de filamento puede ser inducida por limitación o ausencia de nitrógeno, crecimiento en baja fuente de nitrógeno [11] o crecimiento en presencia de bajas concentraciones de alcoholes aromáticos [9].
Este cambio morfológico recibe el nombre de dimorfismo fúngico, que está definido como la habilidad que tienen ciertos hongos de alternar entre un crecimiento unicelular (levaduriforme) y otro multicelular (filamentoso), en respuesta a cambios del entorno [12]. Los estímulos ambientales generan una variedad de signos detectables, que cuando percibidos por los hongos dimórficos, desencadenan cambios morfológicos como una forma de adaptarse a estas nuevas condiciones prevalecientes [13].
Numerosos parámetros químicos y ambientales pueden inducir el dimorfismo fúngico, entre ellos están: la temperatura, pH, niveles de glucosa, fuente de nitrógeno, niveles de dióxido de carbono, metales de transición, agentes quelantes y el tamaño del inóculo o densidad celular inicial [14].
La morfología de los cultivos (levadura o micelio), que representa dos estrategias de crecimiento diferentes relacionadas con ambientes y condiciones particulares, es un factor importante y determinante de los mecanismos de acción de los hongos incluyendo su virulencia o patogenicidad [15]. Los hongos filamentosos, caracterizados por su agresividad y capacidad de penetración de la superficie celular donde crecen, son un claro ejemplo de este fenómeno [16]. Además, constituye un elemento fundamental a la hora de evaluar aplicaciones biotecnológicas de este tipo de organismos.
Para esta investigación el microorganismo de interés fue aislado a partir de Panicum maximun, un zacate o pasto común en plazas de futbol, en orillas de carreteras, en potreros, en lotes baldíos. Es una hierba perenne, que puede alcanzar hasta 3 m de altura. Los tallos son erectos y ascendentes con una vena central pronunciada. La inflorescencia se presenta en forma de panoja abierta de 12 a 40 cm de longitud. Las raíces son fibrosas y largas. Se utiliza generalmente para el pastoreo, corte y acarreo y como barreras vivas, aunque muchos la consideran maleza [17].
El objetivo de este estudio fue analizar el efecto de diferentes condiciones de crecimiento en la morfología de Candida guillermondi aislado de Pannicum maximun, así como su identificación molecular, con el fin de caracterizar cepas de este organismo para aplicaciones biotecnológicas futuras.
Materiales y métodos
Aislamiento y purificación de la levadura
Se colectó Panicum maximun de la zona central y al noroeste de Costa Rica. Se tomaron solo las plantas cuya inflorescencia presentaba crecimiento de un hongo tipo carbón en sus panículas (figura 1). Una vez en el laboratorio, el material fue inoculado en medio papa dextrosa agar (PDA) a 25°C, para el aislamiento de los hongos presentes. Al cabo de tres días, se realizó la identificación al microscopio (40X) de cada uno de los hongos presentes, y se aislaron los que presentaban una morfología macroscópica y microscópica esperada para Candida guillermondi. Dichas evaluaciones y aislamientos se realizaron periódicamente durante ocho días de cultivo, hasta lograr su purificación en placas con medio PDA nuevo.
Establecimiento en medio líquido
Se probaron varios medios líquidos Papa Dextrosa Broth (PDB), Vogels [18] y Medio Mínimo [18]. Se inocularon cinco colonias aisladas por cada 50 mL de medio de cultivo nuevo, en matraces de 250mL. Los cultivos se mantuvieron a 100 rpm a temperatura ambiente, durante 48 horas.
Análisis genético e identificación de la levadura
Los cultivos se mantuvieron en medio líquido Papa Dextrosa Broth (PDB), en agitación durante 7 días, al cabo de los cuales, se colocaron en tubos para centrífuga de 50mL. Se centrifugaron a 5000 revoluciones por minuto (rpm) durante 5 minutos, se descartó el sobrenadante. De cada uno de los cultivos montados, se tomaron muestras entre 100 y 250 mg de tejido. Las muestras se lavaron tres veces con agua peptonada al 1%, con el fin de eliminar los restos de medio de cultivo. Una vez limpio se procedió a hacer la extracción de ADN con el kit Wizard® Genomic DNA Purification de la marca Promega, utilizando el protocolo 3.F., descrito en el manual del fabricante para extracción de ADN genómico a partir de Levadura, con las siguientes modificaciones: en el punto 4 del protocolo, se utilizó la enzima lisozima de la cual se agregaron 560 unidades. La incubación posterior fue a 37 °C por 45 minutos. Y en el punto 7, luego de agregar el Buffer de lisis nuclear, se maceró durante 1 minuto a 30 golpes por segundo, al macerado se le agregaron nuevamente 560 unidades de lisozima, se incubó a 37°C por 15 minutos, y se volvió a macerar durante 1 minuto a 30 golpes/segundo.
La cuantificación del ADN de cada una de las muestras se hizo por medio del Nanodrop Lite de Thermo Scientific, y de forma paralela se hizo una electroforesis en gel de agarosa al 1%, para verificar su integridad. Para la amplificación por PCR (Reacción en Cadena de la Polimerasa), se sintetizaron los imprimadores descritos por White et al [19] y Bakkeren [20] con la empresa IDT, que amplifican las regiones ITS 1 y 2. Sin embargo, estos imprimadores son universales para hongos, por lo que no discriminan entre los diferentes géneros. Razón por la cual se realizó la secuenciación de los amplicones obtenidos para su posterior identificación. Las secuencias de los imprimadores utilizados se muestran en el Cuadro 1.
Se realizaron las pruebas de PCR con los diferentes imprimadores, utilizando un perfil térmico de 95°C por 2 minutos para la desnaturalización inicial, seguido de 30 ciclos de 94°C de desnaturalización por 30 segundos, 49°C para la unión de los imprimadores durante 45 segundos y finalmente 72°C para la elongación por 2 minutos, finalizando con un ciclo de 72°C durante 10 minutos para la elongación final. La concentración del amplicón obtenido se determinó con el nanodrop lite, mientras que la integridad se evaluó por electroforesis en gel de agarosa al 1,5%.
Cuadro 1 Secuencias de imprimadores para regiones ITS 1 y 2 en hongos, descritos por White et al [19] y Bakkeren [20].
| Nombre | [61]Secuencia | [62]Tamaño esperado (bp) | [63]Autor |
|---|---|---|---|
| ITS 4 | [66]TCCTCCGCTTATTGATATGC | [67]500-750 | [68]White |
| ITS 6 | [71]GAAGGTGAAGTCGTAACAAGG | [72]500-750 | [73]White |
| UNUP18S42 | [76]CGTAACAAGGTTTCCGTAGGTGAAC | [77]~1000 | [78]Bakkeren |
| UNLO28S576B | [81]CTCCTTGGTCCGTGTTTCAAGACG | [82]~1000 | [83]Bakkeren |
Las bandas generadas por los imprimadores ITS 4 y 6 brindaron una banda más intensa y en el caso de los imprimadores descritos por Bakkern, generan bandas cercanas a las 1000 pares de bases (bp), lo que permitió tener mayor número de bases para hacer el análisis comparativo para identificación del hongo.
Los amplicones obtenidos se enviaron a secuenciar mediante la empresa Macrogen en Estados Unidos y un segundo análisis con el Centro Nacional de Investigaciones Biotecnológicas (CENIBiot) en Costa Rica. Las secuencias generadas fueron comparadas mediante un alineamiento utilizando la herramienta Blast del National Center for Biotechnology Information (NCBI) y comparando con la base de datos del Gen Bank, con el fin de obtener un porcentaje de similitud que indicó el género al que pertenecía el organismo aislado.
Inducción de crecimiento pseudomicelial de C. guillermondi
Para logar un crecimiento micelial a partir de las levaduras, se realizaron pruebas en cuanto a la composición del medio de cultivo (Cuadro 2) y las condiciones de crecimiento, en medio sólido.
Entre las condiciones de cultivo que se probaron fue la temperatura, pH, luz. En cuanto a la temperatura se evaluaron diferentes temperaturas de incubación: 25°C, 28°C y 30°C, la prueba se realizó por triplicado, durante siete días. El pH se probó a 3,5 y a 5,8. Se probó luz difusa y a la oscuridad tapados con una manta oscura, ambos durante 7 días de evaluación.
Cuadro 2 Medios utilizados para inducción de pseudomicelio en los aislamientos obtenidos.
| Prueba | [98]Medio de cultivo | [99]Composición |
|---|---|---|
| 1 | [102]Medio agar-agua | [103]Agua y agar |
| 2 | [106]Medio con carbón activado | [107]Agua-carbón activado - agar |
| 3 | [110]Diferentes fuentes de carbono | [111]Glucosa-sacarosa-aceite-sin nada* |
| 4 | [114]Medio con diferentes fuentes de nitrógeno | [115]Peptona-extracto de levadura-arginina-sin nada* |
| 5 | [118]Medio pobre (sin algunos macro o micro) | [119]CuSO4, ZnCl2,MnCl2, FeCl3, H3BO3 y Na2MoO4 |
| 6 | [122]Medio Vogels | [123]Handbook of microbiological media (Atlas, 2010) |
| 7 | [126]Medio PDA | [127]Papa Dextrosa-Agar |
| 8 | [130]Medio Mínimo | [131]glucosa, KNO3 , solución salina, sin fuente de nitrógeno ni vitaminas |
*Utilizando como medio base Agar-agua
Cada una de las levaduras con presencia del diformismo, se observaron al estereoscopio y al microscopio para su identificación.
Resultados y discusión
El Panicum maximun recolectado fue identificado por el botánico Armando Ruiz Boyer del Herbario Nacional del Museo Nacional de Costa Rica. Se procuró que el zacate, presentara en sus inflorescencias un hongo negro, tipo carbón (Figura 1).
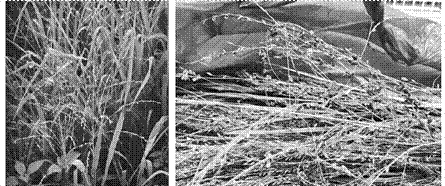
Figura 1 Panicum maximun en la provincia de Cartago colectado con hongo tipo carbón en su inflorescencia.
Como era lo esperado, del Panicum maximun (zacate callejero) crecieron varios hongos (Figura 2-A), se purificaron los que presentaban características morfológicas similares a una levadura. La observación macroscópica de las levaduras aisladas a las 48h fueron colonias pequeñas (de 3 a 5 µm, aproximadamente), ovoides y color crema (Figura 2-B). Posteriormente se observaron conglomerados característicos a las 96 h de incubación (Figura 2-B), color rosa. En la figura 3, se observan el aspecto microscópico de C. guillermondii en el medio PDA.
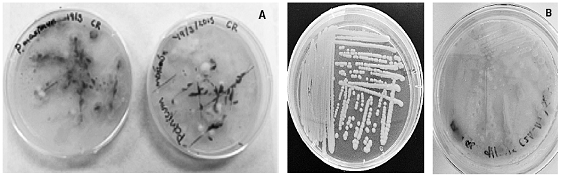
Figura 2 A) Placas de PDA con segmentos de P. maximum a los 3 días de crecimiento. B) Colonias aisladas características de levadura a las 48h y a las 120h
Aunque los métodos tradicionales de identificación de microorganismos se basan en la determinación de sus características morfológicas macroscópicas y microscópicas, así como por sus requerimientos nutricionales, no confirman en un 100% que es el microorganismo de interés. Por ejemplo; Pinoni, N. y colaboradores [21], reportan que tanto la Candida glabrata como Candida guilliermondii y C. famata presentan una coloración crema-rosado en sus morfología, lo cual no resulta útil para la diferenciación entre especies ya que este color también es observado en Candida parapsilosis, Candida lusitaniae, Saccharomyces cerevisiae, Prototheca wickerhamii. Es por lo anterior, que en esta investigación se realizaron técnicas moleculares para logar la identificación molecular.
Con el análisis molecular realizado las concentraciones de ADN obtenidas por el protocolo de extracción descrito, fueron muy variables, obteniéndose una concentración mínima de 12,2 ng/mL y una máxima de 903,4 ng/mL. Los resultados de las electroforesis, donde se obtuvo una banda definida después de la amplificación por PCR para la mayoría de las muestras se resumen en el Cuadro 3 y en la Figura 4, donde se observan como los amplicones obtenidos al utilizar los imprimadores 4 y 6 generaron bandas de mayor intensidad, las mismas se ubicaron entre los 500 y 750 pares de bases (bp) que coincide con lo esperado.
Los alineamientos mediante Blast, dieron como resultado que la secuencia para las levaduras aisladas, correspondieron todas al microorganismo Meyerozyma guilliermondii, también denominado Candida guilliermondii, con un 100% de identidad y cobertura. De acuerdo a Savini y colaboradores [22]C. guilliermondii en su estado telemórfico también recibe el nombre de Pichia guilliermondii[23]. Se encuentra distribuido ampliamente en la naturaleza y puede ser aislado del suelo, plantas, insectos, agua de mar y exudados de varios árboles, siendo además parte la microflora humana. Esto apoya el hecho de que las levaduras fueron aisladas a partir de material vegetal del campo.
Cuadro 3 Concentración y pureza obtenida en las muestras del hongo aislado después de la extracción de ADN. Datos generados mediante análisis con nanodrop lite.
| Muestra | [158]Concentración (ng/mL) | [159]Relación 260/280 |
|---|---|---|
| 1 | [162]91,0 | [163]1 , 60 |
| 2 | [166]200,9 | [167]1 , 72 |
| 3 | [170]740,5 | [171]1 , 96 |
| 4 | [174]903,4 | [175]1 , 96 |
| 5 | [178]84,4 | [179]1 , 74 |
| 6 | [182]54,7 | [183]1 , 67 |
| 7 | [186]38,7 | [187]1 , 69 |
| 8 | [190]12,2 | [191]1 , 86 |
| 9 | [194]93,1 | [195]1 , 38 |
| 10 | [198]140,2 | [199]1 , 61 |
| 11 | [202]64,9 | [203]1 , 57 |
| 12 | [206]116,1 | [207]1 , 60 |
| 13 | [210]129,5 | [211]1 , 51 |
| 14 | [214]108,2 | [215]1 , 52 |
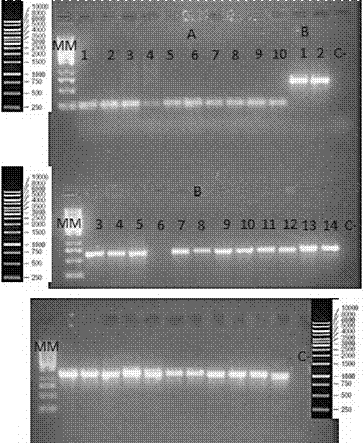
Figura 4 Electroforesis en gel de agarosa al 1,5% de producto de PCR de hongo aislado a partir de Panicum máximum.
La primera línea muestra las amplificaciones obtenidas con los imprimadores ITS 1 y 2 correspondientes a las muestras 1 a 10 (A). Mientras que las muestras señaladas como 1 a 14 (B) muestran las amplificaciones obtenidas con los imprimadores 4 y 6. La tercera línea muestra los amplicones obtenidos con los imprimadores UNUP18S42 y UNLO28S576B. MM señala el marcador de peso molecular Gene Ruler 1Kb de Fermentas y C- indica el control negativo.
Así mismo, la prueba ureasa confirmó la presencia de dos grupos de microorganismos en el aislamiento, donde el resultado dio “ureasa positivo” lo cual indica presencia de ascomycetes, coincidiendo con C. guilliermondii quienprecisamente es un ascomycete [2].
Del Panicum maximun también se aisló e identificó por medio del análisis de las secuencias en Blast con un 99% de identidad y 100% de cobertura: Fusarium sterilihyphosum, Fusarium succisae y Fusarium sp. Dado a que se necesitan más estudios para determinar la especie se concluye que otros organismos aislados del P.maximun corresponden al género Fusarium.
El uso de estas técnicas para el análisis molecular permitió la identificación con exactitud del microorganismo de interés. Desde 1990, White y colaboradores [19], publicaron un artículo en el que se describía la secuencia de 23 imprimadores, cinco de los cuales se basan en la región ITS (Internal Transcribed Spacer), para la identificación de hongos, los cuales han sido ampliamente usados con este fin.
Una vez identificado el microorganismo se procedió a la inducción del crecimiento dimórfico del hongo ascomicete. No todos los microorganismos tienen la capacidad de sufrir un cambio morfológico, en esta investigación para lograrlo se estresó el microorganismo, proporcionándole condiciones de cultivo y medios de cultivo pobres, diferentes a los que está acostumbrado a crecer. En cuanto a las condiciones de cultivo se probaron diferencias en temperatura, pH y luz, en general, los estímulos ambientales generan una variedad de signos detectables, que cuando son percibidos por los hongos dimórficos, desencadenan cambios morfológicos como una forma de adaptarse a estas nuevas condiciones prevalecientes [13]. El pH y la luz, no fueron condiciones determinantes para la aparición de hifas en las levaduras. Por otro lado, con la variación de la temperatura en los cultivos del hongo en los medios probados si se observaron colonias con pseudomicelio, pero aun así ninguno presentó micelio. En el Cuadro 4, se resumen las pruebas realizadas a diferentes temperaturas, con el fin de inducir la formación de micelio, el crecimiento levaduriforme o ambas (levaduras con pseudomicelio). Como el hongo C.guillermondii crece a temperatura ambiente, en su ambiente natural, se realizaron las pruebas aumentando temperatura 25°C a 30°C.
Cuadro 4 Evaluación del crecimiento exhibido por C. guillermondii a diferentes temperaturas.
| T °C | [229]PDA | [230]Agar agua | [231]Vogels | [232]Sin carbono | [233]Sin nitrógeno | [234]Medio Pobre | [235]Carbón activado |
|---|---|---|---|---|---|---|---|
| 25°C | [238]+/+ | [239]+/+ | [240]+/+ | [241]+/- | [242]+/+ | [243]+/+ | [244]+/- |
| 28°C | [247]+/++ | [248]+/++ | [249]+/++ | [250]+/+ | [251]+/+ | [252]+/+ | [253]+/- |
| 30°C | [256]+/+ | [257]+/+ | [258]+/++ | [259]+/+ | [260]+/+ | [261]+/+ | [262]+/- |
+/: Crecimiento levaduriforme. /+: Crecimiento pseudomicelial. /++: Abundante crecimiento pseudomicelial. /- : Ausencia de crecimiento pseudomicelial.
Hornby y colaboradores [24], recomienda además, de las condiciones ambientales como influyentes en el dimorfismo fúngico, también menciona niveles de glucosa, fuente de nitrógeno, niveles de dióxido de carbono, metales de transición, agentes quelantes y el tamaño del inóculo o densidad celular inicial.
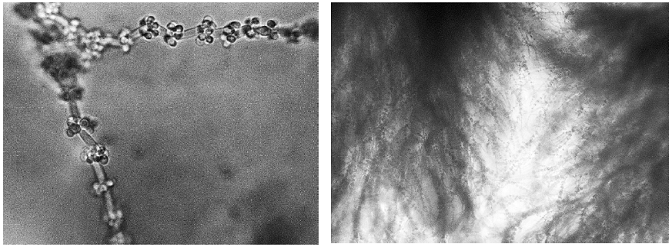
Se observó crecimiento pseudomicelial en los medios sólidos agar agua, suplementado con sacarosa, el que no contenía fuente de nitrógeno y Vogels, los otros medios únicamente exhibieron crecimiento levaduriforme (Figura 5). Así mismo, en la prueba con medio agar-agua con secciones de P. máximum (hoja + tallo + inflorescencias), fueron las que exhibieron mayor crecimiento pseudomicelial en comparación a las cepas en medios con secciones de cada tipo individual.
Entre la composición de los medios de cultivo en los que creció más pseudomicelio, no existe relación entre ellos, todos son medios sencillos, carentes de muchas cosas, más unos que otros. Sin embargo, aunque se logró observar que las colonias empezaron a presentar pseudomicelio, en estos medios, nunca se logró el crecimiento de un micelio completo, en ninguno. Harrison y Sherwood [24] también, utilizaron el medio mínimo Vogels con 1% de carbón activado para determinar la habilidad de los diploides para formar micelio. Sus resultados muestran que los diploides fueron capaces de formar micelio en un medio con carbón activado; mientras que en esta investigación no exhibieron crecimiento alguno.
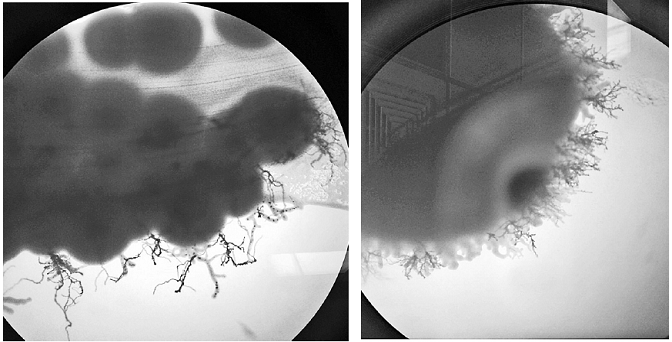
Para asegurar el crecimiento de la levadura en el P. maximun se cultivó un inoculo de las levaduras en medio agar agua con P. maximum autoclavado, permitiendo observar un crecimiento pseudomicelial a los seis días de realizado el inóculo. Donde, dicho comportamiento se presentó en todas las colonias exhibidas en el tratamiento que contenía hoja, fruto maduro e inmaduro (Figura 6). El medio agar agua fue muy útil para evaluar el crecimiento del hongo y observar etapas del mismo.
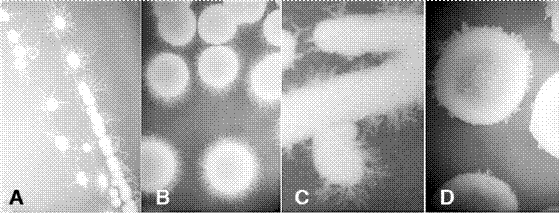
Figura 5 Crecimiento pseudomicelial en los medios sólidos: A) agar agua, B) suplementado con sacarosa, C) sin ninguna fuente de nitrógeno y D) medio Vogels.
Conclusiones
Se logró aislar y purificar Candida guillermondii a partir de un zacate común. El microorganismo fue identificado por medio de técnicas moleculares, siendo un método efectivo para evidenciar que el microorganismo aislado, corresponde al organismo de interés. Y por medio de variaciones en las condiciones normales de crecimiento se observó el dimorfismo característico para estos géneros. Es recomendable evaluar otras composiciones del medio de cultivo como variaciones en las condiciones de crecimiento para lograr el desarrollo del micelio.