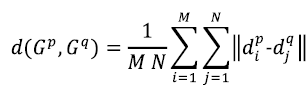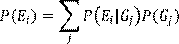Introducción
Las abejas son insectos con alas de la familia Apoidea, orden Hymenoptera. Existen más de 20.000 especies documentadas alrededor del mundo (Michener, 2007). Se consideran el grupo de polinizadores más importantes, tanto de plantas silvestres como de cultivos (Quesada Euan, 2009). Existen numerosos estudios que demuestran su importancia económica en la industria agrícola, pero su aporte al ecosistema natural es difícil de cuantificar. Según proyecciones de la Organización de las Naciones Unidas (ONU), para el año 2050 la población mundial será de más de 9.000 millones de personas, lo que implicará una gran demanda de alimentos, y de ahí la importancia de las abejas para la supervivencia humana y la conservación de la biodiversidad. No obstante, la sobreexplotación de los bosques y el uso de pesticidas han causado la disminución en el número de colonias de Apis mellifera (Apidae: Apini) y efectos adversos no cuantificados en otras especies, entre ellas las abejas nativas sin aguijón (Apidae: Meliponini) (Meléndez, Meneses & Kevan, 2013; Thompson, 2012; Whitehorn, O’Connor, Walkers & Goulson, 2012).
Las abejas nativas sin aguijón tienen un aguijón atrofiado que no pueden utilizar para la defensa. Se conocen más de 400 especies, de las cuales 300 se encuentran en los trópicos y subtrópicos de América. En Costa Rica hay al menos 20 géneros y 59 especies (Camargo & Pedro, 2007) y la mayoría habitan por debajo de los 1500 msnm. Las especies más utilizadas en Costa Rica para la meliponicultura son Tetragonisca angustula y Melipona beecheii.
El cuerpo de una abeja se compone de tres partes: cabeza, tórax y abdomen. El tórax está formado por el protórax, el mesotórax y el metatórax. Cada una de estas partes tiene un par de patas. Las alas de las abejas son membranosas y crecen en el tórax, un par en el mesotórax y otro par en el metatórax. Las membranas superior e inferior de las alas cubren una red de tubos endurecidos, llamados nervios o venas, que le dan rigidez a éstas.
Trabajos relacionados
Tradicionalmente, para la identificación de especies, el experto -melitólogo- utiliza una clave dicotómica (clave taxonómica) que consta de un número de pasos organizados, cada uno de los cuales tiene dos opciones mutuamente excluyentes. Conforme se avanza en la identificación, estas opciones se van descartando. Las claves dicotómicas utilizan características fáciles de observar en el espécimen y con pares de afirmaciones contrapuestas como, por ejemplo, “ojos labros” contra “ojos con pelos”.
Se ha demostrado que es posible identificar especies de insectos usando únicamente la morfometría del ala, ya que su variabilidad entre especies es mayor que dentro de la misma especie. Incluso las diferencias permiten discriminar entre subespecies (Hall, 2011; Tofilski, 2008a; Tofilski, 2008b; Tüzün & Yüksel, 2011). Las alas de los insectos son membranosas y poseen venas o nervaduras visibles, lo que permite la creación de un diagrama claro de su estructura. El uso de indicadores morfométricos ha cobrado relevancia recientemente (Breuker, Gibbs, Van Dongen, Merckx & Van Dyck, 2010; Hall, 2011; Hall, MacLeod & Wardhana, 2014), lo que ha motivado el desarrollo de sistemas de clasificación que utilizan las características morfométricas del ala (Abou Shara & Al Ghamdi, 2012; Arbuckle, Schröder, Steinhage & Wittmann, 2001; Breuker, Gibbs, Van Dongen, Merckx & Van Dyck, 2010). En general, estos sistemas identifican las especies mediante métodos morfométricos basados en el análisis multivariado de distancias y ángulos. En el cuadro 1 se muestra un resumen de algunos de estos sistemas y sus principales características.
Como se puede observar, existen varios sistemas capaces de clasificar especies de abejas de forma eficiente. Sin embargo, todos tienen limitaciones, entre ellas están que ninguno se ha desarrollado específicamente para la discriminación de abejas sin aguijón, por lo general discriminan pocas especies y géneros, en su mayoría no son completamente automáticos, requieren intervención activa de un usuario, requieren de una gran cantidad de información y algunos demandan muchas horas de entrenamiento.
Debido a esto, se considera que el desarrollo de un sistema automático de discriminación de abejas sin aguijón a partir de una fotografía del ala es relevante. En el presente trabajo se propone un sistema de fácil uso, ya que únicamente requiere una fotografía de un ala derecha anterior y con ella se logra identificar la especie más probable a la que pertenece el ala.
Cuadro 1 Sistemas para la clasificación automática de los insectos.
| Sistema | Observaciones | Características |
|---|---|---|
| Fast Africanized Bee Identification System (FABIS) | Uno de los primeros sistemas, se desarrolló en la década de los 80 para clasificar abejas africanizadas. | Requiere alrededor de 25 medidas morfométricas y un proceso de entrenamiento. |
| Automated Bee Identification System (ABIS) | Propuesto en 1991, sin duda es el mejor sistema de identificación de abejas. Diseñado para las alas anteriores. | Reporta un 99,8% de exactitud en la identificación por género y un 95% de exactitud en la identificación por especies. Utilizaba una gran cantidad de información del ala con la que realizaba luego un análisis estadístico para la posterior identificación. Actualmente, el proyecto está inactivo (Arbuckle, Schröder, Steinhage & Wittmann, 2001). |
| MelittO Biotaxis System (MOBS) | Propuesto en 2011. A diferencia del ABIS, redujo considerablemente las características requeridas para la identificación, utilizando alrededor de 20 puntos clave. | Reporta una exactitud de 90% a 100% en la identificación de géneros. Utiliza las características anatómicas más relevantes y la venación del ala, junto con técnicas como ACP (Hall, 2011). |
| Draw Wing | Su uso fue impulsado en 2007. Se utiliza como un complemento de los sistemas anteriores. Diseñado para alas anteriores de insectos. | Extrae información de la imagen de un ala que se puede utilizar para la identificación de especies. Reporta 95% de exactitud. Requiere escanear la muestra. (http://www. drawwing.org/ ). |
| tpsBias, tpsDig, tpsPLS, tpsPower, tpsRelw, tpsRegr | Conjunto de herramientas para el análisis morfométrico y estadístico de alas de insectos. | Desarrolladas por la Universidad de Stony Brook. (http://life.bio.sunysb.edu/morph/ ) |
Base de datos y colección de especímenes
Las colecciones de especímenes de abejas son fundamentales para realizar estudios taxonómicos y constituyen la base de muchos tipos de estudio sobre biodiversidad y población. La falta de colecciones entomológicas apropiadas que cubran el inventario de todo el país es una de las debilidades que se presenta al realizar este tipo de estudios.
En esta investigación, la creación de la base de datos incluyó la preparación de tres colecciones: una de especímenes de abejas montadas en alfileres entomológicos, otra de alas derechas anteriores y posteriores montadas en un medio de preservación inerte y una de fotografías de estas placas.
Colección de especímenes
Parte de los especímenes utilizados se tomó de la colección de abejas nativas sin aguijón del Centro de Investigaciones Apícolas Tropicales (CINAT). Se revisaron todos los especímenes para seleccionar aquellos cuya ala derecha, anterior y posterior estuviera en perfectas condiciones. Para las especies de las que no se obtuvieron individuos con sus alas en perfectas condiciones, fue necesario realizar visitas de campo para ubicar nidos, de donde se recolectaron los especímenes necesarios para la investigación.
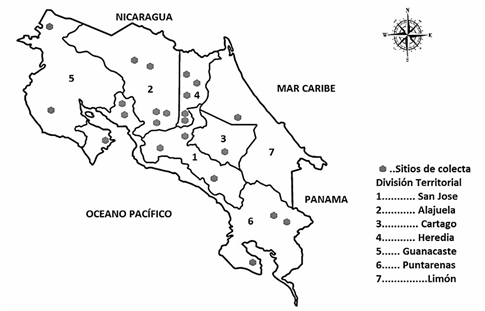
Las abejas se recolectaron directamente de los nidos en diferentes zonas geográficas de Costa Rica (figura 1), utilizando red entomológica y succionadores de plástico. Se usó una “cámara de muerte” con acetato de etilo para matar rápidamente al insecto y evitar daños en las alas. Las muestras se trasladaron al CINAT, donde se realizó el proceso de preservación de los ejemplares, el montaje y la identificación respectiva. Para cada ejemplar se registró la información necesaria para su identificación taxonómica: país, provincia, cantón, fecha, nombre del recolector, muestra en nido o en flor, etc.
Colección de alas
Se extrajo el ala derecha anterior y posterior de las abejas, se cortó en la base del mesotórax con navajilla y tijeras de disección para facilitar el desprendimiento y no dañarla. Se quitó el residuo de la base del ala para su montaje en un portaobjetos con una gota de bálsamo de Canadá. Se usó una lámpara de 50 W para suavizar el bálsamo y permitir que la muestra quedara inmersa en el sustrato sin ser dañada. Se colocó el cubreobjetos con presión moderada para extraer las burbujas y evitar el desplazamiento del ala. Las alas en estudio se dejaron reposar sobre papel toalla por 24 horas antes de ser fotografiadas y almacenadas. La colección de alas comprende un total de 879 placas.
Colección de fotografías
Para la toma adecuada de las fotografías se estableció un protocolo (Herrera et al., 2013). Las imágenes fueron tomadas con un estereoscopio Olympus modelo SZX16 con una cámara integrada marca Olympus modelo DP72, con una fuente de luz circular intralux 4000-1 con una intensidad de entre 1400 y 1600fc, dependiendo de la muestra. La resolución en el milimétrico fue de 2x con una apertura focal de 1/4. Para la captura de imágenes se utilizó un sistema TWAIN con exposición manual, tiempo de exposición 60%, sensibilidad ISO 200 y una resolución Live 1360 x 1024 y un snap 4140 x 3096.
Las fotografías se almacenaron en una base de datos, agrupadas por especie. La colección comprende un total de 1758 fotografías.
Procesamiento de las imágenes
Para la sustracción del fondo de cada una de las fotografías se utilizaron histogramas, discriminación por umbrales y operadores morfológicos. Las fotografías presentaban dos tipos de fondos: uno con un tono gris y otro con presencia de azul. Para determinar el tipo de fondo de cada fotografía se utilizó el espacio de color HSV y una cota, calculada empíricamente. Una vez identificado el tipo de fondo, se hizo un filtrado mediante umbralización (Otsu) ajustado experimentalmente, para determinar cuáles pixeles se convertirían en blancos y cuáles en negros. En la figura 2 se muestra este proceso; la región en blanco de la fotografía (b) corresponde a lo que se identificó como ala y la fotografía (c) es el resultado del proceso de eliminación del fondo.
Algunas fotografías presentaban dos alas, siendo la de interés la ubicada más al centro de la imagen, según el protocolo establecido en Herrera et al. (2013). Para resolver esto, se calcularon los centros de masa de todas las regiones blancas y se eligió como el ala de interés aquella cuya distancia del centro de masa al centro de la imagen fuese menor.

Figura 2 (a) fotografía original de la colección, (b) fotografía obtenida luego del filtrado por umbral, conversión a blanco y negro y operaciones morfológicas, (c) resultado del proceso de eliminación de fondo.
Aprendizaje
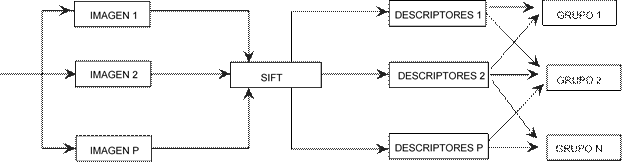
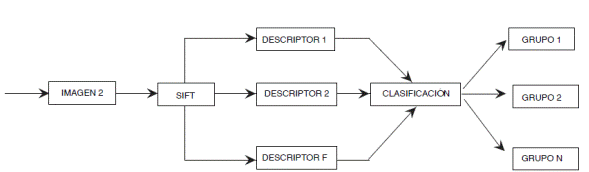
En la figura 3 se muestra el esquema del proceso de entrenamiento propuesto:
Para cada imagen de entrenamiento se extraen sus descriptores SIFT.
Se realiza un agrupamiento aglomerativo jerárquico de los descriptores extraídos.
Para cada descriptor en cada grupo se almacena su correspondiente especie.
Se espera que cada grupo sea representativo de una única especie, pero la práctica demuestra que esto no siempre sucede, ya que existen grupos que comparten descriptores de varias especies. Por lo tanto, fue necesario resolver esta ambigüedad en la etapa de clasificación.
Descriptores SIFT de puntos clave
El Scale Invariant Feature Transform (SIFT) es un transformación propuesta por David Lowe en 1999, cuya idea básica es representar una imagen mediante puntos clave (Lowe, 1999).
El método SIFT reduce la imagen a un conjunto de parámetros invariantes a la escala y rotación. Esto lo logra mediante la creación de pirámides de scale-space y la convolución con funciones gaussianas. La imagen obtenida por diferencia de gaussianas logra resaltar los bordes y esquinas de la imagen.
Una vez calculadas las diferencias gaussianas, se identifican los candidatos a máximos y mínimos entre los puntos que no se anularon. La comparación de cada punto con sus vecinos se hace en la escala próxima. Luego estos se discriminan por estabilidad.
Para asignar la orientación a los puntos clave (x, y) calculados en la etapa anterior, se utilizan la magnitud del gradiente y la orientación. Con la información obtenida se asigna a cada punto clave un conjunto de características, entre las que se destacan la posición, la orientación y las rotaciones del gradiente (para lograr invariancia respecto a rotaciones en 2D o cambios de puntos de perspectiva en 3D). Así, a cada punto clave se le asocia una matriz de 4x4x8, es decir, un vector de tamaño 128, llamado descriptor del punto clave.
Agrupamiento de los descriptores
Para construir los grupos de descriptores se usó el agrupamiento aglomerativo jerárquico. Según este método, dados F descriptores extraídos de las imágenes de entrenamiento, el agrupamiento se inicia con F grupos, cada uno conteniendo un único descriptor. En cada iteración, se unen los dos grupos con mayor proximidad.
Para determinar la proximidad entre grupos se utilizó la agregación de salto promedio con la métrica euclídea (Trejos, Castillo & González, 2014). Esta se define como la distancia promedio de todo descriptor de un grupo a todo descriptor del otro grupo:
Aquí, M y N son el número de descriptores de cada uno de los grupos G p y G q respectivamente, d p i es el i-ésimo descriptor del grupo G p y d p j es el j-ésimo descriptor del grupo G q .
El agrupamiento aglomerativo en cada iteración va uniendo grupos hasta que un criterio de parada detiene el proceso, después de lo cual se obtienen grupos donde la proximidad entre cada par de grupos está por encima de un valor dado. Este valor determina el criterio de parada, que en nuestro caso fue determinado experimentalmente.
Las principales desventajas del agrupamiento aglomerativo son sus excesivos requerimientos en tiempo de ejecución y de memoria, que son del orden de O (- F 2 ) .
Clasificación de una nueva imagen
Dada una nueva imagen, la clasificación se lleva a cabo de la siguiente forma:
Primero se extraen los descriptores SIFT de la imagen.
Luego, cada uno de estos descriptores se clasifica en uno de los grupos del modelo aprendido.
Por último, se determina la especie más probable a la que pertenece la imagen de entrada.
En la figura 4 se muestra el esquema del proceso de discriminación propuesto. Primero se extraen los descriptores d 1 , d 2 ,..., d F de la imagen que se desea identificar. Luego, mediante análisis discriminante lineal (Johnson, 2000), se clasifica cada uno de estos descriptores d i en uno de los grupos G j obtenidos en el proceso de entrenamiento. Por último, se seleccionan las k especies más probables para la imagen de entrada. Para esto se calculan las sumas de las probabilidades de ocurrencia dado cada uno de los grupos seleccionados en la etapa anterior:
Luego se eligen los k valores mayores para cada imagen de entrada. Aquí P(E i |G j ) es la probabilidad de que la especie sea E i dado el grupo G j y P(G j ) es la probabilidad del grupo G j .
El algoritmo fue programado en MatLab y se utilizó la biblioteca VLFeat para la obtención de los descriptores SIFT de los puntos clave de una imagen.
Resultados
La obtención y análisis de los resultados se hizo de la siguiente forma:
Se usó una muestra pequeña de imágenes para calibrar los procesos de eliminación de fondo y la obtención de los descriptores SIFT.
Se entrenó el algoritmo con una muestra de 8 imágenes por especie y se utilizaron 18 especies para un total de 144 imágenes, como se muestra en el cuadro 2.
Se validó el algoritmo con una muestra de 436 imágenes totalmente diferentes a las utilizadas en las etapas anteriores.
Cuadro 2 Totalidad de especies utilizadas en la etapa de entrenamiento.
| Cant. | Especie | Cant. | Especie |
|---|---|---|---|
| 8 | Cephalotrigona zexmeniae | 8 | Scaptotrigona pectoralis |
| 8 | Geotrigona lutzi | 8 | Scaptotrigona subobscuripennis |
| 8 | Lestrimelitta danuncia | 8 | Tetragona ziegleri |
| 8 | Melipona costaricensis | 8 | Trigona corvina |
| 8 | Nannotrigona melleria | 8 | Trigona fulviventris |
| 8 | Nannotrigona perilampoides | 8 | Trigona fuscipennis |
| 8 | Partamona orizabaensis | 8 | Trigona muzoensis |
| 8 | Plebeia frontalis | 8 | Trigona nigerrima |
| 8 | Plebeia jatiformis | 8 | Trigona silvestriana |
Para la etapa de validación se utilizó una muestra de 436 fotografías que corresponden a 18 especies de 10 géneros. Debido a que no se pudo recolectar una cantidad suficiente de especímenes para 6 de las especies, el algoritmo se entrenó con 24 especies y se validó con 18.
Los resultados del proceso de validación con k=1 se tabularon en una matriz de discriminación cruzada, como se muestra en el cuadro 3. Por ejemplo, de las 30 imágenes de la especie Partamona orizabaensis, 27 fueron identificadas correctamente, 2 fueron identificadas como de la especie Trigona fulviventris y 1 como de la especie Geotrigona lutzi. Se usaron colores para agrupar las especies de un género, por ejemplo, para las de Trigona se utilizó el color naranja.
En el cuadro 4 se resumen los resultados de la etapa de validación usando un valor de k=1 (considerando únicamente la especie más probable). El porcentaje promedio de éxito para la discriminación por especie fue de 76,3%, mientras que para la discriminación por género alcanzó 80,7%.
En el cuadro 4 observamos que la discriminación por género para el caso de Trigona es del 100%, no así la discriminación por especie, que es de un 70,7%, pero es importante notar que ninguna de las imágenes de esta especie está clasificada como de otra especie (cuadro 3). Esto sugiere que las alas de las especies de este género son muy similares entre sí, pero tienen cierto grado de diferencia con las alas de las especies de otros géneros.
Esto lleva a pensar que, con el propósito de poder discriminar entre especies de ciertos géneros, como en el caso de Trigona, tal vez sea necesario considerar otros discriminadores que no se encuentran en las alas como, por ejemplo, color del abdomen, vellosidad en las patas o antenas, etc.
Obsérvese que para 10 de las 18 especies y 7 de los 10 géneros el porcentaje de discriminación es superior o igual al 90%; en algunos de los trabajos consultados se obtienen porcentajes similares pero para menos especies (Francoy, Silva, Nunes-Silva, Menezes & ImperatrizFonseca, 2007; Abou Shara & Al Ghamdi , 2012; Tofilski, 2008b).
Los porcentajes de discriminación más bajos que se obtuvieron corresponden a las especies Melipona costaricensis y Tetragona ziegleri, las cuales en su mayoría son identificadas como alguna de las especie del género Trigona, 9 y 16, respectivamente (cuadro 3).
Es usual que los algoritmos de este tipo respondan con las dos opciones más probables; al permitir esto los resultados mejoran, siendo de 84,41% para discriminación por género y de 83,95% para discriminación por especie, como se muestra en el cuadro 5.
Cuadro 3 Matriz de discriminación cruzada.
| - | Ce. Ze | Ge. Lu | L. da | Me. co | Na. me | Na. pe | Par. Or | Pl. fr | Pl. ja | Scp. pe | Scp. su | Te. zi | Tri. co | Tri. Ful | Tri. fus | Tri. mu | Tri. ni | Tri. si |
| Ce. ze | 10 | - | - | - | - | - | - - | - | - | - | - | - | - | - | - | - | - | - |
| Ge. lu | - | 12 | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | |
| L. da | - | - | 30 | - | - | - | - | - | - | - | - | - | - | - | - | - | - | 2 |
| Me. co | - | - | 5 | - | - | - | - | - | 1 | - | - | - | 9 | - | - | - | - | |
| Na. me | - | - | - | 33 | - | - | - | - | - | - | - | - | - | - | - | - | ||
| Na. Pe | - | - | - | - | 4 | - | - | - | 1 | - | - | - | - | - | - | - | - | |
| Par. Or | - | 1 | - | - | - | 27 | - | - | - | - | - | - | 2 | - | - | - | - | |
| Pl. fr | - | - | - | - | - | - | 15 | - | - | - | - | - | - | - | - | - | - | |
| Pl. ja | - | - | - | - | - | - | - | 25 | - | - | - | - | - | - | - | - | - | |
| Scp. Pe | - | - | - | - | - | - | - | - | 34 | - | - | - | - | - | 1 | - | - | |
| Scp. Su | - | - | 1 | 4 | - | - | 1 | - | - | 4 | 12 | - | - | 4 | - | 3 | 1 | 1 |
| Te. zi | - | - | 2 | - | - | - | - | - | 2 | - | 4 | 4 | 6 | 3 | 1 | - | 2 | |
| Tri. co | - | - | - | - | - | - | - | - | - | - | - | 10 | 16 | - | - | 2 | 1 | |
| Tri. Ful | - | - | - | - | - | - | - | - | - | - | - | - | 32 | - | 2 | - | ||
| Tri. Fus | - | - | - | - | - | - | - | - | - | - | - | 2 | 17 | 9 | - | 2 | 1 | |
| Tri. Mu | - | - | - | - | - | - | - | - | - | - | - | 6 | - | 26 | - | |||
| Tri. Ni | - | - | - | - | - | - | - | - | - | - | - | 1 | 1 | - | - | 12 | ||
| Tri. Si | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | 1 | 28 |
Cuadro 4 Porcentaje de éxito considerando las especies más probables, k=1 .
| # | Género | Especie | Código | Muestras | Errores | Éxito por especie | Éxito por género |
|---|---|---|---|---|---|---|---|
| 1 | Cephalotrigona | zexmeniae | Ce.ze | 10 | 0 | 100 | 100 |
| 2 | Geotrigona | lutzi | Ge.lu | 12 | 0 | 100 | 100 |
| 3 | Lestrimelitta | danuncia | L.da | 32 | 2 | 93.75 | 93.75 |
| 4 | Melipona | costaricensis | Me.co | 15 | 10 | 33.33 | 33.33 |
| 5 | Nannotrigona | melleria | Na.me | 33 | 0 | 100 | 97.37 |
| 6 | perilampoides | Na.pe | 5 | 1 | 80 | ||
| 7 | Partamona | orizabaensis | Par.or | 30 | 3 | 90 | 90 |
| 8 | Plebeia | frontalis | Pl.fr | 15 | 0 | 100 | 100 |
| 9 | jatiformis | Pl.ja | 25 | 0 | 100 | ||
| 10 | Scaptotrigona | pectoralis | Scp.pe | 35 | 1 | 97.14 | 75.6 |
| 11 | subobscuripennis | Scp.su | 31 | 19 | 38.7 | ||
| 12 | Tetragona | ziegleri | Te.zi | 24 | 20 | 16.7 | 16.7 |
| 13 | Trigona | corvina | Tri.co | 29 | 19 | 34.48 | 100 |
| 14 | fulviventris | Tri.ful | 34 | 1 | 97.06 | ||
| 15 | fuscipennis | Tri.fus | 31 | 22 | 29.03 | ||
| 16 | muzoensis | Tri.mu | 32 | 6 | 81.25 | ||
| 17 | nigerrima | Tri.ni | 14 | 2 | 85.71 | ||
| 18 | silvestriana | Tri.si | 29 | 1 | 96.55 | ||
| Totales | 436 | 107 | 76.32 | 80.69 | |||
Cuadro 5 Porcentajes de éxito considerando las dos especies más probables, k= 2.
| # | Género | Especie | Código | Muestras | Errores | Éxito por especie | Éxito por género |
|---|---|---|---|---|---|---|---|
| 1 | Cephalotrigona | zexmeniae | Ce.ze | 10 | 0 | 100 | 100 |
| 2 | Geotrigona | lutzi | Ge.lu | 12 | 0 | 100 | 100 |
| 3 | Lestrimelitta | danuncia | L.da | 32 | 1 | 96.88 | 96.88 |
| 4 | Melipona | costaricensis | Me.co | 15 | 9 | 40 | 40 |
| 5 | Nannotrigona | melleria | Na.me | 33 | 0 | 100 | 97.37 |
| 6 | perilampoides | Na.pe | 5 | 1 | 80 | ||
| 7 | Partamona | orizabaensis | Par.or | 30 | 1 | 96.67 | 96.67 |
| 8 | Plebeia | frontalis | Pl.fr | 15 | 0 | 100 | 100 |
| 9 | jatiformis | Pl.ja | 25 | 0 | 100 | ||
| 10 | Scaptotrigona | pectoralis | Scp.pe | 35 | 1 | 97.14 | 75.6 |
| 11 | subobscuripennis | Scp.su | 31 | 15 | 51.61 | ||
| 12 | Tetragona | ziegleri | Te.zi | 24 | 15 | 37.5 | 37.5 |
| 13 | Trigona | corvina | Tri.co | 29 | 5 | 82.76 | 100 |
| 14 | fulviventris | Tri.ful | 34 | 0 | 100 | ||
| 15 | fuscipennis | Tri.fus | 31 | 19 | 38.71 | ||
| 16 | muzoensis | Tri.mu | 32 | 1 | 96.88 | ||
| 17 | nigerrima | Tri.ni | 14 | 1 | 92.86 | ||
| 18 | silvestriana | Tri.si | 29 | 0 | 100 | ||
| Totales | 436 | 69 | 83.95 | 84.41 | |||
Conclusiones
Hemos podido comprobar que es posible identificar el género y la especie de abejas nativas sin aguijón a partir de la fotografía del ala derecha anterior, con porcentajes de éxito que rondan el 84%.
Con base en la información dada por expertos en estudios apícolas, existen especies cuyas alas son muy similares entre sí, por lo que su identificación se realiza considerando otros aspectos como: color de ciertas partes del cuerpo, vellosidad, tamaño de las patas o antenas, etc. Por ejemplo, las alas de las especies Trigona silvestriana y Trigona fulviventris son muy similares pero se diferencian por la coloración del abdomen. Por eso, al incorporar algunos de estos discriminadores, los porcentajes de éxito podrían mejorar.
Aún quedan algunas tareas pendientes, como investigar las razones de que los porcentajes de discriminación para las especies Melipona costaricensis y Tetragona ziegleri sean los más bajos; evaluar otras técnicas para el proceso de clasificación de descriptores e incorporar más especies en los procesos de entrenamiento y validación.