Introducción
El pargo mancha (Lutjanus guttatus) es una especie de alta importancia comercial en pesquerías y cultivos acuícolas en Costa Rica, que se ha visto sometida a una fuerte presión pesquera (Soto-Rojas et al., 2018). Es un pez marino demersal que se distribuye desde el Golfo de California en México hasta Ecuador (Fischer et al., 1995). Esta especie se reproduce durante todo el año en dos picos que difieren según los sitios de muestreo (Arellano-Martínez et al., 2001; Rojas, 1996; Soto-Rojas et al., 2009) y posee desoves por periodos con desarrollo ovárico asincrónico (Pérez-Enríquez et al., 2020). En el país existe una empresa que lo cultiva de forma comercial (Peña-Navarro et al., 2019) y se han dado liberaciones programadas de alevines en programas de repoblamiento (Chacón-Guzmán et al., 2019).
Los estudios de L. guttatus en Costa Rica se han enfocado en el análisis de parámetros poblacionales (Soto-Rojas et al., 2018), hábitos alimenticios (Rojas et al., 2004; Soto-Rojas et al., 2018), hábitos reproductivos (Rojas, 1996; Soto-Rojas et al., 2009), crecimiento (Soto-Rojas et al., 2009) y fecundidad (Rojas, 1996). En el ámbito genético de las poblaciones, no existen reportes de muestras obtenidas en Costa Rica; sin embargo, un análisis basado en ADN mitocondrial indica homogeneidad genética entre las localidades de El Salvador y Panamá (Hernández-Álvarez et al., 2020).
La estructura genética y los niveles de variabilidad de las poblaciones de peces es un indicador del estado poblacional y pueden ser tomadas como referencia para programas de conservación (Muñoz et al., 2019). Dentro de los factores que pueden afectar los niveles de variabilidad genética de las poblaciones se encuentran las características del ambiente en el que viven, eventos históricos que ocurren en el sitio (Palma et al., 2007), sobrepesca, introducción de especies exóticas, deterioro del hábitat y fragmentación de los ecosistemas (Mancera et al., 2013). Los marcadores moleculares son herramientas de suma importancia para el estudio de poblaciones salvajes que se pueden utilizar para conocer las estructuras genéticas y establecer medidas de manejo, programas de restauración e identificación de stocks como posible fuente de reproductores para el desarrollo de tecnologías acuícolas (Povh et al., 2008).
Los microsatélites son marcadores moleculares que se caracterizan por ser secuencias de regiones de ADN que presentan repeticiones en tándem de bases nitrogenadas que pueden tener distinto tamaño. Se identifican mediante técnicas de amplificación de reacción de polimerasa en cadena (PCR) con M13 y distintos fluoróforos para sitios específicos seleccionados con cebadores diseñados para estas regiones (Schuelke, 2000). Estos han dado resultados adecuados para el estudio poblacional tanto en el medio silvestre como en cultivo de especies como camarón blanco (Pérez-Enríquez & Max-Aguilar, 2016) y en peces de la familia Lutjanidae, tales como Lutjanus peru (Molina-Quirón, 2022; Reguera-Rouzaud et al., 2021), Lutjanus synagris (Landínez et al., 2009), Lutjanus argentiventris (Reguera-Rouzaud et al., 2021) y Lutjanus guttatus (Pérez-Enríquez et al., 2020), pues presenta valores representativos según los objetivos de los estudios.
Debido a la poca información de la estructura genética del pargo mancha (L. guttatus) en Costa Rica, se pretende evaluar la estructura genética poblacional de dos poblaciones en el pacífico costarricense en el Golfo de Nicoya y el Golfo Dulce.
Materiales y métodos
Para este estudio, las muestras del pargo mancha (L. guttatus) se tomaron en el Golfo de Nicoya (9º44'21.3'' N & 84º48'55'' W) en septiembre de 2018 y marzo de 2019 y en el Golfo Dulce (8º28'32.2'' N & 83º12'29.4'' W) en agosto de 2019 (Tabla 1), las cuales se encuentran en la costa del Pacífico de Costa Rica separadas por aproximadamente 225 km. Se utilizaron 44 muestras de los peces capturados de cada una de las localidades, se obtuvo la longitud total y se estimó la edad de cada grupo por fecha de muestreo mediante la ecuación descrita por Soto-Rojas et al., (2018). Asimismo, se tomó una sección de la aleta caudal de aproximadamente 2 cm2 que se preservó con etanol al 80 % en tubos de microcentrifugación de 1.5 ml. Estas muestras se encuentran custodiadas por el Laboratorio de Patología Acuícola (LAPA) de la Universidad Técnica Nacional, Sede Pacífico. Para la extracción del ADN, se utilizó un kit comercial (Qiagen, DNeasy Blood and Tissue Kit) y se procedió a seguir la metodología propuesta por el fabricante. Se realizó la cuantificación del ADN y la medición de la pureza por fotometría mediante un biofotómetro (Eppendorf, D30).
Tabla 1 Localidades de muestreo de Lutjanus guttatus en la costa del Pacífico de Costa Rica. / Table 1. Sampling locations of Lutjanus guttatus in the Pacific coast of Costa Rica.
| Región | Localidad | Fecha | N* | Talla (cm) |
| Golfo de Nicoya | Playa San Lucas Tumbabote | 10-oct-2018 | 14 | 16.9 (1.9) |
| Los Garrobos | 08-mar-2019 | 11 | 24.8 (3.7) | |
| Gitana | 08-mar-2019 | 6 | 19.5 (2.3) | |
| Chiquita | 08-mar-2019 | 12 | 22.7 (3.2) | |
| Chincheta | 08-mar-2019 | 1 | 21.0 (nd) | |
| Golfo Dulce | Los Chorros | 21-ago-2019 | 4 | 19.6 (2.2) |
| Manchadito | 21-ago-2019 | 26 | 21.2 (2.7) | |
| Playa Esperanza | 21-ago-2019 | 14 | 22.8 (3.3) |
* N = Número de organismos muestreados por sitio. / * N = Number of sampled organisms per site.
La preparación de las muestras para la amplificación de los microsatélites se llevó a cabo mediante PCR según Pérez-Enríquez et al., (2020). Brevemente, en un volumen final de reacción para el PCR de 11 µl, se colocaron 5.5 µl de máster mix TopTaq (Qiagen) y 0.5 µl del ADN de las muestras; adicionalmente se les agregó 0.1 µM de cebador forward (INVITROGEN, USA), 0.4 µM de cebador reverse (INVITROGEN, USA) y M13 con el fluororóforo (6-FAM, VIC, PET y NED) y el volumen faltante se completó con agua nanopura. Los cebadores empleados en este estudio fueron Lgut15, Lgut18, Lgut19, Lgut21, Lgut26, Lgut34, Lgut37, Lgut38, Lgut43 y Lgut44 siguiendo lo mencionado por Pérez-Enríquez et al., (2020), con sus respectivas temperaturas de alineamiento.
La amplificación se llevó a cabo con el termociclador (Thermo Fisher Scientific, EN61326, USA) con las siguientes condiciones térmicas: 94 ºC por 5 min; 15 ciclos de 94 ºC por 30 s, temperatura de alineamiento por 30 s, 72 ºC por 30 s; después 30 ciclos de 94 ºC por 30 s, 53 ºC por 30 s y 72 ºC por 30 s; finalmente se mantuvo a 72 ºC por 10 min para la extensión final.
Para comprobar los resultados de los protocolos de amplificación modificados, los productos de PCR obtenidos para cada muestra se agruparon en tres POOLPLEX y posteriormente se enviaron a genotipificar al Centro de Investigaciones en Biología Celular y Molecular (CIBCM) de la Universidad de Costa Rica (UCR), para lo cual se utilizó el equipo secuenciador ABI 3500 (Applied Biosystem) usando GeneScan™ 600 LIZ™ como estándar de tamaño (Paz-García et al., 2017). Los resultados se analizaron con el software GeneMarker V 3.0.1 (SoftGenetics) con el objetivo de identificar los alelos para cada locus y se agruparon por localidad.
Se realizó una selección de datos y se eliminaron muestras que presentaban datos perdidos en tres o más loci, y se eliminaron los loci que presentaban más del 10 % de datos perdidos con el análisis del programa Arlequin (Excoffier et al., 2005). Se estimaron las frecuencias alélicas y los parámetros de diversidad (número de alelos, número efectivo de alelos y heterocigosidad observada y esperada). Se estimó la conformación al Equilibrio de Hardy-Weinberg por locus y localidad mediante el programa GenAlEx (Peakall & Smouse 2012); la significancia de los valores de c2 dentro de cada localidad (total y grupos por fecha de muestreo en el Golfo de Nicoya; total y grupos por cohorte en Golfo Dulce), se ajustaron de acuerdo con el método secuencial de Bonferroni (Rice, 1989). Se establecieron las diferencias entre localidades para las especies muestreadas mediante análisis molecular de varianza (AMOVA) con un 95 % de confianza, basado en el índice de fijación F ST con el programa Arlequin (Excoffier et al., 2005). Se implementó un análisis de componentes principales (PCoA) para visualizar la estructura poblacional utilizando el programa Adegenet 2.1.5 (Jombart & Ahmed, 2011). También, se infirió la estructura genética mediante el método bayesiano de agrupamiento instrumentado en el programa Structure 2.3.4 (Pritchard et al., 2000). Las condiciones de las corridas del método Monte Carlo de la Cadena de Markov fueron 20 000 pasos de precalentamiento y 50 000 simulaciones para cada número posible de grupos (K= 1 hasta 8). El valor de K más probable se determinó mediante el método de Evanno (Evanno et al., 2005) implementado en el programa STRUCTURE HARVESTER (Earl & von Holdt, 2012). Finalmente, se realizó un análisis de distancias genéticas mediante remuestreo utilizando el programa Phylip 3.698 (Felsentein, 1998). Las condiciones de corrida fueron 10 000 simulaciones de las frecuencias alélicas en el programa Seqboot, las matrices de distancias genéticas de Nei y Cavalli-Sforza se obtuvieron con la subrutina Gendist, se generaron dendrogramas con el programa Neighbor utilizando las configuraciones Neighbor Joining y UPGMA y finalmente se obtuvo el dendrograma consenso con el programa CONSENSE. Los resultados se visualizaron en el programa Treeview 1.6.5 (Roderic, 1996)
Resultados
Se recolectaron 44 individuos por sitio de muestreo, que presentaron una distribución de longitudes de 20 ± 4 cm y 21 ± 3 cm, para el Golfo de Nicoya y el Golfo Dulce, respectivamente. Se observó que las muestras representaron las cohortes de edad 0 +, 1 y 1 + (Fig. 1).
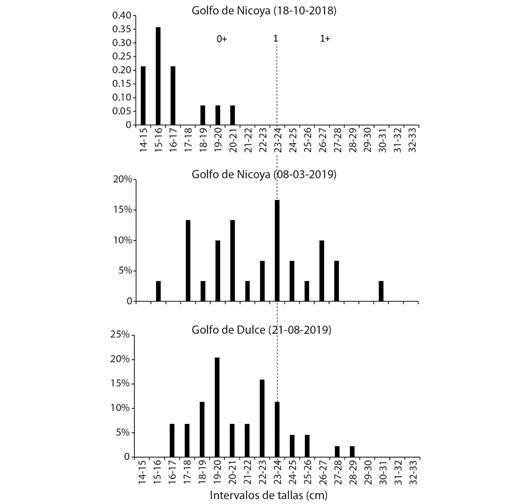
Fig. 1 Estructura de tallas de los muestreos de Lutjanus guttatus en las localidades de Golfo de Nicoya y Golfo Dulce. La línea punteada indica la talla esperada para la edad de 1 año de acuerdo con Soto-Rojas et al., (2018); las tallas menores y mayores a ésta, corresponden a las edades 0+ y 1+, respectivamente. / Fig. 1. Size structure of Lutjanus guttatus samples in the Golfo de Nicoya and Golfo Dulce localities. The dashed line indicates the expected size for 1-year-old individuals according to Soto-Rojas et al., (2018); sizes smaller and larger than this corresponds to ages 0+ and 1+, respectively.
De la extracción de ADN, se obtuvo una concentración promedio de 254 ± 107 ng/µl para las muestras del Golfo de Nicoya y 114 ± 105 ng/µl, y para las del Golfo Dulce. Del análisis de datos perdidos, se eliminaron en total 4 muestras para la población del Golfo de Nicoya y dos muestras para la población del Golfo Dulce, para un total de 40 y 42 muestras analizadas, respectivamente. Por otro lado, del análisis de datos perdidos por loci se encontró que los loci Lgut19 y Lgut36 presentaban más del 10 %, por lo que se procedieron a eliminar para los análisis posteriores.
Cuando se evaluó el equilibrio Hardy-Weinberg para el conjunto de muestras por localidad, ver columnas ''total'' en tabla 2 (Tabla 2) se observó desviación significativa del equilibrio (P < 0.0063) en ambas localidades, con la única excepción del locus Lgut18. Sin embargo, cuando los datos se manejaron por sitio de muestreo o cohorte, se observa que únicamente grupo B-D del Golfo de Nicoya y la cohorte 0 + de Golfo Dulce se desviaron del equilibrio, indicando un posible efecto Wahlund en éstas.
Tabla 2 Valores de probabilidad para los valores de c2 obtenidos en las pruebas de Equilibrio de Hardy-Weinberg por localidad (total) y por grupos dentro de localidades (ver Fig. 1). / Table 2. Probability values for the c2 values obtained in the Hardy-Weinberg Equilibrium tests by locality (total) and by groups within localities (see Fig. 1).
| Locus | Golfo de Nicoya1 | Golfo Dulc | ||||||
| Total | Sitio A | Sitios B-D | Sitios C-E | Total | Cohorte 0 + | Cohorte 1 | Cohorte 1 + | |
| Lgut18 | 0.198 | 0.778 | 0.239 | 0.412 | 0.125 | 0.527 | 0.279 | 0.261 |
| Lgut37 | 0.000* | 0.534 | 0.001* | 0.050 ns | 0.000* | 0.000* | 0.039ns | 0.423 |
| Lgut15 | 0.000* | 0.251 | 0.000* | 0.820 | 0.000* | 0.001* | 0.026ns | 0.261 |
| Lgut43 | 0.000* | 0.214 | 0.001* | 0.680 | 0.221 | 0.532 | 0.316 | 0.423 |
| Lgut21 | 0.003* | 0.159 | 0.185* | 0.491 | 0.000* | 0.009* | 0.142 | 0.317 |
| Lgut26 | 0.003* | 0.348 | 0.125 | 0.516 | 0.000* | 0.010* | 0.179 | 0.423 |
| Lgut44 | 0.000* | 0.171 | 0.000* | 0.092 | 0.000* | 0.004* | 0.000* | 0.157 |
| Lgut34 | 0.026ns | 0.129 | 0.199 | 0.472 | 0.002* | 0.015* | 0.267 | 0.317 |
| Fis | 0.184 | 0.112 | 0.201 | 0.074 | 0.160 | 0.165 | 0.121 | -0.200 |
1 Sitio A: Playa San Lucas Tumbabote; Sitios B-D: Los Garrobos-Chiquita; Sitios C-E: Gitana-Chincheta. El valor crítico del ajuste secuencial de Bonferroni es P = 0.0063. Fis: Coeficiente de endogamia para cada grupo. * Valor estadísticamente significativo; ns: menor a 0.05 pero no significativo. / 1 Site A: Playa San Lucas Tumbabote; Site B-D: Los Garrobos-Chiquita; Site C-E: Gitana-Chincheta. * Statistically significant value; ns: under 0.05 but not significant. The critical value for the sequential Bonferroni adjustment is P = 0.0063. Fis: Inbreeding coefficient for each group.
La diversidad genética se analizó simplificando a las dos localidades, es decir, Golfo de Nicoya vs Golfo Dulce. Se observó una alta diversidad genética en ambas localidades con un número de alelos por locus de 34.12 y 35.75 (Na) (Fig. 2A), un número efectivo de alelos por locus de 23.92 y 23.25 (Ne) (Fig. 2A), una heterocigosidad observada de 0.774 y 0.800 (Ho) (Fig. 2B) y una heterocigosidad esperada de 0.948 and 0.954 (He) (Fig. 2B), en promedio para el Golfo de Nicoya y el Golfo Dulce respectivamente. En ningún caso hubo diferencias significativas entre localidades. Sin embargo, la heterocigosidad promedio observada para los diferentes loci fue menor a la heterocigosidad esperada en ambas localidades (Fig. 2C, Fig. 2D). El déficit de heterocigotos fue más evidente en los loci Lgut37, Lgut15 y Lgut44 en las dos localidades.
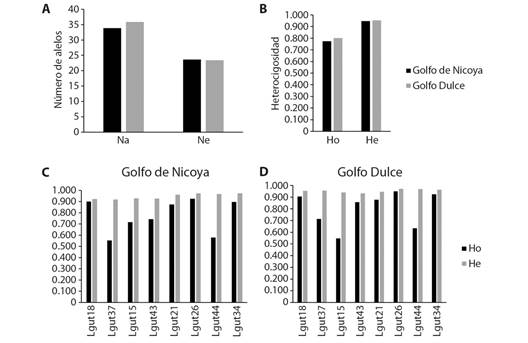
Fig. 2 Diversidad genética de Lutjanus guttatus en las localidades de Golfo de Nicoya y Golfo Dulce. A. Número promedio de alelos por locus (Na) y número efectivo de alelos por locus (Ne); B. Heterocigosidad observada (Ho) y esperada (He); C., D. Heterocigosidad por locus en Golfo de Nicoya y Golfo Dulce, respectivamente. / Fig. 2. Genetic diversity of Lutjanus guttatus in the Golfo de Nicoya and Golfo Dulce localities. A. Average number of alleles per locus (Na) and effective number of alleles per locus (Ne); B. Observed (Ho) and expected (He) heterozygosity; C., D. Heterozygosity per locus in Golfo de Nicoya and Golfo Dulce, respectively.
El índice de endogamia (Fis) total fue mayor al 16 % en ambas localidades (Tabla 2). El análisis a nivel de grupos del Golfo de Nicoya los valores indican una reducción a menos de 11.5 % en los sitios A y C-E; los valores de Fis entre cohortes muestran que en la 0 + el nivel se mantiene en el 16.5 %, una reducción al 12 % en la cohorte 1 y un exceso de heterocigotos en la 1 + (Tabla 2).
Al realizar la comparación de las distancias genéticas evaluadas entre las localidades del Golfo de Nicoya y el Golfo Dulce mediante el AMOVA, se obtuvo que la distancia genética (F ST ) es de 0.00264, la cual no fue significativamente diferente de cero (P > 0.05), por lo que no hay evidencias para considerar que se trata de poblaciones distintas. El resultado del análisis de componentes principales (PCA) (Fig. 3A) y el método de grupos (Fig. 3B) muestran la misma situación, es decir que no hay evidencias de que Golfo de Nicoya y Golfo Dulce sean poblaciones genéticamente distintas; sin embargo, en el PCA se aprecian ejemplares con valores relativamente extremos (fuera de las elipses), que pudieran deberse a dos factores: individuos con dos o más loci homocigotos o de origen distinto. De acuerdo con los dendrogramas de distancia genética, el punto de muestreo A (Golfo de Nicoya), que contiene a los ejemplares colectados en octubre de 2018, se agrupa como ligeramente distinto al resto (Fig. 3C, Fig. 3D).
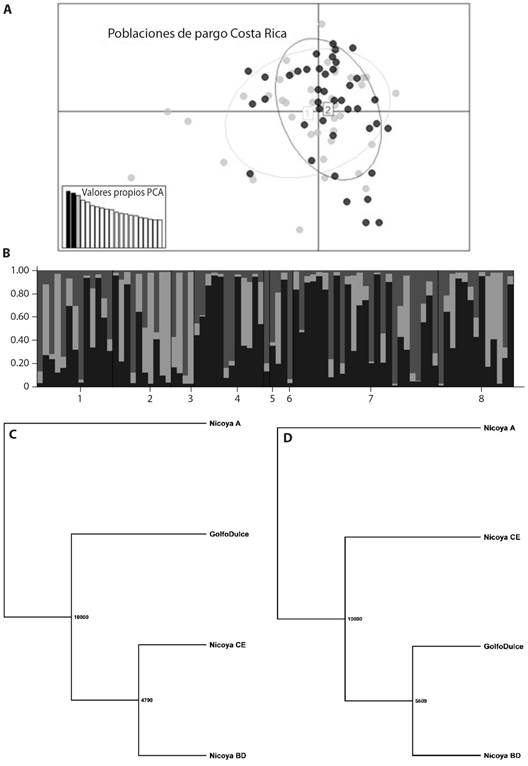
Fig. 3 A. Análisis de componentes principales de las poblaciones de L. guttatus del Golfo de Nicoya (1, color gris) y del Golfo Dulce (2, color negro). B. Análisis de agrupación genética de localidades con agrupamiento a posteriori con K = 3, donde las poblaciones del uno al cinco pertenecen al Golfo de Nicoya, y del seis al ocho al Golfo Dulce. C., D. Árboles con configuración UPGMA de distancias genéticas del remuestreo con 10 000 simulaciones. C. Distancia de Cavalli-Sforza y D. Distancia de Nei. / Fig. 3. A. Principal Component Analysis of L. guttatus populations from Golfo de Nicoya (1, gray color) and Golfo Dulce (2, black color). B. Genetic clustering analysis of localities with posterior assignment using K = 3, where populations one to five belong to Golfo de Nicoya and populations six to eight belong to Golfo Dulce. C., D. UPGMA trees of genetic distance based on resampling with 10 000 simulations. C. Cavalli-Sforza distance and D. Nei distance.
Discusión
Los resultados de este estudio demostraron un elevado grado de polimorfismo asociado a la cantidad de alelos que se encontraron en cada localidad para cada locus analizado, esto indica que estos loci son informativos para la especie L. guttatus (Pérez-Enríquez et al., 2020). Además, es probable que el potencial de viabilidad de las poblaciones a largo plazo se pueda conservar a pesar de factores ambientales o humanos como la pesca (Pérez-Enríquez et al., 2018).
El análisis de diferenciación genética entre las dos poblaciones estudiadas indica que pertenecen a una sola población panmíctica con flujo genético entre ellas. Pese a que ambos sitios presentan topografías y composiciones planctónicas distintas según la estación (seca o lluviosa), en donde el Golfo de Nicoya es un estuario tectónico de 1 543 km2 con alta influencia estacional, mientras que el Golfo Dulce es un estuario tectónico de 750 km2 con menos influencia por la parte estacional (Wangelin & Wolff, 1996), no parecen existir barreras a la conectividad genética entre dichos sitios, de manera que, para efectos de manejo se podrían considerar como una sola población (Gomes et al., 2012). El fenómeno de panmixia también se observó en L. guttatus entre localidades aún más distantes (El Salvador y Panamá), pero utilizando un marcador genético menos variable como el ADN mitocondrial (Hernández-Álvarez et al., 2020). La falta de diferenciación en la región del Pacífico del sur de México, Centroamérica y Colombia se ha reportado también para el Lutjanus peru (Molina-Quirós, 2022; Reguera-Rouzaud et al., 2021), así como en otras especies de pargo en distintos sitios del Atlántico como L. campechanus (Garber et al., 2004), y L. purpureus (Gomes et al., 2012).
Por otro lado, en este estudio, se encontró que los menores niveles de heterocigosidad observada respecto de la esperada en la mayoría de loci, es consecuente con la desviación del EHW.
La desviación en el equilibrio de Hardy-Weinberg, aun cuando exista el supuesto de panmixia, se puede deber a muchos factores, como errores en la genotipificación por la presencia de picos fantasmas, dominancia de alelos pequeños o la presencia de alelos nulos (Ellegren, 2000); otra posible explicación es el efecto Wahlund, que señala que la mezcla de poblaciones con frecuencias alélicas distintas puede ocasionar un déficit de heterocigotos (Pérez-Enríquez et al., 2018).
En referencia a los errores de genotipificación, esto se ha observado en distintos organismos marinos tanto vertebrados como invertebrados (Landínez et al., 2009). De hecho, dentro de los microsatélites empleados para este estudio, el Lgut18, Lgut 21 y Lgut 34 se ha sugerido que presentan una alta tasa de mutación y la presencia de alelos nulos (Pérez-Enríquez et al., 2020).
En cuanto al efecto Wahlund, este tipo de mezcla pudo haber ocurrido debido al programa de repoblación del pargo mancha (L. guttatus) que maneja un centro de reproducción en Costa Rica, quienes durante el 2017 y 2018, realizaron ocho liberaciones de 31 000 individuos de entre 2-10 g en el Golfo de Nicoya (Chacón-Guzmán et al., 2019). Esta posible mezcla es más evidente en las muestras obtenidas en marzo de 2019, las cuales contienen ejemplares de las cohortes 0 +, 1 y 1 + (Fig. 2). En el caso de Golfo Dulce, también se tiene una aparente mezcla de individuos de las cohortes 0 + y 1 que resultó en desviación del EHW, sin embargo, no hay evidencias de ejemplares liberados por el programa de repoblación arriba mencionado o por una granja de cultivo comercial en jaulas marinas que tiene lugar aproximadamente 150 km al noroeste (HFM, 2022). La desviación del EHW fue también utilizado como criterio para determinar la presencia de camarón de cultivo en poblaciones silvestres del noroeste de México (Pérez-Enríquez et al., 2018). Sin embargo, no se puede descartar la posibilidad de que la propia mezcla de cohortes en parte explique la desviación al EHW; en el pez Plectropomus leopardus, una especie asociada a arrecifes de coral, Taboun et al., (2021) detectaron diferencias genéticas significativas entre cohortes, poniendo en relevancia la importancia del seguimiento temporal de la estructura genética.
Estos resultados indicarían, por una parte, que los marcadores genéticos utilizados pueden ser una herramienta adecuada para la detección de organismos provenientes de programas de repoblación o de cultivo comercial en el medio silvestre y, por el otro, que hubo un potencial éxito del programa de repoblación, al menos para la región del Golfo de Nicoya, sitio en el cual se documentó la liberación de juveniles de L. guttatus (Chacón-Guzmán et al., 2019). Un análisis más preciso de esta situación requeriría de la caracterización genética de los lotes de reproductores e incluso un diseño de los programas de repoblación que tomen en cuenta sus posibles efectos negativos en términos de los cambios que pueden ocurrir en las poblaciones silvestres debido a la pérdida de diversidad genética, endogamia, o capacidad adaptativa, entre otros (Pérez-Enríquez et al., 2013).
Declaración de ética: los autores declaran que todos están de acuerdo con esta publicación y que han hecho aportes que justifican su autoría; que no hay conflicto de interés de ningún tipo; y que han cumplido con todos los requisitos y procedimientos éticos y legales pertinentes. Todas las fuentes de financiamiento se detallan plena y claramente en la sección de agradecimientos. El respectivo documento legal firmado se encuentra en los archivos de la revista.











 uBio
uBio