Introducción
Los Cnidarios son un filo de animales invertebrados con una anatomía simple, que se compone de dos capas celulares conocidas como ectodermo (capa más exterior) y la gastrodermis, capa interna con funciones digestivas, que en algunos cnidarios simbióticos alberga a microalgas dinoflageladas de la familia Symbiodiniaceae en una estructura subcelular conocida como simbiosoma (Peng et al., 2010; Roth et al., 1988). Estas dos capas celulares están separadas por una capa no celular llamada mesoglega, cuya función es de sostén y de intercambio de sustancias (Veron, 2000). Este filo incluye las clases Anthozoa (corales), Hydrozoa (hidras y anémonas), Scyphozoa y Cubozoa (medusas) (Barnes, 1974) y se caracterizan por poseer células, llamadas nemotocistos, especializadas en la producción de venenos (Birsa et al., 2010; Ozbek et al., 2009). Estos venenos, de naturaleza proteica y compuestos de bajo peso molecular, son utilizadas por los Cnidarios para su protección y para cazar sus presas (Schendel et al., 2019).
Estos invertebrados han sido de gran interés de bioprospección por las posibles aplicaciones de las sustancias que componen sus venenos (Stabili et al., 2021), por la producción de metabolitos secundarios con gran potencial farmacológico (Raimundo et al., 2018), como los aminoácidos tipo micosporinas (MAAs, por sus siglas en inglés “Mycosporine-like amino acids”) importantes en fotoprotección (Banaszak et al. 1998), y por las sustancias que componen su repertorio de inmunidad. De este modo, estos invertebrados podrían ser una fuente inagotable de productos con actividad biológica de importancia biotecnológica y farmacéutica.
Bajo el escenario anterior se plantea revisar el repertorio inmune de los Cnidarios, como un sistema complejo digno de bioprospección para la búsqueda de biomoléculas con actividad farmacéutica. En este sentido, se conoce que la respuesta inmune de manera general se clasifica convencionalmente en dos tipos: 1) la inmunidad innata, la cual representa la primera línea de defensa, tiene limitada variabilidad, especificidad y es ancestral (de origen evolutivo basal); y 2) la inmunidad adaptativa, que está presente sólo en vertebrados mandibulados y se caracteriza por la alta diversidad y especificidad de sus respuestas mediadas por linfocitos y receptores de antígeno (Cadavid, 2016).
En los cnidarios la inmunidad innata se da por: 1) reconocimiento inmunológico, 2) señalización intracelular, y 3) respuesta efectora (Mydlarz et al., 2016). La detección de patógenos está mediada por receptores de reconocimiento (PRR), proteínas de membrana y a nivel intracelular, que reconocen patrones moleculares asociados a patógenos (PAMP). Estos PAMP son estructuras moleculares conservadas, expresadas por virus, bacterias, hongos, protozoos y helmintos (Buchmann, 2014). En el caso de Cnidaria, las cuatro familias de PRR mejor estudiadas son los receptores de tipo Toll (TLR), Lectinas de tipo C(CTL), los receptores de tipo dominio de oligomerización por unión de nucleótidos (NLR) y los receptores tipo RIG-I (RLR). Además, cumple un papel importante en el manejo de microbios beneficiosos, y en la simbiosis microbiana (Parisi et al.,2020). En la señalización actúan mediadores de traducción de señales, donde factores de trascripción y las ascadas de señalización intracelular inducen la transcripción de genes inmunes para respuestas efectoras (Cerenius et al., 2010). En Cnidaria existen vías de señalización NF-kB, ECSIT, de interferón, y lectina-complemento. Adicionalmente, de sistema de la profenoloxidasa y de complemento. También se da la fagocitosis, opsonización, lisis y producción de péptidos antimicrobianos. En esta revisión se describen los hallazgos realizados en Cnidaria con respecto a los tres procesos inmunológicos principales de la inmunidad innata, de igual forma, en la primera sesión se discute el papel de sistema inmune en el proceso de establecimiento de la simbiosis, mecanismo presente en cnidarios fotosimbióticos. También se hace alusión a la importancia del mucus (Bythell & Wild, 2011) como barrera protectora, y a compuestos bioactivos con aplicaciones biotecnológicas descubiertos en Cnidaria.
A pesar de la morfología primitiva de los cnidarios, sus genomas presentan una gran complejidad que se ve reflejada en el repertorio de genes que conforman su sistema inmunológico, los cuales manifiestan similitud a los de los mamíferos, convirtiendo a este filo en candidatos excepcionales para investigaciones sobre la evolución inmune innata (Miller et al., 2007). Otro motivo por el cual resulta importante estudiar estos mecanismos de defensa primitivos, son las enfermedades de carácter infeccioso que ponen en riesgo la estructura y función de los arrecifes de coral, por la pérdida de tejido vivo en las colonias de corales constructores (hermatípicos) (Alvarez-Filip et al., 2019; Estrada-Saldívar et al., 2020; Weil et al., 2006). Del mismo modo, se ha planteado que estos organismos ofrecen un potencial como fuente de nuevas sustancias antimicrobianas para el tratamiento de infecciones causadas por bacterias multirresistentes (Augustin et al., 2009). Por lo tanto, resulta fundamental comprender los mecanismos de respuesta inmune exhibidos por estos invertebrados, a través de esta revisión, con los objetivos de describir su repertorio molecular, y explorar las aplicaciones biotecnológicas que estas moléculas pudieran tener.
Materiales y métodos
Se recurrió a la revisión de artículos publicados en revistas científicas que abordan el tema de la inmunidad en cnidarios. Los artículos fueron buscados en los motores de búsqueda del NCBI, Scielo y Google Scholar, utilizando palabras claves en dos idiomas (español e inglés): inmunidad, reconocimiento inmunológico, mecanismos de defensa inmunológicos y aplicaciones biotecnológicas en cnidarios (immunity, immunological recognition, immunological defense mechanisms and biotechnological applications in cnidarians, bioprospection in cnidaria). Se abordaron los temas: simbiosis e inmunidad, reconocimiento inmunológico en cnidarios, señalización inmunitaria, respuestas efectoras enfocadas en péptidos antimicrobianos, y aplicaciones biotecnológicas de compuestos bioactivos. Se tuvieron en cuenta tanto artículos en inglés como en español de una ventana de tiempo de la última década (2011-2021), incluyendo la bibliografía clásica sobre el tema de estudio, dando un total de 90 artículos revisados. La búsqueda se hizo desde el 20 de julio del 2021 hasta el primero de noviembre del 2021.
Resultados y discusión
Simbiosis e inmunidad: Los cnidarios como algunos corales escleractínios, anémonas y medusas, establecen una simbiosis de tipo mutualista con microalgas dinoflageladas de la familia Symbiodiniaceae que generalmente residen dentro de las células gastrodérmicas del huésped cnidario (Davy et al., 2012). En esta asociación los cnidarios aportan nutrientes como amonio, fosfatos y dióxido de carbono a las microalgas, mientras que estas les transfieren azúcares, lípidos y oxígeno, compuestos que contribuyen a la producción de energía para la actividad metabólica del huésped (Allemand et al., 1998). El establecimiento de la simbiosis se sustenta en cuatro fases: a) reconocimiento y fagocitosis de la microalga simbionte, b) regulación de la división celular del simbionte, c) intercambio metabólico y tráfico de nutrientes, d) calcificación, en el caso de los corales escleractíneos (Davy et al., 2012).
La primera fase de reconocimiento es crucial, debido a que el hospedero ha desarrollado mecanismos celulares y moleculares que discriminan sus simbiontes potenciales de agentes infecciosos, mientras que los posibles simbiontes deben tener la capacidad para invadir y evadir la respuesta inmune del hospedero para sobrevivir dentro de él (Neubauer et al., 2017). Esta fase está mediada por interacciones lectina-glucano, una relación común entre patrones moleculares asociados a patógenos y receptores de reconocimiento de patógenos (PAMP-PRR), en repertorios inmunes innatos de animales (Davy et al., 2012). Un estudio con la anémona de mar Aiptasia pulchella demostró que la eliminación de glucanos en la superficie de Symbiodinium disminuyó significativamente el éxito de la infección de este sobre la anémona (Lin et al., 2000). En tres especies de Symbiodiniaceae se midió la afinidad de glicoconjugados presentes en su superficie celular con diferentes tipos de lectinas, y se pudo determinar que tanto los residuos de D-manosa como D-glucosa son probablemente componentes funcionales de la superficie del simbionte que participan en el reconocimiento de Symbiodiniaceae y el mantenimiento del mutualismo. Además, se encontró que lectinas específicas para manosas BC2L-A, CALSEPA, GRFT y ORYSATA reconocieron glicoproteínas en las tres especies de simbiontes (Tortorelli et al., 2021). En el coral Pocillopora damicornis, mediante experimentos de inmunolocalización se pudo encontrar evidencia de inmunoreactividad de PdC-Lectina en células de coral pertenecientes al tejido endodérmico en contacto con simbiontes dinoflagelados libres presentes transitoriamente en el coelenteron, lo cual sugiere un papel putativo para la PdC-Lectina en la interacción y adquisición de zooxantelas (Vidal-Dupiol et al., 2009). En este mismo estudio se demostró que la PdC-Lectina muestra grandes similitudes con lectinas denominadas Millectinas, aisladas en el coral Acropora millepora, con capacidad para unirse a patógenos bacterianos como a ciertos miembros de la familia Symbiodiniaceae (Kvennefors et al., 2008).
La regulación negativa de la expresión de algunos genes de la inmunidad parece ser una condición necesaria para la simbiosis en cnidarios. Esto se evidencia en Exaiptasia diaphana (=Exaiptasia pallida) cuya expresión y actividad del factor de transcripción NF-kB estuvo regulado negativamente al introducir células de Symbiodinium en larvas aposimbióticas (libres de microalgas simbióticas), y regulado positivamente con la pérdida de la simbiosis (Mansfield et al., 2017). Por otra parte, se ha planteado que la regulación de citocinas del factor de crecimiento transformante beta (TGF-b) cumple un papel importante en el establecimiento de la simbiosis. En E. diaphana, la adición de TGF-b humano exógeno suprimió la respuesta inmune inducida por lipopolisacáridos (LPS), mientras que la adición anti TGF-b previno la aparición de simbiosis, lo cual sugiere que los simbiontes dinoflagelados promueven la tolerancia del huésped a través de la activación de las vías inmunes tolerogénicas (Detournay et al., 2012). En esta especie también se caracterizó la glucoproteína trombospondina (TSR), donde se demostró que el dominio TSR estimula la adquisición de simbiontes (Neubauer et al., 2017). Sin embargo, ciertos corales blanqueados se encuentran inmuno-suprimidos, esto puede observarse en el coral Orbicella faveolata, donde se demostró que vías relacionadas con el sistema inmunitario, como la apoptosis y el sistema de complemento, se suprimen durante el blanqueamiento y se mantienen así después de un año del evento (Pinzón et al., 2015).
Estos hallazgos sugieren que los genes de la inmunidad innata pueden estar regulados positiva o negativamente de acuerdo con las condiciones necesarias para el establecimiento de la simbiosis. Por ejemplo, la regulación positiva de genes relacionados con la expresión de lectinas puede resultar crucial para el reconocimiento y unión del simbionte, y la inhibición del factor de trascripción NF-kB puede conducir a una supresión de diferentes vías inmunes, lo cual puede facilitar su adaptación. Estudios futuros deben estar encaminados en evaluar la actividad de diferentes genes y vías de señalización inmunes durante el blanqueamiento, con el fin de establecer si la pérdida de la simbiosis aumenta la vulnerabilidad a enfermedades de carácter infeccioso.
A continuación, se describen los hallazgos realizados en Cnidaria con respectos a los cuatro procesos inmunológicos principales de la inmunidad innata 1, reconocimiento inmunológico, 2) señalización intracelular, y 3) respuesta efectora.
Reconocimiento inmunológico de patógenos en cnidarios: Los PRR son proteínas claves de la inmunidad innata presentes en la membrana celular y a nivel intracelular que detectan PAMP (Buchmann, 2014). Activan la especificidad inmunitaria en invertebrados, ya que su diversidad permite generar respuestas inmunitarias específicas (Emery et al., 2021). Una vez es reconocido el patógeno, estos receptores inducen una respuesta en el organismo afectado en tres niveles: 1) estimula la fagocitosis e ingestión enzimática, 2) moviliza moléculas a los sitios donde se produce la infección y 3) induce moléculas efectoras para respuestas inmunes efectivas (Cadavid, 2016; Dunn, 2009) (Fig. 1). A continuación, se describen los principales PRR presentes en cnidarios.
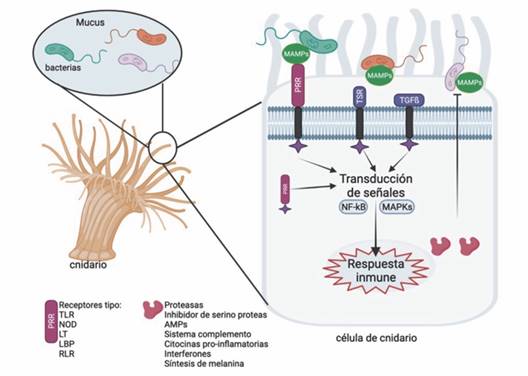
Fig. 1 Principales receptores de reconocimiento de patógenos (PRR) y posibles respuestas efectoras de la inmunidad en invertebrados identificados en Cnidaria. Receptores PRR: Receptor de tipo Toll (TLR), Receptor de tipo NOD (NLR), Lectinas (LT), Receptor de tipo RIG-I (RLR) y proteínas de unión a polisacáridos (LBP). Receptores asociados con la regulación del sistema inmune: glucoproteína trombospondina (TSR) y el receptor del factor de crecimiento transformante beta (TGF-b). Respuestas efectoras asociadas con la síntesis de proteasas e inhibidores de Serino proteasas, péptidos antimicrobianos, sistema de complemento, citocinas proinflamatorias, interferones, fagocitosis, opsonización y encapsulación. Esquema creado en BioRender.com / Fig. 1. Main pathogen recognition receptors (PRR) and possible effector responses of immunity in invertebrates identified in Cnidaria. PRR receptors: Toll-like receptor (TLR), NOD-like receptor (NLR), Lectins (LT), RIG-I-like receptor (RLR), and polysaccharide-binding proteins (LBP). Receptors associated with the regulation of the immune system: thrombospondin glycoprotein (TSR) and the receptor for transforming growth factor beta (TGF-b). Effector responses associated with the synthesis of proteases and inhibitors of serine proteases, antimicrobial peptides, the complement system, proinflammatory cytokines, interferons, phagocytosis, opsonization, and encapsulation. Outline created on BioRender.com
Receptores tipo Toll (TLR): Los TLRs son proteínas transmembranales presentes en el huésped, que poseen dominios específicos para unirse a (PAMP) (Parisi et al., 2020). Mediante genómica, Miller et al. (2007) identificaron TLR en Nematostella vectensis. Estas proteínas incluyen a NvTLR-1. También, hay proteínas receptoras con dominios de inmunoglobulinas (NvlL-1R1, NvlL-2R2, NvlL-1R3), y una proteína homóloga de MyD88 (NvMyD88). Para Hydra magnipapillata, se encontraron cuatro proteínas HyLRR-1 y LyTRR-2. Las proteínas HyTRR-1 y HyTRR-2 se relacionan con la producción de péptidos antimicrobianos cómo Hydramacin-1 (Augustin et al., 2010).
El genoma de Acropora digitifera muestra un sistema inmune complejo. En A. digitifera existen cuatro Toll/TLR, cinco proteínas tipo IL-1R, y proteínas TIR con dominios de inmunoglobulinas (Shinzato et al., 2011). Emery et al. (2021) evidenciaron TLR en los antozoos Dendronephyta gigantea y A. millepora.
Brennan et al. (2017) caracterizaron funcionalmente Nv-TLR- 1 en N. vectensis, que activa la señalización de NF-kB en respuesta a patógenos. En E. diaphana la exposición a patógenos evidenció una fuerte expresión de MyD8 (Roesel & Vollmer, 2019), y ante la infección con Vibrio parahaemolyticus se identificaron varios componentes de la vía TLR- NF-kB, donde resaltan homólogos con dominios conservados como MyD88, TRAF, TBK1, NIK, IRF, NF-kB y AP-1 (Seneca et al., 2020).
Lectinas: Las lectinas son receptores en cnidarios de unión a carbohidratos que activan el complemento y opsonizan (Dunn, 2009). En la vía de las lectinas se une al azúcar del patógeno formando el C3 y proteínas efectoras del complejo de ataque de membrana/perforina (MACPF) (Parisi et al., 2020).
Unas lectinas comunes en cnidarios es la Taquilectina que se une a LPS y peptidoglucanos (Beisel et al., 1999). Burge et al. (2013) determinaron que la Taquilectina-2 se expresa en Gorgonia ventalina ante parásitos. Hay un homólogo de Taquilectina-2 en Oculina varicosa (Hayes et al., 2010).
Como respuesta a V. parahaemolyticus, E. diaphana expresa lectinas como la colectina-12 (colec12), lectinas de unión a L-ramnosa (Ep_RBL) y lectina de tipo C (Seneca et al., 2020). Emery et al., (2021) identificó lectinas tipo C. En los medusuzoos C. hemisphaerica y M. virulenta existen factores de Von Willebrand y dominios secretores ricos en cisteína, además de lectinas de unión a manosa (MBL) en Cassiopea xamachana y C. hemisphaerica.
Receptores similares a NOD: Los NLR son receptores intracelulares con dominios LRR C-terminal para el reconocimiento de PAMP, un dominio central de unión a nucleótidos denominado dominio NACHT, y un dominio efector N-terminal (Parisi et al., 2020). Los NLR activan vías NF-kB, interferón, proteína cinasa activada por mitógenos (MAPK), e inflamasoma (Elinav et al., 2011).
Se han caracterizado NLR en Cnidaria. Hamada et al. (2013) determinaron que Acropora digitifera incluye NLR putativos con receptores tipo NOD, con una diversidad amplia de dominios proteicos comparado con los vertebrados, evidenciando que los NLR son esenciales en la inmunidad de los antozoos. Esto se observa en Emery et al. (2021), donde se describe la presencia de NLR en los antozoos analizados. También, hay ausencia de NLR en especies de medusozoos (Tabla 1).
Tabla 1 Componentes de diferentes vías de señalización asociados con la respuesta inmune en cnidarios / Table 1. Components of different signaling pathways associated with the immune response in cnidarians
| Anthozoa | Medusozoa | |||||
| Vías/Elementos de señalización | N. vectensis | A. millepora | H. magnipapillata | Referencia | ||
| Vía NF-kB | Miller et al., 2007 | |||||
| LBP | + | - | + | |||
| TLR prototípicos (LRR/TIR) | + | - | - | |||
| TIR/TRAF6/TAK1/TRAF6 | + | + | + | |||
| MyD88/IRAK/IkK | + | - | + | |||
| NF-kB | + | + | - | |||
| Vía ECSIT | ||||||
| ECSIT/MKK/JNK | + | - | + | |||
| P38/AP1/MEKK1 | + | + | + | |||
| Vía de IFN | ||||||
| TRAM | + | + | + | |||
| IRF3 | + | - | + | |||
| Vía lectina-complemento | ||||||
| C3/MACPF | + | + | + | |||
| Vía NF-kB | Emery et al., 2021 | |||||
| TLR prototípicos (TIR, LRR, y dominio transmembrana) | A. tenebrosa, A. millepora, M. capitata, N. vectensis, P. damicornis, O. faveolata, D. gigantea | Ausente en todas las especies de medusozoos analizadas | ||||
| NLR prototípicos (NACHT y LRR) | Todas las especies de la casilla anterior más Xenia sp. y E. diaphana | Ausente en todas las especies de medusozoos analizadas | ||||
| NF-kB | Todas las especies de antozoos estudiadas | Aurelia sp., C. xamachana, C. hemisphaerica, H. vulgaris, M. virulenta, C. cruxmelitensis | ||||
| Vía de IFN | ||||||
| RLR (RIG-I/MDA5) | Todas las especies de antozoos analizadas | Ausente en todas las especies de medusozoos | ||||
| RLR (LGP2) | Xenia sp., A. tenebrosa, A. millepora, M. capitata | Ausente | ||||
| Vía lectina-complemento | ||||||
| C2 | Todas las especies de antozoos analizadas | Aurelia sp., C. xamachana, M. virulenta, C. cruxmelitensis, | ||||
| C3 | Todas las especies de antozoos analizadas | C hemisphaerica, H. vulgaris, M. virulenta, C. cruxmelitensis | ||||
| MASP | Todas las especies de antozoos analizadas | Aurelia sp., C. xamachana, C. hemisphaerica, M. virulenta | ||||
| Vía NF-kB | E. diaphana | Seneca et al., 2020 | ||||
| MyD88, TRAF6, IRF, AP-1, NF-kB, NLR prototípicos | ||||||
| Vía de IFN | RLR-3 (RIG-I), IFIH1 | |||||
| Vía lectina-complemento | C2, C3, C4 | |||||
+ indica presencia, - indica ausencia. / + indicates presence, - indicates absence.
Los receptores tipo RIG-I (RLR): Son PRR que detectan ARN viral (Loo & Gale, 2011). Tres proteínas altamente relacionadas constituyen la familia de las RLR: El RIG-I (gen I inducible por ácido retinoico), MDA5 (el gen 5 asociado a la diferenciación de melanoma), que contiene el dominio C helicasa inducida por interferón (IFIH1), y LGP2 (La ARN helicasa DHX58) (Dixit & Kagan, 2013). Se caracterizan por la presencia de una ATPasa central de caja DExD/H que funcionan como sensores citoplasmáticos (Loo & Gale, 2011); también comparten un dominio helicasa de ARN funcional común cerca del C-terminal (HELICc) que se une específicamente a las moléculas de ARN viral (Yoneyama et al., 2005). RIG-1 y MDA5 contiene dominios CARD en tándem N-terminales que median la señalización aguas abajo, para desencadenar la respuesta del sistema de interferón. LGP2 al igual que RIG-1, albergan un dominio represor (RD) en sus dominios reguladores C-terminales (Dixit & Kagan, 2013).
En N. vectensis se identificaron homólogos de RIG-I/MDA5 (Zou et al., 2009). En E. diaphana, existen dominios IFIH1 y receptores RIG (RLR-3) (Seneca et al., 2020). Emery et al. (2021) encontraron RIG-I/MDA5 en antozoos, y LGP2 en las especies Xenia sp., Actinia tenebrosa, A. millepora y Montipora capitata.
Otros PRR: En Cnidaria se reportan proteínas de unión a lipopolisacáridos (LBP), que reconoce LPS de bacterias Gram negativas, y activan la vía NF-kB (Parisi et al., 2020). Estas proteínas han sido detectadas en los genomas de H. magnipapillata y N. vectensis (Miller et al., 2007). En Acropora palmata y O. faveolata se han identificado LRR fundamentales durante la simbiosis (Schwarz et al., 2008). Para E. diaphana (= Aiptasia pallida) existe un receptor del TGF-b, el cual como ya se mencionó, posiblemente está implicado en la regulación de la simbiosis Cnidario-microalgas (Detournay et al., 2012).
Señalización inmunitaria: La activación de la respuesta inmune se da por moléculas en las vías de señalización intracelular que activan las moléculas efectoras (Cadavid, 2016). Se han identificado mediadores intracelulares de la vía de señalización Toll/TLR en Nematostella y Acropora, asociados con la localización nuclear de NF-kB, y con la activación de la vía ECSIT que conduce a la trascripción de una variedad genes diana a través de los factores AP1. También se han encontrado elementos de señalización asociados a la vía interferón (Miller et al., 2007) (Tabla 1).
Se han identificado profenoloxidasas para la síntesis de melanina, esencial en la encapsulación de patógenos y curación de heridas (Parisi et al., 2020). Esta vía es iniciada por TLR y lectinas que desencadenan varias reacciones proteolíticas que inducen melanina (Mydlarz et al., 2016). La activación de la cascada induce proteólisis de la profenoloxidasa inactiva (PPO) para formar fenoloxidasa activa (PO). La enzima cataliza la oxigenación de monofenoles a o-difenoles y la oxidación de o-difenoles a quinonas, necesarias para la síntesis de melanina (Satoh et al., 1999). En P. damicornis el gen lacasa-3 y un homólogo de una enzima de la profenoloxidasa se activan por V. coralliilyticus (Vidal-Dupiol et al., 2014). También se han identificado tirosinasas en H. magnipapillata y Nematostella vectensis (Esposito et al., 2012). Mydlarz et al. (2008), encontraron amebocitos granulares acidófilos en la mesoglea de Gorgonia ventalina al ser infectado con el hongo Aspergillus sydowii.
Otro sistema proteico en la inmunidad cnidaria es el complemento, el cual es citotóxico, opsonización, regula respuestas inflamatorias y lisis bacteriana (Sarma & Ward, 2011). Se activa mediante tres cascadas proteolíticas como la vía clásica, de la lectina, y la vía alternativa (Parisi et al., 2020). En última instancia, todas estas vías activan el complejo de la proteína C3 para aumentar las respuestas inflamatorias como la fagocitosis, la lisis celular y la coagulación (Mydlarz et al., 2016). La vía de la lectina se ha detectado en cnidarios y se une a un azúcar del patógeno, lo cual conduce a la señalización serina, proteasas asociadas a lectinas de unión a manosa (MASP), que luego activan las proteínas C2 y similares a C4 para iniciar la formación del complejo C3 (Endo et al., 2006). Una vez formado el complejo, las perforinas (MACPF) son secretadas para formar un agujero en la membrana microbiana lisando el patógeno (Endo et al., 2006). En cnidarios se han identificado componentes del complemento de la lectina, MASP, C3 y MACPF (Mydlarz et al., 2016). En N. vectensis se reportan dos genes C3, dos factores de serina proteasa de vía alternativa B y uno de serina proteasa (Endo et al., 2003). El C3 se registra en especies de coral como, Swiftia exserta, A. millepora, A. digitifera, Porites lobata (Dishaw et al., 2005) y en las anémonas Diadumene lineata (= Haliplanella lineata) y Anemonia viridis (Fujito et al., 2010). Miller et al. (2007) identificó proteínas que contienen el dominio MACPF similar al presente en el componente C6 del sistema de complemento en Hydra magnipapillata y Nematostella vectensis.
E. diaphana ataca a Vibrio parahaemolyticus, mediante el complemento C2, C3, C4 y el factor B de Exaiptasia (Ep_Bf-1) lo cual sugiere una defensa activa contra el patógeno (Seneca et al., 2020). En un estudio realizado a 15 especies de cnidarios, en la mayoría se encontraron homólogos de MASP, C2 y C3. Sin embargo, en ninguna especie se pudo detectar la proteína de la familia C6, lo cual indicaría que los cnidarios no usan el complejo de ataque a la membrana (MAC), y en cambio utilizan el complemento para la opsonización a través de C3 (Emery et al., 2021).
Respuestas efectoras en cnidarios: El sistema inmune de los cnidarios presentan diversidad de respuestas efectoras contra patógenos, incluyendo una capa superficial de mucus que impide el acceso de patógenos, actividad microbiana debida a péptidos antimicrobianos, especies reactivas de oxígeno, y productos formados en diversas vías de señalización (Mydlarz et al., 2016).
El mucus representa la primera línea de defensa contra patógenos en antozoos como corales y anémonas, y está compuesto por lípidos, proteínas y polisacáridos que recubren el cuerpo del animal (Ducklow & Mitchell, 1979). Está involucrado en la locomoción, captura de alimento y defensa contra depredadores patógenos (Parisi et al., 2020). La capa de mucosa superficial sirve como nutrientes para muchos microorganismos beneficiosos o patógenos (Stabili et al., 2018). Se han aislado diferentes biomoléculas que conforman esta barrera protectora. En los corales Goniopora djiboutiensi, A. millepora y O. faveolata, existen mucinas poliméricas de alto peso molecular similares en vertebrados (Jatkar et al., 2010). Rivera-Ortega y Thomé (2018) identificaron colágeno, melanina y fenoloxidasa en el mucus de Pseudodiploria strigosa, E. diaphana y C. xamachana. Además, determinaron que el mucus tiene actividad antimicrobiana contra Serratia marcescens y Aurantimonas sp.
En cuanto a las especies reactivas de oxígeno (ROS), en dosis bajas actúan como moléculas de señalización en la respuesta inmune y la apoptosis, y en altas dosis producen estrés oxidativo peligroso para los componentes celulares del huésped y los patógenos. Las ROS son liberadas por fagocitos o amibocitos móviles para ayudar a matar a los agentes infecciosos (Parisi et al., 2020). Las anémonas contienen fagocitos productores de ROS que junto al mucus forman mallas complejas para capturar microbios (Robb et al., 2014).
Otra defensa innata son los inhibidores proteasas de serina. Las proteasas de serina actúan directamente en la patogénesis, con capacidad de dañar el tejido del huésped destruyendo células y proteínas del mismo (Lantz et al., 1997).
Otro mecanismo es la fagocitosis. Los cnidarios generan inflamación para destruir células infectadas, esto implica una infiltración del tejido lesionado por las células inmunes y el inicio de la fagocitosis (Mydlarz et al., 2016). Un ejemplo de esto lo encontramos en G. ventalina, especie en la cual se han identificado células especializadas en fagocitosis (amebocitos) contra Aspergillus sydowii (Mydlarz et al., 2008). También, se ha identificado un mecanismo de defensa en corales escleractíneos y gorgonias relacionado con la encapsulación. Los microbios son encapsulados con material proteico, como colágeno o gorgonina, para rodear al microbio (Mullen et al., 2004).
Péptidos antimicrobianos (AMP): Los AMP son catiónicos con regiones hidrofóbicas y bactericidas (Smith et al., 2010). Todos están genéticamente codificados específicamente (Rivas et al., 2006). Su tamaño puede afectar de manera diferente a los componentes estructurales del patógeno. Por ejemplo, los péptidos pequeños (23 aminoácidos de longitud) destruyen la integridad de la membrana celular, mientras que los más grandes lisan o secuestran nutrientes esenciales de los microbios (Stabili et al., 2018).
Los péptidos se unen a la superficie bacteriana y luego a la membrana citoplasmática, mediante interacciones electrostáticas entre los péptidos cargados positivamente y las moléculas cargadas negativamente de la pared celular bacteriana (Smith et al., 2010). Los péptidos destruyen estructuras de la membrana bacteriana produciendo lisis celular (Teixeira et al., 2012). Según la composición de aminoácidos de los péptidos, el tamaño y la estructuras que lo conforman, estos se clasifican en categorías: 1) péptidos con estructura de hélice alfa; 2) péptidos con estructura de hojas betas estabilizadas con puentes disulfuro; 3) péptidos con estructuras extendidas; y 4) péptidos ricos en glicina (Ortiz-López, 2019). Características como la carga, el tamaño, la conformación, estructura secundaria, la hidrofobicidad y la antipaticidad son fundamentales para determinar la actividad antibacteriana (Smith et al., 2010).
Diferentes PAM se han identificado en cnidarios (Mydlarz et al., 2016). Damicornina, el primer PAM informado en un coral escleractinio (P. damicornis), es catiónico de 39 residuos plegado por tres puentes disulfuro intramoleculares que implica seis dominios de cisteína en su secuencia con una amidación C-terminal; presenta actividad antimicrobiana contra bacterias Gram-positivas y el hongo Fusarium oxysporum (Vidal-Dupiol et al., 2011). En un estudio realizado por Franzenburg et al. (2013) se detectaron armininas en Hydra oligactis, Hydra viridissima y en Hydra vulgaris, implicado en la selección de parejas bacterianas adecuadas. Estudios en Hydra magnipapillata identificaron el péptido arminina 1a, cuyo dominio C-terminal cargado positivamente de 31 aminoácidos mostró un amplio grado de eficacia contra patógenos humanos multirresistentes (Augustin et al., 2009). Además, se observó su baja eficacia contra células eucariotas.
En Aurelia aurita se purificó el péptido antimicrobiano aurelina, el cual exhibe actividad contra bacterias Gram positivas y Gram negativas, y se caracteriza por la presencia de seis cisteínas que forman tres enlaces disulfuro (Ovchinnikova et al., 2006). En H. vulgaris se identificó Perculina (Fraune et al., 2010), PAM hallado en H. magnipapillata con actividad bactericida contra Bacillus megaterium (Bosch et al., 2009).
Los péptidos antimicrobianos presentan características que les permiten ser modelos para estudios de evolución y bioprospección. Son moléculas altamente conservadas codificadas a partir de genes específicos, que en cnidaria han mostrado una alta eficiencia contra bacterias multirresistentes. Se han encontrado homólogos de péptidos antimicrobianos en diferentes especies, por ejemplo, las defensinas, presentes en plantas, invertebrados, y vertebrados (Rivas et al., 2006). Sin embargo, los estudios para organismos primitivos aún siguen siendo escasos, y Cnidaria puede ser un modelo que contribuya en gran medida al conocimiento sobre el estado de conservación de estas moléculas, y su respuesta ante diferentes patógenos que muestran alta capacidad de resistencia.
Aplicaciones biotecnológicas: Hasta el 2015 se conocían cerca de 30 mil productos de origen marino, caracterizados por su diversidad estructural y complejidad (Hu et al., 2015; Kiuru et al., 2014). Cnidaria ha demostrado que es una fuente importante de productos bioactivos de carácter nutracéuticos, cosmecéuticos, biomédicos y biomateriales (Merquiol et al., 2019). Por ejemplo, el colágeno tipo II de Stomolophus meleagris es útil para el tratamiento de la artritis reumatoide (Hsieh, 2005). También, se ha demostrado que el colágeno de la medusa Stomolophus nomuraise presenta actividad de inmunoestimulación aumentando la producción de inmunoglobulinas, de interferón y factor de necrosis tumoral (TNF) (Sugahara et al., 2006). La administración a largo plazo de péptidos de colágeno de la medusa Rhopilema esculentum en ratas reduce la presión arterial sistólica y la presión arterial diastólica (Zhuang et al., 2012). Esta proteína extraída principalmente de las campanas de medusa, tienen capacidad protectora ante la exposición a rayos UV, debido a que disminuye la destrucción de la piel y la formación de arrugas, además de tener la capacidad de reparar fibras proteicas endógenas de colágeno y elastina, y de mantener la proporción natural de colágeno de tipo I a tipo III (Fan et al., 2013).
En las especies Aurelia sp., Cotylorhiza tuberculata y Rhizostoma pulmo se han caracterizado las propiedades bioquímicas y antioxidantes de las biomasas gelatinosas, en las cuales se encontró altas cantidades de colágeno, péptidos antioxidantes y otras moléculas bioactivas que resaltan su valor nutracéutico, cosmecéutico y farmacológico (Leone et al., 2015). El extracto de medusa Chiropsalmus quadrumanus (Cq) incubado en neuronas SH-SY5Y humanas genera una mayor longitud en el crecimiento de neuritas y uniones ramificadas, amplificando el contacto entre neuronas SH-SY5Y, sin afectar el cuerpo celular ni la viabilidad, lo cual proyecta esta proteína como una herramienta prometedora para la recuperación de la conexión neuronal, condición que es esencial en el tratamiento de enfermedades neurodegenerativas (Arruda et al., 2021).
En el coral Cladiella pachyclados se han aislado diterpenos de eunicelina que exhiben una función anti-migratoria y anti-invasiva de células de cáncer de próstata PC3, siendo la paquicladina A el compuesto que presenta mayor efectividad (Hassan et al., 2010). De igual forma, han sido aislados varios tipos de diterpenos en el coral Sarcophyton crassocaule, con actividad citotóxica significativa contra las líneas celulares de carcinoma Daoy, HEp-2, MCF-7 y WiDr, además de actividad antiinflamatoria en la línea celular de macrófagos RAW264.7 (Lin et al., 2011). También, se han evaluado los efectos antitumorales de 11-deshidrosinulariolide, un compuesto bioactivo aislado del coral blando Sinularia leptoclados, el cual induce apoptosis en células cancerosas escamosas orales CAL-27. Además, tuvo efectos sobre la regulación de algunas proteínas específicas que pueden estar involucradas en la proliferación celular (Liu et al., 2011). La proteína verde fluorescente (GFP) es probablemente el compuesto derivado de las medusas con más aplicaciones en el campo biomédico. Obtenida de la hydromedusa Aequorea victoria, esta biomolécula tiene grandes aplicaciones en el campo de la oncología y desarrollo de células nerviosas debido a su capacidad para marcar células (Merquiol et al., 2019).
Retomando el cóctel principal de compuestos activos producto de los cnidarios, comúnmente conocido “veneno”, quien aunque merece una revisión aparte vale la pena abordarlo aquí, contienen compuestos no proteicos como prostaglandinas, palitoxinas, psuedopterosina, sarcodictinas, y compuestos proteicos como citotoxinas, toxinas formadoras de poros, hidralisinas, fosfolipasas, metaloproteasas, neurotoxinas, inhibidores de transportadores de iones regulados por voltaje y protones, inhibidores de proteasas, y péptidos inductores de necrosis (Mariottini & Pane, 2010; Menezes & Thakur, 2022). Dichos compuestos, poseen un gran abanico de aplicaciones a nivel farmacológico, por su actividad apoptótica y de despolarización de membranas, como antitumorales, analgésicos, anestésicos y antibióticos (Mariottini & Pane, 2010; Menezes & Thakur, 2022). Un ejemplo es el Dalazatide, un derivado del péptido ShK aislado de la anémona del caribe Stichodactyla helianthus (Castañeda et al., 1995), que inhibe específicamente canales de potasio, actualmente está siendo probado contra varias enfermedades autoinmunes (Tarcha et al.,2017; Menezes & Thakur, 2022). Mientras que otras biomoléculas derivadas del veneno de Palythoa caribaeorum, retrasan e inhiben el proceso de inactivación de canales de iones activados por voltaje en ratas, sugiere una posible aplicación en la modulación del sistema neuronal (Lazcano-Pérez et al., 2016).
En conjunto, esta gran diversidad de compuestos producidos por los cnidarios, revisados aquí a través de los mecanismos de protección propios de estos invertebrados, abre una gran puerta hacia el hallazgo de nuevos antibióticos y compuestos bioactivos contra diferentes patógenos y enfermedades.
Declaración de ética: los autores declaran que todos están de acuerdo con esta publicación y que han hecho aportes que justifican su autoría; que no hay conflicto de interés de ningún tipo; y que han cumplido con todos los requisitos y procedimientos éticos y legales pertinentes. Todas las fuentes de financiamiento se detallan plena y claramente en la sección de agradecimientos. El respectivo documento legal firmado se encuentra en los archivos de la revista.












 uBio
uBio 


