Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista de Biología Tropical
On-line version ISSN 0034-7744Print version ISSN 0034-7744
Rev. biol. trop vol.59 n.1 San José Mar. 2011
Variabilidad genética de Brycon henni (Characiformes: Characidae) en la cuenca media de los ríos Nare y Guatapé, sistema Río Magdalena, Colombia
Julio César Hurtado-Alarcón1, Néstor J. Mancera-Rodríguez2 & Clara I. Saldamando-Benjumea3
1. Departamento de Ciencias Forestales, Universidad Nacional de Colombia, Calle 59A No 63-20, Bloque 11 Oficina 213, Medellín, Colombia. Fax: (54) (4) 4309080; jchurtad@unal.edu.co
2. Postgrado en Bosques y Conservación Ambiental, Universidad Nacional de Colombia, Sede Medellín, Colombia; njmancer@unal.edu.co
3. Escuela de Biociencias, Universidad Nacional de Colombia, Sede Medellín, Colombia; cisaldam@unal.edu.co
Dirección para correspondencia
Abstract
Genetic variability of Brycon henni (Characiformes: Characidae) in the middle basin of Nare and Guatapé rivers, Magdalena River system, Colombia. Brycon henni is a native species in Magdalena´s River basin, and because of its cultural and economic importance, is strongly overexploited. This study aimed to describe the genetic variability and population structure of this species from Nare and Guatapé rivers basins. A total of 195 individuals were collected and DNA extractions were obtained from muscle and blood tissue. Fourteen primers were evaluated with the RAPD technique, being four of them polymorphic, and produced 66 different fragments (63% polymorphism). Besides, using the molecular variance (AMOVA) analysis, the population structure was described for all sites ( ΦST=0.297, ΦST=0.163; Nare and Guatapé river basins, respectively), and suggested the importance of the migratory behaviour of the species in the genetic differentiation. Genetic distances among sampled sites showed that most of the genetic differentiation occurred between sites Guatapé and El Cardal with respect to the others. A Mantel Test demonstrated a correlation between genetic and geographic distances (r=0.431 both of the basins evaluated; r=0.377, Nare river middle basin), and suggested isolation by distance. The outcomes obtained in this study have valuable implications in species conservation and the genetic variability of natural populations of B. henni, and should be complemented with morphological analyses. Rev. Biol. Trop. 59 (1): 269-282. Epub 2011 March 01.
Key words: Neotropical fish, population structure, RAPD, Characid conservation, Magdalena River.
Resumen
Brycon henni es una especie nativa de la cuenca del río Magdalena, Colombia, donde es fuertemente explotada, por lo cual en este trabajo se estudió la variabilidad genética y estructura poblacional en individuos provenientes de la cuenca media de los ríos Nare y Guatapé, Antioquia. Se recolectaron 195 ejemplares y se les extrajo ADN genómico total del tejido muscular y/o sanguíneo. Se evaluaron 14 cebadores para la técnica RAPD (Random Amplifyed Polimorphic DNA), siendo cuatro de ellos polimórficos, los cuales generaron 66 fragmentos diferentes (63% polimorfismo). Por medio de un análisis de varianza molecular (AMOVA), se determinó la estructuración poblacional para todos los sitios evaluados y la cuenca del río Nare ( ΦST =0.297 y ΦST =0.163, respectivamente). Las distancias genéticas de Nei mostraron que todas las poblaciones diferían entre sí y que la mayor diferenciación genética se dio para las poblaciones del río Guatapé y la quebrada El Cardal. Un test de Mantel mostró correlación entre las distancias genéticas y geográficas (ambas cuencas, r=0.431; cuenca del río Nare, r=0.377). Esto sugiere aislamiento por distancia y separación de grupos genéticos producidos por los embalses sobre los ríos Nare y Guatapé, lo cual puede tener implicaciones relevantes en la conservación de la variabilidad genética de las poblaciones naturales de esta especie en el oriente antioqueño.
Palabras clave: pez Neotropical, estructura poblacional, RAPD, conservación de Carácidos, río Magdalena.
En Colombia, los ríos andinos están sometidos a problemáticas asociadas con la alteración de ecosistemas por fragmentación y pérdida de hábitat, alteración en las dinámicas de regulación hídrica, pérdida de la cobertura vegetal de sus laderas, presión sobre recursos hidrobiológicos, contaminación, entre otros problemas, lo cual conlleva a una alta pérdida de biodiversidad (Mojica 1999, Mojica et al. 2002). El país posee un enorme potencial en recurso íctico, debido a que se han estimado en más de 1 400 las especies de peces de agua dulce solo a partir de información secundaria y de ejemplares de museo, lo que da cuenta de la alta diversidad de la ictiofauna nativa (Maldonado-Ocampo et al. 2008).
Brycon henni, conocida popularmente como sabaleta, es una especie nativa con una amplia distribución en Colombia, con alta importancia para la seguridad alimentaria de los pobladores ribereños. Además es una alternativa promisoria para realizar reintroducciones como opción para la conservación de la especie y el desarrollo de pesca deportiva (Montoya et al. 2006). B. henni no aparece en ninguna de las categorías de riesgo propuestas por el libro rojo de peces dulceacuícolas de Colombia (Mojica et al. 2002), ni en la lista roja de especies amenazadas de la Unión Internacional para la Conservación de la Naturaleza (UICN 2003). Sin embargo, su sobreexplotación podría conllevar a la pérdida de su variabilidad genética.
Las modificaciones de las condiciones ambientales en sistemas lóticos pueden reducir las poblaciones de peces. En particular, la construcción de presas puede producir serios impactos en sus comunidades, debido a que perturba o bloquea sus migraciones (Morita & Yamamoto 2002; Katano et al. 2003, 2006). Tales modificaciones ocasionadas por la construcción de embalses (Juárez & Varsi 1992), como ocurre en los ríos Nare y Guatapé (oriente del Departamento de Antioquia, Colombia), podrían originar impactos sobre las poblaciones de sabaleta presentes en este río. Esta especie es considerada un nadador de potencia, con preferencia por zonas de ríos con corrientes fuertes y turbulentas (Maldonado-Ocampo et al. 2005). No obstante, dado que hay información relacionada con migración de la sabaleta a cortas distancias dentro de los ríos pequeños y riachuelos o quebradas (Perdomo 1978, Jiménez et al. 1998), este comportamiento podría ser una causa adicional que genere estructuración genética de sus poblaciones.
Por ello, el propósito de este trabajo es utilizar marcadores moleculares altamente polimórficos para realizar una evaluación genético-poblacional de la sabaleta presente en el oriente antioqueño y determinar si existe una relación del efecto de los embalses de las cuencas medias de los ríos Nare y Guatapé sobre la variabilidad genética de esta especie. Así mismo, se busca determinar si el comportamiento migratorio parcial de ésta especie es una de las causas principales de su diferenciación. Se utilizaron marcadores RAPD´s (Random Amplifyed Polimorphic DNA – Polimorfismos de ADN Amplificados Aleatoriamente) (Williams et al. 1990), puesto que han sido previamente usados con éxito en varias especies de peces del género Brycon - (B. cephalus, Wasko et al. 2004; B. henni, Pineda et al. 2004, 2007; B. orbignyanus, Lopera-Barrero et al. 2006, 2008), así como en otras especies de peces (Pineda et al. 2004, Povh et al. 2005, Vieira et al. 2005) y de vertebrados (Soto-Marambio 2003). Este marcador molecular es una herramienta útil para evaluar polimorfismo genético en las poblaciones estudiadas y obtener información sobre su diversidad genética, sin tener un conocimiento previo de la genética de la especie en cuestión (Barman et al. 2002, Barfai et al. 2003), como lo exigen marcadores moleculares como los microsatélites y RFLP’s (Hartl & Clark 1997, Kang et al. 2006, Moreira et al. 2007). Este trabajo puede ser de utilidad como una medida inicial de evaluación genética de esta especie en el oriente antioqueño, con relación a los planes de conservación y manejo de la misma.
Materiales y métodos
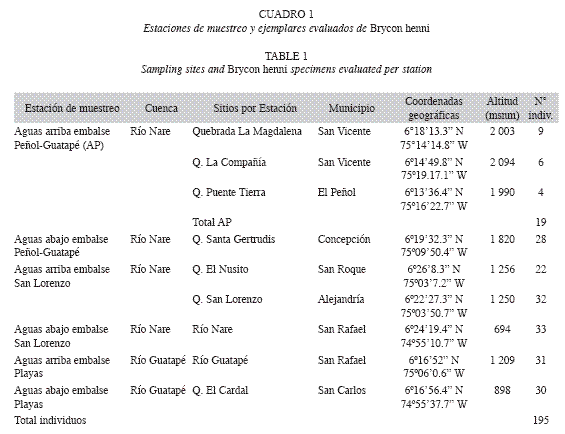
Área de estudio y colección de muestras: Las colecciones de peces se realizaron entre octubre de 2007 y mayo de 2008 en diferentes sectores de las cuencas medias de los ríos Nare y Guatapé, en Antioquia, Colombia. Los sitios de muestreo se encuentran interrumpidos por los embalses Peñol-Guatapé (quebradas La Compañía, Puente Tierra, La Magdalena y Santa Gertrudis), Jaguas-San Lorenzo (quebradas El Nusito y San Lorenzo, y río Nare) y Playas (río Guatapé y quebrada El Cardal) (Cuadro 1). Una de las estaciones de muestreo (Aguas arriba embalse Peñol-Guatapé), estuvo conformada por tres sitios diferentes, debido a que son muy cercanos entre sí y drenan al mismo embalse (Peñol-Guatapé), están localizados aproximadamente a 2 000msnm y fueron los sitios con menor número de muestras (19 en total) (Cuadro 1). Los ejemplares se capturaron utilizando diferentes artes de pesca (eléctrica, atarraya, nasa, señuelos) y de cada uno de ellos se colectó aprox. 1.0g de tejido de la región anterior del músculo epiaxial y/o 0.5-1ml de sangre periférica obtenida mediante punción de la vena caudal. Los tejidos colectados se introdujeron en viales para microcentrífuga (AMERICAN SCIENTIFIC, USA) de 2.0ml, los cuales contenían solución buffer de colecta (0.1M Tris, 0.1M EDTA, 0.01M NaCl y 0.5% p/v SDS) y se refrigeraron a 4°C. Se transportaron a los Laboratorios de Ecología Aplicada -LECA-, Área de Fauna Silvestre (Departamento de Ciencias Forestales), y de Biología Molecular y Celular (Escuela de Biociencias), de la Universidad Nacional de Colombia Sede Medellín. Allí se almacenaron a -20°C, antes de someterse a procesos de extracción de ADN.
Extracciones de ADN genómico: Para la extracción de ADN genómico total a partir de los tejidos de los ejemplares de sabaleta capturados (sangre y músculo), se utilizó el método de fenol:cloroformo:alcohol isoamílico (Sambrook et al. 1989, Taggart et al. 1992), con algunas modificaciones. Se tomaron aproximadamente 0.3g de tejido, se maceraron y resuspendieron en 500μl de soluciσn de extracción STE (0,1M Tris, 0,01M NaCl, 0,5M EDTA) en viales nuevos y estériles de 2.0ml. Se adicionaron 30μl SDS 10% y 15μl de proteinasa K (FERMENTAS UAB, CANADΑ) (20mg/ml) y se dejaron incubando por 14-16 hr a una temperatura de 55°C. Posteriormente, se adicionaron 400μl de cloroformo:alcohol isoamνlico (24:1), se agitaron suavemente por dos minutos y se centrifugaron a 13 000rpm durante 15min. Se tomó el sobrenadante, se adicionaron 400μl de isopropanol frνo, se homogenizó suavemente y se centrifugó nuevamente a 13 000rpm por 15 min. Se descartó el sobrenadante y el tubo se resuspendió con 100μl de agua ultrapura estéril, se almacenó por 24 hr a 4°C para la completa hidratación y dilución de las extracciones, finalmente se adicionaron a cada muestra 2μl de RNAsa A (FERMENTAS UAB, CANADÁ) y se incubó por 30 min a 37°C. Por último el ADN se almacenó a 4°C.
Evaluación de la calidad y cantidad de ADN obtenido en las extracciones: La cantidad y calidad del ADN extraído se evaluó en geles de agarosa (0.8%) teñidos con bromuro de etidio (EtBr), se verificó en transiluminador de luz U.V. y se cuantificó con ayuda de un espectrofotómetro (ThermoScientific, USA), para lo cual la concentración del ADN genómico se determinó mediante un análisis de absorbancia a una longitud de onda de 260nm.
Estandarización del proceso de amplificación de loci de RAPD (Polimorfismos de ADN Amplificados Aleatoriamente): Inicialmente, se probaron 14 cebadores decámeros para RAPD de las series OPBA, OPAN y OPE (OPERON TECHNOLOGIES, USA) (Cuadro 2), los cuales se sometieron a evaluación para detectar como primera medida contaminación de los mismos, seguido de la identificación de los más polimórficos al momento de efectuar las reacciones de amplificación aleatoria de ADN mediante PCR (Polymerase Chain Reaction, Amplificación en Cadena de la Polimerasa). Para obtener productos de amplificación de secuencias RAPD (Random Amplifyed Polimorphic DNA), se tuvieron en cuenta diferentes condiciones, a partir de las recomendadas por Williams et al. 1990, Wasko et al. 2004, Pineda et al. 2004, 2007 y Lopera-Barrero et al. 2008. Además, se incluyeron variaciones a las condiciones mencionadas por los autores señalados, en las concentraciones usadas de cada reactivo y en los perfiles térmicos de amplificación. Las reacciones de PCR se llevaron a cabo en un termociclador PT-100 (MJ RESEARCH, USA) en viales de 200μl, en cada uno de los cuales se adicionó la mezcla de reacción que contenía 1μl de ADN molde (30-60ηg), 0.5U de Taq polimerasa (FERMENTAS UAB, CANADÁ), 0.2mM de una solución de los cuatro deoxinucleótidos trifosfatados (dNTPs; FERMENTAS UAB, CANADÁ), 2.0mM MgCl2 (FERMENTAS UAB, CANADÁ), 10ρmol de cebador (primer) (IDT, OHIO- USA) y 2.5μl de solución buffer 10X (200mM Tris-HCl pH 8.4, 500mM KCl y 1.5mM MgCl2) (FERMENTAS UAB, CANADÁ). El volumen final por reacción fue 25μl. Se estandarizaron las concentraciones de ADN, de cloruro de magnesio y las temperaturas de hibridización de los cebadores. El perfil térmico para las reacciones de PCR incluyó: temperatura inicial de 94°C durante cinco minutos, seguida de 35-40 ciclos de un minuto a 95°C, entre 35-45s a una temperatura óptima de alineamiento (36°C) y un minuto a 72°C. Por último, una extensión final de dos minutos a 72°C. Los productos de amplificación se almacenaron a -20°C.
Determinación del tamaño de los fragmentos obtenidos por PCR: Los productos de amplificación se cargaron en geles de agarosa (1.5%) en tampón TBE 1X (0.089M Tris Base, 0.089M ácido bórico y 0.002 M EDTA) y se utilizó una escalera de peso molecular, la cual contiene 14 bandas entre 100-3 000 pares de bases (GeneRuler™ 100 bp Plus DNA Ladder, FERMENTAS CANADA INC.). Para su visualización, los geles se tiñeron con bromuro de etidio (0.5μg/ml) y se sometieron a electroforesis en cámaras horizontales (BIO-RAD, USA). Las condiciones de corrido fueron 80V y 400mA durante dos horas. Los geles se observaron sobre una pantalla de luz ultravioleta para confirmar la efectividad de la amplificación. Posteriomente, los geles se fotografiaron en un equipo de captura de imágenes (BIODOC ANALYZE -BIOMETRA, GERMANY) y las imágenes se guardaron en formato JPG para el análisis de las bandas (fragmentos) obtenidas. Debido a los potenciales problemas de replicabilidad de la amplificación relacionados con la técnica de RAPDs, aproximadamente el 25% de las muestras fueron amplificadas y corridas electroforéticamente por duplicado.
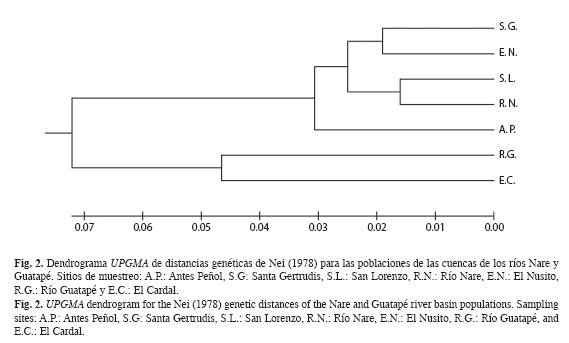
La presencia (1) o ausencia (0) de fragmentos de tamaños moleculares idénticos obtenidos por la técnica de RAPDs, se usó para la construcción de una matriz de distancias genéticas de Nei (Nei 1978). Estas distancias fueron agrupadas con el algoritmo UPGMA (Unweighted Pair Group Method with Arithmetic Mean, Método No Ponderado de Apareamiento por Grupos con Media Aritmética), obtenido con el programa MEGA 4.0 (Kumar et al. 2008). La variabilidad genética se determinó por el porcentaje de loci (fragmentos) polimórficos. El análisis de estructura poblacional se obtuvo mediante un análisis de varianza molecular (AMOVA), ΦST, descrito por Excoffier et al. (1992) y Michalakis & Excoffier (1996). La diferenciación genética entre poblaciones se estableció por medio de la definición propuesta por Wright (1965), en la cual valores entre 0.00-0.05; 0.05-0.15; 0.15-0.25 y >0.25 indican pequeña, moderada, alta y elevada diferenciación genética, respectivamente (Hartl & Clark 1997). Adicionalmente, se llevaron a cabo análisis de distancia e identidad genética insesgados, según lo descrito por Nei (1978). Luego, se realizó una prueba de Mantel con 999 permutaciones, para evaluar la posible correlación entre las matrices de distancias genética y geográfica. Mediante la prueba de Mantel, se pueden mostrar patrones de aislamiento por distancia (Wright 1943). La matriz de distancias genéticas, los análisis de distancias e identidades genéticas y la prueba de Mantel fueron realizados con el programa GenAlEx (Peakall & Smouse 2006).
Resultados
Extracción de ADN genómico total: En la cuantificación de ADN de todas las muestras, se encontró que sus concentraciones eran adecuadas para realizar la amplificación de marcadores RAPD, debido a que sus valores de absorbancia a 260nm oscilaron entre 0.012-0.212.
Amplificación aleatoria de Polimorfismos de ADN
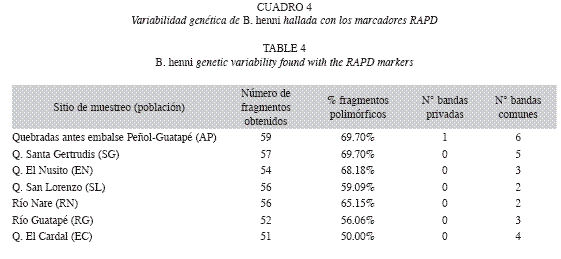
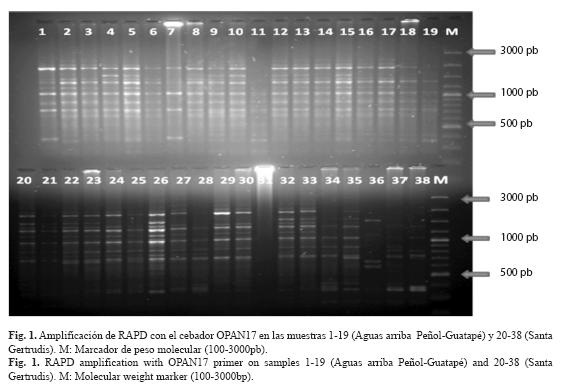
Variabilidad genética: 195 ejemplares de Brycon henni fueron genotipificados mediante oligonucleótidos para la técnica RAPD con el uso de cuatro secuencias de oligonucleótidos (OPBA13, OPBA10, OPE07, OPAN17) escogidas de un total inicial de 14 secuencias cebadoras. Esta escogencia se basó en la replicabilidad de los perfiles obtenidos y el éxito de amplificación en todos los 195 individuos muestreados. Con estos marcadores, se obtuvo un total de 66 fragmentos de amplificación reproducibles, con tamaños entre 350 y 3 000pb (pares de bases), de los cuales 41 fragmentos (62.55 %) fueron polimórficos. El menor número de fragmentos polimórficos lo aportaron los cebadores OPBA13 y OPBA10 (nueve cada uno) y el mayor número de fragmentos polimórficos se obtuvo con el cebador OPAN17 (12 fragmentos) (Cuadro 3). Los valores de variabilidad genética arrojados por la amplificación de las secuencias RAPD en los siete sitios de muestreo, se pueden observar en el Cuadro 4. Adicionalmente, entre el total de bandas amplificadas, solo se encontró un alelo privado para los sitios de muestreo localizados aguas arriba del embalse Peñol-Guatapé (estación de muestreo Antes Peñol). En la Fig. 1 se puede observar uno de los geles de los amplificados obtenidos de secuencias RAPD en geles de agarosa de algunas de las muestras de B. henni, en los sitios aguas arriba Peñol-Guatapé (1-19) y Santa Gertrudis (20-38). Se diferencian algunos patrones polimórficos para los individuos 3, 4, 6, 7, 15, 27, 34, 36, 37, con el cebador OPAN17 (Fig. 1).
Distribución de la variabilidad genética: El análisis de varianza molecular (AMOVA) en las cuencas de los ríos Nare y Guatapé en conjunto, arrojó un valor de 30% de variabilidad genética producida por las diferencias genéticas entre los sitios de muestreo. El 70% restante de esta variabilidad se debió a diferencias dentro de los sitios de muestreo. Sin embargo, cuando se realizó un AMOVA para los sitios de la cuenca del río Nare exclusivamente, se encontró que solo el 16% de la variabilidad genética se explica por diferencias entre los sitios de muestreo y que el resto (84%) de la variabilidad era explicado por diferencias dentro de sitios. Además, los estimados de estructuración genética ( ΦST) obtenidos en este análisis, mostraron que para los sitios de muestreo de las dos cuencas se presentó diferenciación genética, debido a que el valor de Phi (Φ) obtenido fue de 0.297 (g.l.=194, p<0.001). De acuerdo con Excoffier et al. (1992), los valores de Phi se pueden interpretar como análogos del índice de fijación FST descrito por Wright (1965), por lo que se puede inferir que el nivel de estructuración genética obtenido con estos marcadores RAPD es muy alto. Similarmente, al analizar solo las poblaciones de la cuenca del río Nare (estaciones 1-5) se halló un valor ΦST de 0.163 (g.l.=133, p<0.001). Este valor, con relación a lo planteado por Excoffier et al. (1992), se sitúa en un rango medio-alto de estructuración genética (Hartl & Clark 1997).
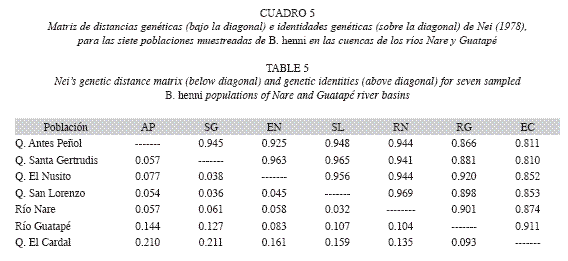
Distancia genética entre poblaciones: En el Cuadro 5 se encuentran las matrices de distancias genéticas de Nei (1978) (debajo de la diagonal) y de identidades genéticas (sobre la diagonal). En la comparación por pares de poblaciones, cuando se evaluaron las dos cuencas muestreadas (ríos Nare y Guatapé) se encontró que para la especie B. henni, la menor distancia genética observada entre poblaciones fue de 0.032 (Quebrada San Lorenzo y río Nare) y la mayor distancia genética fue de 0.211 (entre las quebradas Santa Gertrudis y El Cardal). Así mismo, en la matriz de distancias genéticas de Nei (1978), se pudo discriminar entre ejemplares provenientes de las dos subcuencas de donde se extrajeron (río Nare, poblaciones 1-5, vs río Guatapé, poblaciones 6-7). Adicionalmente, los datos de identidad genética obtenidos con estos marcadores moleculares indican que las poblaciones de la subcuenca del río Nare son muy similares entre sí, produciendo valores entre 0.925-0.969. El valor obtenido de identidad genética de Nei (1973) para las poblaciones del río Guatapé fue de 0.911, siendo inferior a los obtenidos para las poblaciones del río Nare. Al comparar los drenajes evaluados, se diferenciaron las poblaciones de B. henni de la cuenca media del río Guatapé (ríos Guatapé y quebrada El Cardal), con respecto a las poblaciones de la cuenca media del río Nare (quebradas La Magdalena, Santa Gertrudis, San Lorenzo y El Nusito; Río Nare), debido a que compartieron distancias genéticas más similares entre sí (Fig. 2).
Test de Mantel: Para todas las poblaciones evaluadas en los ríos Nare y Guatapé, así como para la cuenca del río Nare, se encontró una correlación positiva significativa entre la distancia genética y la geográfica (coeficiente de correlación Rxy=0.431, p<0.001, ambas cuencas; Rxy=0.377, p<0.001, cuenca del río Nare). Estos resultados sugieren que la especie estudiada presenta aislamiento por distancia (Wright 1943).
Discusión
La caracterización genotípica de diferentes especies del género Brycon, así como la evaluación de su estructura poblacional mediante marcadores moleculares RAPD, ha sido demostrada en B. cephalus (Wasko et al. 2004), B. henni (Pineda et al. 2004, 2007) y B. orbignyanus (Lopera-Barrero et al. 2006, 2008), lo que refleja la validez y la vigencia de esta técnica para la realización de estudios sobre la variabilidad genética de B. henni y el análisis de su estructura genético-poblacional.
En el presente estudio se halló un nivel de polimorfismo promedio cercano al 63% y para todas las poblaciones muestreadas entre 50-70%. Para el río Nare, en las poblaciones muestreadas antes y después del embalse Peñol-Guatapé así como en la quebrada El Nusito y el río Nare, se encontraron valores entre 65-70%. En la quebrada San Lorenzo, se observaron valores de polimorfismo cercanos al 60%. Para el río Guatapé, los valores de polimorfismo obtenidos fueron inferiores (50-55%). Estos valores de variabilidad genética en B. henni son altos dentro de cada población y bajos entre poblaciones, lo que podría demostrar en parte la sobre-explotación de la especie en los lugares muestreados. La pérdida de variabilidad genética siempre es esperada cuando no hay un buen manejo genético en lotes de reproductores debido al cruzamiento de individuos emparentados, originados por endogamia (Kang et al. 2006, Moreira et al. 2007), causando reducción de la variabilidad genética de una población (Povh et al. 2005). Sin embargo, un bajo tamaño de una población también es una de las causas principales de su baja variabilidad genética (Hartl & Clark 1997). En el caso de B. henni, uno de los sitios muestreados en este trabajo, tuvo una baja densidad poblacional (estación Antes Peñol -AP), lo cual puede ser considerado como una causa adicional del bajo polimorfismo encontrado en este estudio.
Por otro lado, las reducciones en el número de loci polimórficos presentes en peces pueden estar correlacionadas con un bajo número de ejemplares en su hábitat natural, y/o por ser poblaciones que se utilizan para establecer parentales para cría (Winkler et al. 1999). Para la zona del oriente antioqueño, no se cuenta con información de este tipo. Sin embargo, Pineda et al. (2004, 2007), mostraron que la diversidad genética en poblaciones de B. henni en Antioquia es relativamente alta y similar para todos los sitios, expresada tanto en número de fragmentos totales como en el número de fragmentos únicos (ríos Piedras y Humarada y estación piscícola San Carlos, 201, 209, 190 fragmentos amplificados, respectivamente; 40, 20, 54 fragmentos únicos, respectivamente). Los valores superiores de polimorfismo genético en la estación piscícola San Carlos, podrían estar relacionados con potenciales cruzamientos entre ejemplares de B. henni de procedencia desconocida, lo cual podría ayudar a explicar los resultados obtenidos (Pineda et al. 2004, 2007). Para el establecimiento de la estación piscícola San Carlos, la cual se localiza en el área de influencia del presente proyecto, se trasladaron ejemplares de B. henni de ríos con mayor abundancia poblacional hacia ríos con menos presencia de dichos peces, sin tener las precauciones necesarias para la identificación correcta de los parentales en cautiverio (Pineda et al. 2007).
Este tipo de introducciones se deben realizar con cautela, ya que se pueden mezclar individuos con una constitución genética diferente a la de las poblaciones receptoras, lo cual podría tener una implicación compleja en el mantenimiento de B. henni debido a que la incorporación de nuevos ejemplares no necesariamente implica una fácil adaptabilidad de éstos a su nuevo hábitat y a su vez se podrían reproducir con las sabaletas nativas de cada localidad.
Adicionalmente, introducciones de nuevos ejemplares de peces de una localidad a otra pueden generar la propagación de enfermedades. Un ejemplo de ello, es la introducción de los peces Pseudorasbora parva, Pungtungia herzi, Pseudogobio esocinus, Acheilognathus intermedia, Odontobutis interrupta, Zacco temmincki, Zacco platypus, y Hemibarbus labeo de China a Corea, lo cual a su vez provocó la propagación de clonorquiasis, producida por la bacteria parásita Clonorchis sinensis en Corea, agente causal de una enfermedad que era inexistente en este país (Kim et al. 2008).
Los valores de polimorfismo genético hallados en este estudio son similares a los hallados por otros autores (Wasko et al. 2004, Vieira et al. 2005, Lopera-Barrero et al. 2008), al utilizar esta misma clase de marcadores aleatorios en especies relacionadas y peces de aguas continentales, indicando que los marcadores RAPD utilizados en este trabajo reflejan un buen panorama de la variabilidad genética de la sabaleta ubicada en la cuenca del río Nare. Wasko et al. (2004) encontraron valores de polimorfismo de 85% en cuatro poblaciones amazónicas de B. cephalus. Lopera-Barrero et al. (2008), hallaron niveles de polimorfismo entre 54-59% en B. orbygnianus. Así mismo, en Tilapia Nilótica (Oreochromis niloticus), se hallaron niveles entre 47-80 % de polimorfismo. Para esta última especie, la amplitud en los rangos obtenidos se relaciona con el hecho de que las poblaciones con menores valores de variabilidad genética provienen de criaderos. Además, en el anterior trabajo las tilapias tuvieron procesos de introducciones en sus linajes en los sitios de cría, ya que es una especie que presenta una buena adaptación a los efectos del mejoramiento genético. Los menores valores de polimorfismo genético hallados en ese estudio están directamente relacionados con el tiempo de introducción de nuevos linajes (Vieira et al. 2005).
Se encontró una gran diferenciación genética entre las poblaciones muestreadas en ambas cuencas, lo cual podría demostrar el efecto de los embalses sobre la estructuración de poblaciones de B. henni genéticamente diferenciadas en la zona de muestreo, y por lo tanto, el bajo flujo genético entre ellas. Sin embargo, esta estructuración también puede ser explicada por los movimientos de migración que realiza esta especie (laterales cauce principal-quebradas-cauce principal y movimientos río arriba), los cuales pueden considerarse como movimientos de protección y no de migración reproductiva (Perdomo 1978, Jiménez et al. 1998). Estos movimientos podrían ser el efecto de estructuración más válido para esta especie respecto al efecto de separación geográfica generada por los embalses, debido a que éstos se construyeron hacia el año de 1971 (Juárez & Varsi 1992). Este es un tiempo relativamente corto para producir estructuración genética en la sabaleta del oriente antioqueño, B. henni, debido a que su ciclo de vida es relativamente largo (tiempo generacional 1.2-2.3 años) (Builes & Urán 1974, Perdomo 1978), como para que los embalses actúen como una barrera geográfica que genere divergencia genética en sus poblaciones.
Adicionalmente, es evidente la diferencia existente entre las dos cuencas muestreadas, con relación a los valores obtenidos de distancias genéticas, debido a que los datos muestran bajos valores entre las poblaciones El Nusito, San Lorenzo y Río Nare, de la cuenca del río Nare (0.054-0.077), así como altos niveles de distancia genética en la cuenca del río Guatapé (poblaciones Río Guatapé y El Cardal, 0.144-0.210 respectivamente). La explicación de este acontecimiento se puede deber a la ausencia de flujo genético entre ambas cuencas. Aunque en el presente trabajo no se estimó de forma directa el número de migrantes (Nm) entre las poblaciones evaluadas, se utilizó un método indirecto con el uso de la siguiente ecuación: FST = 1/(1+4Nm) (Wright 1931). A partir de esta fórmula, se puede tomar como análogo al valor FST el valor obtenido en los cálculos de estructuración genética (ΦST para las poblaciones de las dos cuencas muestreadas=0.297;ΦST cuenca del río Nare=0.163). Al despejar de la ecuación el número de migrantes, arrojó un valor bajo para todas las poblaciones en las dos cuencas bajo estudio (ríos Nare y Guatapé, Nm=0.59), así como para el caso de la cuenca del río Nare (Nm=1.0), lo cual corrobora los resultados obtenidos de estructuración genética, y demuestra que hay un bajo flujo genético entre las poblaciones de este estudio. Sin embargo, es importante tener en cuenta que, para confirmar estos resultados, es necesario evaluar otros marcadores moleculares (i.e. regiones de ADN microsatélite). Estos marcadores pueden demostrar los posibles efectos de la endogamia y podrían ayudar a determinar cuellos de botella, efecto Wahlund, deriva genética, entre otros (Frankham et al. 2002). Adicional a esto, un análisis de dispersión directa podría ser utilizado en esta especie mediante marca-recaptura, lo cual podría proveer elementos para un uso más adecuado de estimados de estructura genética en la búsqueda de indicadores de flujo genético (Whitlock & McCauley 1999).
El test de Mantel estima la correlación existente entre dos matrices de distancias, típicamente una matriz de distancias genéticas y una matriz de distancias geográficas, con un test de significancia por permutación. De acuerdo con los resultados obtenidos al correlacionar las matrices triangulares de distancias genética y geográfica, la significancia de este test fue alta, demostrado por la correlación positiva hallada (coeficiente de correlación Rxy=0.431, p<0.001, cuencas medias de los ríos Nare y Guatapé; Rxy=0.377, p<0.001, cuenca del río Nare), sugiriendo procesos de aislamiento por distancia en las poblaciones evaluadas (Peakall et al. 2003). En el modelo de aislamiento por distancia de Wright (1943), la población no está subdividida en subunidades donantes o receptoras de migrantes, ni es una unidad panmíctica. Los cruzamientos al azar están limitados por la distancia, de modo que los individuos tendrán una mayor probabilidad de aparearse con vecinos que con individuos más lejanos. De este modo, se pueden agrupar a los individuos en "vecindarios", áreas definidas por "individuos centrales" cuyos progenitores se pueden tratar como extraídos al azar (Wright 1943) y por lo tanto, si se requiere realizar repoblamientos de esta especie, se deberá tener más en cuenta las poblaciones vecinas que las poblaciones distanciadas genéticamente, para evitar una contaminación genética de la sabaleta de las cuencas medias de los ríos Nare y Guatapé en el oriente antioqueño.
El modelo de aislamiento por distancia asume que la discontinuidad en las poblaciones se produce por diversas causas. En este trabajo la discontinuidad en las poblaciones de B. henni se podría explicar por las barreras geográficas entre los sitios muestreados, sus barreras bióticas, pendientes de ríos muy altas y migraciones a corta distancia que realiza la especie (Jiménez et al. 1998), siendo el comportamiento migratorio una de las causas más plausibles observadas en este trabajo. Se han realizado estudios de aislamiento por distancia en otras especies de peces. En Columbia Británica (Canadá), se encontró un fuerte aislamiento por distancia en poblaciones del Salmón Oncorhynchus tshawytscha, en 22 localidades ubicadas entre Alaska y Columbia Británica, al evaluarse 32 sistemas de aloenzimas. Se encontró que la pendiente de la regresión fue -0.299 y r2=0.385; p<0.001 (Guthrie & Wilmot 2004). Otro estudio en el norte de Europa, el cual evaluó poblaciones de salmón del Atlántico (Salmo salar), un análisis con aloenzimas y microsatélites también demostró un aislamiento por distancia entre las poblaciones bálticas y del atlántico (r=0.477; p=0.001 (Tonteri et al. 2005). En ambos casos, las barreras geográficas fueron las causas del aislamiento por distancia. También puede citarse el trabajo de Mulvey et al. (2002), quienes realizaron un test de Mantel de tres vías usando distancia genética, distancia geográfica y distancia de contaminantes entre pares de sitios. Este test se utilizó para intentar correlacionar diferencias en contaminación de sedimentos con PAH (polycyclic aromatic hydrocarbons; hidrocarburos policíclicos aromáticos), con la distancia genética obtenida por marcadores isoenzimáticos entre las poblaciones evaluadas del pez Fundulus heteroclitus en diferentes sitios del estuario del río Elizabeth (Virginia, USA). El test de Mantel indicó una correlación positiva pero no significativa estadísticamente (r=0.47, α=0.05), entre la diferencia en contaminación de PAH en sedimentos y la distancia genética entre poblaciones de nueve sitios evaluados. No obstante, coeficientes de correlación muy bajos y no significativos estadísticamente indicaron que no existía relación entre la distancia genética y geográfica en peces capturados a una escala geográfica inferior (Mulvey et al. 2002).
En otra investigación, Bay et al. (2008), evaluaron la diversidad genética y estructura genética en la región control I del ADN mitocondrial entre regiones y arrecifes en el pez arrecifal Acanthochromis polyacanthus, el cual tiene amplio rango de distribución en la Gran Barrera de Coral de Australia. En este estudio se evaluaron mediante test de Mantel (1 000 permutaciones), correlaciones entre matrices de distancias genéticas y geográficas de pares de poblaciones, utilizando para ello el programa GenAlEx 6 (Peakall & Smouse 2006). Los resultados de análisis para el modelo de aislamiento por distancia de Wright (1943), indicaron que se halló correlación entre distancias genéticas y geográficas solo para algunas de las áreas muestreadas (ΦST vs. km: r=0.77, p=0.001). Sin embargo, esta correlación permaneció estadísticamente significativa cuando el número de arrecifes muestreados se redujo a seis (dos de cada una de las regiones evaluadas: norte, centro y sur), para obtener un número de comparaciones similar al evaluado dentro de regiones (ΦST vs. km: r=0.91, p=0.02). En contraste, las distancias genéticas y geográficas no se correlacionaron dentro de regiones (p>0.05 para todos los casos) (Bay et al. 2008). En el caso de las poblaciones de B. henni analizadas en el presente trabajo, los datos sugieren que se presenta aislamiento por distancia, lo cual podría ser explicado en parte por su comportamiento migratorio en cortas distancias (Builes & Urán 1974, Perdomo 1978, Jiménez et al. 1998).
En conclusión, en este trabajo los marcadores moleculares RAPD fueron útiles en la determinación de la variabilidad genética de B. henni, hallándose resultados de polimorfismo genético similares a trabajos previos realizados en esta especie. La estructuración genética y el aislamiento por distancia de B. henni, podrían explicarse por el comportamiento migratorio del pez en cortas distancias y por agrupaciones (clusters) genéticas en los dendrogramas de distancias que separaron las cuencas de los ríos Naré y Guatapé. Esta evaluación genética inicial es relevante para el conocimiento de la biología de B. henni en el oriente antioqueño, ya que aporta información útil para los planes de conservación y manejo de esta especie. Es recomendable que en futuras investigaciones con B. henni se utilicen marcadores moleculares codominantes, que puedan determinar niveles de endogamia y de heterocigosidades de sus poblaciones, entre otros análisis. Así mismo, se podrían implementar estudios morfológicos y morfométricos de la especie, análisis de depredadores potenciales de la sabaleta y estudios de calidad del agua de los sitios analizados, entre otros, que puedan ser usados como complemento de los resultados obtenidos en esta investigación.
Agradecimientos
A ISAGEN E.S.P., a la Corporación Autónoma Regional de los Ríos Negro y Nare -CORNARE y a la Universidad Nacional de Colombia Sede Medellín, por la financiación y logística necesaria para llevar a cabo esta investigación. Así mismo, a las Empresas Públicas de Medellín–EPM, por la logística brindada en el Campamento "La Araña" de la Central Hidroeléctrica Guatapé, en el municipio de San Rafael, Antioquia, Colombia.
Referencias
Barfai, R., S. Egedi, G.H. Yue, B. Kovacs, B. Urbanyi, G. Tamas, L. Horvath & L. Orban. 2003. Genetic analysis of two commom carp broodstocks by RAPD and microsatellite markers. Aquaculture 219: 157-167. [ Links ]
Barman, H.K., A. Barat, B.M. Yadav, S. Banerjee, P.K. Meher, P.V. Reddy & R.K. Jana. 2002. Genetic variation between four species of Indian major carps as revealed by random amplified polymorphic DNA assay. Aquaculture 202: 1-9. [ Links ]
Bay, L., J. Caley & R. Crozier. 2008. Meta-population structure in a coral reef fish demonstrated by genetic data on patterns of migration, extinction and re-colonisation. BMC Evol. Biol. 8: 248-264. [ Links ]
Builes, J. & A. Urán. 1974. Estudio del ciclo sexual de la Sabaleta Brycon henni Eigenmann: Su comportamiento y fecundación artificial. Actual. Biol. 7: 2-12. [ Links ]
Excoffier, L., P.E. Smouse & J.M. Quattro. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data. Genetics 131: 479-491. [ Links ]
Frankham, R., J. Ballou & D. Briscoe. 2002. Introduction to conservation genetics. Cambridge, Cambridge, England. [ Links ]
Guthrie III, C.M. & R. Wilmot. 2004. Genetic structure of wild chinook salmon Populations of Southeast Alaska and northern British Columbia. Environ. Biol. Fish. 69: 81-93. [ Links ]
Hartl, D.L. & A.G. Clark. 1997. Principles of population genetics. Sinauer, Sunderland, Massachussets, EEUU. [ Links ]
Jiménez, J.L., C. Román-Valencia & M. Cardona. 1998. Distribución y constancia de las comunidades de peces en la quebrada San Pablo, cuenca del Río La Paila, Alto Cauca, Colombia. Rev. Actual. Biol. 20: 21-27. [ Links ]
Juárez, J.R. & E. Varsi. 1992. Avances en el manejo y aprovechamiento acuícola de embalses en América Latina y el Caribe. Capítulo 3. Aprovechamiento Acuícola de Embalses en Colombia. Proyecto Aquila II. FAO, Organización de las Naciones Unidas para la Agricultura y la Alimentación, Roma, Italia. (Consultado: 28 marzo, 2009, http://www.fao.org/docrep/field/003/ab488s/AB488S00.htm#TOC). [ Links ]
Kang, J.H., J.K. Noh, J.H. Kim, J.H. Lee, H.C. Kim & K.K. Kim. 2006. Genetic Relationship Between Broodstocks of Olive Flounder, Paralichthys olivaceus (Temminck & Schlegel) Using Microsatellite Markers. Aquac. Res. 37: 701-707. [ Links ]
Katano, O., K. Hosoya, K. Iguchi, M. Yamaguchi, Y. Aonuma & S. Kitano. 2003. Species diversity and abundance of freshwater fishes in irrigation ditches around rice fields. Environ. Biol. Fish. 66: 107-121. [ Links ]
Katano, O., T. Nakamura, S. Abe, S. Yamamoto & Y. Baba. 2006. Comparison of fish communities between above- and below-dam sections of small streams; barrier effect to diadromous fishes. J. Fish Biol. 68: 767-782 [ Links ]
Kim, E.M., J.L. Kim, S.Y. Choi, J.W. Kim, S. Kim, M.H. Choi, Y.M. Bae, S.H. Lee & S.T. Hong. 2008. Infection Status of Freshwater Fish with Metacercariae of Clonorchis sinensis in Korea. Korean J. Parasitol. 46: 247-251. [ Links ]
Kumar, S., J. Dudley, M. Nei, M. Tamura. 2008. MEGA: A biologist-centric software for evolutionary analysis of DNA and protein sequences. Brief. Bioinform. 9: 299-306. [ Links ]
Lopera-Barrero, N.M., R.P. Ribeiro, S.N. Sirol, J.A. Povh, P.C. Gomes, L. Vargas & D.P. Streit Jr. 2006. Genetic diversity in piracanjuba populations (Brycon orbignyanus) with the RAPD (Random Amplified Polimorphic DNA) markers. J. Anim. Sci. 84: 170-170. [ Links ]
Lopera-Barrero, N., R. Ribeiro, S. Sirol, J. Povh, P. Gomes, L. Vargas & C. Mangolin. 2008. Variabilidad genética de lotes de Brycon orbignyanus utilizados en programas de repoblamiento: manejo y conservación. Acta Biol. Col. 13: 107-118. [ Links ]
Maldonado-Ocampo, J.A., A. Ortega, J.S. Usma, G. Galvis, F.A. Villa, L. Vásquez, S. Prada & C. Ardila. 2005. Peces de los Andes de Colombia. Instituto de Investigación de Recursos Biológicos Alexander von Humboldt, Bogotá D.C., Colombia. [ Links ]
Maldonado-Ocampo, J.A., R.P. Vari & J.S. Usma. 2008. Checklist of the Freshwater Fishes of Colombia. Biota Col. 9: 143-237. [ Links ]
Michalakis, Y. & L. Excoffier. 1996. A generic estimation of population subdivision using distances between alleles with special reference to microsatellite loci. Genetics 142: 1061-1064. [ Links ]
Mojica, J.I. 1999. Lista preliminar de las especies de peces dulceacuícolas de Colombia. Rev. Acad. Col. Ciencias Ex. Fis. Nat. 23: 547-566. [ Links ]
Mojica, J.I., C. Castellanos, J.S. Usma & R. Álvarez. 2002. Libro rojo de peces dulceacuícolas de Colombia. Serie Libros Rojos de Especies Amenazadas de Colombia. Instituto de Ciencias Naturales, Universidad Nacional de Colombia y Ministerio del Medio Ambiente, Bogotá, Colombia. [ Links ]
Montoya, A., L. Carrillo & M. Olivera. 2006. Algunos aspectos biológicos y del manejo en cautiverio de la sabaleta Brycon henni Eigenmann, 1913 (Pisces: Characidae). Rev. Col. Cienc. Pec. 19: 180-186. [ Links ]
Moreira, A.A., A.W.S. Hilsdorf, J.V. Silva & D.A. Souza. 2007. Variabilidade genética de duas variedades de tilápia nilótica por meio de marcadores microssatélites. Pesqui. Agropecu. Bras. 42: 521-526. [ Links ]
Morita, K. & S. Yamamoto. 2002. Effects of habitat fragmentation by damming on the persistence of stream-dwelling charr populations. Conserv. Biol. 16: 1318-1323. [ Links ]
Mulvey, M., M. Newman, W. Vogelbein & M. Unger. 2002. Genetic structure of Fundulus heteroclitus from PAH-contaminated and neighboring sites in the Elizabeth and York Rivers. Aquat. Toxicol. 61: 195-209. [ Links ]
Nei, M. 1973. Analysis of Gene Diversity in Subdivided Populations. P. Natl. Acad. Sci. USA. 70: 3321-3323. [ Links ]
Nei, M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics 89: 583-590. [ Links ]
Peakall, R., M. Ruibal & D.B. Lindenmayer. 2003. Spatial autocorrelation analysis offers new insights into gene flow in the Australian bush rat, Rattus fuscipes. Evolution 57: 182-1195. [ Links ]
Peakall, R. & P.E. Smouse. 2006. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research. Mol. Ecol. Notes 6: 288-295. [ Links ]
Perdomo, J. 1978. La sabaleta (Brycon henni) observaciones bioecológicas y su importancia como especie de cultivo. Rev. Div. Pesq. 11: 1-46. [ Links ]
Pineda, H., D. Pareja, M. Olivera & J. Builes. 2004. Contribución a la relación taxonómica entre cuatro especies de peces de la familia Characidae mediante el Polimorfismo de ADN Amplificado al Azar (RAPD). Rev. Col. Cienc. Pec. 17: 30-37. [ Links ]
Pineda, H., L. Arboleda, A. Echeverry, S. Urcuqui, D. Pareja, M. Olivera & J. Builes. 2007. Caracterización de la diversidad genética en el pez Brycon henni (Characiformes: Characidae) en Colombia central por medio de marcadores RAPD. Rev. Biol. Trop. 55: 1025-1035. [ Links ]
Povh, J.A., H.L.M. Moreira, R.P. Ribeiro, A.P. Prioli, L. Vargas & D.V. Blanck. 2005. Estimativa da variabilidade genética em tilápia do Nilo (Oreochromis niloticus) com a técnica de RAPD. Acta Sci. An. Sci. 27: 1-10. [ Links ]
Sambrook, J., E.F. Fritsch & T. Maniatis. 1989. Molecular cloning: a laboratory manual. Cold Spring Harbor Laboratory, Cold Spring Harbor, Nueva York, EEUU. [ Links ]
Soto-Marambio, E.R. 2003. Variación morfológica, variación genética y estructura poblacional en poblaciones alopátricas de Bufo spinulosus (Anura: Bufonidae) en Chile. Tesis de Maestría, Universidad de Chile, Santiago de Chile, Chile. (Consultado: 13 septiembre, 2008, http://www.cybertesis.cl/tesis/uchile/2003/soto_e/html/index-frames.html). [ Links ]
U.I.C.N. 2003. Lista Roja de Especies en Peligro. Unión Internacional para la Conservación de la Naturaleza, Gland, Suiza. (Consultado: 13 febrero 2009, http://www.redlist.org). [ Links ]
Taggart, J.B., R.A. Hynes, P.A. Prodohl & A. Ferguson. 1992. A simplified protocol for routine DNA isolation from salmonid fishes. J. Fish Biol. 40: 963-965. [ Links ]
Tonteri, A., S. Titov, A. Veselov, A. Zubchenko, M.T. Kosikinen, D. Lesbarreréres, S. Kaluzhin, I. Bakhmet, J. Lumme & C.R. Primmer. 2005. Phylogeography of anadromous and non-anadromous Atlantic salmon (Salmo salar) from Northern Europe. Ann. Zool. Fennici 42: 1-22. [ Links ]
Vieira, V., R. Ribeiro, L. Vargas, H. Marques, J. Povh & N. Lopera. 2005. Avaliação da variabilidade genética de linhagens de tilápia do nilo (Oreochromis niloticus) com o uso do marcador de RAPD. Rev. Acad. Curitiba 3: 41-49. [ Links ]
Wasko, A.P., C. Martins, C. Oliveira, J.A. Senhorini & F. Foresti. 2004. Genetic monitoring of the Amazonian fish matrinchã (Brycon cephalus) using RAPD markers: insights into supportive breeding and conservation programmes. J. Appl. Ichthyol. 20: 48-52. [ Links ]
Whitlock, M. & D. MacCauley. 1999. Indirect measures of gene flow and migration: FST≠1/(4Nm+1). Heredity 82: 117-125. [ Links ]
Williams, J.G.K., M.K. Kubelik, K.J. Licak, J.A. Rafalski & S.V. Tingey. 1990. DNA polymorphism amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res. 18: 6531-6535. [ Links ]
Winkler, F.M., D. Bartley & N.F. Díaz. 1999. Genetic differences among year classes in a hatchery population of coho salmon (Oncorhynchus kisutch (Walbaum, 1792)) in Chile. Aquaculture 173: 425-433. [ Links ]
Wright, S. 1931. Evolution in mendelian populations. Genetics 16: 97-158. [ Links ]
Wright, S. 1943. Isolation by distance. Genetics 38: 114-138. [ Links ]
Wright, S. 1965. The interpretation of population structure by F-statistics with special regards to systems of mating. Evolution 19: 395-420. [ Links ]
Correspondencia a: Julio César Hurtado-Alarcón. Departamento de Ciencias Forestales, Universidad Nacional de Colombia, Calle 59A No 63-20, Bloque 11 Oficina 213, Medellín, Colombia. Fax: (54) (4) 4309080; jchurtad@unal.edu.co
Néstor J. Mancera-Rodríguez. Postgrado en Bosques y Conservación Ambiental, Universidad Nacional de Colombia, Sede Medellín, Colombia; njmancer@unal.edu.co
Clara I. Saldamando-Benjumea. Escuela de Biociencias, Universidad Nacional de Colombia, Sede Medellín, Colombia; cisaldam@unal.edu.co
Recibido 07-IV-2010. Corregido 14-IX-2010. Aceptado 12-X-2010.