Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista de Biología Tropical
On-line version ISSN 0034-7744Print version ISSN 0034-7744
Rev. biol. trop vol.55 n.2 San José Jun. 2007
Relaciones filogenéticas entre géneros de ciervos neotropicales (Artiodactyla: Cervidae) mediante secuenciación de ADN mitocondrial y marcadores microsatelitales
Manuel Ruiz-García1, Ettore Randi2, María Martínez-Agüero1 & Diana Alvarez1
1 Unidad de Genética (Genética de Poblaciones-Biología Evolutiva), Departamento de Biología, Facultad de Ciencias, Pontificia Universidad Javeriana, Cra 7ª No 43-82, Bogotá DC, Colombia; mruiz@javeriana.edu.co
2 Instituto Nazionale per la Fauna Selvática, Ozzano dell´Emilia, Bologna, Italia.
Recibido 06-VII-2003. Corregido 18-IX-2006. Aceptado 10-XI-2006.
Abstract: Phylogenetic relationships among Neotropical deer genera (Artiodactyla: Cervidae) by means of DNAmt sequences and microsatellite markers. The current work shows two molecular phylogenetic analyses on Neotropical deers. In the first analysis, the mitochondrial control region (D-loop) was sequenced in six Odocoileinae species from Latin America, using the sequences of two Muntiacinae as outgroups. The results obtained were as follows: A sequence of Mazama americana showed a striking relationship with several sequences of Odocoileus in contrast to that expected, since this M. americana haplotype, from a Mexican origin, was not associated with several Bolivian Mazama sequences analyzed. This could put forward that this genera is not monophyletic. On the other hand, these Bolivian Mazama formed a clade with Pudu puda and Ozotoceros bezoarticus. Likely, an Odocoileus virginianus sequence from the Central area of Colombia showed a more strong relationship with a Northamerican O. heminonus sequence than with the other O. virginianus sequences of Colombian origin as well. This could be explained by means of various different hypotheses. The first is the existence of common ancestral haplotypes between both species. Another one is the reiterative hybridization among both Odocoileus species before the migration of O. virginianus from North America to South America. Moreover, the maximum parsimony analysis showed an intense relationship between the Muntiacinae and this Neotropical Cervidae clade. In addition, and adding credence to the relevant polyphyletism found in Mazama by means of the mitochondrial control region DNA sequences, a second analysis with 16 DNA microsatellite loci also showed a higher genetic relationship between M. americana and O. virginianus, than between the first species regard to Mazama gouazoubira. Rev. Biol. Trop. 55 (2): 723-741. Epub 2007 June, 29.
Key words: molecular phylogeny, neotropical deers, DNAmt, microsatellites, Mazama, Odocoileus.
La sistemática de los cérvidos neotropicales ha sido poco estudiada y, además, es profundamente controvertida. Tradicionalmente los ciervos neotropicales han sido divididos en tres grupos de acuerdo con el tamaño corporal y a la forma de la ramificación de los cuernos. De este modo, los ciervos de alto porte (Odocoileus, Blastocerus y Ozotoceros) formarían un grupo, las dos especies del género Hippocamelus conformarían el grupo de mediano porte y las especies de los géneros Mazama y Pudu constituirían el tercer grupo (ciervos de pequeño porte) (Duarte y Merino 1997). Sin embargo, no todos los autores coinciden con este esquema. Algunos ejemplos ponen en evidencia esta afirmación. Para diversos auto res los géneros de ciervos Blastocerus (Wagner, 1844) y Ozotoceros (Ameghino, 1891), cuya distribución geográfica, para el primero, se da desde la Amazonía peruana, extendiéndose por el centro de Brasil y este de Bolivia, alcanzan do Paraguay y norte de Argentina, mientras que para el segundo se da por el centro y sur de Brasil, Bolivia, Paraguay, Uruguay y noreste y centro de Argentina, se deberían fusionar al género Odocoileus (Rafinesque, 1832). Este ultimo posee una distribución que abarca la mayor parte de Estados Unidos, buena parte de Canadá, Centroamérica y Suramérica al norte del Amazonas (Colombia, venezuela, Guyana, Surinam, Ecuador y Perú) (Ximenes et al. 1972). Por el contrario, otros autores basándose en otros datos morfológicos (Wilson y Reeder 1993) diferencian claramente los tres géneros. Más compleja, todavía, es la sistemática intragenérica de Mazama (Rafinesque 1817). Este género de pequeños cérvidos es enormemente controvertido en cuanto al número de especies que puede contener en su seno. Allen (1915) separó a Mazama en dos grandes ensambles en función de la coloración del pelaje. El grupo de coloración rojiza contuvo las especies M. americana, M. trinitatis, M. rufa, M. sheila, M. rufina, M. bricenii, M. sartorii, M. zetta, M. gualea, M. fuscata y M. zamora. El grupo de coloración grisácea estuvo integrado por las especies M. simplicicornis, M. murelia, M. tschudii, M. nemorivagus, M. superciliaris, M. cita y M. pandora. Cabrera (1960) propuso la clasificación más aceptada todavía en nuestros días: Mazama contendría las especies M. americana, M. gouazoubira, M. chunyi y M. rufina. Las especies citadas por Allen (1915) serían simplemente subespecies de las cuatro especies nominadas por Cabrera (1960). Czernay (1987), más recientemente, definió la existencia de 6 especies para el género en cuestión: M. americana, M. gouazoubira, M. rufina, M. bricenii, M. chunyi y M. nana. Esta última substituiría a la población aislada de M. rufina en el sur del Brasil. Duarte (1992) y Duarte y Merino (1997) definieron cromosómicamente una nueva especie a partir del material analizado de un individuo capturado en la región de Capao Bonito (Sao Paulo). Este taxón fue denominado M. bororo. Es evidente que se hace necesario el análisis de representantes de esos géneros para intentar dilucidar las respectivas relaciones inter e intraespecíficas de los cérvidos sudamericanos desde una perspectiva molecular.
Caben resaltar algunos trabajos moleculares, que han incluido especies de cérvidos neotropicales, como los de Douzery y Randi (1997) y Smith et al. (1986). En el primero se analizó la región de control (CR) del ADN mitocondrial, también conocida como D-loop, en seis especies de Cervidae, representantes de las subfamilias Cervinae (Cervus elaphus, 2 individuos; Cervus nippon, 1 y Dama dama, 1), Hydropotinae (Hydropotes inermis, 1) y Odocoileinae (tribu Odocoileini: Odocoileus virginianus, 1; Odocoileus hemionus,1 y Mazama americana, 1; tribu Capreolini: Capreolus capreolus, 1 y Capreolus pygargus, 1). En el referido trabajo se determinó una fuerte relación entre las dos especies de Odocoileus, ambas de procedencia norteamericana, sustentada por un árbol de máxima parsimonia, y con un "bootstrap" para ese clado del 100 %. El taxón hermano de ese clado fue M. americana con un "bootstrap" del 99 %. Sin embargo, las restantes especies analizadas no pertenecieron a la fauna de cérvidos neotropicales, con lo que no se profundizó en la relación filogenética entre los géneros sudamericanos de cérvidos. En el segundo trabajo mencionado, utilizando datos morfológicos e isoenzimáticos, se constató una fuerte relación entre M. americana y O. virginianus del Surinam, mientras que la primera especie presentó menor relación con M. gouazoubira, y mientras la segunda especie presentó menor semejanza de la prevista con ejemplares de O. virginianus de Estados Unidos. Es decir, en ese estudio no se encontraron relaciones monofiléticas entre diferentes poblaciones, para ninguno de ambos géneros, Odocoileus y Mazama. Para intentar dilucidar algunas de esas incertidumbres filogenéticas, se muestra en el presente estudio un análisis a partir de la secuenciación de la región de control del ADN mitocondrial (1 133 pares de bases) de cuatro O. virginianus procedentes de la región central de Colombia, de un O. hemionus proveniente de Estados Unidos, de una M. americana procedente de México analizada por Douzery y Randi (1997), de dos individuos "a priori" clasificados morfológicamente como M. americana, muestreados en Bolivia en 1997 por dos de los autores (M. R-G y D. A), de un "a priori" clasificado morfologicamente M. gouazoubira, muestreado, también, en Bolivia por los mismos autores, de un Ozotoceros bezoarticus de procedencia boliviana, y de un Pudu puda de origen chileno. Como especies externas ("outgroups") se utilizaron las secuencias de otros dos cérvidos, Muntiacus reevesi y Elaphodus cephalophus pertenecientes a la subfamilia de los Muntiacinae (= Cervulinae).
Igualmente, se construyeron árboles de máxima parsimonia a partir de 16 loci microsatélites analizados obtenidos en diversas especies de bóvidos, del reno (Rangifer tarandus) y del ciervo coli-blanco (O. virginianus). Estos marcadores microsatélites fueron RM012, BM757, INRA131, FCB193, TGLA337, HUJ175, BOvIRBP, TGLA127, FEB, IDvGA055 (Bovidae; Slate et al. 1998), Cervid 1, Cervid 3 (O. virginianus; Dewoody et al. 1995), y NvHRT16, NvHRT30, NvHRT71 y NvHRT73 (renos; Roed y Midthjel 1998). La presencia-ausencia de los diversos alelos encontrados en esos 16 microsatelites fueron utilizados en conjunto para evaluar el grado de similitud con las filogenias obtenidas a partir de la región de control mitocondrial y de las filogenias obtenidas mediante el uso de isoenzimas y características biométricas por Smith et al. (1986). Aunque diferentes autores han mostrado que los marcadores microsatélites son excelentes marcadores para la realización de mapas de ligamiento y para estudios forenses, genético poblacionales y de parentesco, otros análisis han mostrado posibles restricciones de estos marcadores en tareas de reconstrucción filogenética. Esto estaría motivado, principal-mente, por cambios en la estructura interna de los motivos de repetición sin afectar los tamaños globales de los alelos (Garza y Freimer 1996) o, por el hecho, de que mutaciones independientes pueden producir alelos de idénticos tamaños partiendo de alelos de diferentes tamaños (homoplasias) (Goldstein y Pollock 1997). Sin embargo, ciertos autores como Morin et al. (1993), Blanquer-Maumont y Crouau-Roy (1995), Fitzsimmons et al. (1995) y Goldstein et al. (1995), han mostrado la utilidad potencial en casos determinados de los microsatélites para realizar inferencias filogenéticas entre géneros relativamente diferenciados. En el estudio de Goldstein et al. (1995), se mostró como 30 loci microsatélites reconstruyeron adecuadamente las relaciones filogenéticas entre humanos, chimpancés y gorilas. Morin et al. (1993) mostraron, utilizando microsatélites, que las subespecies actualmente reconocidas de chimpancés podrían, en realidad, ser reconocidas como especies diferentes debido a su fuerte diferenciación genética. Blanquer-Maumont y Crouau-Roy (1995) mostraron que tres microsatélites localizados en el cromosoma humano 6p están conservados en diferentes especies de dos superfamilias de Primates, los Hominoidea y los Cercopithecoidea. Los referidos autores concluyeron en la enorme utilidad de esos marcadores desde un punto de vista filogenético. Por último, Fitzsimmons et al. (1995) mostraron la existencia de loci microsatélites homólogos en seis especies de tortugas marinas pertenecientes a dos familias diferentes (Cheloniidae y Dermochelyidae), al igual que en una familia de tortugas dulceacuícolas (Emydidae), lo que indicó una conservación de las secuencias flanqueantes de esos microsatélites de, al menos, 300 millones de años. Igualmente, Ruiz-García (2007) ha mostrado que un microsatélite, AP68, reconstruye adecuadamente la evolución de la familia Atelidae en los últimos 16 millones de años. Aunque no se muestra con detalle aquí, las simulaciones de Di Rienzo et al. (1994), Nielsen (1997) y Nielsen & Palsboll (1999) revelaron que la mayoría de los marcadores microsatélites analizados en este estudio, al igual que los aplicados en otras especies neotropicales, como el oso andino (Tremarctos ornatus), el jaguar (Panthera onca), el delfin rosado (Inia sp.) o en algunas especies de Primates, como los aulladores (Alouatta) y monos araña (Ateles) (Ruiz-García 2003, 2005, Ruiz-García et al. 2005, 2006a,b,c,d), evolucionan preferentemente siguiendo el modelo mutacional de un paso ("one-step"), mientras que los modelos mutacionales en dos fases ("two-phase") o multipasos ("multistep") son mucho menos frecuentes. Si estos dos modelos mutacionales fueran importantes podrían causar sobrelapamiento de alelos con el mismo tamaño sin coalescencia común (homoplasia). Sin embargo, como estos modelos mutacionales son relativamente poco frecuentes, esto reduce la posibilidad de alelos homoplásicos. Adicionalmente, los resultados filogenéticos obtenidos con microsatélites fueron muy similares a los obtenidos con otros marcadores para los géneros Odocoileus y Mazama. Por ello son presentados en este estudio.
Materiales y métodos
Análisis de la región de control del ADN mitocondrial: para el análisis filogenético llevado a cabo con cérvidos neotropicales se obtuvieron las siguientes muestras: Se obtuvo una muestra de ADN de M. americana (Y08209), de origen mexicano, del mismo tejido que utilizaron Douzery et al. (1995) en un estudio previo. Cuatro muestras de O. virginianus procedentes del área central de Colombia. Estas muestras consistieron en mechones de pelos con sus respectivos bulbos. La secuencia de un individuo norteamericano de O. hemionus fue obtenida por Feng et al. (no publicado, número de acceso U12865 en el banco de datos EMBL). Una muestra de pelos con bulbos de O. bezoarticus obtenida en la zona de Otuquis en Bolivia. Dos muestras de M. americana, de la misma naturaleza que la anterior, procedente de la región de Santa Cruz en Bolivia, una muestra de pelos de una M. gouazoubira procedente de la región de Isoso en Bolivia y una muestra de tejido de un P. puda procedente de Chile. Como grupos externos fueron utilizadas las secuencias obtenidas de tejidos de dos Muntiacinae, M. reevesi y E. cephalophus.
Las muestras de ADN, en el caso de los tejidos, fueron extraídas con el método estándar del fenol-cloroformo (Sambrock et al. 1989), mientras que las procedentes de los bulbos de pelo se obtuvieron con una solución de 200 µl Chelex al 10 %. Las secuencias de 1 133 pb de la región de control del ADN mitocondrial (D-loop) se amplificó vía PCR utilizando los cebadores ("primers") L-Pro 5-CGTCAGTCT CACCATCAACCCCCAAAGC-3 y H-Phe 5GGGAGACTCATCTAGGCATTTTCAGTG3, los cuales están adyacentes a las posiciones 15 740 y 420 de los genes mitocondriales bovinos tRNAPro y tRNAPhe. Las amplificaciones se realizaron con AmpliTaq DNA polimerasa y con MgCl2 3 mM, utilizando un termociclador 9.600 Perkin Elmer con los siguientes ciclos: 94 ºC durante 2 min, y 30 ciclos a 94 ºC durante 15 s, 55 ºC durante 15 s y 72 ºC durante 1 min. Finalmente, 10 min a 72 ºC. Los productos obtenidos de esos PCR se purificaron en geles de agarosa de baja temperatura de fusión. Las secuencias fueron obtenidas por ciclos de ADN de dos cadenas con Δtaq Sequenase, utilizando los cebadores (primers) externos ya citados (L-Pro y H-Phe) y los cebadores adyacentes a la zona media de la regiσn central conservada de los artiodáctilos (L-362 5AATCACCATGCCGCGTGAAACC-3y H493 5-TGAGATGGCCCTGAAGAAAGAACC-3).
Una vez obtenidas las secuencias, éstas fueron alineadas utilizando el programa Clustal W (Thompson et al. 1994). En regiones altamente variables, se introdujeron huecos ("gaps") cuando implicaron al menos dos substituciones y fueron agrupados para maximizar la similitud global. Inserciones grandes (47 y 75 pb) fueron excluidas de los análisis filogenéticos. Se utilizaron otros programas, como Aligner, Alignseq, DNANALYZE 1.8, MultiDNA, Seqvu 1.1 y Threealign 2.0, y siempre se obtuvieron los mismos resultados en la alineación de las secuencias.
Análisis de los marcadores microsatélites: como se comentó anteriormente, aunque los marcadores microsatélites pueden presentar problemas para desempeñar tareas de análisis filogenético, los resultados en el presente estudio coinciden notablemente con los obtenidos por Smith et al. (1986) haciendo uso de variables morfométricas e isoenzimáticas. Los microsatélites empleados fueron RM012, BM757, INRA131, IDvGA055, FCB193, TGLA337, HUJ175, BOvIRBP, TGL127 y FSHB (diseñados para bóvidos; Slate et al. 1998), Cervid 1 y Cervid 3 (diseñados para O. virginianus; Dewoody et al. 1995), y NVHRT 16, NVHRT 30, NVHRT 71 y NVHRT 73 (diseñados para renos; Roed y Midthjell 1998). En otro trabajo (Ruiz-García et al. 2006e), se muestran las condiciones empleadas en las reacciones de PCR, las temperaturas utilizadas y el origen geográfico preciso de las 120 muestras de cérvidos neotropicales (71 muestras pertenecientes a M. americana, M. gouzaoubira, M. rufina, O. virginianus, Hippocamelus antisensis, Pudu mephistopheles, O. bezoarticus, Blastoceros dichotomus) y europeo-asiáticos (49 muestras pertenecientes a Cervus elaphus, C. nippon, Capreolus capreolus, C. pygargus y Dama dama).
Análisis matemáticos de las secuencias mitocondriales: en primer lugar, se obtuvieron los estadísticos π (diversidad nucleotídica, número promedio de diferencias nucleotídicas por sitio entre dos secuencias escogidas al azar) con su varianza de muestreo y error estándar, theta (θ) por nucleótido, K (= número promedio de diferencias nucleotídicas, Tajima 1983) con sus varianzas de muestreo y estocásticas, y M (=2Ne 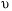
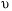
A partir de todas las secuencias nucleotídicas obtenidas, se calcularon las matrices de distancias genéticas entre pares de secuencias utilizando los métodos de los dos parámetros de Kimura (1980) y el de Tajima & Nei (1984). Para el primer procedimiento, se tienen en consideración posibles diferencias entre transiciones (P) y transversiones (Q), siendo el número de substituciones por sitio nucleotídico entre dos secuencias dadas igual a
K = (1/2) ln (a) + (1/4) ln (b), dónde a = 1/(1 - 2P - Q) y b = 1/(1 - 2Q). En el segundo método, en el cual las frecuencias de los 4 nucleótidos pueden ser diferentes,
K=-b ln (1 -(p/b)2), dónde b=(b1 + p2 / h)/2, siendo b1=1 - Σ qi , con qi siendo la frecuencia en equilibrio del nucleótido ith (i = 1, 2, 3, 4, esto es A, G, T, C), y
h=Σi = 1-3 Σj = i+1 - 4 (xij)2/(2 qi qj), dónde xij es la proporción de pares de nucleótidos i y j entre dos secuencias de ADN homólogas comparadas (Li 1997), siendo p la probabilidad que dos secuencias sean diferentes en un sitio nucleotídico dado en un tiempo t determinado.
Los resultados con otras distancias genéticas, como la de Jukes-Cantor (1969), la del modelo de los 6 parámetros de Kimura (1981), o la de Tamura y Nei (1993), ofrecieron resultados idénticos a los mostrados en este trabajo.
A esas matrices de distancias genéticas se les aplicaron los algoritmos UPGMA (Sneath y Sokal 1973) y neighbor-joining (Saitou y Nei 1987) para obtener diversos árboles filogenéticos entre los géneros de cérvidos americanos analizados. Estos análisis se llevaron a cabo con el programa MEGA 1.2 (Kumar et al. 1993). Para analizar la significación de los diversos clados obtenidos se aplicó el método "bootstrap" con 1 000 permutaciones. valores por encima del 75 % se consideran altamente significativos. También, se obtuvieron árboles filogenéticos utilizando el método de máxima parsimonia con los procedimientos "heuristic search" y "branch and bound search" con el programa PAUP 3.1.1 (Swofford 1993). El porcentaje de "bootstrap" fue utilizado, de nuevo, para determinar la significación de los diversos clados encontrados.
Análisis filogenéticos con marcadores microsatélites: basándose en los alelos detectados (presencia-ausencia) para los diferentes marcadores microsatélites estudiados, se construyeron filogenias a partir de diversos métodos de máxima parsimonia. Esta técnica permite minimizar la longitud de la filogenia obtenida. Dos procedimientos principales fueron utilizados: "heuristic search" y "branch and bound search". Con el primero, las estrategias empleadas fueron el procedimiento general con almacenamiento en cada paso del árbol, utilizando adición paso a paso. El método "branch and bound" fue computado vía "stepwise". Para elegir los árboles más parsimoniosos se utilizaron la longitud del árbol, el índice de consistencia (CI), el índice de retención (RI), el índice de consistencia rescalado (RC) y el índice de homoplasia (HI). Para determinar la consistencia de los diferentes clados internos se realizaron análisis de "bootstraps" con 500 replicaciones. Con los diversos árboles de máxima parsimonia encontrados se construyeron árboles consenso con los métodos estricto, semiestricto, regla de la mayoría y Adams.
Resultados
Análisis filogenético de la región control mitocondrial: para los géneros representados por más de un individuo (Odocoileus y Mazama), se estimaron una serie de estadísticos básicos. En el caso de O. virginianus se presentaron 106 gaps en los 1 133 pb analizados. De los 1 027 pb restantes, 84 fueron sitios polimórficos y 943 monomórficos. De esos 84 sitios polimórficos, 24 fueron informativos filogenéticamente con dos variantes y dos fueron informativos con tres variantes nucleotídicas. En el Cuadro 1 se muestran los estadísticos moleculares encontrados para esa especie. La diversidad nucleotídica (π) fue de 0.0454, θ por nucleσtido de 0.0446, k fue 46.67 y M fue de 45.82. El análisis de desequilibrio de ligamiento para los 24 sitios nucleotídicos que fueron polimórficos para dos nucleótidos diferentes mostró 276 pares de comparaciones. Con el test exacto de Fisher no se detectó ningún par de nucleótidos que presentara desequilibrio significativo. Por el contrario, al utilizar el test ji-cuadrado se detectaron 193 pares de comparaciones significativas (χ2 =4.0, 1 g.l., p<0.05) (193/276=69.93 %). Este valor es significativamente mayor que el error I del 5 % (χ2 =248.31, 1 g.l., p<0.001). Sin embargo, si utilizamos la corrección estándar de Bonferroni (α=0.05/276=0.000181), ninguno de los valores de desequilibrio individuales entre pares de nucleótidos resultó ser significativo.

En el caso del género Mazama procedente de Bolivia, sin incluir la muestra Mazama americana de origen mexicano, se presentaron 220 huecos en las 1 133 pb analizadas. De los 913 pb restantes, 30 fueron polimórficos para dos variantes nucleotídicas. En este caso no se encontraron sitios polimórficos para tres o cuatro variantes. Los niveles encontrados de diversidad genética a nivel de la región de control del ADN mitocondrial en esos Mazama de Bolivia fueron claramente inferiores a los mostrados por los ejemplares de O. virginianus procedentes de Colombia (Cuadro 1), aun cuando, supuestamente, pertenecen a dos especies morfológicamente diferentes (M. americana y M. gouazoubira). La diversidad nucleotídica fue 0.0219, es decir, menos de la mitad de la encontrada en la especie anterior, mientras que K y M fueron igual a 20. No se presentó ningún caso de desequilibrio de ligamiento. También, se analizaron esos mismos estadísticos, incluyendo la secuencia del ejemplar de M. americana de origen mexicano. El número de huecos encontrados fue de 241 en la secuencia de 1 133 pb analizadas. La introducción de esta secuencia aumentó considerablemente el número de sitios polimórficos a 106 en los restantes 892 pb. Cinco de ellos correspondieron a variantes de dos nucleótidos que fueron filogenéticamente informativos. Además, se dieron 4 sitios polimórficos con tres variantes nucleotídicas, todas ellas informativas. En el cuadro 1 se muestran los parámetros moleculares correspondientes a esta agrupación. Como puede observarse, el incremento de la diversidad nucleotídica fue elevada (π=0.0618), tres veces superior a la encontrada sin incluir esa muestra. También se incrementaron los valores de θ por nucleótido, K y M (0.062, 55.17 y 57.82). Esto demuestra que la citada Mazama diverge considerablemente a nivel molecular en la región de control del ADN mitocondrial respecto de los ejemplares de Mazama de origen boliviano. Es posible, que estos últimos ejemplares, aun cuando presentaron aspectos diversos, unas más rojizas otras más grisáceas, en realidad pertenezcan únicamente al taxón designado como M. gouzaoubira. Adicionalmente, la introducción de la muestra mexicana generó una cierta cantidad de desequilibrio de ligamiento entre los 5 sitios nucleótidos polimórficos para dos variantes (10 pares de combinaciones), al utilizar el test ji-cuadrado (4/10=40 %; cada par significativo con χ2=4.00, 1 g.l., p<0.05). Sin embargo, con el test exacto de Fisher ningún caso fue significativo estadísticamente y, tampoco, la aplicación de la corrección estándar de Bonferroni (α=0.05/10=0.005) a los tests ji-cuadrado, determinó algún caso de desequilibrio de ligamiento significativo.
El test de neutralidad de Tajima (1989) mostró valores iguales a 0.1936 (caso de O. virginianus), 0 (caso de Mazama bolivianas) y -0.4799 (caso de todas las Mazama consideradas simultáneamente). En los dos primeros casos, esos valores no fueron significativos, asumiendo que seguían una distribución beta. Luego se estima que el modelo mutacional es plena-mente neutral. En el tercer caso, el valor fue significativo y negativo, lo que podría indicar la presencia ligera de mutaciones deletéreas.
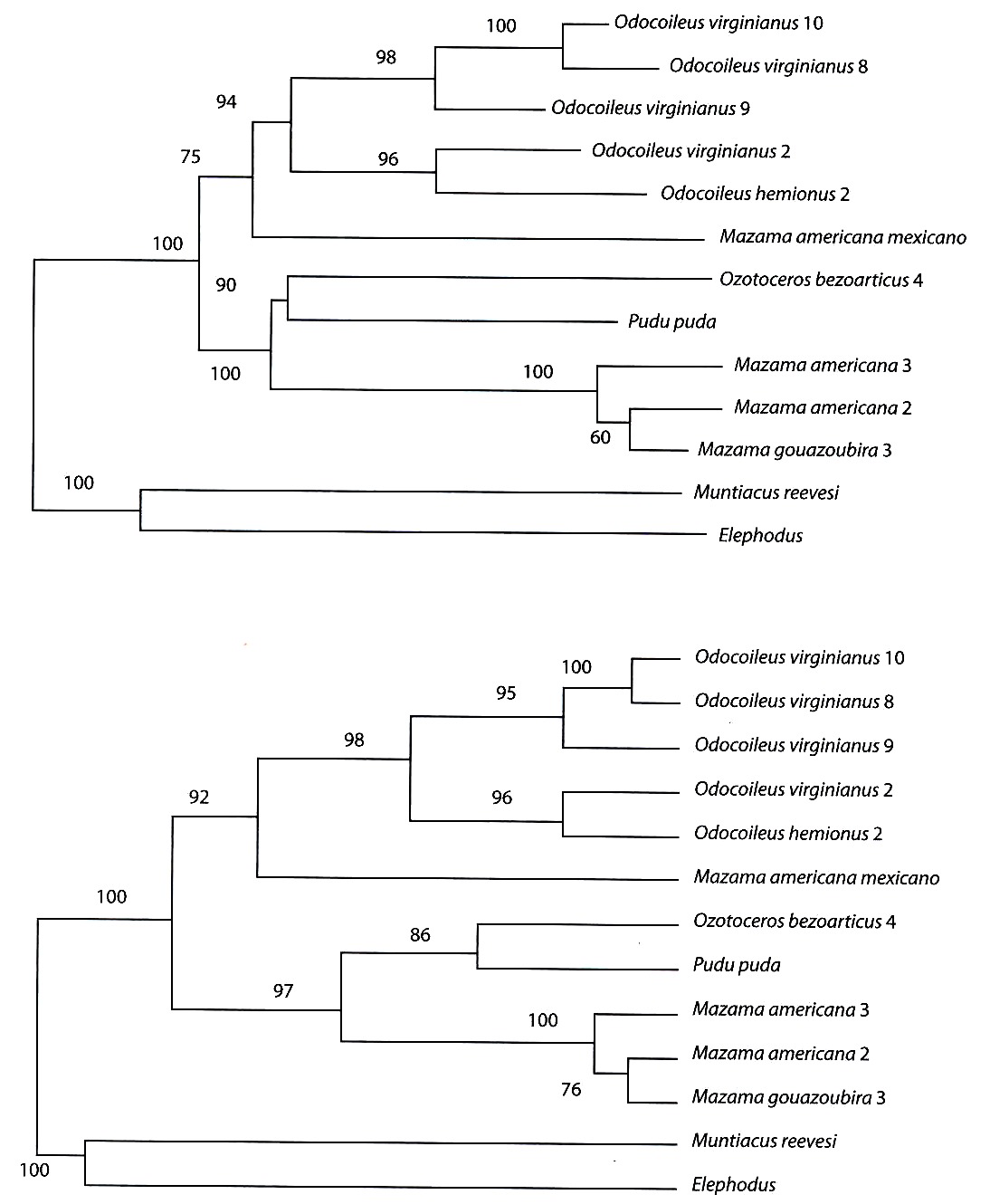
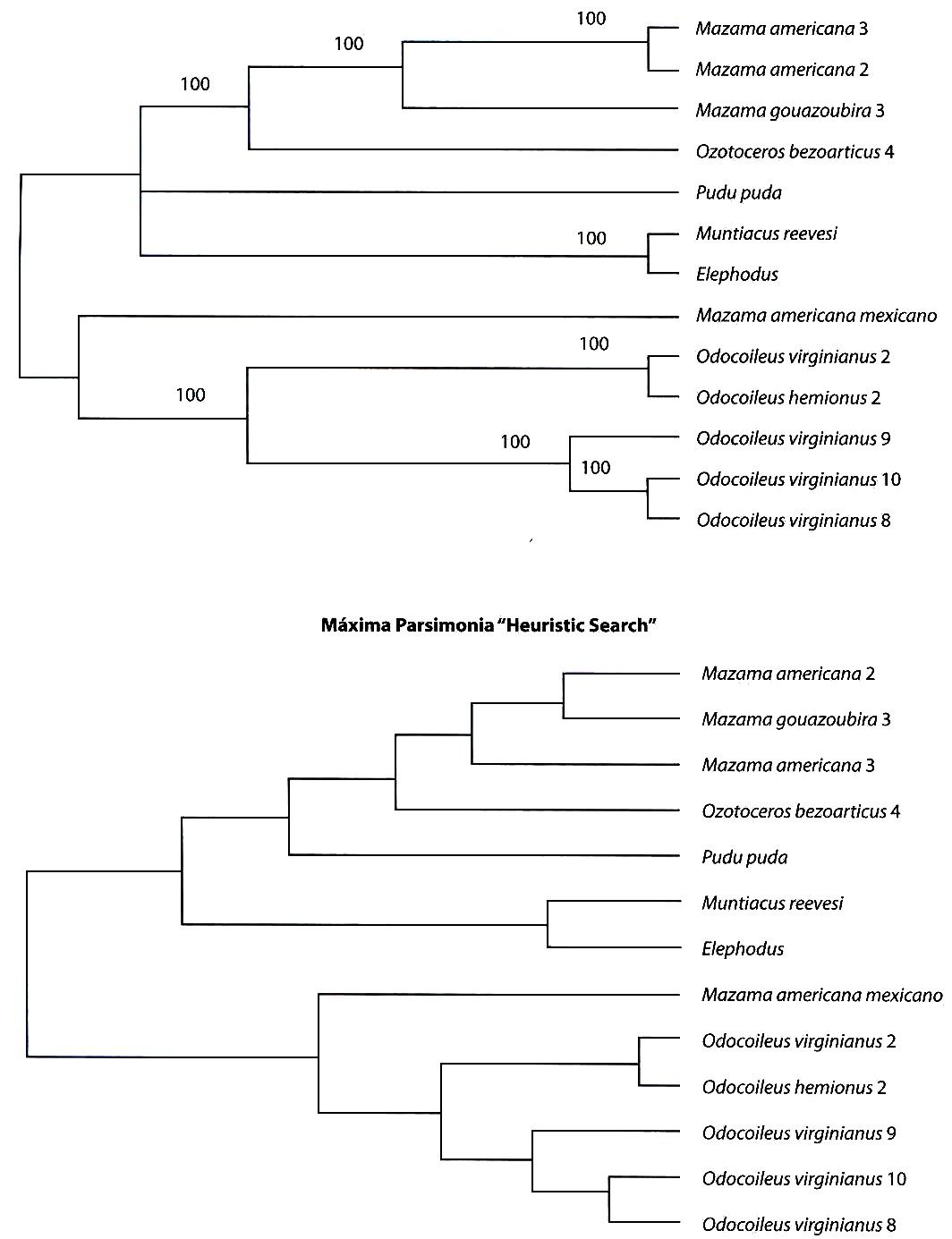
Los análisis filogenéticos llevados a cabo con los métodos de matrices de distancias genéticas (Kimura 1980, Tajima y Nei 1984), y los obtenidos con los métodos de máxima parsimonia pueden observarse en la Fig. 1. Todos los árboles generados con los métodos de distancias muestran que el primer clado en divergir corresponde al integrado por las secuencias de los Muntiacinae asiáticos (Muntiacus y Elaphodus) respecto a las restantes secuencias, todas ellas de Odocoileinos americanos. Esta separación es robusta ya que todos los porcentajes de "bootstraps" son del 100 %. En el seno de los Odocoileinos encontramos dos clados principales. En uno de ellos se observaron todas las secuencias de los O. virginianus colombianos, la del O. hemionus norteamericano y la del M. americana de origen mexicano ("bootstraps" del 75-92 %). Cabe resaltar que la secuencia del O. virginianus 2, procedente de la cordillera andina central de Colombia, estuvo significativamente ("bootstrap" del 96 %) más relacionada con la secuencia del O. hemionus de origen norteamericano que con las restantes secuencias de O. virginianus, también procedentes de Colombia. El otro gran clado ("bootstraps" del 97-100 %) presentó otros dos sub-clados. Uno constituido por un cérvido, de tamaño mediano de 28 a 35 kg de peso, O. bezoarticus leucogaster y el pequeño P. puda, de no más de 8 kg de peso, con un porcentaje de "bootstrap" que osciló entre el 86 y el 90 %. El otro sub-clado agrupó a las tres secuencias procedentes de los ejemplares de Mazama de origen boliviano. La secuencia, del morfológicamente reconocible como M. americana 2, presentó una más intensa relación con el M. gouazoubira 3 que con la secuencia del otro M. americana 3 ("bootstrap" 60-76 %). Esto puede concordar con la explicación anteriormente referida de que en realidad esos tres especimenes bolivianos pertenecen a un único taxón (M. gouzaoubira). Los análisis de máxima parsimonia, sin embargo, mostraron algunas diferencias importantes respecto a lo descrito con los métodos de distancias genéticas. Es de resaltar que los análisis filogenéticos basados en distancias y en máxima parsimonia asumen diferentes hechos con respecto a los procesos evolutivos que han podido acontecer en y entre las especies analizadas de cérvidos. Se obtuvieron dos árboles de máxima parsimonia con un número mínimo de 714 pasos. Se presentan los árboles consenso con la regla de la mayoría del 50 %. Utilizando el procedimiento "heuristic search" se observaron dos grandes agrupaciones. La primera gran diferencia, respecto al análisis anterior, viene dada por la pertenencia de los géneros Muntiacinae a uno de los dos clados integrados por Odocoileinos. En esa agrupación se encontraron las secuencias de los ejemplares de Mazama procedentes de Bolivia, O. bezoarticus y P. puda. En este caso, Ozotoceros presentó mayor relación con Mazama que con Pudu. Al igual que con la técnica anterior, no existió una diferenciación clara entre ambas formas de Mazama. En el segundo clado se obtuvieron relaciones idénticas a las encontradas con los métodos de distancias (O. virginianus, O. hemionus y M. americana de origen mexicano). Utilizando el procedimiento "branch and bound search" se obtuvo un panorama muy similar. La diferencia principal fue que se obtuvo una tritomía no resuelta en el primer clado constituida por Mazama-Ozotoceros, Pudu y Muntiacus-Elaphodus. La otra diferencia fue, que, en este caso, sí se diferenciaron las secuencias de M. americana y M. gouazoubira ambas procedentes de Bolivia. Todos los porcentajes de "bootstraps" obtenidos en este análisis fueron del 100 %, lo cual significa que las agrupaciones obtenidas son altamente significativas.
mitocondrial en varias especies de Odocoileinae, utilizando métodos de matrices de distancias
(A y B) y máxima parsimonia (C y D). A/Árbol "neighbor-joining" con la distancia de Tajima y
Nei (1984). B/Árbol UPGMA con la distancia de Tajima y Nei (1984). Los valores sobre los nódulos
indican los porcentajes de "bootstrap".
Fig. 1. Phylogenetic analysis of the nucleotide sequences of the mtDNA control region in several
Odocoileinae species, using distance matrix procedures (A and B) and maximum parsimony
(C and D). A/ Neighbor-joining tree with the Tajima y Nei (1984)`s distance. B/ UPGMA tree with
the Tajima y Nei (1984)`s distance. The values on the nodules show the bootstrap percentages.
Análisis filogenético de los marcadores microsatélites: Con los resultados para los loci microsatélites, y tomando los tamaños alélicos reportados para 5 especies de cérvidos europeos y asiáticos y para ocho especies de cérvidos neotropicales (Ruiz-García et al. 2006e), se obtuvieron árboles de máxima parsimonia utilizando la presencia-ausencia de alelos de los 16 loci microsatélites empleados. Se obtuvieron 15 árboles de máxima parsimonia, todos con 191 pasos de longitud sin importar cual fuera el método de análisis utilizado. Los índices fueron idénticos para todos los árboles obtenidos, con los siguientes valores: CI=0.702, HI=0.298, RI=0.430, RC=0.302. A partir de estos árboles se procedió a realizar el análisis consenso por diferentes procedimientos, obteniéndose dos modelos de árboles diferentes, uno con los métodos estricto y de Adams (Fig. 2A) y otro para los métodos semiestricto y regla de la mayoría (Fig. 2B). Como se puede observar, al igual, que se detectó en el análisis de la región de control mitocondrial, M. americana presentó una más alta relación genética con O. virginianus que con M. gouzaoubira. Identica relación fue detectada por Smith et al. (1986) mediante isoenzimas y características morfométricas.
en varias especies de Odocoileinae, utilizando métodos de matrices de distancias (A y B) y máxima parsimonia
(C y D). C/ Con el procedimiento de máxima parsimonia "branch and bound". Núnero mínimo de pasos
714. CI=0.867, RI= 0.668. D/ Con el procedimiento de máxima parsimonia "heuristic search".
Fig. 1. Phylogenetic analysis of the nucleotide sequences of the mtDNA control region in several Odocoileinae
species, using distance matrix procedures (A and B) and maximum parsimony (C and D). C/ Tree with the
maximum parsimony "branch and bound" procedure. Minimum step number 714. Ci= 0.867, RI= 0.668.
D/ Tree with the maximum parsimony "heuristic search" procedure.
Discusión
En los dos taxones en los que se midió la variabilidad genética a nivel de la región de control mitocondrial (O. virginianus y ejemplares de Mazama obtenidos en Bolivia) se observó la existencia de mayor diversidad nucleotídica en la primera especie (π=0.0454) que en la segunda (π=0.0219), aun cuando, en este segundo género, aparentemente, se distinguieron dos morfotipos atribuibles, a M. americana y M. gouazoubira. Las muestras de ambas especies se obtuvieron en la Chiquitania de Santa Cruz (Bolivia) en un lugar simpátrico. La escasa diferenciación molecular entre ellas podría sugerir dos posibles explicaciones, al menos. La primera de ellas es que existió hibridación entre ambas especies de Mazama. De hecho, las muestras obtenidas pertenecen a una población, dónde ambos morfotipos se encontraban en una situación geográfica muy cercana. Los ejemplares de M. americana, reconocibles morfológicamente como tales, podrían portar ADN mitocondrial de alguna hembra ancestral de M. gouazoubira que se reprodujo con algún macho de M. americana en el seno de ese linaje, o viceversa. También pudo existir introgresión genética en M. gouazoubira proveniente de alguna hembra de M. americana. Quizá, sin embargo, el primer caso de introgresión pudiera ser más factible que el segundo, debido al tamaño de los animales. Los machos de M. americana de mayor porte pueden alcanzar los 40 kg, excepcionalmente hasta 55 kg en el Surinam (Smith et al. 1986), mientras que los de M. gouazoubira alcanzan hasta los 25 kg de peso, por lo que resultaría más complejo que pudiera cruzarse con una hembra de M. americana. Una segunda posibilidad es que, en realidad, los animales muestreados pertenezcan a una única especie con una variabilidad morfológica muy amplia y que se sobrelape con las características de esta naturaleza que han utilizado los zoólogos tradicionalmente para distinguir ambas especies de Mazama. Si fuera así, probablemente, las muestras bolivianas analizadas pertenecerían a la forma M. gouazoubira. Esta segunda hipótesis puede ser muy importante como se mostrará más adelante. Este hecho debe tenerse en consideración para el manejo de las poblaciones de Mazama en Bolivia.
La cantidad de diversidad nucleotídica estimada en esos dos géneros de cérvidos puede compararse a la encontrada en otras especies de Mamíferos. Recordemos, que un valor mínimo de π=0.02-0.03 entre especies diferentes de un mismo género es usual. Para otros cérvidos se han encontrado los siguientes valores. Entre el alce (Alces alces) y el reno (Rangifer tarandus), π=0.111; entre tres especies de Cervinae (Cervus elaphus elaphus, C. e. canadiensis y C. nippon), los valores de π oscilaron entre 0.0502 y 0.056. Entre wapitis de Norteamérica y Siberia (C. e. manitobensis vs. C. e. sibirecus), π=0.0123 (Polziehn y Strobeck 1998). El valor de π fue de 0.084 entre O. virginianus y Cervus elaphus (Cronin 1986), por citar algunos ejemplos en cérvidos. En otros mamíferos algunos de los valores encontrados han sido los siguientes. Entre los bóvidos, π fue de 0.065 entre la vaca y el bisonte (Bison bison), o de π=0.0056 para Rupicapra rupicapra y R. pyrenaica); entre poblaciones de la nutria Enhydra lutris (π=0.00004-0.0041); para poblaciones de liebre europea (Lepus europaeus), π=0.01 (Hartl et al. 1993); para Ovis canadiensis, π=0.0043 (Luikart y Allendorf 1996). De este modo, la diversidad nucleotídica encontrada para los ejemplares de Mazama procedentes de la región de Chiquitania en Bolivia está dentro de los límites intraespecíficos.
Sin embargo, cuando se introdujo en el análisis la secuencia perteneciente a la M. americana de origen mexicano, la diversidad nucleotídica y otros estadísticos genéticopoblacionales aumentaron considerablemente (π=0.0618), superando, incluso, a los obtenidos cuando las secuencias de O. virginianus colombiano fueron analizadas conjuntamente con O. hemionus norteamericano (datos no mostrados en este estudio). Esta es una primera indicación de que el género Mazama podría no ser monofilético, si no más bien polifilético. El análisis de los árboles filogenéticos, tanto utilizando las distancias de los dos parámetros de Kimura (1980) o de Tajima y Nei (1984), como los métodos de máxima parsimonia, pusieron en evidencia este hecho. El Mazama de origen mexicano siempre quedó afiliado de forma robusta a las secuencias de Odocoileus, mientras que las tres secuencias de Mazama procedentes de Bolivia siempre estuvieron en otra agrupación bien diferenciada y separada del anterior, mostrando mucha mayor relación con las secuencias de Ozotoceros y Pudu, y sin mostrar especial filiación con las secuencias de Odocoileus. El análisis de máxima parsimonia con los loci microsatélites también reveló una conspicua asociación entre M. americana y O. virginianus, estando M. gouzaoubira más distantemente relacionada. En el estudio de Smith et al. (1986), con isoenzimas y datos biométricos, también se encontró mayor relación entre M. americana y O. virginianus en Surinam, que de esa M. americana con M. gouazoubira, también de la misma zona del norte de Sudamérica. Incluso, esa fuerte relación entre M. americana y O. virginianus en ese país neotropical fue mayor que entre la segunda especie y representantes de O. virginianus procedentes del Sudoeste de los Estados Unidos. Aunque para algunas características, tales como los arcos supraorbitales, hueso frontal, espacio de la parte posterior de las suturas frontales y la bulla auditiva, éstas son mayores en Odocoileus que en Mazama, además de las diferencias ostensibles en la cornamenta de los machos, y el hecho de que un análisis discriminante lineal basado en 31 variables reclasificó con una probabilidad del 100 % los 39 cráneos analizados en tres grupos "a priori" designados (O. virginianus del Sudeste USA, O. virginianus del Surinam y M. americana del Surinam), otras características diferenciales esperadas entre ambas especies no se dieron. Por ejemplo, con las mismas 31 variables craneométricas, un cráneo de M. americana del Surinam reclasificó con un 100 % de probabilidades con los O. virginianus del Surinam (con datos directos no transformados), y con un 99.75 % con los O. virginianus de Sudeste USA (datos ajustados por tamaño), y no con la agrupación compuesta por M. gouzaoubira. Los ejemplares de O. virginianus del Surinam poseen una anchura maxilar más estrecha que los especimenes de la misma especie procedente del Sudeste de USA, mientras que ocurre, exactamente, lo mismo, entre las dos especies de Mazama de ese mismo país. M. americana posee una menor anchura maxilar que M. gouazoubira. Esos mismos autores (Smith et al. 1986), usando 19 loci bioquímicos y la distancia genética de Nei (1972), encontraron que ésta era igual a 0.236 entre las dos especies de Mazama, mientras que esa distancia genética era notablemente inferior entre O. virginianus del Sudeste USA y M. americana (D=0.148), y entre O. virginianus del Surinam y M. americana (D=0.063). Las distancias entre las dos formas de Odocoileus respecto a M. gouazoubira del Surinam fueron considerablemente mayores (D=0.378 y D=0.271, respectivamente). Los resultados de esos autores y, especialmente, los datos reportados en el presente análisis con la región de control mitocondrial y los loci nucleares microsatélites, induce a pensar que se debe realizar una profunda revisión taxonómica del género Mazama, ya que podría ser un género polifilético. Esto es, el ejemplar de Mazama de origen mexicano podría ser un verdadero representante de M. americana, mientras que los animales bolivianos pudieran ser únicamente representativos de M. gouazoubira. Eso indicaría que esos dos taxones, no únicamente son especies diferentes, si no que el género Mazama podría ser desdoblado en dos géneros diferentes, al menos. Se conocen restos asignables a M. gouazoubira procedentes del Pleistoceno de Lagoa Santa y del valle del Rio das velhas (Brasil) (Lund 1950). Luego, este es un taxón antiguo. Además, aunque en algunos lugares se puede dar simpatría entre ambas formas, por ejemplo, en Tucumán (Argentina) o en la zona boliviana objeto de estudio, M. gouazoubira vive hasta los 600-900 m de altitud, a partir de la cual, es reemplazada por M. americana (Richard et al. 1995). La primera forma prefiere zonas abiertas y alteradas, mientras que la segunda prefiere zonas primarias y selvas más cerradas. En las zonas secas como El Chaco, la forma gouazoubira prefiere zonas xerófitas, y la forma americana zonas húmedas. Todo ello conjuntamente nos induce a pensar en que ambos taxones forman, en realidad, dos géneros diferentes. El primer género estaría más íntimamente relacionado con Odocoileus, y el segundo estaría más relacionado con Ozotoceros y Pudu y, quizá, presumiblemente, con Blastocerus.
Un segundo aspecto filogenético interesante hace referencia a la mayor relación existente entre el haplotipo mitocondrial de O. virginianus 2 (Colombia) con el haplotipo mitocondrial de O. hemionus norteamericano que con los restantes haplotipos de O. virginianus (también colombianos). Este es un género que presenta una extensa variabilidad genética en su seno revelada por diversos trabajos llevados a cabo con poblaciones y subespecies norteamericanas. Por ejemplo, Cronin et al. (1988) descubrieron que dos O. virginianus procedentes de Illinois (π=0.0051) presentaban mayor divergencia genética mitocondrial entre ellos que con respecto a algunos ejemplares de Illinois y Georgia (π=0.0026-0.0055). En las muestras de los especímenes colombianos, la divergencia genética es todavía más considerable a nivel mitocondrial que en el caso citado anteriormente con ejemplares norteamericanos de O. virginianus, aunque no así a nivel de los microsatélites nucleares para los que no fueron heterogéneos significativamente. Las diferencias moleculares detectadas, para la región de control del ADN mitocondrial, entre ejemplares norteamericanos de O. virginianus y O. hemionus columbianus fueron 69 transiciones, 21 transversiones y seis deleciones/inserciones con π=0.0758 y un tiempo de divergencia estimado entre ambos taxones de 351 000 años (Polziehn y Strobeck 1998). valores de divergencia genética similares, entre esas dos formas biogeográficamente separadas, han sido encontradas por otros autores (Carr et al. 1986, 1993, Cronin et al. 1988). También, utilizando 18 isoenzimas, vaughan et al. (1994) encontraron distancias genéticas elevadas entre muestras de O. virginianus procedentes de Georgia (USA), la isla San Lucas (Costa Rica) y Surinam, especialmente entre las muestras de Estados Unidos y Costa Rica. Sin embargo, entre otras subespecies de O. virginianus en Norteamérica, y entre éstas y algunas poblaciones de O. hemionus, las diferencias genéticas son muy pequeñas. Por ejemplo, Hillestad (1984) encontró una distancia de Nei (D=0.023) muy pequeña entre cuatro subespecies de O. virginianus norteamericanos. Incluso, Baccus et al. (1983) detectaron una distancia genética no excesivamente grande entre O. virginianus y O. hemionus en Norteamérica (D=0.149). Este valor encontrado corresponde a una distancia genética típicamente de especies congenéricas o subespecies. Cronin et al. (1988), haciendo uso de un análisis con endonucleasas de restricción aplicadas al ADN mitocondrial, encontraron un valor de diversidad nucleotídica sumamente pequeña (π=0.0057) entre ambas especies, valor parecido al encontrado por Carr et al. (1986) entre ambas formas en Texas (p=0.008). Cruzamientos en cautiverio entre esas dos especies han sido reportados por diversos autores (Whitehead 1972, Wallmo 1981, Derr et al. 1991). Pero, también, se ha encontrado hibridación interespecífica entre ambas formas de Odocoileus en poblaciones naturales de la Columbia Británica (Cowan 1962), Alberta (Wishart 1980, Hughes 1990), Sur Oeste USA (Derr 1991), Pacífico Noroeste (Cronin 1991) y Texas (Carr et al. 1986, Ballinger et al. 1992). Carr y Hughes (1993) amplificaron 359 pb del gen citocromo b mitocondrial de 157 ejemplares de Odocoileus. Estos autores mostraron que en el oeste de Texas, dónde viven simpátricamente las formas O. virginianus texanus y O. hemionus crooki, todos los individuos analizados poseyeron un único haplotipo mitocondrial común y que perteneció al clado de los haplotipos de O. hemionus. Esto indica que existió introgresión genética de ADN mitocondrial desde O. hemionus a O. virginianus. Las poblaciones del Sur Oeste USA y, especialmente, las de Texas son las que muestran un mayor grado de hibridación entre ambas especies de Odocoileus. Para el locus Albúmina, ambas especies están fijadas para diferentes alelos (Cronin et al. 1988). Sin embargo, en la localidad texana de Pecos Co, el 13 % de O. hemionus y el 24 % de los O. virginianus mostraron genotipos heterocigotos con ambos alelos (Ballinger et al. 1992) y todos los O. hemionus, y la mayoría de los O. virginianus, compartieron un mismo mapa de restricción del ADN mitocondrial (Carr et al. 1986). Una posibilidad es que algunos de esos haplotipos mitocondriales (linajes maternos) estuvieran ya presentes en las formas ancestrales que darían, posteriormente, a ambas especies de Odocoileus y que se hubieran repartido asimétricamente. Una segunda posibilidad es que se haya dado extensiva hibridación entre ambas especies y en el proceso de colonización de O. virginianus hacia Centro y Suramérica, esta especie haya portado haplotipos mitocondriales característicos de los O. hemionus norteamericanos. Una tercera posibilidad es que O. virginianus se haya derivado recientemente de O. hemionus, aunque los datos paleontológicos no parecen estar en concordancia con esta última hipótesis (Kurtén y Anderson 1980). Cualquiera de las diversas hipótesis invocadas podría explicar la existencia de una fuerte relación entre un haplotipo mitocondrial en un O. virginianus colombiano y O. hemionus.
Con los métodos de máxima parsimonia, se observó que los Muntiacinae (Muntiacus y Elaphodus) formaron un clado sólido con cierto grupo de Odocoileinae, concretamente con los Mazama bolivianos, Ozotoceros y Pudu. Polziehn y Strobeck (1998) observaron que otro género de Cervidae, Hydropotes (subfamilia Hydropotinae), en algunos análisis, se agrupaba con Mazama. Ellos justificaron este hecho porque la estructura telemetacarpal, la gran apertura del canal temporal, tamaño similar, hembras con cuatro mamas, caninos como colmillos y gran habilidad para nadar son comunes para el Odocoileinae, Mazama y el Hydropotinae, Hydropotes. Algo similar podría ocurrir entre los Muntiacinae y algunos Odocoileinae. De hecho, Comincini et al. (1996), utilizando RAPD con ocho cebadores diferentes, detectaron una considerable relación entre O. hemionus hemionus y M. muntjac vaginalis, aunque la relación de este último con C. capreolus, un Odocoileinae europeo, fue ostensiblemente menor.
A partir de los árboles de máxima parsimonia obtenidos con los alelos de los loci microsatélites se observó también la citada y considerable relación entre O. virginianus y M. americana, más otras relaciones filogenéticas interesantes. Por ejemplo, se presenta una relación estrecha entre M. gouazoubira y C. capreolus. Estos dos clados siempre forman un grupo hermano y, a continuación, encontramos la divergencia de B. dichotomus seguido por la divergencia de H. antisensis. Las relaciones entre las demás especies no fueron tan claras, especialmente debido al bajo número de muestras analizadas para cada una de ellas. Aunque los marcadores microsatélites no se utilizan tradicionalmente para la configuración de filogenias intergenéricas, observamos en este caso una fuerte congruencia con lo encontrado a partir de la secuenciación de la región de control mitocondrial. Un exhaustivo análisis molecular, empleando genes nucleares y mitocondriales, es requerido para clarificar definitivamente la sistemática y la filogenia de los ciervos neotropicales.
Agradecimientos
Los autores agradecen la ayuda prestada por K.H. Roed, de Noruega, por el envío de cebadores de microsatélites diseñados para Rangifer tarandus, los cuales fueron aplicados en los ciervos neotropicales. A la Pontificia Universidad Javeriana agradecemos los apoyos prestados por la Decanatura de la Facultad de Ciencias y de la vicerectoría Académica en la financiación parcial de un viaje a Bolivia, en 1997, que permitió obtener buena parte de las muestras aquí analizadas.
Resumen
El presente trabajo muestra dos análisis moleculares sobre la filogenia de los cérvidos neotropicales. En uno se secuenció la región control del mtDNA (D-loop) de seis especies de Odocoileinae, utilizándose las secuencias de dos Muntiacinae como elementos externos. Se evidenciaron los siguientes resultados: La secuencia de una Mazama americana, de origen mexicano, presentó una fuerte relación filogenética con las diversas secuencias estudiadas de Odocoileus, contrario a lo esperado ya que, a priori, debería haberse asociado con las secuencias analizadas de otros ejemplares de Mazama de origen boliviano. Esto pone en evidencia que este género no es monofilético. A su vez, las secuencias de los ejemplares bolivianos de Mazama formaron una agrupación con secuencias de Pudu puda y O. bezoarticus. Una secuencia de O. virginianus, del área central de Colombia, presentó más relación con la secuencia de un O. hemionus norteamericano que con las restantes secuencias analizadas de O. virginianus, también de origen colombiano. Esto puede reflejar varias explicaciones hipotéticas, tales como la existencia de haplotipos ancestrales comunes entre ambas especies de Odocoileus, hasta la hibridación en Norteamérica entre ambos taxones antes de su penetración en Sudamérica. Los análisis de máxima parsimonia presentan una especial relación entre los Muntiacinae y el clado de los ciervos sudamericanos. El segundo análisis filogenético hizo uso de 16 marcadores nucleares microsatélites. Aunque, en principio, estos marcadores no son los más recomendables para estudios filogenéticos intergenéricos, los resultados muestran, al igual que el ADN mitocondrial, una mayor relación entre M. americana y O. virginianus que entre la primera especie y M. gouzaoubira.
Palabras clave: filogenia molecular, ciervos neotropicales, ADN mitocondrial, microsatélites, Mazama, Odocoileus.
Referencias
Allen, J.A. 1915. Notes on American deer of the genus Mazama. B. Am. Mus. Nat. Hist. 34: 521-553. [ Links ]
Baccus, R., N. Ryman, M.H. Smith, C. Reuterwall & D. Cameron. 1983. Genetic variability and differentiation of large grazing mammals. J. Mammal. 64: 109-120. [ Links ]
Ballinger, S.W., L.H. Blankenship, J.W. Bickham, & S.M. Carr. 1992. Allozyme and mitochondrial DNA analysis of a hybrid zone between white-tailed deer and mule deer (Odocoileus) in West Texas. Biochem. Genet. 30: 1-11. [ Links ]
Blanquer-Maumont A., & B. Crouau-Roy. 1995. Polymorphism, monomorphism, and sequences in conserved Microsatellites in Primate species. J. Mol. Evol. 41: 492-497. [ Links ]
Cabrera, A. 1960. Catálogo de los mamíferos de América del Sur. Rev. Mus. Argent. Cien. Nat. "Bernardino Rivadavia" 4: 309-732. [ Links ]
Carr, S.M., S.W. Ballinger, J.N. Derr, L.H. Blankenship & J.W. Bickham. 1986. Mitochondrial DNA analysis of hybridization between sympatric white-tailed deer and mule deer in West Texas. Proc. Nat. Acad. Scien. USA 83: 9576-9580. [ Links ]
Carr, S.M. & G.A. Hughes. 1993. Direction of introgressive hybridization between species of North American deer (Odocoileus) as inferred from mitochondrial-Cytochrome-b sequences. J. Mammal. 74: 331-342. [ Links ]
Comincini, S., M. Sironi, C. Bandi, C. Giunta, M. Rubini & F. Fontana. 1996. RAPD analysis of systematic relationships among the Cervidae. Heredity 76: 215-221. [ Links ]
Cowan, I.M. 1962. Hybridization between the black-tail deer and the white-tail deer. J. Mammal. 43: 539-541. [ Links ]
Cronin, M. 1986. Genetic relationships between white-tailed deer, mule deer and other large mammals inferred from mitochondrial DNA analysis. Tesis de MSc, Montana State University, Bozeman, Montana, EEUU. 49 p. [ Links ]
Cronin, M. 1991. Mitochondrial DNA phylogeny of deer (Cervidae). J. Mammal. 72: 553-556. [ Links ]
Cronin, M., E. vyse & D. Cameron. 1988. Genetic relationships between mule deer and white-tailed deer in Montana. J. Wildlife Manage. 52: 228-237. [ Links ]
Czernay, S. 1987. Die spiesshirsche und pudus. Die Neue Brehm Bucherei 581: 1-84. [ Links ]
Derr, J.N. 1991. Genetic interactions between white-tailed and mule deer in the southwestern United States. J. Wildlife Manage. 55: 228-237. [ Links ]
Derr, J.N., D.W. Hale, D.L. Ellesworth & J.W. Bickham. 1991. Fertility in an F1 male hybrid of white-tailed deer (Odocoileus virginianus) x mule deer (O. hemionus). J. Reprod. Fertil. 93: 111-117. [ Links ]
DeWoody J.A., R.L. Honeycutt & L.C. Skow. 1995. Microsatellite markers in White-Tailed Deer. J. Hered. 86: 317-319. [ Links ]
DiRienzo A., A.C. Peterson, J.C. Garza, A. valdes, M. Slatkin & N.B. Freimer. 1994. Mutational proceses of simple-sequence repeat loci in human populations. Proc. Nat. Acad. Scien. USA 91: 3166-3170. [ Links ]
Douzery, E., J.D. Lebreton & F.M. Catzeflis. 1995. Testing the generation time hypothesis using DNA/DNA hybridization between artiodactyls. J. Evolution. Biol. 8: 511-529. [ Links ]
Douzery, E. & E. Randi. 1997. The mitochondrial control region of Cervidae: Evolutionary Patterns and Phylogenetic content. Mol. Biol. Evol. 14: 1154-1166. [ Links ]
Duarte, J.M.B. 1992. Aspectos taxonomicos e citogenéticos de algumas espécies de cervídeos brasileiros. Tesis de Maestría, Facultade de Ciencias Agrárias e veterinárias, Jaboticabal, Brasil. 153 p. [ Links ]
Duarte, J.M.B. & M.L. Merino. 1997. Taxonomia e evolucao. In J.M.B. Duarte (ed). Biologia e conservacao de Cérvidos Sul-americanos: Blastocerus, Ozotoceros e Mazama. FUNEP, Jaboticabal, Brasil. 238 p. [ Links ]
FitzSimmons N.N., C. Moritz & S.S. Moore. 1995. Conservation and dynamics of Microsatellite loci over 300 million years of marine turtle evolution. Mol. Biol. Evol. 12: 432-440. [ Links ]
Garza, J.C. & N.B. Freimer. 1996. Homoplasy for size at Microsatellite loci in humans and Chimpanzees. Genome Res. 6: 211-217. [ Links ]
Goldstein D.B. & D.D. Pollock. 1997. Launching Microsatellites: a review of mutation processes and methods of phylogenetic inference. J. Hered. 88: 335-342. [ Links ]
Goldstein D.B., A. Ruiz-Linares, L.L. Cavalli-Sforza & M.W. Feldman. 1995. Genetic absolute dating based on Microsatellites and the origin of modern humans. Proc. Nat. Acad. Scien. USA 92: 6723-6727. [ Links ]
Hartl, G.B., F. Suchentrunk, K. Nadlinger & R. Willing. 1993. An integrative analysis of genetic differentiation in the brown hare Lepus europaeus based on morphology, allozymes, and mitochondrial DNA. Acta Theriologica 38: 33-57. [ Links ]
Hill, W.G. & A. Robertson. 1968. Linkage disequilibrium in finite populations. Theor. Appl.Genet. 38: 226-231. [ Links ]
Hillestad, H.O. 1984. Stocking and genetic variability of white-tailed deer in the southeastern United States. Disertación de PhD, University of Georgia, Athens, EEUU. 121 p. [ Links ]
Hughes, G.A. 1990. A molecular genetic analysis of hibridization between two species of deer (Odocoileus) in western Canada. Tesis MSc, Memorial University of Newfoundland, St. John, Newfoundland, Canada 93 p. [ Links ]
Jukes, T.H. & C.R. Cantor. 1969. Evolution of protein molecules, p. 21-132. In H.N. Munro (ed). Mammalian Protein Metabolism. Academic, Nueva York, Nueva York, EEUU. [ Links ]
Kimura, M. 1980. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences. J. Mol. Evol. 16: 111-120. [ Links ]
Kimura, M. 1981. Estimation of evolutionary distances between homologous nucleotide sequences. Proc. Nat. Acad. Sci. USA 78: 454-458. [ Links ]
Kumar, S., K. Tamura & M. Nei. 1993. MEGA: Molecular Evolutionary Genetics Analysis software for microcomputers. Comput. Appl. Biosci. 10: 189-191. [ Links ]
Kurten, B. & E. Andersson. 1980. Pleistocene mammals of North America. Columbia University, Nueva York, Nueva York, EEUU. 442 p. [ Links ]
Lewontin, R.C. 1964. The interaction of selection and linkage. I. General considerations: heterotic models. Genetics 49: 49-67. [ Links ]
Lewontin, R.C. & K. Kojima. 1964. The evolutionary dynamics of complex polymorphisms. Evolution 14: 458-472. [ Links ]
Li, W.H. 1997. Molecular Evolution. Sinauer Associates, Sunderland, Massachusetts, EEUU. 515 p. [ Links ]
Luikart, G. & F. Allendorf. 1996. Mitochondrial -DNA variation and genetic-population structure in Rocky Mountain bighorn sheep (Ovis canadiensis canadiensis). J. Mammal. 77: 109-123. [ Links ]
Lund, P.W. 1950. Memórias sobre a palentologia Brasileira. Instituto Nacional do Livro. Rio de Janeiro, Brasil. 592 p. [ Links ]
Morin P.A., J. Wallis, J. Moore, R. Chakraborty & D. Wooddruff. 1993. Non-invasive sampling and DNA amplification for paternity exclusion, community structure, and phylogeography in wild chimpanzees. Primates 34: 347-356. [ Links ]
Nei, M. 1972. Genetic distance between populations. The American Naturalist 106: 283-292. [ Links ]
Nielsen R. 1997. A likelihood approach to populations samples of Microsatellite alleles. Genetics 146: 711-716. [ Links ]
Nielsen R & P.J. Palsbol. 1999. Single-locus tests of Microsatellite evolution: Multi-step mutations and contrains on allele size. Mol. Phylogenet. Evol. 11: 477-484. [ Links ]
Polziehn, R.O. & C. Strobeck. 1998. Phylogeny of Wapiti, red deer, sika deer, and other North American Cervids as determined from mitochondrial DNA. Mol. Phylogenet. Evol. 10: 249-258. [ Links ]
Richard, E., J.P. Julia, J. Samaniego & P. Aceñolaza. 1995. La Corzuela Parda. Serie Monográfica y Didáctica N° 22. Facultad de Ciencias Naturales e Instituto Miguel Lillo, Universidad Nacional de Tucumán, Tucumán, Argentina. [ Links ]
Roed K.H. & L. Midthjell. 1998. Microsatellites in reindeer, Rangifer tarandus, and their use in other cervids. Mol. Ecol. 7: 1773-1776. [ Links ]
Rozas, J. & R. Rozas. 1995. DnaSP, DNA sequence polymorphisms: an interactive program for estimating population genetics parameters from DNA sequence data. Comput. Appl. Biosci. 11: 621-625. [ Links ]
Ruiz-Garcia, M. 2003. Molecular population genetic analysis of the spectacled bear (Tremarctos ornatus) in the Northern Andean Area. Hereditas 138 :81-93. [ Links ]
Ruiz-Garcia, M. 2005. The use of several microsatellite loci applied to 13 Neotropical Primates revealed a strong recent bottleneck event in the woolly monkey (Lagothrix lagotricha) in Colombia. Primate Report 71: 27-55. [ Links ]
Ruiz-Garcia M., A. Parra, N. Romero-Alean, P. Escobar-Armel & J. Shostell. 2006c. Genetic Characterization and phylogenetic relationships between Ateles species (Atelidae, Primates) by means of DNA microsatellite markers and craniometric data. Primate Report 73: 3-47. [ Links ]
Ruiz-Garcia M., C.E. Payan, A. Murillo & D. Alvarez. 2006a. DNA Microsatellite characterization of the Jaguar (Panthera onca) in Colombia. Genes and Genetics Systems 81: 115-127. [ Links ]
Ruiz-Garcia M., M. Gaviria, M. Martinez-Aguero, P. Escobar-Armel & E. Banguera. 2006b Genetic structure of Inia geoffrensis and Inia boliviensis by means of heterologous DNA microsatellites. (sometido). [ Links ]
Ruiz-Garcia, M., M. Martinez-Aguero, D. Alvarez, S. Goodman. 2006e Análisis de la variabilidad genética en diversos géneros de Cervidae neotropicales mediante el uso de loci microsatélites (sometido). [ Links ]
Ruiz-Garcia M., P. Escobar-Armel, D. Alvarez, M. Mudry, M. Ascunce, G. Gutierrez-Espeleta & J.M. Shostell. 2006d Genetics variability in four Alouatta species measured by means of nine DNA microsatellites markers: Genetic structure and recent bottlenecks. Folia Primatologica 78: 73-87. [ Links ]
Ruiz-Garcia, M., P. Orozco-ter Wengel, A. Castellanos & L. Arias. 2005. Microsatellite analysis of the spectacled bear (Tremarctos ornatus) across its range distribution. Genes Genetics Syst. 80: 57-69. [ Links ]
Saitou, N. & M. Nei. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 4: 406-425. [ Links ]
Sambrook, J., E.F. Fritsch & T. Maniatis. 1989. Molecular cloning: a Laboratory Manual. Cold Spring Harbor Laboratory, Nueva York, Nueva York, EEUU. [ Links ]
Slate, J., D.W. Coltman, S.J. Goodman, I. Macclean, J.M. Pemberton & J.L. Williams. 1998. Bovine microsatellite loci are highly conserved in red deer (Cervus elaphus), sika deer (Cervus nippon) and Soay sheep (Ovis aries). Anim. Genet. 28: 307-315. [ Links ]
Smith, M.H., W.v. Branan, R.L. Marchinton, P.E Johns & M. Wooten. 1986. Genetic and Morphologic comparisons of red brocket, brown brocket, and white-tailed deer. J. Mammal. 67: 106-111. [ Links ]
Sneath, P.H.A. & R.R. Sokal. 1973. Numerical Taxonomy. W.H. Freeman, San Francisco, California, EEUU. 718 p. [ Links ]
Swofford, D.L. 1993. PAUP 3.1.1: Phylogenetic analysis using parsimony. Illinois Natural History Survey, Champaign, Illinois, EEUU. [ Links ]
Tajima, F. 1983. Evolutionary relationship of DNAsequences in finite populations. Genetics 105: 437-460. [ Links ]
Tajima, F. 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphisms. Genetics 123: 585-595. [ Links ]
Tajima, F. & M. Nei. 1984. Estimation of evolutionary distance between nucleotide sequences. Mol. Biol. Evol. 1: 269-285. [ Links ]
Tamura, K. & M. Nei. 1993. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol. Biol. Evol. 10: 512-526. [ Links ]
Thompson, J. D., D. G. Higgins, & T. J. Gibson. 1994. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Res. 22: 4673-4680. [ Links ]
Vaughan, C., K. Fisher, M.A. Rodriguez, P. Johns & M. Smith. 1994. variabilidad genética en la población de venado de cola blanca en la isla San Lucas, Costa Rica, y las implicaciones para su manejo. In C.Vaughan & M.A. Rodríguez (eds.). Ecología y Manejo del venado de cola blanca en México y Costa Rica, Heredia, Costa Rica. [ Links ]
Wallmo, O.C. 1981. Mule and black-tailed deer distribution and habitats. In O.C. Wallmo (ed). Mule and black-tailed deer of North-America. University Nebraska, Lincoln, Nebraska, EEUU. [ Links ]
Whitehead, G.K. 1972. Deer of the world. Constable, Londres, Inglaterra. 194 p. [ Links ]
Wilson,D.E. &D.Reeder. 1993. Mammal species of the world: A taxonomic and geographic reference. Smithsonian Institution, Washington DC, EEUU. 1312 p. [ Links ]
Wishart, W.D. 1980. Hybrids of white-tailed and mule deer in Alberta. J. Mammal. 61: 716-720. [ Links ]
Ximenez, P., A. Langguth & R. Praderi. 1972. Lista sistemática de los mamíferos del Uruguay. An. Mus. Hist. Nat. Montevideo 5: 1-49. [ Links ]