Revista de Biología Tropical
versión On-line ISSN 0034-7744versión impresa ISSN 0034-7744
Rev. biol. trop vol.52 no.3 San José sep. 2004
Comparación de tres métodos de extracción de ADN a partir de restos óseos
Christian Del Valle*, Anayanci Rodríguez & Marta Espinoza
Complejo de Ciencias Forenses del Organismo de Investigación Judicial, Heredia, Costa Rica.
* Este trabajo fue requisito para optar por el título de Bachiller en Ingeniería en Biotecnología, Escuela de Biología, Instituto Tecnológico de Costa Rica; chrdvs@hotmail.com
Recibido 18-V-2003. Corregido 12-VII-2003. Aceptado 16-VIII-2004.
Abstract
Comparison of three methods for DNA extraction from bone remains. Extraction of amplifiable DNAis a frequent problem when working with degraded specimens like bone samples. The possibility of obtaining as much information as possible from these samples has a particular significance in many forensic investigations. The present investigation was aimed to assess the efficiency of three organic extraction methods for purifying amplifiable DNA from bone samples. The amount of nucleic acids obtained, the success rate in the amplification of DNA microsatellite (STR) markers and amelogenin by PCR, the influence of PCR inhibitors and environmental conditions, and where the samples were found before their processing in the laboratory, were all evaluated in this investigation for the three methods. Results showed that method A (a modification of FBI method for DNA extraction) performed better in producing not a higher amount but a better quality amplifiable DNA, in comparison with the other two methods evaluated. It was also demonstrated that the quality of the DNA to be amplified by PCR was influenced by the presence of inhibitors and/or contaminants and the environmental conditions where the bone sample was taken from. The worst conditions were observed from aquatic environments. The results suggest that the implementation of some specific modifications in the method A (use of purification columns, reliable quantification methods and different dilutions) would help to obtain better DNA extracts intended to be used in different molecular identification tests. Rev. Biol. Trop. 52(3): 717-725. Epub 2004 Dic 15.
Key words: DNA extraction, PCR, STRs, bones, inhibitors, Costa Rica.
Palabras clave: Extracción ADN, PCR, hueso, STRs, inhibidores, Costa Rica.
El descubrimiento de las regiones hipervariables del ADN en el genoma humano por Wyman y White en 1980, hizo posible el reconocimiento del potencial del ADN en la producción de perfiles genéticos útiles para identificar individuos o indicios biológicos de cualquier índole. En Costa Rica así como en otros países, se ha incorporado el uso de marcadores genéticos para la creación de estos perfiles, los cuales hoy día son evidencia clave para esclarecer temas médico-legales (Rojas 1995, Morera et al. 1998, Jiménez et al. 1999, Morera et al. 2001, Morales et al. 2004). El uso de marcadores basados en repeticiones cortas en tandem (STR) ha adquirido mucha popularidad en la labor genético-forense porque permite trabajar con muestras más degradadas o con poco ADN (Nakamura 1987, Higuchi et al. 1988, Buttler 2001).
La posibilidad de utilizar restos óseos como fuente para la obtención de ADN ha significado un importante impulso en la aplicación de las técnicas de biología molecular para múltiples fines forenses cuando no se cuenta con otro tipo de muestras (Hochmeister et al. 1991, Hochmeister et al. 1995, Lassen et al. 1996, Cattaneo et al. 1997). El hueso, a diferencia de los tejidos conjuntivos, posee componentes extracelulares calcificados que lo hacen un material duro, capaz de resistir en el tiempo más que el resto de los tejidos, excluyendo a los cabellos (Fawcett 1990). Sin embargo, su descomposición ocurre normalmente con una pérdida de la estructura integral, la exposición de las células internas y su consecuente degradación a nivel molecular. En el período post mortem la presencia y estabilidad del ADN en el hueso está determinada por diferentes factores, principalmente ambientales (Lindahl 1993). Esto resulta de suma importancia puesto que la cantidad y calidad del ADN que pueda ser extraído de un hueso determina el potencial de las pruebas de biología molecular que se pueden aplicar al mismo.
En el presente estudio se procedió a evaluar tres métodos de extracción fenólica de ácidos nucleicos a partir de restos óseos con el fin de determinar cual reúne las mejores condiciones de aplicabilidad y eficiencia para las pruebas de cuantificación de ácidos nucleicos y su amplificación utilizando la reacción en cadena de la polimerasa (PCR). También se evaluó la relevancia de otros factores relacionados (procedencia de la muestra, presencia de inhibidores, método de cuantificación) sobre el rendimiento final de los procedimientos.
Materiales y métodos
Selección de muestras: Se seleccionaron seis huesos de diferentes individuos del banco de muestras congeladas a –20º C del Laboratorio de Bioquímica del Complejo de Ciencias Forenses del Organismo de Investigación Judicial (OIJ) de Costa Rica. Los mismos correspondieron a cinco fémures (cuatro masculinos y uno femenino) y un cráneo femenino. De acuerdo con la procedencia, las muestras fueron clasificadas como contexto "exhumado" (para huesos enterrados), "acuático" (para huesos encontrados en ríos y represas) y "otros" (para los huesos expuestos sobre una superficie).
Pulverización de muestras: Por ser muestras ya analizadas, no se necesitó eliminar de los huesos residuos carnosos o impurezas. Cada hueso se trabajó por separado, en una pequeña cámara de plástico transparente la cual fue lavada con detergente y tratada con etanol antes de ser usada, con el fin de mantener las condiciones de asepsia.
En el procesamiento de los fémures se removió inicialmente la médula con un taladro especial (Dremel) y se cortaron del centro de cada hueso aproximadamente 20 discos (con forma de dona) que luego se colocaron en una placa de Petri estéril. El tamaño promedio de los discos fue de 2.5 cm de diámetro por 0.3 cm de grosor. La cantidad de discos estuvo sujeta a la posibilidad de obtener al menos 20 gramos de muestra para generar resultados significativos y hacer las respectivas evaluaciones de reproducibilidad. El cráneo fue cortado con la misma sierra para obtener varios fragmentos de 3 cm de largo por 1 cm de ancho.
Cada una de las muestras fue cortada por aparte con una cizalla plana a fin de obtener fragmentos lo suficientemente pequeños para pulverizarlos en el Freezer/Mill (este es un mortero electromagnético enfriado por nitrógeno líquido, capaz de reducir los fragmentos de hueso a polvo). Los mismos fueron colocados en tubos plásticos de 50 ml (tipo Falcon) debidamente identificados y se almacenaron a –20º C sin pulverizar.
Los tubos utilizados para pulverizar las muestras fueron previamente irradiados con luz ultravioleta o autoclavados. El Freezer/Mill se ajustó a 15 minutos de pre-enfriamiento con nitrógeno líquido y tres ciclos de pulverizado a 14 golpes por segundo durante 2 minutos. Cada ciclo de pulverización estuvo intercalado por 2 minutos de enfriamiento de la muestra. Cada muestra se pulverizó por separado.
Extracción del ADN: Se utilizaron 9 tubos plásticos de 50 ml por muestra y se repartió 2 g de polvo de hueso en cada tubo debidamente rotulado. La extracción del ADN de cada muestra se realizó por triplicado para cada uno de los métodos evaluados (A: modificación del método del FBI [Federal Bureau of Investigation] de los Estados Unidos); B: método de rutina utilizado en el Laboratorio de ADN del OIJ de Costa Rica y C: modificación del método del Laboratorio de la Universidad de Santiago de Compostela, España, los cuales se resumen en el cuadro 1.
Cuantificación y pureza de los ácidos nucleicos: Una vez obtenidos los extractos, se cuantificaron los ácidos nucleicos por 3 métodos diferentes: el fluorométrico Hoefer Dyna Quant 200, el Quantiblot (método de cuantificación mediante hibridización por sonda específica de Perkin Elmer y el kit DNA Dipstick de Invitrogen. La pureza de los extractos y la cantidad de proteínas presentes fue evaluada por un cuarto método; se utilizó el Gene Quant.
Amplificación por PCR: Para evaluar la calidad de los productos de los diferentes métodos de extracción, los mismos fueron analizados por PCR utilizando los ensayos de STR y amelogenina, de acuerdo con los procedimientos de rutina usados en el Laboratorio de ADN del OIJ (Matamoros 1999, Anónimo 1994-1998). Los paquetes de amplificación de STR utilizados fueron: "Silver STR TM III triplex" (incluye los marcadores D16S539, D7S820 y D13S317) y STR Multiplex (incluye los marcadores D16S539, D7S820, D13S317 y D 5S818) de Promega. Se montaron 4 diluciones (1:1, 1:2, 1:10 y 1:100) por extracto para un total de 294 amplificaciones. También se montó un control positivo (K562) de Promega y un control negativo (mezcla de reactivos sin muestra de ADN) por cada gradilla de PCR. La amelogenina se analizó con la mejor dilución que se determinó luego del análisis de STR.
Electroforesis en poliacrilamida con tinción de plata: Los productos de amplificación se separaron en geles de poliacrilamida al 4% según el método previamente descrito (Maniatis 1982). Las corridas de electroforesis se hicieron a aproximadamente 100 watts, 1850 voltios y 54 mili amperios, por 1 hora y 30 minutos. Para las lecturas se tiñeron los geles con una solución de nitrato de plata.
Búsqueda de inhibidores de la PCR: En aquellas reacciones donde no se obtuvo resultados de amplificación, se realizó un ensayo cruzado (muestra-ADN control) para buscar posibles inhibidores de la PCR. Para la amplificación se utilizó el paquete STR III, el cual provee el control K562, utilizando 2.5 ul de ADN control y 3 µl de la muestra.
Análisis estadístico: Los datos fueron tabulados en una base de datos elaborada en el programa SPSS 11.0. Los datos que presentaron una distribución normal fueron sometidos a un análisis de varianza (ANOVA) y los que no, fueron analizados de acuerdo con el análisis de varianza de una vía de Kruskal-Wallis (K-W). En ambos casos se utilizaron intervalos de confianza de un 95%.
Resultados
Los resultados promedio de las extracciones de los huesos incluidos en este estudio para los diferentes parámetros (cuantificación de ácidos nucleicos, ácidos ribonucleicos, proteínas y pureza de los extractos), con los 3 métodos utilizados, se resumen en el cuadro 2.
Rendimiento de la extracción de ácidos nucleicos, según el método de cuantificación: Solo el método de "DNA Strips" mostró una diferencia significativa (p= 0.003) en la comparación estadística de la cuantificación de los ácidos nucleicos. En general, para los 3 métodos de cuantificación se obtuvo un mejor rendimiento con el método B pero la diferencia no resultó estadísticamente significativa (p= 0.063 para Dyna Quant y p= 0.599 para Quantiblot). Como puede observarse en la cuadro 2, el análisis de ARN presente en los extractos mostró diferencias estadísticamente significativas (p= 0.003 de K-W) entre los métodos de extracción, siendo el método B el que extrajo la mayor cantidad de ARN.
Por su parte, el análisis de proteínas también mostró una diferencia significativa entre los métodos (p= 0.011 K-W), detectándose una mayor cantidad en el método A(ver Cuadro 2).
El análisis de los valores de porcentaje de pureza emitidos por el Gene Quant para cada uno de los extractos no presentó diferencias significativas entre los métodos de extracción (p= 0.164 de ANOVA); sin embargo, el análisis de la proporción (A260 nm/A280 nm) reveló posibles problemas de contaminación en los extractos de los métodos B y C por presencia de proteínas ya que las proporciones eran menores a 1.7 (Miesfeld 1999). Para la proporción (A260 nm/A230 nm), el resultado del método A fue de 2.7 y para los métodos B y C se obtuvo valores inferiores a 2.0
Amplificación del ADN por PCR, según el método de extracción: La comparación general entre los diferentes métodos de extracción según la tasa de éxito en las amplificaciones de STRs (porcentaje de amplificaciones positivas versus amplificaciones negativas) mostró que las mismas fueron de 50%, 40% y 33% para los métodos A, B y C respectivamente, pero las diferencias entre los métodos no fueron estadísticamente significativas según el análisis de Chi cuadrado (p= 0.589). Tampoco lo fueron para las amplificaciones de amelogenina en las cuales incluso la tasa de éxito fue mayor para los 3 métodos (72%, 44% y 44%, respectivamente).
Inhibidores de la PCR: La presencia de inhibidores de la PCR resultó nula en el caso de los extractos obtenidos por el método A (0%), siendo significativamente diferentes (p= 0.0001) con relación a los métodos B (15.4%) y C (84.6%) del 100% de las reacciones inhibidas.
Contexto de las muestras: Como se puede observar en el cuadro 3, se obtuvo mayor cantidad de ADN de las muestras provenientes del contexto "otro" (p= 0.002) en las cuantificaciones con DyNa Quant 200. La cuantificación de los extractos con "DNA Strips", si bien mostró un mejor rendimiento con las muestras provenientes del contexto "acuático", las diferencias no fueron significativas (p= 0.123). En el caso de las cuantificaciones por Quantiblot, el contexto "otro" fue la fuente de las muestras que mostraron una concentración significativamente mayor de ADN (p= 0.0001).
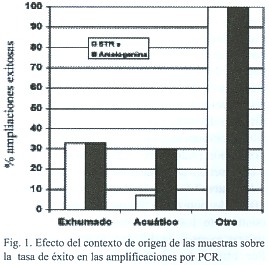
En la evaluación de la tasa de éxito en las amplificaciones por PCR, según el contexto de origen de la muestra se encontró que el mayor número de amplificaciones se logró en los extractos del contexto "otro" (100% tanto para los STR como para la amelogenina) donde las diferencias fueron significativas (p= 0.0001) para ambas comparaciones (ver Fig. 1).
Finalmente, no se detectó la presencia de inhibidores en las muestras provenientes del contexto "otro", pero los mismos sí estuvieron presentes de forma significativa en los contextos "exhumado" y "acuático" donde los inhibidores se detectaron en un 44% y 33% de los extractos, respectivamente.
Discusión
Según ha sido demostrado en otros estudios (Hochmeister et al. 1991, 1995, Lassen et al. 1996) se requiere que los métodos de extracción de ADN brinden resultados satisfactorios cuando las muestras podrían estar degradadas, situación bastante común en los casos forenses. De allí el objetivo de este estudio de valorar la eficiencia de tres métodos de extracción orgánica de ácidos nucleicos a partir de huesos y su aplicabilidad en los análisis de ADN amplificable por la técnica de PCR.
En el presente estudio las comparaciones de las cuantificaciones de ácidos nucleicos según el método de extracción sólo mostraron ser significativamente diferentes cuando las mismas se realizaron por el método de "DNA Strips", el cual cuantifica la cantidad total de ácidos nucleicos en la muestra (tanto ADN como ARN); esto en comparación con el Quantiblot que detecta ADN humano y el DyNa Quant 200 que detecta cualquier tipo de ADN. La diferencia en la cantidad de ácidos nucleicos observada en los extractos podría deberse, a la presencia de ARN en los mismos, lo cual pareció comprobarse con las determinaciones por Gene Quant que también mostraron valores más elevados de ARN en los extractos elaborados con el método B. Por ser el ADN no codificante donde se encuentran las secuencias STR que interesan en las pruebas de identificación forenses, la presencia de ARN podría ser un factor interferente, dependiendo del método de cuantificación que se utilice por cuanto, pese a contar con una buena cantidad de ácidos nucleicos totales la concentración de ADN amplificable en la muestra sería proporcionalmente menor. Además, según ha sido descrito (Maniatis 1982, Miesfeld 1999) el ARN podría funcionar como un obstáculo en la amplificación de las secuencias de interés. Las reacciones moleculares dependen de la cantidad de choques efectivos entre partículas o moléculas. La reacción de PCR depende de que se forme el complejo ADN-"primer" para que pueda iniciarse la extensión de los amplicones. Los sitios donde se unen los "primers" son muy pequeños en relación con el ADN nuclear, de manera que el ARN u otro tipo de ADN podrían bloquear o interferir con la hibridación de esos "primers" con las secuencias específicas de interés.
El análisis de las proporciones de A260 nm/A280 nm en los extractos del presente estudio indicó que los métodos B y C presentaban una posible contaminación por altos niveles de proteínas (los promedios de las proporciones para los métodos B y C están por debajo de 1.7), pero tal problema no se presentó con el método A con el cual los extractos tampoco presentaron contaminación con carbohidratos o degradación significativa.
Teóricamente, una proporción de 0.6 para la relación (A260 nm / A280 nm) corresponde a la presencia única de proteínas; y una proporción entre 1.8–2.0, corresponde a un 90% -100% de pureza de los ácidos nucleicos. En contraparte, los valores mayores a 2.0 podrían indicar excesos de ARN (Schultz et al. 1994). Por otro lado, el análisis de la proporción (A260 nm/A230 nm) indica la presencia de carbohidratos (Misfeld 1999) cuando los valores son menores a 2.0; también existen estudios en donde esta proporción se utiliza para indicar la integridad de los ácidos nucleicos (ARN). Los valores inferiores a 2.0 permiten suponer la presencia de degradación de las moléculas.
En general, el análisis de amplificación de STRs y amelogenina no presentó diferencias significativas entre los diferentes métodos de extracción. Esto obedece, probablemente, a la semejanza de los métodos y al hecho que debieron ser modificados para ajustarlos a la disponibilidad de recursos en el laboratorio sede del estudio. En los métodos A y C, por ejemplo, se sustituyó el último paso de purificación por una precipitación de ácidos nucleicos en presencia de acetato de sodio y etanol, lo cual incrementó las semejanzas.
La comparación entre los promedios de amplificación de STRs y amelogenina aunque sin significación estadística en la diferencia, permitió observar una relación inversamente proporcional con la presencia de inhibidores la cual si presentó diferencias significativas. Este hallazgo parece reforzar la noción de que el éxito de la amplificación por PCR radica en poder remover los contaminantes que inhiben la reacción de la polimerasa (Cattaneo et al. 1997). La diferencia de los promedios, relacionada con los inhibidores, parece marcar una jerarquía entre los métodos, la cual estaría determinada por la habilidad para remover inhibidores. Si se hubiesen utilizado los métodos A y C originales, podría haberse observado un aumento en el porcentaje de resultados positivos en las amplificaciones, puesto que el paso final de purificación original se fundamenta en la extracción del ADN a partir del extracto, en lugar de una precipitación de los ácidos nucleicos y extracción de los contaminantes por medio de lavados, como se practicó en los tres métodos utilizados en este estudio.
Es un hecho conocido que una técnica que permita extraer el ADN de un medio que contenga contaminantes es mucho más prometedora que un método que busque extraer los contaminantes de la preparación (Cattaneo et al. 1997, Buttler 2001). El extraer el ADN del medio (con una herramienta afín al ADN) minimiza los problemas de contaminación puesto que no se requiere extraer todo el ADN allí presente sino obtenerlo en una cantidad y calidad suficiente para las pruebas moleculares. La remoción de los contaminantes del extracto, por el contrario, es usualmente una tarea difícil que con frecuencia lleva a inhibición de las reacciones o errores en la amplificación (Buttler 2001).
Tratándose de métodos fenólicos de desproteinización, la presencia de proteínas en los extractos para la obtención de ADN es un indicador de deficiencia del procedimiento (Maniatis 1982). Sin embargo, en este estudio el análisis de la pureza (A260/A280) no indicó diferencias entre los métodos. Aún así, el método B mostró los mejores resultados en las amplificaciones sin poseer la mayor concentración de ADN y mostrando la mayor contaminación de proteínas de los tres métodos. De allí que los datos parecen confirmar los hallazgos de que las proteínas no son los principales contaminantes responsables de la inhibición observada en las amplificaciones. Por el contrario, el análisis de la proporción de absorción entre A260nm y A230nm en los métodos B y C (promedios inferiores a 2.0 según Miesfeld 1999) podrían indicar que la contaminación por carbohidratos sería al menos parcialmente responsable de las inhibiciones observadas en las amplificaciones por PCR.
El análisis estadístico del efecto del contexto de origen de la muestra con relación a la cantidad de ADN y al éxito en las amplificaciones por PCR indica que el medio donde se encontraban los huesos influye diferencialmente sobre el rendimiento en la extracción de los ácidos nucleicos, el estado en que se encuentran y la cantidad de contaminantes (al menos proteínas y carbohidratos) presentes en los extractos. Sin embargo, en vista de que fue posible detectar ADN por los diferentes métodos de cuantificación en los extractos provenientes de todos los contextos, los fallos en las amplificaciones no pueden achacarse a la ausencia de ADN aunque sí podría deberse a la presencia de ADN no humano o degradado.
En este estudio los huesos provenientes del contexto "otro" dieron el mejor rendimiento al evaluar la contaminación del ADN y la capacidad de amplificación de los diferentes marcadores. La presencia de condiciones adecuadas para la proliferación bacteriana en los otros contextos puede haber tenido un importante efecto sobre la estructura de los huesos y, directamente, sobre la condición de sus ácidos nucleicos.
Aunque en esta investigación no se evaluó este factor, la edad de las muestras puede haber tenido influencia sobre los resultados obtenidos, como ha sido demostrado por otros autores. Cattaneo et al. (1997), en un estudio de comparación de métodos de extracción demostró una diferencia significativa al comparar rendimientos obtenidos con huesos frescos en comparación con huesos provenientes de estudios forenses. Las diferencias fueron igualmente influenciadas por las condiciones de almacenamiento de las muestras y el tipo de marcadores que se intentó amplificar (por ejemplo, genes de copia única versus genes de copia múltiple).
En conclusión, los resultados de este estudio muestran que los tres métodos de extracción orgánica evaluados produjeron ADN amplificable por PCR, pero la calidad del mismo fue diferencialmente influenciado por la presencia de inhibidores, degradación y/o contaminantes; esto, a su vez, estuvo determinado, entre otros, por el contexto en el cual se encontró la muestra previo su análisis en el laboratorio. Las muestras encontradas en medios acuáticos, presentaron la mayor degradación y contaminación de los contextos estudiados. La combinación del método A (una modificación del usado por el FBI), con el uso de columnas de purificación, la aplicación de un método para cuantificar ADN humano (como el Quantiblot), un método para estimar la calidad del extracto (como el que brinda Gene Quant) y diluciones de las muestras en las amplificaciones (para disminuir la cantidad de contaminantes), podrían mejorar la tasa de éxito en los estudios de marcadores moleculares a partir de las muestras de hueso remitidas al Laboratorio de ADN del Poder Judicial de Costa Rica.
Agradecimientos
Al personal del Departamento de Bioquímica: Ana I. Morales, Gladis Núñez, Silvia Fallas, Concepción Morelli y José Luis Peraza. A Luis Del Valle y Luis González Salas por su gran ayuda en la elaboración del manuscrito.
Resumen
Diferentes investigaciones han demostrado que la posibilidad de llevar a cabo estudios de marcadores genéticos está muchas veces limitada por la capacidad de obtener ácidos desoxirribonucleicos (ADN) a partir de muestras degradadas como los restos óseos. Asu vez, la información que de allí se derive resulta de suma importancia en diferentes situaciones médico-legales. El presente estudio se diseñó para comparar la eficiencia de tres métodos orgánicos de extracción de ADN amplificable por la técnica de Reacción en cadena de la Polimerasa (PCR) a partir de muestras de hueso. En esta investigación se procedió a evaluar, comparativamente, la cantidad de ácidos nucleicos obtenidos, el éxito en la amplificación de marcadores microsatélites (STR) y amelogenina por PCR, la posible presencia de inhibidores y el contexto de donde provenían las muestras para los tres métodos. Una modificación del método de extracción utilizado por el FBI (Federal Bureau of Investigation) de los Estados Unidos, denominado método A, aunque no produjo la mayor cantidad de ADN, mostró el mejor rendimiento en cuanto a la calidad requerida para la amplificación por PCR. A la vez, se demostró que dicho rendimiento es influenciado por la posible presencia de inhibidores o contaminantes y por el contexto en el cual permanecieron las muestras previo a su análisis en el laboratorio. Los hallazgos sugieren que la introducción de algunas modificaciones específicas al método A (la incorporación de columnas de purificación, la cuantificación específica de ADN humano en los extractos y el uso de diluciones) podría mejorar sustancialmente el rendimiento de este método de extracción para que pueda ser utilizado con mayor éxito en diferentes investigaciones médico-legales que requieran la disponibilidad de ADN en calidad y cantidad adecuadas.
Bibliografia
Anónimo. 1994-1998. GenePrint STR Systems(Silver Stain Detection). Technical Manual. Promega Co. Madison, WI, USA. 52 p. [ Links ]
Buttler, J. 2001. Forensic DNA Typing; Biology & Technology behind STR Markers. Academic, California, USA. 322 p. [ Links ]
Cattaneo, C. Craig, O. James, & N. Sokol, R. 1997. Comparison of Three DNA Extraction Methods on Bone and Blood Stains up to 43 years old and Amplification of Three Different Gene Frequencies. J. Forensic Sci. 42: 1126-1135. [ Links ]
Fawcett, D. 1990. Tratado de Histología. Bloom-Fawcett. México. Nueva Editorial Interamericana, S.A. 1026 p. [ Links ]
Higuchi, R., C.H. Von Beroldingen, GF. Sensabaugh & HA. Erlich. 1988. DNA typing from single hairs. Nature 332: 543-546. [ Links ]
Hochmeister, M.N., B. Bodowle, U.V. Borer, O. Rudin, M. Bohnert & R. Dirnhofer. 1995. Confirmation of the identity of human skeletal remains using multiplex PCR amplification and typing kit. J. Forensic Sci. 40: 701-705. [ Links ]
Hochmeister, M.N., B. Budowle, UV. Borer, U. Eggman, C.E. Comey & R. Dirnhofer. 1991. Typing of deoxyrribonucleic acid (DNA) extracted from compact bone from human remains. J. Forensic Sci. 36: 1649-1661. [ Links ]
Jiménez-Arce, G. & B. Morera. 1999. Revisión sobre la extracción de ADN a partir de huesos humanos. Med. Legal Costa Rica 16(1/2): 11-14. [ Links ]
Lassen, C., S. Hummel & B. Herrman. 1996. PCR Based Sex Identification of Ancient Human Bones by Amplification of X- and Y- Chromosomal Sequences: A Comparison. Ancient Biomolecules. (Netherlands) 1(1): 25-33. [ Links ]
Lindahl, T. 1993. Instability and decay of the primary structure of DNA. Nature 362: 709-715. [ Links ]
Maniatis, T., EF. Fritsch & J. Sambrook. 1982. Molecular cloning: a laboratory manual. Cold Spring Harbor Lab, New York, 468 p. [ Links ]
Matamoros, M. 1999. Frecuencias alélicas de los loci de repeticiones cortas en tandem en la población hondureña: su aplicación forense. Tesis de Maestría. Universidad de Costa Rica. [ Links ]
Morales C., A.I., B. Morera & G. Jiménez-Arce. 2004. La implementación forense de la tecnología del ADN en Costa Rica: Un análisis retrospectivo. Rev. Biol. Trop. 52(3): 291-308. [ Links ]
Miesfeld, R. 1999. Applied Molecular Genetics. John Wiley, New York, USA. 293 p. [ Links ]
Morera, B. & G. Jiménez-Arce. 1998. Identificación de restos óseos mediante análisis de ADN. Med. Legal Costa Rica 15 (1/2): 6-7. [ Links ]
Morera, B., R. Marín & R. Barrantes. 2001. Análisis de varios marcadores genéticos clásicos en la población de Costa Rica. Rev. Biol. Trop. 49: 1237-1252 [ Links ]
Nakamura, Y. 1987. Variable number of tandem repeats (VNTR) markers for human gene mapping. Science 235: 1616-1622. [ Links ]
Rojas, E. 1995. Análisis de marcadores polimórficos de ADN para pruebas de identidad. Tesis Magíster Scientiae. Universidad de Costa Rica. San José, Costa Rica. [ Links ]
Schultz, D.J., R. Craig, D.L. Cox-Foster, R.O. Mumma & J.I. Medford. 1994. RNA isolation from recalcitrant plant tissue. Plant Mol. Biol. Rep. 12: 310-316. [ Links ]











 uBio
uBio