El reemplazo de células, tejidos u órganos humanos es utilizado como terapia para restaurar o establecer la función normal de un tejido u órgano dañado.1
Desde que se realizó el primer trasplante con éxito se ha generado una vasta experiencia en el tema, desarrollando una serie de procedimientos que dependen del tipo de trasplante que se realice.
Cuando se habla de trasplante de órganos sólidos como es el caso del riñón, el órgano por lo general proviene de un donante que puede ser vivo o también de un donante muerto, que sea compatible con el receptor o persona trasplantada.2
En el caso de los trasplantes de células progenitoras hematopoyéticas o de médula ósea, los criterios se vuelven más selectivos y por la misma complejidad, se ha establecido una clasificación con el fin de escoger el más apropiado para el paciente enfermo. Varias enfermedades relacionadas con células progenitoras hematopoyéticas han sido tratadas exitosamente por alotransplante de médula ósea.2
Los virus pertenecientes al grupo de los Herpesvirus, son los que más frecuentemente infectan al paciente trasplantado. La importancia de los Herpesvirus en esta población se justifica por su ubicuidad y por su capacidad de producir latencia, lo que permite al virus reactivarse y comenzar su ciclo replicativo en condiciones en las que existe una disminución de la vigilancia inmunológica, como sucede en los trasplantes.3
Existen factores asociados al trasplante a partir de los cuales se toman decisiones para el manejo terapéutico del paciente. Dentro de estos factores están los estados de neutropenia e inmunosupresión desencadenados por los medicamentos aplicados para que el injerto se logre con éxito y se disminuya la posibilidad de rechazo del trasplante.4 Otro factor es la aparición de infecciones dadas por la reactivación de virus en estado de latencia que se haya establecido previamente, aún desde etapas tempranas de la niñez.5 La latencia de las infecciones virales se caracteriza por tres propiedades generales: los productos de los genes virales que promueven la reproducción de los virus en pocas cantidades, las células que albergan al genoma latente y son poco reconocidas por el sistema inmune y la persistencia del genoma viral intacto para que se inicie una infección productiva posterior, garantizando la difusión de la progenie viral hacia nuevos hospederos.6 La latencia de ciertos genomas de Herpesvirus se puede mantener en forma de episoma en células en división, como es el caso del virus Epstein-Barr (EBV) en las células B o en la infección por Citomegalovirus en las células salivales y de glándula mamaria. La infección por Citomegalovirus (HCMV) es una de las causas más significativas de morbilidad y mortalidad en individuos que se ven afectados por episodios de inmunodepresión como es el caso de los trasplantes.7 El HCMV representa la primera causa de infección primaria en humanos y se mantiene en latencia durante toda la vida, por lo que puede darse una reactivación que provoque una reinfección.6 La tasa de seropositividad de la población en general en países desarrollados, oscila entre el 30% y 70%, y es mayor en sujetos de mayor edad y bajo nivel socioeconómico.8
La glicoproteína B del HCMV (gB) es una proteína altamente inmunogénica incorporada en la envoltura viral, que ejerce un papel biológico esencial en la interacción con el hospedero del virus, ya que participa en la entrada, propagación y replicación del virus en diferentes células hospederas.9, 10. Las cepas de HCMV de tipo silvestre se pueden clasificar en cuatro variantes genotípicas gB mayores (gB 1-4), basadas en la secuencia de gB. Cada una de ellas tiene tropismo por líneas celulares distintas, lo que conduce a diferencias en patogénesis y gravedad de la enfermedad.
Por lo tanto, la caracterización genotípica de cepas de HCMV que infectan a individuos inmunocomprometidos puede contribuir a estudios moleculares epidemiológicos y a la definición del papel de la variabilidad genética viral en la expresión clínica y en el pronóstico.11
El objetivo del presente estudio fue determinar el curso de infecciones virales por un periodo de un año, mediante la medición de la carga viral de Adenovirus, virus BK, virus Epstein- Barr, Citomegalovirus y Herpesvirus humano 6, en pacientes sometidos a trasplante de riñón o células progenitoras hematopoyéticas, atendidos en el Hospital San Juan de Dios (HSJD).
Métodos
Pacientes: Se estudiaron treinta pacientes mayores de 12 años con diagnóstico de trasplante de riñón (TR) y trasplante de células progenitoras hematopoyéticas o medula ósea (TMO). Se analizaron las muestras de pacientes del Servicio de Nefrología y del Servicio de Hematología del HSJD de la Caja Costarricense del Seguro Social (CCSS), durante un período de un año (septiembre 2015 a setiembre 2016). La cantidad de pacientes trasplantados en el estudio se calculó con base en el comportamiento histórico anual que se ha registrado en el HSJD en años anteriores (Fuente: Servicio de Nefrología y Hematología, HSJD-CCSS). Todos los pacientes realizaron el proceso de consentimiento informado, de acuerdo con los procedimientos y el protocolo establecido por el Comité Local de Bioética e Investigación del HSJD, CCSS (CLOBI) y el Comité Ético Científico del Centro de Investigación de Enfermedades Tropicales (CIET), Facultad de Microbiología y Vicerrectoría de Investigación, Universidad de Costa Rica.
Método: Se recolectó sangre a través de punción venosa en tubos Vacutainer con anticoagulante EDTA. Se centrifugó a 2000 g (4400 r.p.m.) por 20 minutos, con el fin de separar el plasma con el cual se trabajó. La medición realizada fue de 10 muestras por paciente que incluyeron: una muestra pretrasplante que correspondió a la medición 1 y luego 8 muestras cada quince días posteriores al trasplante por un período de 4 meses (mediciones 2 a 9) y una última muestra a los 6 meses posteriores al trasplante (medición 10).
Cuantificación y detección del ácido nucleico de los virus: Se realizó la cuantificación de virus BK, HCMV, EBV y HHV6 por medio de la técnica de reacción en cadena de la polimerasa (PCR) en tiempo real, para conocer la carga viral de cada uno. Solamente en el caso de Adenovirus, la detección se realizó de manera cualitativa, por PCR de punto final.
Para HCMV, se utilizó la técnica y equipos de la casa comercial ABBOTT. La extracción del ácido nucleico fue a través de partículas electromagnéticas que atrapan los ácidos nucleicos en el equipo de Abbott m2000sp. Posteriormente se realizó el montaje de la mezcla de reacción y finalmente se realizó la detección y cuantificación de la carga viral del HCMV en el equipo Abbott m2000rt. Para el resto de los virus, se realizó una sola extracción del ácido nucleico a través de columnas de la marca QIAGEN en el instrumento QIAcube. El principio de la técnica se basa en varios ciclos de centrifugación a través de los cuales el material genético viral queda atrapado en las columnas que son de gel de sílice y luego el ADN es liberado por medio de una solución de elución, para ser recolectado en tubos de polipropileno. El volumen final del ácido nucleico extraído fue de 100 uL. A partir de ese ADN extraído, se realizó el montaje de cada mezcla de reacción según fuera el virus a determinar y por medio de PCR en tiempo real se determinó la carga viral de cada virus. Las reacciones de PCR en tiempo real se realizaron en el equipo Rotor Gene de QIAGEN. En el caso de Adenovirus, se utilizaron reactivos de la casa comercial SECASE para el montaje de la mezcla de reacción y se realizó la amplificación del ácido nucleico mediante un PCR de punto final, en un termociclador de la casa comercial Applied Biosystems. Luego se hizo la corrida electroforética del fragmento amplificado en un gel de agarosa teñido con bromuro de etidio y se visualizó a través de un transiluminador con luz ultravioleta. Las muestras fueron clasificadas cualitativamente como positivas o negativas. Las cargas virales del resto de los virus fueron reportadas en términos de copias/mL. Toda carga viral mayor al límite inferior de detección se consideró prueba positiva y los límites superiores e inferiores de detección fueron determinados por lo establecido por las casas comerciales y recomendaciones del fabricante.
PCR anidado y detección del genotipo del HCMV: Las cargas virales positivas por HCMV se guardaron en un congelador que mantiene muestras a -80 °C. Para efectos de la determinación de genotipos de HCMV, se trabajó únicamente con cargas virales superiores a 1000 copias/mL6 y se realizó un PCR anidado para verificar la presencia de material genético viral mediante un gel de agarosa. Se realizó una extracción de ácido nucleico a partir de las muestras guardadas y seleccionadas para el análisis de genotipo, utilizando columnas de la marca QIAGEN en el instrumento QIAcube. Para el PCR anidado se utilizó una mezcla de reacción de la casa comercial Thermo Scientific N°K0171. Para la primera ronda de amplificación, los oligonucleótidos iniciadores externos utilizados fueron gB 1319 (5’TGGAACTGGAACGTTTGGC3’) y gB 1676 (5’TGACGCTGGTTTGGTTGAATG3’).
Para la segunda ronda del PCR anidado se utilizaron los iniciadores gB 1319 (5’TGGAACTGGAACGTTTGGC3’) y gB 1604 (5’GAAACGCGCGGCAATCGG3’). Todos los iniciadores se estandarizaron para ser utilizados a una concentración final de 10 uM. La mezcla de reacción tuvo un volumen final de 50 uL incluyendo la solución de reacción, cada uno de los iniciadores, ADN de cada muestra clínica y agua libre de nucleasas. Las condiciones de PCR fueron las siguientes: una desnaturalización inicial a 95 °C por 3 minutos, 25-40 ciclos con desnaturalización a 95 °C por 30 segundos; hibridación a Tm 55 °C por 30 segundos y extensión a 72 °C por 1 minuto, con una extensión final a 72 °C por 5-15 minutos.
El producto amplificado de 357 pb de la primera ronda, fue visualizado en un gel de agarosa al 2 % teñido con bromuro de etidio. Se realizó un PCR anidado de todas las muestras amplificadas de la primera ronda, para obtener un fragmento amplificado de 285 pb. Las condiciones y cantidades para la segunda ronda fueron las mismas que para la primera, pero con los iniciadores mencionados anteriormente. Para la secuenciación se contrataron los servicios del CIBCM (Centro de Investigación en Biología Celular y Molecular de la Universidad de Costa Rica) donde las muestras fueron analizadas en el equipo ABI 3500. Las secuencias obtenidas de los pacientes se editaron, con el fin de dejar únicamente la secuencia nucleotídica que corresponde a los codones 441-511 del gen para la glicoproteína B (gB) del HCMV humano. Para ello se realizó un alineamiento de las secuencias obtenidas con la secuencia de referencia Human Herpesvirus 5 strain HANRTR5, complete genome (número de accesión KY123652.1), utilizando el programa DNA Baser (Version 4.36.0.2.). Se comparó la secuencia de aminoácidos mediante un análisis Blast en el sitio NCBI (https://blast. ncbi.nlm.nih.gov) y se realizó un alineamiento de Clustal de las secuencias de aminoácidos para comparar las posiciones de los aminoácidos en la proteína. El alineamiento de Clustal se realizó mediante la herramienta Clustal Omega (http:// www.ebi.ac.uk/Tools/msa/clustalo/).
Resultados
De la muestra seleccionada con 30 pacientes, 21 fueron sometidos a TR y 9 fueron sometidos a TMO y la distribución por sexo fue de 15 hombres y 15 mujeres, con solo un paciente menor de 18 años. Además, un paciente presentó un rechazo del injerto al segundo día tras realizado el trasplante y tres fallecieron aproximadamente al mes y medio de haber recibido el injerto. Se encontró que 23 pacientes (77 %) evidenciaron resultados positivos para al menos uno de los virus analizados (Cuadro 1).
Cuantificación y detección del ácido nucleico de los virus:
El cuadro 1 muestra los virus encontrados por cada paciente. En total se encontraron 7 pacientes positivos para adenovirus, de los cuales 3 corresponden a TMO y 4 a trasplante de riñón. El virus BK fue detectado en 5 casos, de los cuales 2 fueron de TR y 3 de TMO.
El virus que más se detectó fue el HCMV, con 17 casos que corresponden a 11 de TR y 6 de TMO. El virus EBV fue el que menos se detectó, con solo 2 casos y finalmente el HHV6 con 3 casos positivos.
En los pacientes positivos por Adenovirus, se obtuvo una medición cualitativa positiva en la medición 3, dos en la 4, una en la 5, uno en la 6, uno en la 7 y uno en la medición postrasplante. El cuadro 2 muestra la cantidad de pacientes positivos según el virus y sexo del paciente. En general hubo más virus presentes en la población femenina que en la masculina. El Adenovirus presentó 6 casos en mujeres y 1 caso en hombres.
De los 5 casos positivos por virus BK, 2 fueron masculinos y 3 femeninos. El HCMV mostró un comportamiento casi igual entre hombres y mujeres, ya que del total de 17 casos positivos 9 fueron masculinos y 8 femeninos. Para el virus Epstein-Barr solo hubo un caso femenino y uno masculino, mientras que para HHV6 se presentaron 2 casos en mujeres y uno en hombres.
La figura 1A muestra el comportamiento en el tiempo de las determinaciones de la carga viral de los pacientes positivos por BKV. Se observó que en el paciente 15 se elevó la carga viral de manera significativa durante la medición 5, luego decayó y volvió a elevarse en la medición 9, mientras que en los demás pacientes se detectaron mediciones de siete a nueve en el estudio.
La figura 1B muestra el comportamiento de la carga viral en el tiempo para el virus Epstein-Barr en los pacientes 23 y 24. En ambos pacientes se detecta el virus en la cuarta medición.
Los tres únicos casos que fueron positivos para HHV6 se muestran en la figura 1C, donde se observa un comportamiento similar. El paciente 3 se detectó en la medición 2 mientras que los pacientes 13 y 19 se detectaron durante la medición tres del estudio.
La figura 1D muestra el comportamiento en el tiempo de las cargas virales de los pacientes positivos por HCMV. Este fue el virus que más casos positivos reportó, con valores elevados de carga viral.
El paciente 15 y el paciente 23 fueron los únicos pacientes a los cuales se les detectaron varios virus simultáneamente, como se observa en la figura 2.
PCR anidado y detección del genotipo del HCMV:
De los 17 casos positivos para HCMV, solo se tomaron en cuenta 13 pacientes que presentaron cargas virales con valores mayores a 1000 copias/ mL. De estos 13 pacientes, solamente se logró obtener amplificación de 11 muestras en la segunda ronda de PCR y 8 de estas muestras correspondieron a pacientes de TR y las otras 3 restantes fueron de pacientes de TMO. Las bandas obtenidas presentaron un tamaño de aproximadamente 285 pb. Finalmente, de las 11 muestras amplificadas en la segunda ronda de PCR, se lograron secuenciar y analizar exitosamente las muestras de 10 pacientes (cuadro 3). Sólo un paciente (número 15) presentó el genotipo 1 de HCMV, mientras que los demás mostraron el genotipo 3. No se detectó ninguna infección mixta en los pacientes.
Cuadro 1 Distribución de los 30 pacientes con trasplante de riñón (TR) y trasplante de células progenitoras hematopoyéticas o medula ósea (TMO) según el tipo de virus encontrado, periodo septiembre del 2015 a septiembre del 2016, Servicio de Nefrología y Servicio de Hematología del Hospital San Juan de Dios, Caja Costarricense de Seguro Social.
| Sexo | Adenovirus | BKV | HCMV | EBV | HHV6 | TOTAL |
|---|---|---|---|---|---|---|
| Masculino | 1 | 2 | 9 | 1 | 1 | 14 |
| Femenino | 6 | 3 | 8 | 1 | 2 | 20 |
| TOTAL | 7 | 5 | 17 | 2 | 3 | 34 |
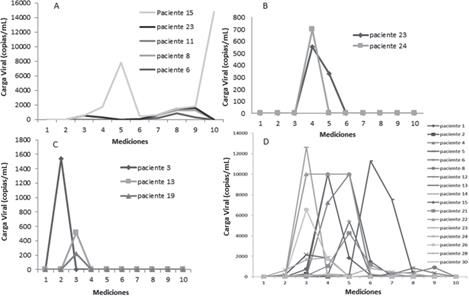
Figura 1 Mediciones en el tiempo de las cargas virales en los pacientes de TMO y TR, con resultados positivos por cuatro virus seleccionados: A. BKV, B. EBV, C. HHV6 y D. HCMV. La medición 1 es pretrasplante, las mediciones 2-9 se realizaron cada 15 días postrasplante y la medición 10 se realizó a los 6 meses posteriores al trasplante; periodo de septiembre del 2015 a septiembre del 2016, Servicio de Nefrología y Servicio de Hematología del Hospital San Juan de Dios, Caja Costarricense de Seguro Social.
Discusión
Durante un año se realizó la detección de cinco virus en treinta pacientes de TR y TMO del Hospital San Juan de Dios, se contó con la aprobación de un consentimiento informado por parte de cada uno de los pacientes.
En particular, los pacientes trasplantados de medula ósea por la medicación previa que reciben, sufren de una alta depleción de células, en especial linfocitos, lo que conlleva a propiciar un ambiente favorable para que se desarrollen muchos microorganismos, en especial los virus. Estas infecciones virales se pueden desarrollar por una primoinfección o bien latencia existente por infecciones pasadas y según lo demuestran ensayos clínicos el 30% de las muertes por infecciones después de un trasplante se deben a los virus.15 También se ha observado en esta población de pacientes, infecciones concomitantes de varios virus a la vez.13
Generalmente la activación de los Herpesvirus comunes como el HCMV, promueve la reactivación de otros Herpesvirus en latencia como son el EBV, virus BK y Adenovirus. Del total de pacientes de este estudio, siete resultaron positivos por Adenovirus es decir, un 23% lo que concuerda con otros estudios que indican que la incidencia de Adenovirus en pacientes trasplantados es superior al 21%.13,14
El manejo terapéutico con antivirales hace que la carga viral disminuya como se observó en este estudio don de los pacientes pasaron varias mediciones positivas pero luego la carga viral cae a niveles indetectables por el suministro de fármacos. Cabe mencionar que estos tratamientos lamentablemente traen efectos secundarios al paciente, sobre todo anormalidades hematológicas, principalmente neutropenia, anemia y trombocitopenia.17 Por lo anterior, las mediciones periódicas de la carga viral en estos procesos son de gran ayuda pues así, solo se suministraría tratamiento a los casos que lo ameriten. La alta toxicidad y la posibilidad de resistencia de estos antivirales un tema que se debe tomar en cuenta para los protocolos institucionales ya que es válido realizar un manejo terapéutico disminuyendo la inmunosupresión y observando la reacción del paciente.6, 13, 16 Además, el costo para la institución por paciente sería menor, ya que estos medicamentos son de alto costo.
Cuadro 2 Distribución de los 30 pacientes según las detecciones virales y el tipo de trasplante, periodo de septiembre del 2015 a septiembre del 2016, Servicio de Nefrología y Servicio de Hematología del Hospital San Juan de Dios, Caja Costarricense de Seguro Social.
| NÚMERO DE PACIENTE | SEXO DEL PACIENTE | TIPO DE TRASPLANTE | Adenovirus | BKV | HCMV | EBV | HHV6 |
|---|---|---|---|---|---|---|---|
| 1 | Femenino | TR | Positivo | ||||
| 2 | Femenino | TR | Positivo | ||||
| 3 | Masculino | TR | Positivo | ||||
| 4 | Masculino | TR | Positivo | ||||
| 6 | Masculino | TR | Positivo | Positivo | Positivo | ||
| 7 | Masculino | TR | |||||
| 9 | Femenino | TR | Positivo | ||||
| 10 | Femenino | TR | |||||
| 11 | Femenino | TR | Positivo | ||||
| 12 | Masculino | TR | Positivo | ||||
| 13 | Masculino | TR | Positivo | Positivo | |||
| 14 | Femenino | TR | Positivo | Positivo | |||
| 17 | Masculino | TR | |||||
| 18 | Masculino | TR | |||||
| 19 | Femenino | TR | Positivo | Positivo | |||
| 20 | Masculino | TR | |||||
| 21 | Masculino | TR | Positivo | ||||
| 22 | Masculino | TR | Positivo | ||||
| 26 | Masculino | TR | Positivo | ||||
| 27 | Masculino | TR | |||||
| 28 | Femenino | TR | Positivo | ||||
| 5 | Femenino | TMO | Positivo | Positivo | |||
| 8 | Femenino | TMO | Positivo | Positivo | |||
| 15 | Femenino | TMO | Positivo | Positivo | Positivo | ||
| 16 | Femenino | TMO | Positivo | ||||
| 23 | Masculino | TMO | Positivo | Positivo | Positivo | ||
| 24 | Femenino | TMO | Positivo | Positivo | |||
| 25 | Femenino | TMO | |||||
| 29 | Femenino | TMO | |||||
| 30 | Masculino | TMO | Positivo |
Trasplante de riñón (TR), trasplante de células progenitoras hematopoyéticas o medula ósea (TMO). Pacientes fallecidos
Cargas virales bajas < 1000 copias/mL
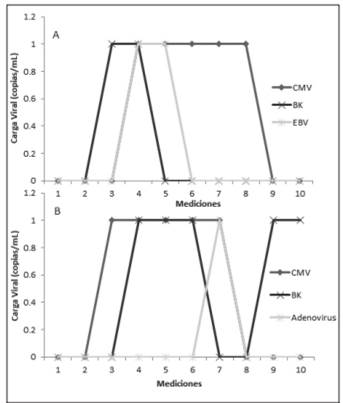
Figura 2 Mediciones en el tiempo de las cargas virales de: A. los virus BKV, HCMV y EBV del paciente TMO número 23 y B. los virus BKV, HCMV y Adenovirus del paciente TMO número 15. La medición 1 es pretrasplante, las mediciones 2-9 se realizaron cada 15 días postrasplante y la medición 10 se realizó a los 6 meses posteriores al trasplante; periodo de septiembre del 2015 a septiembre del 2016, Servicio de Nefrología y Servicio de Hematología del Hospital San Juan de Dios, Caja Costarricense de Seguro Social.
En el estudio se observa una baja incidencia de los virus EBV y HHV6 en comparación con HCMV lo que resulta curioso ya que según la literatura, en pacientes trasplantados la positividad para EBV podría llegar hasta un 20% .16 y para HHV6 el 95% de las personas adultas son seropositivas por este tipo de Herpes virus. 3. El estudio demostró que 2 pacientes del total de 30 resultaron positivos para EBV, es decir un 6% y solamente 3 del total resultaron positivos por HHV6 (10%)
Además el estudio reflejó que el virus que más casos reportó fue el HCMV con 17 pacientes positivos equivalente a un 57% del total de pacientes involucrados en el proyecto lo que hace sentido ya que a nivel mundial, el 70% de la población adulta son seropositivos para HCMV.6 A 10 pacientes positivos por HCMV se les realizó la determinación del genotipo por HCMV mediante secuenciación y se obtuvo que el más predominante fue el genotipo 3 (gB3) el cual se presentó en 9 pacientes. El paciente restante, fue el único caso de genotipo 1 (gB1) y corresponde a un paciente trasplantado de riñón. Estudios señalan que el genotipo 1 no se asocia con enfermedad mortal por HCMV. 18
Cuadro 3 Alineamiento múltiple de secuencias de aminoácidos deducidas a partir de la secuencia de los codones 441-511 del gen para la glicoproteína B (gB) de HCMV, obtenidas a partir de muestras de sangre de los 10 pacientes positivos por HCMV y con una carga viral superior a 1000 copias/mL, periodo de septiembre del 2015 a septiembre del 2016, Servicio de Nefrología y Servicio de Hematología del Hospital San Juan de Dios, Caja Costarricense de Seguro Social.

Nota: Se indican con símbolo de una letra, las posiciones de los aminoácidos que difieren de la secuencia de referencia uti- lizada para HCMV (número de accesión en GenBank: KY123652.1), con un asterisco (∗) las posiciones que coinciden y con un guión (-) las posiciones ausentes. El genotipo de HCMV determinado para cada paciente se muestra en la última columna y el sombreado en gris corresponde a las posiciones más relevantes para determinar el genotipo de HCMV, según Chou y Dennison (1991).
Según otros estudios,19 el genotipo predominante en Costa Rica es el genotipo 2 (gB2), sin embargo, en menor proporción también encontraron el genotipo 1 y 3. Por otro lado, Cunha et al 11 mencionan que los genotipos 2 y 3 son los que se encuentran más frecuentemente en pacientes con trasplantes de médula ósea.
El presente estudio no es concluyente, ya que tiene varias limitaciones tales como: muestreo limitado, el tipo de muestras utilizado solo es de plasma y la población en estudio se remite solo a pacientes trasplantados. Por esta razón se sugiere a futuro, realizar un estudio con un mayor muestreo que sea más representativo de la población en cuestión.
Como conclusión, se puede afirmar que las técnicas moleculares son un excelente instrumento para la detección y cuantificación de los virus y así el médico tratante podrá dar un mejor manejo clínico al paciente que atraviese por alguna infección viral oportunista. Además se podrá tener un mejor pronóstico sin tener que hacer uso de terapias farmacológicas que conllevan a los efectos secundarios indeseables.20
Agradecimientos y colaboradores: A los asesores de la Universidad de Costa Rica: MSc. Sandra Silva De la Fuente, Dr. Rodrigo Mora Rodríguez. Asesor en Bioinformática: Lic. Mario Ulate Solano. A la Dirección del Laboratorio Clínico del Hospital San Juan de Dios: Dr. José Pablo Marín Gómez.











 text in
text in 


